Hans Journal of Computational Biology
Vol.3 No.3(2013), Article ID:12320,4 pages DOI:10.12677/HJCB.2013.33003
FBA and FVA Analysis of Central Carbon Metabolism in E. coli Deletion Mutants*
1School of Life Science, Guizhou Normal University, Guiyang
2Guizhou Province of Key Laboratory of Plant Physiology and Development Regulation, Guiyang
3School of Life Science, Anhui Agricultural University, Hefei
Email: #ruibin@ahau.edu.cn, #swhx1@ahau.edu.cn
Received: Jun. 25th, 2013; revised: Jul. 15th, 2013; accepted: Jul. 30th, 2013
Copyright © 2013 Tie Shen et al. This is an open access article distributed under the Creative Commons Attribution License, which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited.
ABSTRACT:
E. coli has clear genetic background and is easy to be cultured, so it has been widely used in a variety of biochemistry, biotechnology research as the mode microorganisms. In this work, a small E. coli metabolic system has been built in the cobra workspace. 25 enzymes in the metabolic system were separately knocked out and the metabolic system was investigated by FBA (flux balance analysis) and FVA (flux variability analysis). By combing the outcome of the analysis, we could infer the role played by the various enzymes in the E. coli metabolic system.
Keywords: E. coli; Flux Blance Analysis; Flux Variality Analysis; Metabolic Flux; Enzyme
大肠杆菌基因缺失菌株中碳中心代谢系统的通量平衡分析和通量变异分析*
申 铁1,2,金杰军3,乙 引1,2,文 汉3#,芮 斌3#
1贵州师范大学生命科学学院,贵阳
2贵州省植物生理与发育重点实验室,贵阳
3安徽农业大学生命科学学院,合肥
Email: #ruibin@ahau.edu.cn, #swhx1@ahau.edu.cn
摘 要:
大肠杆菌(E. coli)因其遗传信息简单且容易培养而被广泛用作各种生物化学、生物技术研究等的模式微生物。本文首先通过COBRA工具包构建了大肠杆菌碳中心代谢系统的小规模网络,然后分别模拟敲除了其中的25种酶,并利用代谢通量平衡分析和代谢通量变异分析研究了基因敲除后的代谢网络。通过这些结果的综合分析,我们可以推断大肠杆菌碳中心代谢系统的不同的酶对整个体系的作用。
收稿日期:2013年6月25日;修回日期:2013年7月15日;录用日期:2013年7月30日
关键词:大肠杆菌;通量平衡分析;通量变化分析;代谢通量;酶
1. 引言
大肠杆菌(Escherichia coli, E. coli)革兰氏阴性短杆菌,大小0.5 × 1~3微米。周身鞭毛,能运动,无芽孢。能发酵多种糖类产酸、产气,是人和动物肠道中的正常栖居菌,婴儿出生后即随哺乳进入肠道,与人终身相伴,其代谢活动能抑制肠道内分解蛋白质的微生物生长,减少蛋白质分解产物对人体的危害,还能合成维生素B和K,以及有杀菌作用的大肠杆菌素[1]。
大肠杆菌是研究微生物遗传的重要材料,如局限性转导就是1954年在大肠杆菌K12菌株中发现的。莱德伯格(Lederberg)采用两株大肠杆菌的营养缺陷型进行实验,奠定了研究细菌接合方法学上的基础,以及基因工程的研究。大肠杆菌作为外源基因表达的宿主,遗传背景清楚,技术操作简单,培养条件简单,大规模发酵经济,倍受遗传工程专家的重视。目前大肠杆菌是应用最广泛,最成功的表达体系,常作高效表达的首选体系。
系统生物学着重以系统的思路和方法定量研究研究生物过程[1]。由于生物系统的组成关系主要用网络来表示,代谢网络研究就自然地成为热门领域。最初的网络理论往往只涉及一个底物和一个产物,故而与实际网络不符。FELL等学者便致力于线性代数的方法来以更便利地计算代谢途径,其主要思想是:先假设代谢网络的途径是代谢网络计量系数矩阵的线性无关解。通过对这些途径的线性组合或者枚举来产生存在的(但未必是热力学可行的)代谢途径。最近,凸分析被运用到代谢途径分析中,代谢网络理论于是得到了更进一步的发展,渐渐形成了得到普遍认可的两种方法,基元模式(EFMs)和极端途径(EPs)分析。
同时,进一步,在另一个层面上,对代谢网络的研究也逐渐演化出来单独的静态分析和动态分析。静态分析主要包括Schilling等学者提出的极端途径(extreme pathway analysis),基元模式(elementary flux mode analysis),FBA流平衡分析(flux balance analysis)和jeong等学者提出的图论法(graph theory)[3-6]。而动态分析方法,依靠基于动力学信息的,它包括代谢控制分析(metabolic control analysis),控制论模拟方法(cybernetic modeling)。当前在基因组规模的代谢网络来说,许多反应的动力学参数和反应物浓度参数都是不清楚的,故静态的研究方法占据了目前代谢网络研究的主流位置[4,7-10]。
本篇论文在matlab cobra工具箱的基础上对大肠杆菌代谢系统进行FBA和FVA分析,将分析结果进行汇总后,为显示在敲除和不敲除各种酶的结果变化[9],对数据进行绘图,通过直观的图形对比,显示出敲除各种酶和不敲除酶对大肠杆菌代谢系统中每一个反应的影响,以及大肠杆菌代谢系统的全部反应在敲除某个酶后的两项结果变化是否存在共性,以此为依据,大致判断各个酶在大肠杆菌代谢系统中起到的作用。大肠杆菌代谢系统中有很多的酶参与,此篇论文中,我以25个酶为例,分别单独将每一个酶从代谢系统中进行敲除后[11-13],对代谢系统进行FBA和FVA分析,将结果与未敲除任何酶时的代谢系统FVA和FBA结果进行对比,以确定所敲除在代谢系统中起到的作用,这提供了一个全新的分析方法,进行酶分析,这样的结果虽说只是猜测,但是可以对以后的实验起到指导性作用,使研究者可以少走弯路,尽快获得可靠的数据。然而由于时间原因,进行分析后没有再以实验对结果进行验证,在这方面还有待提高。
2. 材料与方法
2.1. 所需操作软件以及文件
Matlab版本7.0以上cobra toolbox版本2.0以上libsbml版本4.0.1以上。
Sbml toolbox版本3.1.1以上gurobi 5.0以上。
大肠杆菌小型中心代谢模型“ecoli_core_model. Xml”,此模型中包含大肠杆菌中心代谢的全部信息[6]。
2.2. 方法:操作过程
2.2.1. 代谢模型读取
在matlab命令窗口中执行指令: model = readCbModel(‘文件名’)其中文件名为模型保存目录[14,15]。
2.2.2. 模型的修饰
执行指令:
model = changeRxnBounds(model,'EX_glc(e)',-16.7,'l');其中'EX_glc(e)'代表了底物摄取速率,“l”是“lower”的缩写,表示模型的下限[2]。
2.2.3. 酶的敲除
执行指令:
model = removeRxns(model,'rxnRemoveList'); 'rxnRemoveList'代表的是含有所敲除反应的反应信息的单元矩阵,酶的敲除中,rxnRemoveList可直接用所需敲除酶的英文缩写代替。如敲除烯醇化酶即ENO酶,直接在命令窗口中输入指令model = removeRxns(model,'ENO')即可,执行命令后表示该酶所直接参与的反应的电子信息从模型中去除,该反应在模型中不再起作用。
2.2.4. FBA最优化值的获取
执行指令: changeCobraSolver('gurobi5','LP'); Fluxdistribution = optimizeCbModel(model); flux = geometricFBA(model)
其中第一步指令是修改cobra solver;第二步是执行标准FBA命令。
2.2.5. FVA(Flux Variability Analysis)值的获取
执行命令:
[minFlux,maxFlux] = fluxVariability(model,90);其中“90”代表一个百分比,为模型所能达到的目标函数相对于最优值得比例。FVA命令的结果有minFlux和maxFlux,分别代表模型中95个反应的通量分布情况。
2.3. 作图说明
作图从两个方向进行,分别为横向和纵向,横向作图在于显示模型中每个单独反应在不同酶被敲出后受到的影响,作图时横坐标代表被敲除的酶,为更好的显示结果,分别用数字代表各种酶,对应情况为:1) 没有敲除任何酶;2) EDD;3) GLUUPT;4) GdH;5) GND;6) TK2;7) PFK;8) HXI;9) TAL;10) TK1;11) SER;12) SHMT;13) GEV;14) ENO;15) PYK;16) PDH;17) AC-EX;18) GLTA;19) ICD;20) AKD;21) FUM;22) MDH;23) GS1;24) GS2;25) PPC;26) MEZ[4]。横向作图时FBA的结果使用折线图显示,FVA的结果选用柱形图来显示区间的变化.纵向作图在于显示敲除某个酶对整个代谢系统的影响,作图时横坐标的数字分别代表大肠杆菌代谢模型中的95个反应。纵向作图时由于反应有95个,在进行FVA分析时,用柱形图难以显示结果的变化,所以将结果进行拆分,分别对minflux和maxflux作图。
3. 结果
3.1. 横向分析
论文中只将反应ATP synthase,Biomass,glutamate transport的流量变化图附上(如图1~6),其他反应可以同理进行分析。
3.2. 纵向分析
横向分析后,会发现在酶14(ENO),15(PYK),16(PDH),21(FUM),25(PPC)等被敲出后,反应受到的影响最大,本次论文只将酶ENO即烯醇化酶,酶

Figure 1. Changes in reaction ATP synthase after the knockoutof different enzyme
图1. 反应ATP synthase在敲除不同的酶后最优流量值的变化
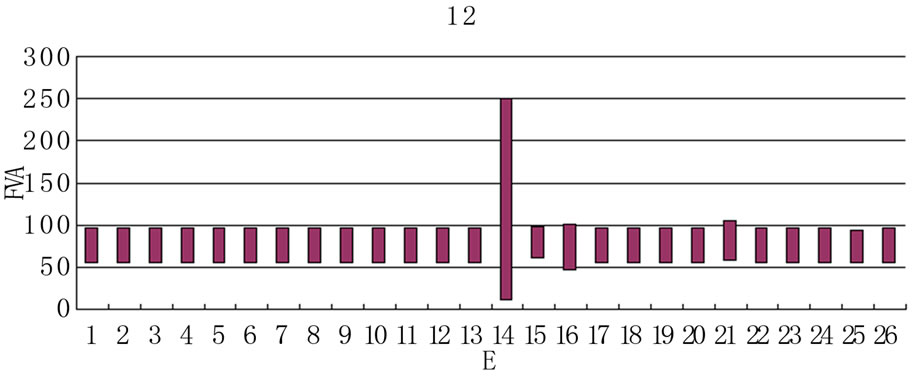
Figure 2. Changes in flux range of reaction ATP synthase after the knockout of different enzyme
图2. 反应ATP synthase在敲除不同的酶后流量范围的变化
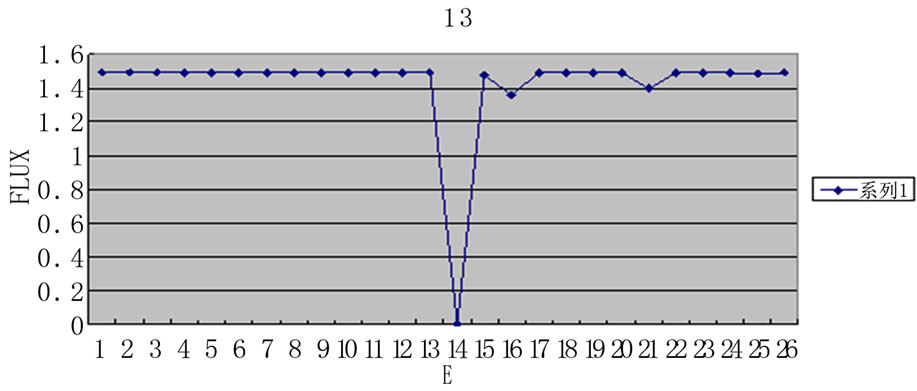
Figure 3. Changes in reaction Biomass after the knockoutof different enzyme
图3. 反应Biomass在敲除不同的酶后最优流量值的变化
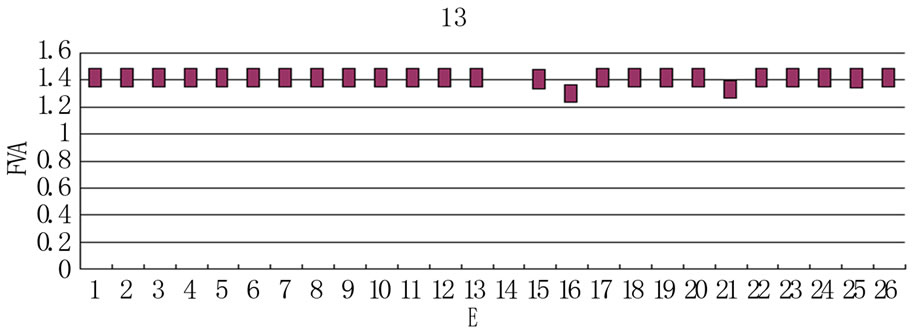
Figure 4. Changes in flux range of reaction Biomass after the knockoutof different enzyme
图4. 反应Biomass在敲除不同的酶后流量范围的变化

Figure 5. Changes in reaction glutamate transport after the knockout of different enzyme
图5. 反应glutamate transport在敲除不同的酶后最优流量 值的变化
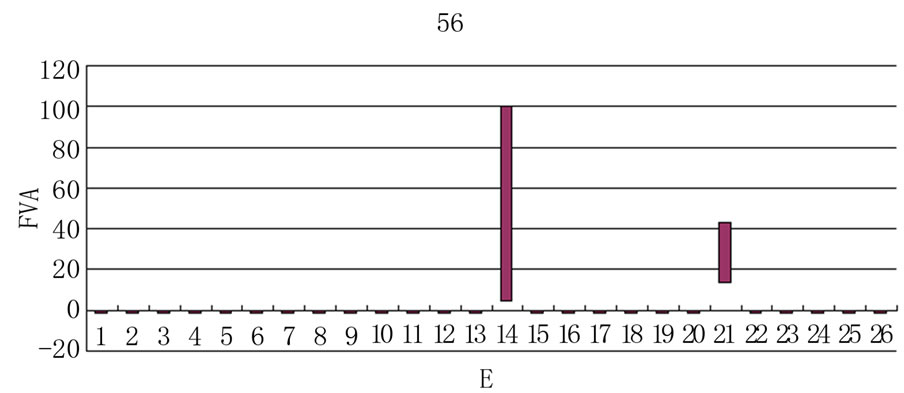
Figure 6. Changes in flux range of reaction glutamate transport after the knockout of different enzyme
图6. 反应glutamate transport在敲除不同的酶后流量范围的变化
PDH即丙酮酸脱氢酶,酶FUM即延胡索酸酶,酶PPC即磷酸烯醇式丙酮酸羧化酶的纵向分析图附上(如图7~12),其它酶可以用同种方法进行分析。
4. 讨论
综合横向分析和纵向分析,我们可以确定酶ENO即烯醇化酶,酶PDH即丙酮酸脱氢酶,酶FUM即延胡索酸酶,酶PPC即磷酸烯醇式丙酮酸羧化酶等对整个代谢系统的影响最大,以酶ENO为例,通过纵向分析来看,会发现,在ENO敲出后,在大肠杆菌代谢系统中有相当多的反应都受到了影响,而酶的催化作用具有专一性,即只催化一种或一类底物,因此我们可以推测出酶ENO所参与的反应应该是位于大肠杆菌代谢系统比较靠前的的位置,可以为代谢系统中之后的反应提供底物等。再返回汇总数据,可以发现,酶ENO,被敲出后反应代谢系统中反应就会少一个,说明在这些酶所直接参与的反应中,他们的作用是不可替代的,即大肠杆菌代谢系统中ENO酶无同工酶,由此我们也可以基本判定定酶ENO所催化的反应。再观察横向分析图与汇总的数据,会发现酶ENO对反应的影响可以分为两类:
1) ENO酶敲除后,反应被抑制,这又可以被分为
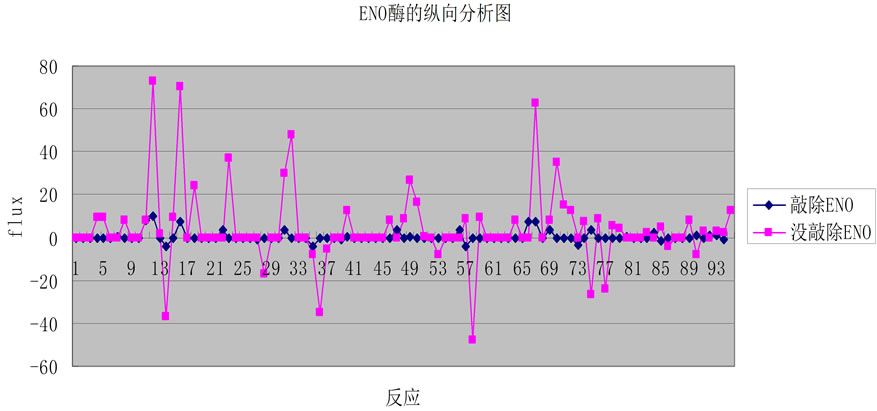
Figure 7. The all reaction FBA knockout enzyme ENO metabolic system results change
图7. 敲除酶ENO代谢系统中全部反应的FBA结果变化
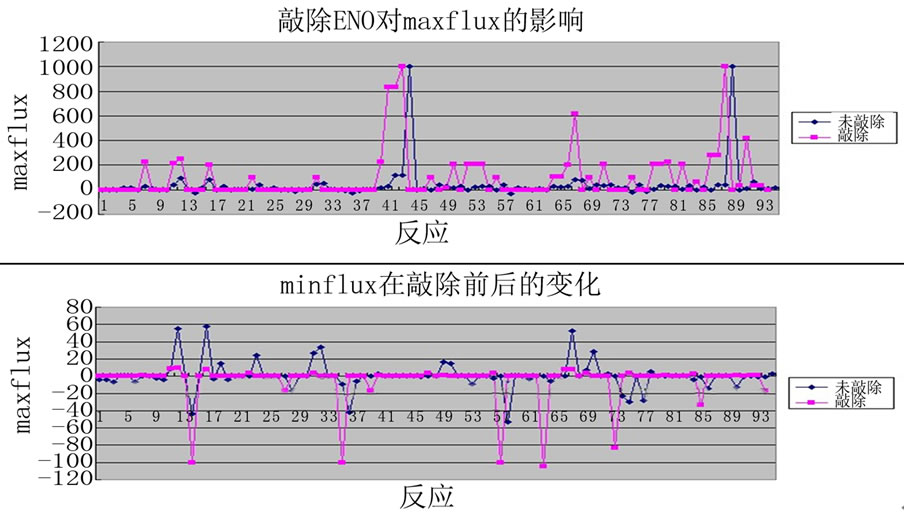
Figure 8. All knockout enzyme ENO metabolic system response changes in FVA results
图8. 敲除酶ENO代谢系统中全部反应FVA结果变化

Figure 9. Changes in the knockout enzyme PDH metabolic system in all the reaction FBA results
图9. 敲除酶PDH代谢系统中全部反应的FBA结果变化

Figure 10. knockout enzyme PDH metabolic system in all reaction FVA results change
图10. 敲除酶PDH代谢系统中全部反应FVA结果变化
两类:a) ENO酶敲出后该反应被抑制,但是并未停止,如反应ATP synthease;b) ENO酶被敲出后该反应完
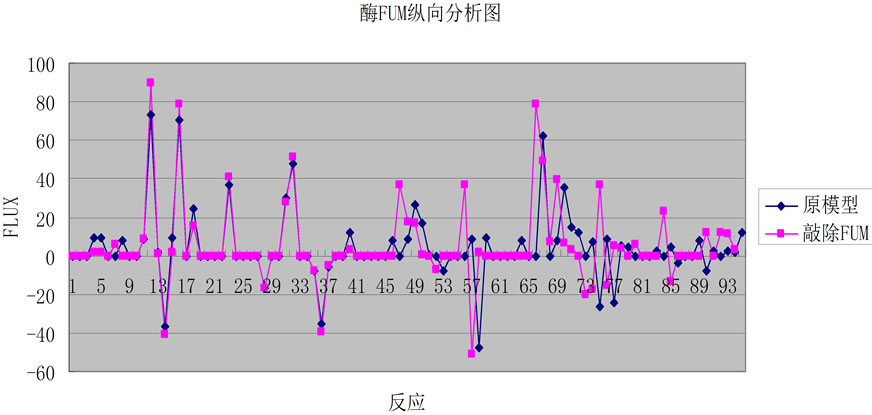
Figure 11. Knockout enzyme the FUM metabolic system, all the reaction the FBA result of changes
图11. 敲除酶FUM代谢系统中全部反应的FBA结果变化
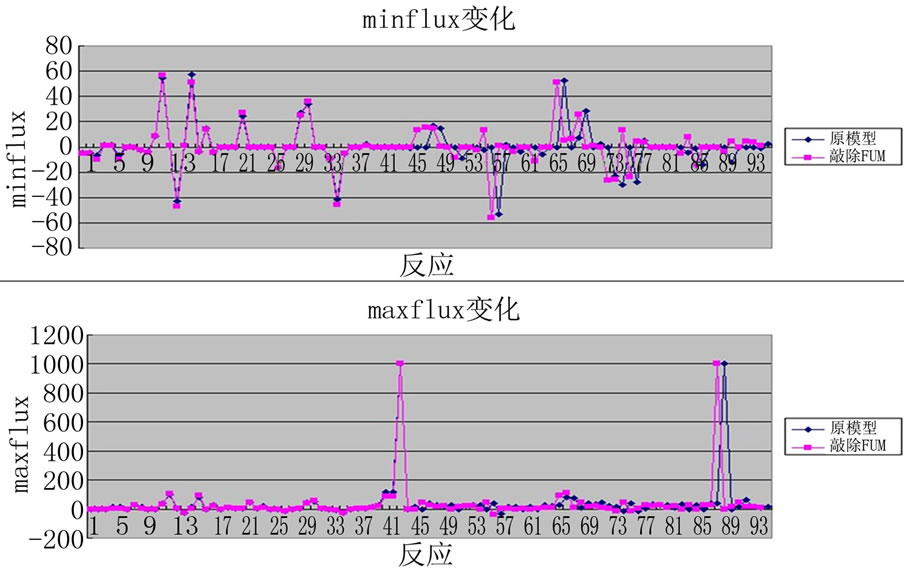
Figure 12. knockout enzyme the FUMF metabolic system, the whole reaction FVA results change
图12. 敲除酶FUM代谢系统中全部反应FVA结果变化
全停止,如反应Biomass。
这说明代谢系统中反应ATP synthease和反应Biomass在酶ENO直接参与的反应后面进行。而且反应Biomass离ENO酶直接参与的反应可能更近一点,这样有利于我们对肠杆菌代谢网络结构的理解。
2) ENO酶被敲出后,反应被促进,如反应glutamate transport。考虑到大肠杆菌是兼性厌氧性微生物,因此猜测:酶ENO可抑制反应glutamate transport的进行或者是抑制反应glutamate transport底物的合成,也可能是酶ENO的产物对反应有抑制作用,即反应glutamate transport可能是大肠杆菌在另一种不同的环境才能进行的。
同理可以应用于大肠杆菌代谢系统中的其他酶和反应的分析,这将有利于我们对大肠杆菌代谢系统的认识。在此所用的大肠杆菌小型中心代谢系统模型,是比较简单的的代谢系统,这样的分析方案不仅适用于这样的小型代谢系统,也可用于对比较复杂的代谢网络分析。
参考文献 (References)
[1] 花强, 杨琛. 代谢流量比率分析及其在代谢工程中的应用[J].生物工程学报, 2009, 25(9): 1303-1311.
[2] 史晋辉. 细胞代谢过程分析及模型优化[J]. 生命的化学, 2001, 21(1): 71-73.
[3] T. Shen, B. Rui, H. Zhou, et al. Metabolic flux ratio analysis and multi-objective optimization revealed a globally conserved and coordinated metabolic response of E. coli to paraquat-induced oxidative stress. Molecular BioSystems, 2012, 9 (1): 121-132.
[4] 杨志杰. 基于代谢流量分析的微生物细胞代谢特性研究[D].华东理工大学, 2011.
[5] 陈琦, 王卓, 魏冬青. 代谢网络流分析进展及应用[J]. 中国科学, 2010, 55(14): 1302-1309.
[6] 丁德武, 何小青, 陆克中等. 用约束法研究大肠杆菌代谢网络核心模型[J]. 计算机与应用化学, 2010, 27(6): 835-838.
[7] 李毅, 郑浩然, 钮俊清等. VFA: 一种可视化代谢网络建模工具[J]. 北京生物医学工程, 2008, 27(5): 490-494.
[8] 谭青乔, 陈宁, 王东洋. 代谢工程发展及应用[J]. 生物技术通讯, 2001, 12(2): 123-126.
[9] 魏春, 陈宁. 代谢网络定量分析研究进展[J]. 生物技术通讯, 2002, 13(3): 234-237.
[10] 马红武, 赵学明, 迟万忠. 应用Excel处理生化过程数据(I)代谢通量分析及代谢网络优化[J]. 计算机与应用化学,1998, 15(6): 274-278.
[11] A. T. Ekaterini. Growth possibilities of E. coli in natural waters. International Journal of Environmental Studies, 1997, 52(1-4): 67-73.
[12] K. Andrejs, S. Egils. Paint4Net.COBRA toolbox extension for visualization of stoichiometric models of metabolism. BioSys-tems, 2012, 109(2): 233-241.
[13] 蒋达. 代谢网络通量和结构分析[D]. 大连理工大学出版, 2006.
[14] 赵静. 细胞代谢网络的结构、功能与进化研究[D]. 上海交通大学, 2008.
[15] J. Schellenberger, R. Que, R. M. T. Fleming, et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox v2.0. Nature, 2011, 21(6): 1290-1307.
NOTES
*基金来源:国家自然科学基金(31200626),贵州省科技厅联合基金(黔科合J字LKS[2012]22号),贵州师范大学博士启动基金。
#通讯作者。