Bioprocess
Vol.2 No.2(2012), Article ID:669,6 pages DOI:10.4236/bp.2012.22009
Overproduction of Lycopene by Metabolic Engineering Escherichia coli
Biotechnology Research Center, School of Life Sciences, Sun Yat-sen University, Guangzhou
Email: *lssljz@mail.sysu.edu.cn
Received: May 4th, 2012; revised: May 21st, 2012; accepted: May 29th, 2012
ABSTRACT:
Lycopene is an effective antioxidant and a potential pharmaceutical drug with anticancer. The knockout of gdhA, aceE or fdhF was beneficial to lycopene production in engineered E. coli. The double (gdhA and aceE) gene knockouts showed a similar effect to the triple (gdhA, aceE and fdhF) on lycopene production. Replacement of native promoter of dxs gene in the double gene knockout strain resulted in 103% increase in lycopene content. In order to avoid application of expensive inducer, we also constructed some constitutive plasmids containing heterologous carotenoid genes. The final engineered E. coli BW25113 (ΔgdhAΔaceE, PT5-dxs, pAC316-WZM4R) produced lycopene of 15.6 mg/g DCW without inducer in a batch shake flask.
Keywords: Lycopene; Escherichia coli; Metabolic Engineering; Promoter Replacement; Constitutive Plasmid
代谢工程大肠杆菌过量生产番茄红素
翁志明,王 玥,刘建忠*
中山大学生命科学学院生物工程研究中心,广州
Email: *lssljz@mail.sysu.edu.cn
摘 要:
番茄红素是一种高效抗氧化剂和潜在抗癌药物。gdhA、aceE和fdhF基因敲除促进了重组大肠杆菌番茄红素的合成,其中gdhA和aceE双基因敲除和gdhA、aceE和fdhF三基因敲除具有相似的作用。在gdhA和aceE双基因敲除的基础上,dxs基因天然启动子被T5启动子置换后,重组大肠杆菌番茄红素的产量提高了103%。为了避免采用诱导剂进行基因表达,构建了一系列组成型质粒,最终构建的代谢工程大肠杆菌BW25113(ΔgdhAΔaceE、PT5-dxs、pAC316-WZM4R)在不需诱导条件下摇床发酵可产番茄红素15.6 mg/g DCW。
收稿日期:2012年5月4日;修回日期:2012年5月21日;录用日期:2012年5月29日
关键词:番茄红素;大肠杆菌;代谢工程;启动子置换;组成型质粒
1. 引言
番茄红素(Lycopene)是类胡萝卜素的一种,是一种很强的抗氧化剂,具有极强的清除自由基的能力,是淬灭单线态氧和清除过氧化氢等自由基能力最强的类胡萝卜素,其猝灭活性是b-胡萝卜素的2倍、维生素E的100倍。对防治前列腺癌、肺癌、乳腺癌、子宫癌等有显著效果,还有预防心脑血管疾病、提高免疫力、延缓衰老等功效,有植物黄金之称,被誉为“21世纪保健品的新宠”。为此,番茄红素的生产引起了各国学者注意。国内研究主要集中在利用三孢布拉氏霉菌(Blakeslea trispom)发酵生产番茄红素。但因三孢布拉氏霉菌(Blakeslea trispom)发酵是一个嗜氧高黏的发酵,生产成本高,而难以与番茄提取法竞争。
近年来,随着代谢工程技术的发展,国际上许多学者通过代谢工程技术将外源番茄红素合成基因簇导入大肠杆菌实现了大肠杆菌生产番茄红素,并取得了可喜的进展[1-11]。这样便可利用大肠杆菌高密度发酵技术高效生产番茄红素,而且因大肠杆菌本身并不能合成番茄红素,当外源番茄红素合成基因簇转入大肠杆菌后,并不会合成其它类胡萝卜素,从而简化了下游提取步骤。但是国际上大都采用诱导性表达载体来表达番茄红素合成基因[1-9],发酵过程需要添加诱导剂,从而会增加生产成本。国内学者在这方面的研究与先进国家的差距,我们在国际学者的研究基础上,采用基因敲除、启动子置换和组成型表达载体的构建等策略,构建了一株不需诱导的代谢工程大肠杆菌以生产番茄红素,其番茄红素的发酵水平达到了国际先进水平。
2. 材料和方法
2.1. 菌株和质粒
表1是本研究所使用的菌株和质粒。大肠杆菌DH5a用于质粒构建,大肠杆菌BW25113为宿主菌。
pAC-LYC04和pAC-LYC由马里兰大学的Cunningham教授惠赠[12]。l-Red重组质粒pKD46、pKD3、pKD4和pCP20用于基因敲除和启动子置换,由普渡大学Wanner教授惠赠[13]。pBAD24由哈佛医学院Beckwith教授惠赠[14]。
2.2. 基因敲除和质粒构建
基因敲除和dxs基因的天然启动子置换采用pKD46表达的l-Red重组系统[13]。本研究所用引物见表2。
利用相应引物从pAC-LYC04中,分别PCR扩增得到crtE、crtI、crtB和ipi基因片段,连接到质粒pBAD24中,分别得到质粒pBAD-crtE、pBAD-crtI、pBAD-crtB和pBAD-ipi。KpnI和XbaI酶切pBAD-crtI、XbaI和SalI酶切pBAD-crtB基因片段连接到KpnI和SalI酶切pBAD-crtE载体片段中,获得质粒pBAD-crtEIB。SalI和HindIII酶切pBAD-ipi基因片段,连接到相同内切酶处理后的pBAD-crtEIB,获得pBAD-crtEIBipi,简称pB-WZM1。EcoRI切pB-WZM1质粒后得到的载体片段(8821 bp),进行CIAP去磷酸化处理后分别同EcoRI酶切pB-rpoS质粒后的rpoS片段、EcoRI酶切pB-DR后的dxs-rpoS片段和EcoRI酶切pB-dxs后的dxs片段连接,得到的质粒经PCR和酶切验证插入方向正确的,分别命名为pBADrpoS-crtEIBipi(简称pB-WZM2)、pBAD-dxs-rpoScrtEIBipi(简称pB-WZM3)和pBAD-dxs-crtEIBipi (简称pB-WZM4)。
BamHI切pAC-LYC后得到载体片段(4893 bp),用Pfu DNA聚合酶进行补平反应后直接连接,得到pACYC315。BamHI-SalI切pAC-LYC后得到载体

Table 1. Strains and plasmids used in the stude
表1. 菌种和质粒


Table 2. Primersa
表2. 引物a
片段(4249 bp),用Pfu DNA聚合酶进行补平反应后直接连接,得到pACYC316。EcoRV分别切pACYC315和pACYC316,得到的4472 bp和3832 bp载体片段分别进行CIAP去磷酸化处理,胶回收产物分别与补平后的NheI酶切自pB-WZM1-4的4种目的片段WZM1、WZM2、WZM3和WZM4进行连接,理论上可以得到正反连接的8种pAC315系列质粒和8种pAC316系列组成型质粒,分别命名为pAC315- WZM1-4F/R和pAC316-WZM1-4F/R(F指WZM1-4正向连接,R指WZM1-4反向连接)。实际得到了13种组成型质粒。
2.3. 培养条件
种子培养基为2YT-KAc,发酵培养基为2YT-KAc和2 × M9-glucose。除了2YT-KAc含0.5% KAc外,其余的发酵培养基在最后发酵的时候均含2 mM KAc。培养基中添加相应抗生素。种子液于37℃,150 rpm中摇过夜后,按1%接种量接种到50 mL发酵培养基中,发酵培养基于30℃,150 rpm中发酵两天后提取番茄红素并定量。
2.4. 分析
发酵液发酵两天后,取少量菌液离心收集菌体,并用蒸馏水洗涤2~3次,沉淀菌体重悬于1 mL丙酮中,抽提液于55℃水浴中保温15 min后,于室温下10,000 rpm离心5 min,取上清液于暗处保存。再用丙酮抽提2次,合并抽提液,通过测定抽提液在472 nm下的吸光度,来计算番茄红素含量。另外,取15 mL以上的发酵液装于50 mL离心管中,于8000 rpm中离心10 min后去上清,沉淀于105℃中烘6 h以上,然后称量菌体干重,根据测出的番茄红素的产量就可算出单位干菌重的番茄红素产量。同时测量发酵液的OD600。
3. 结果与讨论
3.1. 基因敲除
根据Stephanopoulos研究小组代谢通量平衡分析(FBA)显示敲除大肠杆菌的fdhF、aceE和gdhA基因,将促进大肠杆菌番茄红素的合成[2]。为此,我们利用l-Red重组技术,敲除了大肠杆菌BW25113的fdhF、aceE和gdhA基因,并将含番茄红素合成基因簇crtEIB和ipi的质粒pAC-LYC04导入敲除菌中,检测基因敲除对番茄红素合成的影响,结果见图1(a)。从图1(a)可看出,营养丰富的2YT培养基比2M9培养基更适合于番茄红素的合成。为此,以后实验我们采用2YT培养基进行番茄红素的发酵生产。单基因敲除无论在2M9还是在2YT培养基中,对番茄红素合成稍有促进作用,但是多基因敲除对番茄红素合成有较强的促进作用。在2M9培养基中,三基因敲除对番茄红素合成的促进作用最强,这与Stephanopoulos研究小组报道结果一致[2],但是在2YT培养基中,aceE和gdhA双基因敲除和fdhF、aceE和gdhA三基因敲除的促进作用差不多。因此,后面研究中我们以aceE和gdhA双基因敲除菌作为宿主菌。gdhA编码谷氨酸脱氢酶,催化α-酮戊二酸和NADPH转化成谷氨酸,敲除gdhA能够增加NADPH的供应,而合成1 mol番茄红素需要16 mol NADPH[2]。fdhF编码甲醛脱氢酶,催化甲醛转化为CO2,敲除fdhF后可以减少丙酮酸(Pyr)流向甲醛及其副产物,从而提高丙酮酸的供给。aceE编码丙酮酸脱氢复合体的E1亚基(丙酮酸脱氢酶组分),敲除aceE阻止了丙酮酸向乙酰辅酶A的转化,同样可以增加丙酮酸的供给。从图1(b)可看出,基因敲除将抑制菌体的生长,尤其aceE基因的敲除将严重抑制菌体的生长,但当添加KAc后又将削弱因aceE敲除而引起的生长抑制。这主要是由于aceE基因的敲除造成了乙酰辅酶A供给的不足,从而抑制了柠檬酸循环,导致一些氨基酸和ATP的合成受到抑制,因此严重影响了菌体的生长。KAc通过acs编码的乙酰辅酶A合成酶催化生成乙酰辅酶A,添加KAc可弥补因aceE被敲除而导致的乙酰辅酶A供应的不足[15]。实验还发现,添加2 mM KAc能够部分消除敲除aceE对菌体生长造成的影响,而添加0.5% KAc则基本能够完全消除敲除aceE对菌体生长造成的影响(结果未呈现)。
3.2. dxs基因启动子置换
dxs基因编码1-脱氧-D-木酮糖-5-磷酸(DXP)合成酶,催化甘油醛-3-磷酸(G3P)和丙酮酸(Pyr)合成DXP,它是大肠杆菌通过MEP途径合成IPP(合成番茄红素重要前体)的第一个步骤。许多研究发现dxs基因是番茄红素合成的关键基因,dxs基因的过量表达将促进番茄红素的合成[3-6]。可是基于质粒的基因过量表达,虽然能提高基因的表达水平,但同时又将增加代谢
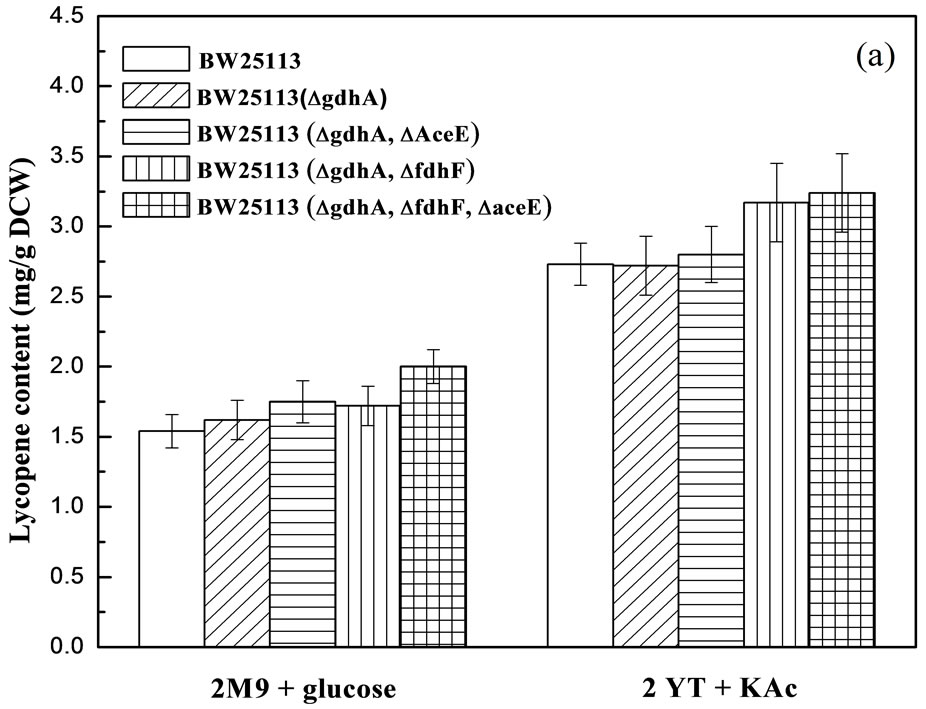
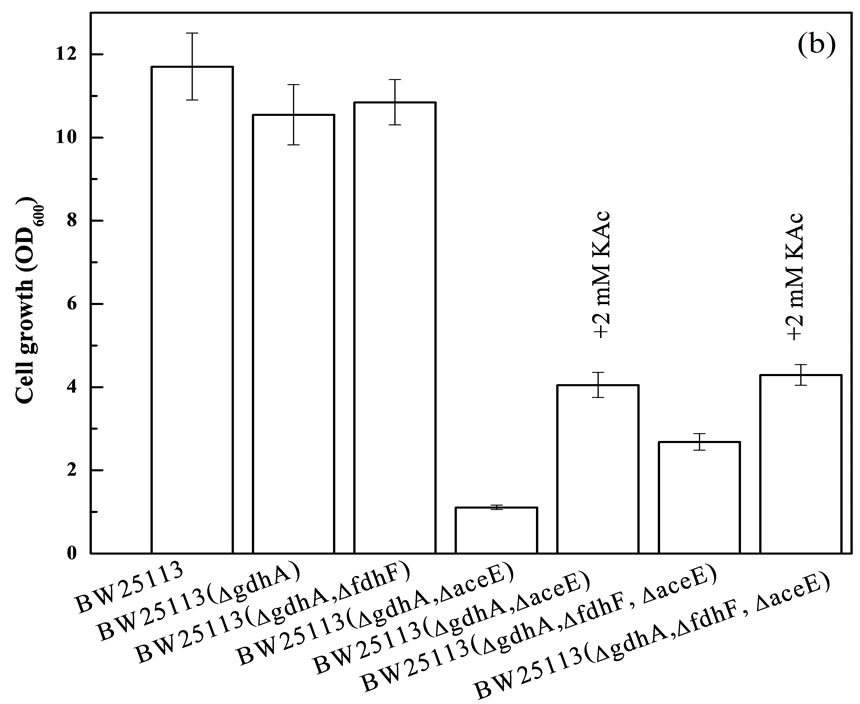
Figure 1. Effects of gene knockout on lycopene production (a) and cell growth (b) in E. coli BW25113 harboring pAC-LYC04
图1. 基因敲除对番茄红素合成(a)及重组大肠杆菌生长(b)的影响
负担、消耗代谢中间产物,而且存在质粒不稳定性(包括结构不稳定性和分离不稳定性)的缺陷[16,17]。增大启动子强度同样是提高基因表达水平的一种方法。近年来,启动子工程已成功应用于代谢工程领域,以调节基因的表达从而提高产物的合成产率[8,9]。为此,我们利用l-Red重组技术,将大肠杆菌BW25113(ΔgdhA ΔaceE)的dxs基因的天然启动子置换成pQE30的T5启动子。结果表明,T5置换导致重组大肠杆菌番茄红素产量提高了103%(表3)。Yuan等在研究b-胡罗卜素时获得了相似的结果,他们研究发现dxs基因的天然启动子置换成T5启动子导致重组大肠杆菌b-胡罗卜素产量提高了1倍[8]。Alper等利用易错PCR技术将PLtetO-1启动子进行随机突变,构建了一个具有不同强度的启动子文库,并将获得的强启动子置换大肠杆菌dxs基因的天然启动子,发现增大启动子强度将有利于重组大肠杆菌番茄红素的合成[9]。
3.3. 组成型质粒的构建
组成型质粒因不需要添加诱导剂,这有利于工业化生产。pAC-LYC04也是一个组成型质粒,它含有草生欧文氏菌(Erwinia herbicola) crtEIB和雨生红球藻(Haematococcus pluvialis) ipi基因,但在crtE和crtI中间存在一段未知基因。Kang等研究发现去除这段未知基因将导致大肠杆菌番茄红素产量提高了3倍[5]。为此,我们从pAC-LYC04中扩增crtE、crtI、crtB和ipi基因,并将其串联在一起,同时与其它关键基因相连接,成功构建获得了13种pAC315(4893 bp)和pAC316(4253 bp)系列的组成型表达载体。图2为代表性的含不同组成型质粒大肠杆菌BW25113产番茄红素的情况。从图2可看出,无论是含pAC315还是含pAC316质粒的重组大肠杆菌合成番茄红素的水平都比对照菌(含pAC-LYC04)高;而且在反向连接表达相同基因时(如WZM1R、WZM3R或WZM4R),采用pAC316系列质粒的番茄红素产量远远高于采用pAC315系列质粒的产量。这主要是由于目的基因距离启动子越远,转录水平相应下降,从而导致代谢终产物产量下降[18]。在正向连接时,基因表达是利用载体的四环素抗性基因的启动子,而反向链接时则利用载体的氯霉素抗性基因启动子。在反向链接时,采用pAC315系列质粒中氯霉素抗性基因启动子与目的基
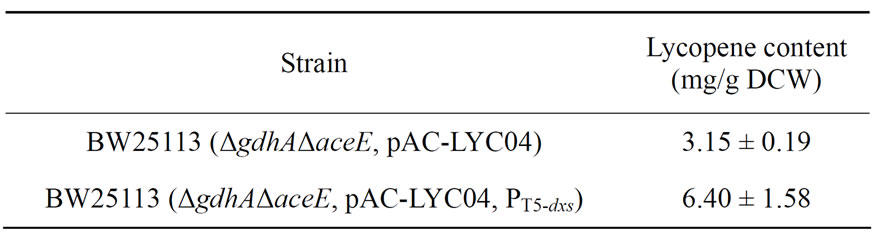
Table 3. Effect of the replacement of native promoter of dxs gene by the promoter of T5 on lycopene production
表3. dxs基因启动子置换的影响

Figure 2. Effects of constitutive plasmids on lycopene production by E. coli BW25113. pAC315(open bars); pAC316 (solid bars). R represents reverse ligation; F represents forward ligation. E. coli BW25113 (pAC-LYC04) was controlled strain
图2. 组成型质粒对野生型大肠杆菌合成番茄红素的影响。pAC315(空白柱);pAC316(黑体柱)。R指反相连接;F指反相连接。大肠杆菌BW25113 (pAC-LYC04)为对照菌
因间距离比pAC316系列质粒的长约640 bp。
将上述筛选得到的番茄红素产量较高的3个质粒pAC315-WZM4F、pAC316-WZM3R和pAC316- WZM4R分别转化到上面的基因敲除菌BW25113 (ΔgdhAΔaceE),考察其番茄红素的合成。结果见图3。结果表明含组成型质粒pAC315-WZM4F、pAC316- WZM3R和pAC316-WZM4R的菌株番茄红素产量都远远高于含pAC-LYC04对照菌,其中含pAC316- WZM3R和pAC316-WZM4R菌产量最高。接着,将它们转化到双基因敲除的dxs启动子T5置换菌BW25113 (ΔgdhAΔaceE,PT5-dxs)中,考察其番茄红素的合成。结果见图3。结果表明,含pAC316-WZM3R和pAC316-WZM4R的dxs启动子T5置换的双基因敲除菌,番茄红素产量得到了进一步提高,其中大肠杆菌BW25113 (ΔgdhAΔaceE,PT5-dxs,pAC316-WZM4R)可产15.47 mg/g DCW)。从图3同样可看出,各种质粒对细胞生长影响不大。
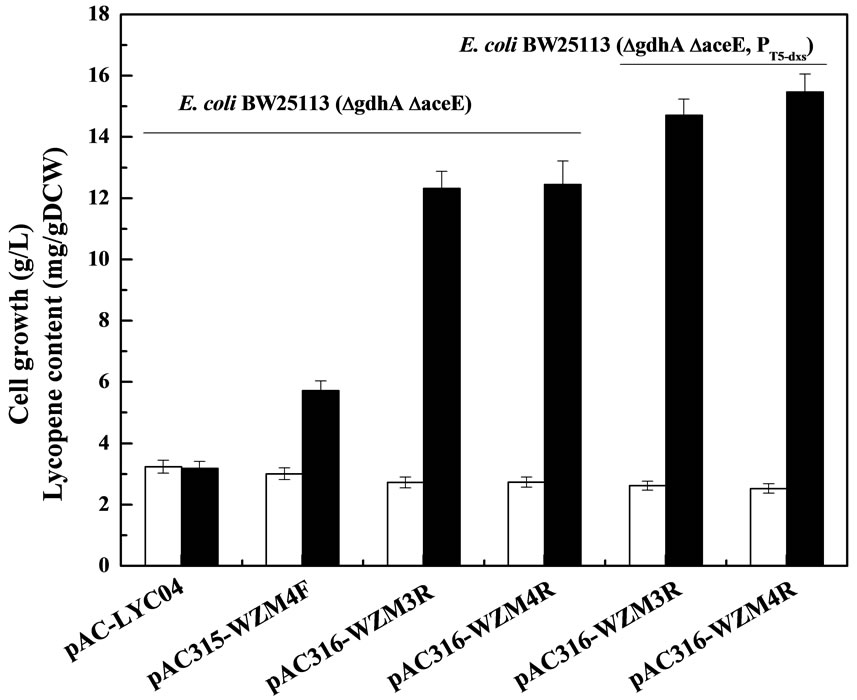
Figure 3. Effects of constitutive plasmids on cell growth (open bars) and lycopene production (solid bars) by engineered E coli
图3. 组成型质粒对工程大肠杆菌生长(空白柱)和合成番茄红素(黑体柱)的影响
3.4. 发酵过程的表征
为了了解最后构建的工程菌培养过程特性,我们测定了大肠杆菌BW25113 (ΔgdhAΔaceE,PT5-dxs,pAC316-WZM4R)不同发酵时间的菌体生长和番茄红素合成,如图4。由图可知,重组大肠杆菌经过短暂的适应期后,很快进入对数生长期,大约26 h细胞生长达到最大值,其后由于营养成分的不足,细胞进入衰老期,细胞浓度开始下降。番茄红素同样在对数生
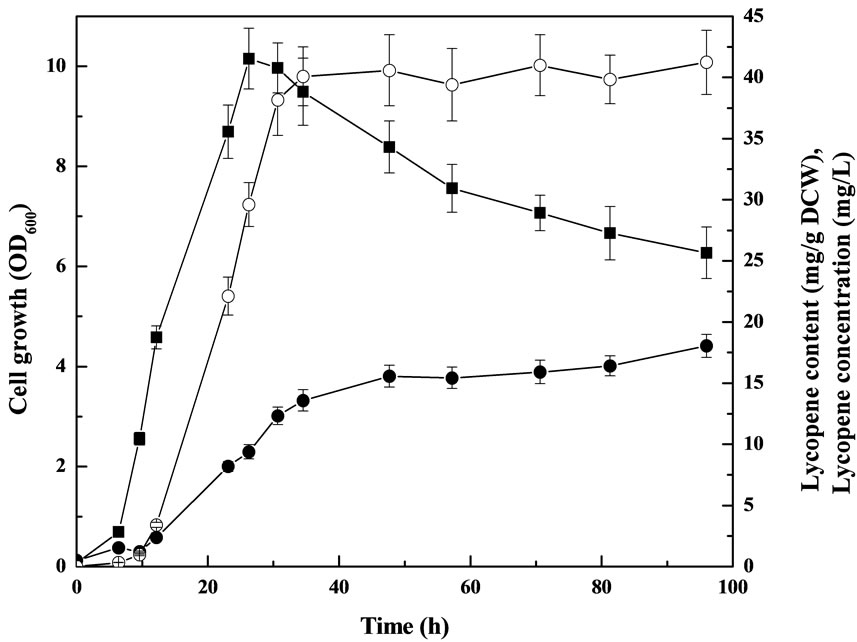
Figure 4. Time course of recombinant E. coli BW25113(ΔgdhAΔaceE, PT5-dxs, pAC316-WZM4R) growth and lycopene production. Cell growth (■), lycopene content (●) and lycopene concentration (○). Data represent the mean of the triplicate cultures ± standard deviation
图4. 重组大肠杆菌BW25113 (ΔgdhAΔaceE,PT5-dxs,pAC316-WZM4R)发酵过程时间曲线。生长(■),番茄红素含量(●)和番茄红素浓度(○),数据为三批发酵的平均值 ± SD
长期开始大量合成,34 h左右番茄红素产量达到最大值(40.1 mg/L),其后番茄红素产量几乎不变,但菌体中番茄红素含量却仍慢速地增加,到48 h菌体中番茄红素含量达到最大值15.6 mg/g DCW,该值接近目前研究重组大肠杆菌产番茄红素最系统的美国麻省理工大学Stephanopoulos教授课题组的先进水平[1-3]。由此可说明,重组大肠杆菌番茄红素的合成是生长耦联的,在发酵后期若能进行营养物质的补充(如采用后期流加补料工艺),番茄红素产量可望进一步提高。
4. 结论
aceE和gdhA基因敲除,同时关键基因dxs天然启动子被强启动子T5置换,大大提高了大肠杆菌合成番茄红素的能力。所构建的大肠杆菌BW25113 (ΔgdhAΔaceE,PT5-dxs)在引入异源胡萝卜素基因后,同样可作为合成其它类胡萝卜素(如b-胡罗卜素、玉米黄素、角黄素、虾青素、海胆酮等)的出发菌株。我们构建的代谢工程大肠杆菌W25113 (ΔgdhAΔaceE,PT5-dxs,pAC316-WZM4R)在发酵培养48 h时,可产番茄红素15.6 mg/g DCW,而且我们构建的工程菌由于采用组成型质粒,不需要诱导剂,从控制生产成本的角度来看,这对放大培养是有利的。为了进一步提高番茄红素的产率,今后的工作一方面可考虑将关键基因整合到染色体上,同时结合其它关键基因启动子的置换,以减轻高拷贝质粒引起的代谢负担,增加工程菌的遗传稳定性;另一方面可尝试对已构建的工程菌W25113 (ΔgdhAΔaceE,PT5-dxs,pAC316-WZM4R)进行发酵培养条件的优化及高密度发酵,以提高番茄红素的产量,为工业化生产奠定基础。
5. 致谢
本项目得到了国际自然科学基金项目(No. 3097 0089)和广东省自然科学基金项目(No. 9351027501 000003,S2011010001396)的资助,在此表示感谢!
参考文献 (References)
[1] H. Alper, G. Stephanopoulos. Uncovering the gene knockout landscape for improved lycopene production in E. coli. Applied Microbiology and Biotechnology, 2008, 78(5): 801-810.
[2] H. Alper, Y. S. Jin, J. F. Moxley, et al. Identifing gene targets for the metabolic engineering of lycopene biosynthesis in Escherichia coli. Metabolic Engineering, 2005, 7(3): 155-164.
[3] Y. S. Jin, G. Stephanoupoulos. Multi-dimensional gene target search for improving locopene biosynthesis in Escherichia coli. Metabolic Engineering, 2007, 9(4): 337-347.
[4] W. R. Farmer, J. C. Liao. Precursor balancing for metabolic engineering of lycopene production in Escherichia coli. Biotechnology Progress, 2001, 17(1): 57-61.
[5] M. J. Kang, S. H. Yoon, Y. M. Lee, S. H. Lee, J. E. Kim, K. H. Jung, Y. C. Shin and S. W. Kim. Enhancement of lycopene production Escherichia coli by optimization of the lycopene synthetic pathway. Journal of Microbiology and Biotechnology, 2005, 15(4): 880-886.
[6] S. H. Yoon, Y. M. Lee, J. E. Kim, S. H. Lee, J. H. Lee, J. Y. Kim, K. H. Jung, Y. C. Shin, J. D. Keasling and S. W. Kim. Enhanced lycopene production in Escherichia coli engineered to synthesize isopentenyl diphosphate and dimethylallyl diphosphate from mevalonate. Biotechnology and Bioengineering, 2006, 94(6): 1025-1032.
[7] L. Z. Yuan, P. E. Rouviére, R. A. LaRossa and W. Suh. Chromosomal promoter replacement of the isoprenoid pathway for enhancing carotenoid production in E. coli. Metabolic Engineering, 2006, 8(1): 79-90.
[8] H. Alper, C. Fischer, E. Nevoigt and G. Stephanopoulos. Tuning genetic control through promoter engineering. Proceedings of the National Academy of Sciences of the USA, 2005, 102(36): 12678-12683.
[9] H. Alper, G. Stephanopoulos. Global transcription machinery engineering: A new approach for improving cellular phenotype. Metabolic Engineering, 2007, 9(3): 258-267.
[10] C. J. Chiang, P. T. Chen and Y. P. Chao. Replicon-free and markerless methods for genomic insertion of DNAs in phage attachment sites and controlled expression of chromosomal genes in Escherichia coli. Biotechnology and Bioengineering, 2008, 101(5): 985-995.
[11] K. E. J. Tyo, P. K. Ajikumar and G. Stephanopoulos. Stabilized gene duplication enables long-term selection-free heterologous pathway expression. Nature Biotechnology, 2009, 27: 760-765.
[12] F. X. Cunningham, Z. Sun, D. Chamovitz, J. Hirschberg and E. Gantt. Molecular structure and enzyme function of lycopene cyclase from the Cyanobacterium Synechococcus sp. Strain PCC7942. The Plant Cell, 1994, 6(8): 1107-1121.
[13] K. A. Datsenko, B. L. Wanner. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy Sciences of the USA, 2000, 97(12): 6640-6645.
[14] L. M. Guzman, D. Belin, M. J. Carson and J. Beckwith. Tight regulation, modulation, and high-level expression by vectors containing the arabinose PBAD promoter. Journal of Bacteriology, 1995, 177(14): 4121-4130.
[15] D. Langley, J. R. Guest. Biochemical genetics of the alpha-keto acid dehydrogenase complexes of Escherichia coli K12: Genetic characterization and regulatory properties of deletion mutants. Journal of General Microbiology, 1978, 106(1): 103-117.
[16] M. O’Connor, M. Peifer and W. Bender. Construction of large DNA segments in Escherichia coli. Science, 1989, 244(4910): 1307-1312.
[17] D. Noack, M. Roth, R. Geuther, G. Müller, K. Undisz, C. Hoffmeier and S. Gáspár. Maintenance and genetic stability of vector plasmids pBR322 and pBR325 in Escherichia coli K12 strains grown in a chemostat. Molecular and General Genetics, 1981, 184(1): 121-124.
[18] T. Nishizaki, K. Tsuge, M. Itaya, N. Doi and H. Yanagawa. Metabolic engineering of carotenoid biosynthesis in Escherichia coli by ordered gene assembly in Bacillus subtilis. Applied and Environmental Microbiology, 2007, 73(4): 1355-1361.
NOTES
*通讯作者。