Biophysics
Vol.1 No.1(2013), Article ID:11835,9 pages DOI:10.12677/biphy.2013.11001
Progress on the EF-Hand Proteins*
Department of Chemistry, Fudan University, Shanghai
Email: 10210220023@fudan.edu.cn, 11210220066@fudan.edu.cn, †yushaoning@fudan.edu.cn
Received: Mar. 15th, 2013; revised: Mar. 19th, 2013; accepted: Apr. 15th, 2013
Copyright © 2013 Ting Yu et al. This is an open access article distributed under the Creative Commons Attribution License, which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited.
ABSTRACT:
Structural the EF-hand Ca2+-binding proteins and calcium have been recognized as the key players in all aspects of cell function, starting with a cell’s birth during mitosis and ending with its apoptotic death. A malfunction in EF-hand proteins-signaling can engender many human diseases. Functionally, EF-hand proteins can be divided into two general classes: the Ca2+ sensors and the Ca2+ buffers. The exceptional versatility of the EF-hand proteins is intimately associated with the diversity of the EF-hand motifs, such as discrepancy in conformations, domain organization, structural responses to calcium and so on. In the present review, we describe the progress on the structure and function of EF-hand proteins, as well as many human diseases caused by signaling dysfunction.
Keywords: EF-Hand Protein; EF-Hand Motif; Calcium
EF-Hand蛋白研究进展*
吁 亭#,赵宇威#,余绍宁†
上海复旦大学化学系,上海
Email: 10210220023@fudan.edu.cn, 11210220066@fudan.edu.cn, †yushaoning@fudan.edu.cn
摘 要:
EF-hand蛋白作为钙离子结合蛋白家族中的特殊成员和钙离子一起参与了从细胞增殖到细胞凋亡各方面的功能调节,EF-hand蛋白调节信号的异常也被认为是人类多种疾病的诱因。按照功能分类,EF-hand蛋白可以分为具有调控功能的Ca2+信号蛋白和只参与Ca2+转运、缓冲的Ca2+缓冲蛋白两大类。EF-hand蛋白功能的多样性与EF-hand结构的构象、组织形式、对钙离子的响应程度等密切相关。本文就EF-hand蛋白结构与功能的差异以及与疾病的关系进行简要综述。
收稿日期:2013年3月15日;修回日期:2013年3月19日;录用日期:2013年4月15日
关键词:EF-Hand蛋白;EF-Hand结构域;钙离子
1. 引言
Krestinger[1]在用X衍射分析小清蛋白(Parval bumin, PV)三维结构时,首次提出了EF-hand结构模型。如图1,这种Ca2+结合蛋白有6个螺旋部分(A、B、C、D、E、F)成对的组成一个类似的结构区域。每个结构区域包括二个含10个氨基酸的α螺旋,中间为一个含10个氨基酸的非螺旋结构的环状区域。每个环状区域内,蛋白质主、侧链上的6~8个氧原子提供电子与Ca2+结合。Krestinger将这种特殊的螺旋–环–螺旋结构称为“EF-hand结构”,拥有EF-hand结构的蛋白称为EF-hand蛋白。这种螺旋结构很像拳头上伸开的

(A) 用右手表示的EF-hand结构的三维结构图:食指表示的是E螺旋(残基1~10),弯曲的中指表示的是经典的钙离子结合环(残基10~21),大拇指表示的是F螺旋(残基19~29)。(B) X射线衍射的小清蛋白结构。(C) 常见的经典EF环的氨基酸序列,1、3、5、6、8和12位为最保守的氨基酸残基,最常见的氨基酸用下划线表示。
Figure 1. The EF-hand motif of parvalbumin[4]
图1. 小清蛋白的EF-hand结构
食指与拇指,E螺旋相当于食指,F螺旋相当于拇指,两者伸出成直角,其根部弯曲的中指,相当于螺旋之间的环,Ca2+正好位于三指的凹陷之中。EF-hand蛋白在钙结合蛋白中非常普遍,对人类全部染色体组的分析表明,共有242个蛋白中含有EF-hand结构[2],同时,从其他物种中确定的含有具有EF-hand结构的蛋白目前已超过600个[3]。其中最著名的成员有钙调蛋白(Calmodulin, CaM)、小清蛋白(Parvalbumin, PV)、肌钙蛋白C (Troponin C, TnC)、calbindin、S100蛋白家族等。
EF-hand蛋白作为钙离子结合蛋白家族中的特殊成员和钙离子一起参与了从细胞增殖到细胞凋亡各方面的功能调节。按照功能分类,EF-hand蛋白可以分为具有调控功能的Ca2+信号蛋白和只参与Ca2+转运、缓冲的Ca2+缓冲蛋白两大类[5]。一般讲,在静息状态时,大多数细胞包括神经细胞和肌肉细胞的Ca2+浓度为50~100 nM,这时EF-hand蛋白在静息细胞中主要以未结合钙离子的形式存在。当细胞受到刺激时,内部储备的钙离子释放或外部的钙离子大量涌入导致钙离子浓度急剧增加,EF-hand蛋白被激活并参与钙离子信号的调节。信号蛋白是EF-hand蛋白家族中最大的一类,它包括CaM、TnC及S100B等大家所熟知的家族成员。信号蛋白能将钙离子浓度增加的化学信号转变为各种生物化学响应,信号转变过程中都会伴随着典型的Ca2+依赖性的构象改变。而缓冲蛋白不会发生Ca2+依赖性的构象改变,它们在结合Ca2+以后主要作为钙离子浓度的调节器,参与对细胞内钙离子浓度的调控,calbindin D9k、calretinin和PV就属于这种类型。EF-hand蛋白功能的多样性与EF-hand结构域的组织形式、构象以及对钙离子的结构响应等密切相关。近年来,随着研究的不断深入,EF-hand蛋白的许多新的功能又相继被发现,它们在人类的免疫系统、神经系统、生殖系统、运动系统等都发挥了突出的作用,EF-hand蛋白功能的失调,也被认为是许多人类疾病如老年痴呆、心肌病、癌症、唐氏综合征的诱因[4,6,7]。本文就EF-hand蛋白结构与功能的差异以及与疾病的关系进行简要综述。
2. EF-Hand蛋白的结构特征
EF-hand蛋白中的EF-hand结构是由一段螺旋–环–螺旋的二级结构组成,EF-hand结构中的钙离子结合区域EF环按照组成和长度的不同可以分为经典与非经典EF环。大部分EF-hand蛋白都含有2、4或6个EF-hand结构,具有很强的成对倾向,形成一个个分离的结构域,不同EF-hand蛋白中的EF-hand结构域可以按照不同的组织形式形成不同的分子构象,这种EF-hand蛋白结构的多样性为EF-hand蛋白对钙离子浓度变化产生不同的信号响应提供了结构基础。
2.1. EF环的结构特征
EF-hand蛋白中的EF-hand结构是由一段螺旋–环–螺旋的二级结构组成的。经典的EF环由12个氨基酸构成,起始和终止氨基酸一般分别为为天冬氨酸和谷氨酸(图1(C))。钙离子与7个氧原子螯合成五角双锥形,其中的5个氧原子由EF环中的9个残基提供,剩下的两个是由出口螺旋侧链中的酸性氨基酸提供的双齿羧酸酯配体。在EF环本身提供的配基中,3或4个来源于负电荷氨基酸侧链的羧基,另一个来源于骨架羰基,与EF环的侧链形成氢键的水分子也常常作为钙离子的配体(图2(A))。EF环的残基一般用两种方式标注:第一种是根据它们的线性位置;第二种是根据它们在五角双锥中的几何分布。与钙离子螯合的配体分布在正交坐标系中:1(+X),3(+Y),5(+Z),
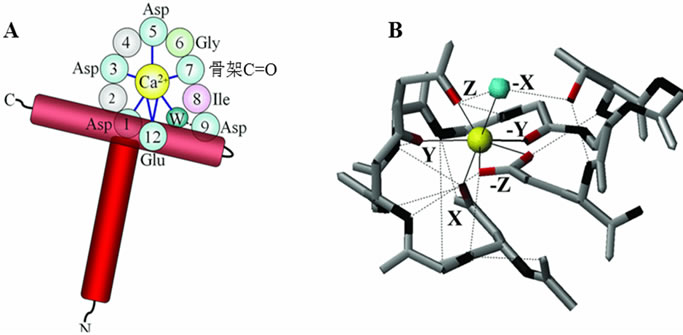
(A) 钙离子螯合结构示意图:入口和出口螺旋用红色标注,蛋白提供的配体用青色标注,水分子(W)用深青色标注,8位的与配对的EF-hand结构形成短的β片的残基用紫红色标注。(B) CaM EF1的钙离子螯合图:钙离子用黄色标注,水分子用青色标注,侧链氧原子用红色标注,骨架的NH基团用黑色。
Figure 2. The co-ordination sphere of the canonical EF-loop[1]
图2. 经典EF环的螯合结构
7(−Y),9(−X),12(−Z)(图2(B))。在所有已知的EF-hand蛋白结构中,EF环-Y位置的残基通过主链羰基氧原子与钙离子结合,和这个残基相邻的疏水氨基酸(大部分是异亮氨酸、缬氨酸或亮氨酸)与配对的EF-hand结构中对应的残基形成两个氢键。这两个氢键是β片的一部分,连接着两个EF环[8]。
尽管大部分的EF-hand蛋白含有的是经典的EF环,但EF环的长度和组成的变化对EF-hand蛋白的多样性意义重大。有些非经典的EF环虽然长度与经典EF环一样,但是结合钙离子方式不同,如调节肌球蛋白轻链(the regulatory light chain of myosin, myosin RLC)12位(−Z)的谷氨酸替换成天冬氨酸,这种氨基酸的替换使得EF-hand蛋白对钙离子的亲和性降低,更倾向于结合镁离子,与钙离子螯合的几何构型为八面体[9]。有些非经典EF环是由于EF环长度的变化,如Calbindin D9k的N端的EF-hand结构的EF环含有14个氨基酸,除了C端的谷氨酸,所有的钙离子配体都来源于主链的羰基氧原子。尽管背离了常规的钙离子结合模式,但Calbindin D9k还是保留了五角双锥的几何构型和对钙离子的高亲和力。除此以外,11个残基的EF环、13个残基的EF环都不断被发现。令人惊讶的是,所有EF环氨基酸的插入或丢失一般都发生在N端,而C端的长度是不变的,且−Y和−Z配体间的氨基酸间距是很保守的[10]。EF环的结构对EF-hand蛋白结合钙离子的性质、结合钙离子后蛋白激活的程度、速度和持续时间等具有重要的影响。
2.2. EF-Hand结构的配对
大部分EF-hand蛋白具有2~8个多拷贝的EFhand结构,而且往往空间上成对排列。成对的EF-hand结构面对面的排列,4个螺旋聚集在一起形成了一个疏水区,两个短的反平行的β片组成的EF环进一步完善了EF-hand结构。结合了Ca2+后EF-hand结构由于Ca2+-配体、极性基团和紧密结合的水分子间的相互作用使得EF-hand结构更加稳定[10]。配对的两个EF-hands结构几乎是2次对称,但这两个EF-hands结构并不是完全一样的[11]。
EF-hand结构的配对现象几乎是无处不在的。首先,假如配对的EF-hands结构中的一个失去了功能,这两个EF-hand结构仍然能够保持配对,这种现象在无脊椎的杂色沙蚕和文昌鱼的肌质Ca2+结合蛋白中(Sarcoplasmic Ca2+-binding proteins,简称SCPs)都可以见到。以沙蚕的SCP为例,由于大量的插入,EF2丧失了功能,但仍然能与具有功能的EF1配对,且几乎是二次对称的[12]。在文昌鱼的SCP中,丧失功能的EF4能够再次和Ca2+结合的EF3配对,但两个EF-hands结构间反平行的β片丢失了[13]。其次,根据对单个的EF-hand结构的肽段的研究得到的EF-hand结构的疏水折叠,为肽段的二聚作用提供了一个很强的驱动力。通过观察不同独立的EF-hand结构肽段聚合形成含有多个EF-hand结构蛋白质的实验,不同蛋白质EF-hand结构二聚的程度已经得到证实,其中包括CaM[14],TnC[15],calbindin D9K[16]和沙蚕SCP[17]等。研究表明,在Ca2+存在下,EF-hand结构肽段能够聚合形成同源二聚体。但将同源二聚体和它们的原来的配对EF-hand结构肽段混合时,EF-hand结构肽段更倾向于形成异源二聚体。异源二聚体和同源二聚体相比对Ca2+的亲和性增加了200倍[18]。有趣的是,除了二聚体的形成,金属离子的结合会诱导EF-hand结构肽段中的无规卷曲向螺旋–环–螺旋的构象转变,这种转变也许对于蛋白质的折叠具有重要的作用[19]。
2.3. EF-Hand的结构域的组织形式
尽管配对的EF-hand的结构是高度保守的,但是通过对两个EF-hands的连结在组成和长度上进行修饰,或者通过改变蛋白质中EF-hand结构域的组织方式,都可以增加EF-hand的结构的多样性。
Calbindin D9k是EF-hand蛋白质家族中最小的蛋白,分子量只有9kDa,只含有一对EF-hand结构。大部分EF-hand蛋白质家族中的成员都含有2对EF-hand结构,每对都与calbindin D9k类似(图3)。根据成对的EF-hands结构域之间的连结的长度和构象的区别,可以将EF-hands结构域的组织形式分为三类:第一类,两个独立的结构域由一个非常灵活的连结连接,以CaM[20]和TnC[21]为例,EF-hands结构域之间柔软的连结使得它们可以采取不同的方位和靶蛋白相互作用;第二种组织方式在恢复蛋白(recoverin)和其他的神经传感器蛋白(Neuronal calcium sensor proteins, NCS)亚家族其他成员中可以见到,两个结构域由一个U型的连结连接,4个EF-hands结构紧凑的串联排布在蛋白质的一个平面上[22]。这种相对的排布方式是由结构域之间和结构域内部的相互作用共同决定的;最后一种组织形式可以在无脊椎SCPs中观察到[13],四个EF-hand结构折叠为紧密的球形结构,两对EF-hand位于球形结构的相反面,事实上,两个结构域高度的非极性表面使得相互间有高度的专一性和亲和性以致于阻碍了对靶蛋白疏水表面的识别和结合[23]。此外,在EF-hand蛋白的大家族中,有些蛋白质含有6个EF-hand结构包括神经保护功能的calbindin D28K和它的同源体calretinin以及内质网调节蛋白亚家族。以calbindin D28K为例,三对EF-hand结构形成一个紧密的、椭圆形的构象,这种构象与NCS类似[24]。
由于EF-hand结构具有很强的成对倾向,因此含有单数EF-hand结构的蛋白质是非常少见的。单个具有功能的EF-hand结构在电压门控钙离子通道1.2的亚基的C端结构域被发现,尽管它的EF环是一段非经典的序列,但是这个EF-hand结构被认为能够结合二价离子且对于维持钙离子的动态平衡具有重要作用[25]。对于含有3对EF-hand结构的小清蛋白亚家族而言,未配对的EF1由于提供氧配体的氨基酸已被其他氨基酸所取代,因此在功能上不能结合Ca2+,但是可以看作结合在C端结构域内生的肽段,以Ca-CaM-肽段的形式稳定配对的Ca2+结合的EF2/EF3结构[26]。有趣的是,如果将EF1结构替换为经典的EF-hand结构并不会恢复其对Ca2+的结合和影响整个结构的稳定性[27]。最后,当一整个亚家族含有5个EF-hand结构,第五个具有功能的EF-hand结构能够发生自缔合作用生成含有10个EF-hand的结构单元[8]。
3. EF-hand蛋白的工作机理
在钙离子调控过程中,许多生物学反应都是通过
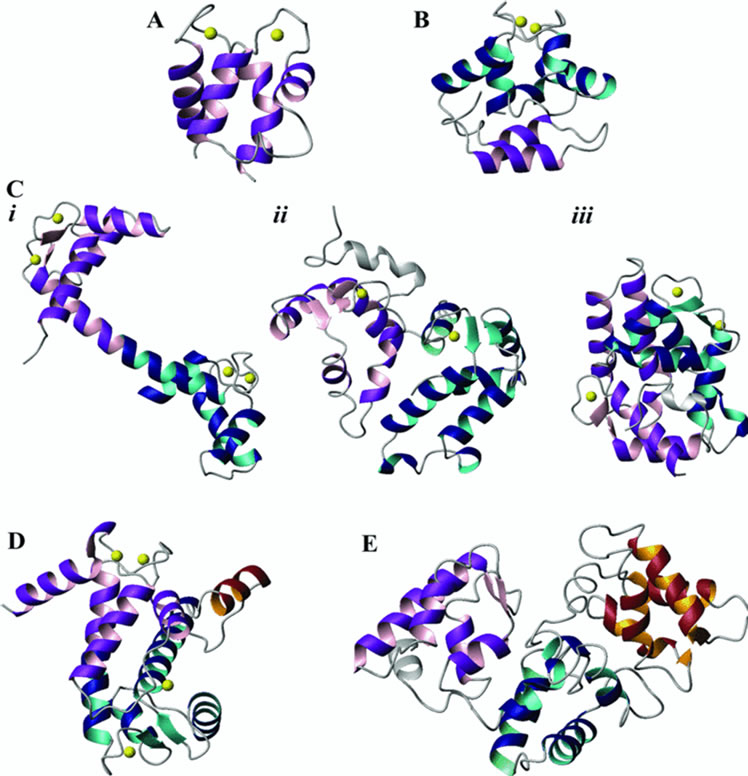
(A) Calbindin D9K (B) Parvalbumin (C) i,CaM; ii,recoverin iii,Nereis SCP (D) Domain VI of calpain (E) Calbindin D28K。在(A)~(E) 第一对配对的EF-hand 结构域用紫红/粉红标注,第二队为蓝色/青绿色,第三对棕色/橘黄色,未配对的EF-hands 结构域用灰色标注。
Figure 3. EF-hand domain organization[6]
图3. EF-hand结构域的组织形式[6]
细胞内钙离子微妙的变化进行触发或调控的。EF-hand蛋白作为细胞内的传感器,在不同范围内,对细胞内钙离子浓度的精确感应是其调控的关键。EF-hand蛋白中的信号蛋白的调节功能主要归功于钙离子诱导的构象变化,导致大量疏水的靶蛋白结合位点暴露。缓冲蛋白作为钙离子浓度的调节器,其功能受到对钙离子的选择性、结合钙离子的动态性等多方面的影响。
3.1. 钙离子诱导的信号蛋白的构象变化
EF-hand蛋白中的信号蛋白的调节功能主要归功于钙离子诱导的构象变化。最早的TnC[28]的晶体结构表明,钙离子只结合在C端结构域。结合了钙离子C端结构域呈现出一个开放的构象,伴随着大面积的疏水区域的暴露,而未结合钙离子的N端结构域呈现出一个封闭的构象,所有的螺旋结构都互相紧密的堆积在一起。Herzberg[29]等认为钙离子的结合会引起结构域的构象由闭合向开放转变,同时伴随着大量疏水基团的暴露,作为和靶蛋白相互作用的位点。这个推论被称为HMJ模型,且已经被大量化学生物和结构数据证明[10]。
HMJ模型同样适用于CaM,CaM是目前研究最多,在所有的真核细胞中分布最广的一个钙离子结合蛋白,介导调控由Ca2+引起的一系列生理生化反应,参与并调节细胞的增生、分化、运动等基本代谢过程[30]。Babu等[20]首次用X光衍射法得到了分辨率0.22 nm的Ca2+-CaM三维晶体结构,从中可以看出CaM为一哑铃型结构,整个分子长度为6.5 nm。柄是一个长α螺旋,每个铃(球区)大约25 × 20 × 20 Å,两铃间无直接接触,一铃上含2个Ca2+结合位,2个Ca2+间距为11.3 Å。CaM共含八段α螺旋,在N端和C端分别含有两个EF环,由这四个EF环与位于两头的部的六个α螺旋区共同形成四个典型的螺旋–环–螺旋EF-hand结构。NMR和X衍射得出的无Ca2+结合的CaM(apo-CaM)的晶体结构与Ca2+-CaM相比[31,32],分子结构有很大的不同(图4)[33]。从整体看,apo-CaM的结构更紧凑、封闭,Ca2+-CaM的结构则较松弛、开放。在apo-CaM中,螺旋Ⅰ与Ⅳ、Ⅴ与Ⅷ在空间上形成分子内相互作用,导致apo-CaM的疏水性残基被α螺旋片段包围,在两端各形成一个结构内部的疏水性核心,只有部分疏水残基暴露在溶液中。apo-CaM C端结构域的这种构象使得细胞在静息状态时,与一些钙不依赖钙调素结合蛋白(Caimodulin binding proteins, CaMBPs)结合,形成有活性的复合物,行使某些重要功能[34]。中心螺旋在Ser-81位置一折为二,两个球部扭转了大约180˚。每个EF-hand结构的2个α螺旋在空间上平行,当EF-hand的钙结合环结合钙后,这2个α螺旋张开成直角,结果导致哑铃球部由沿中心螺旋折叠而伸直,中心螺旋暴露,疏水残基从α螺旋片段之间伸展出去,在每个长把勺末端形成一个面朝外的疏水穴,这两个疏水穴将与中心螺旋共同负责与靶酶及拮抗剂的结合[35]。最近的研究发现,钙离子激活CaM不仅仅是通过每个结构域局部的结构变化,更多的是通过整个分子的形状的改变,针对不同的底物,钙离子能够诱导CaM选择不同的构象与底物相互作用[36]。
Ca2+诱导的这种由封闭到开放的构象变化并不是在所有的EF-hand家族成员中可以看见。S100A6的X衍射的晶体结构表明,结合了钙离子和未结合钙离子的EF1构象非常相似,但是在EF2中,钙离子的结合使得螺旋Ⅲ和螺旋Ⅳ之间的夹角变化了86˚[37],这种钙离子诱导的螺旋Ⅲ的转动在S100B中也可以观察到[38],因此相对于Ca2+诱导的CaM和TnC中EF-hand
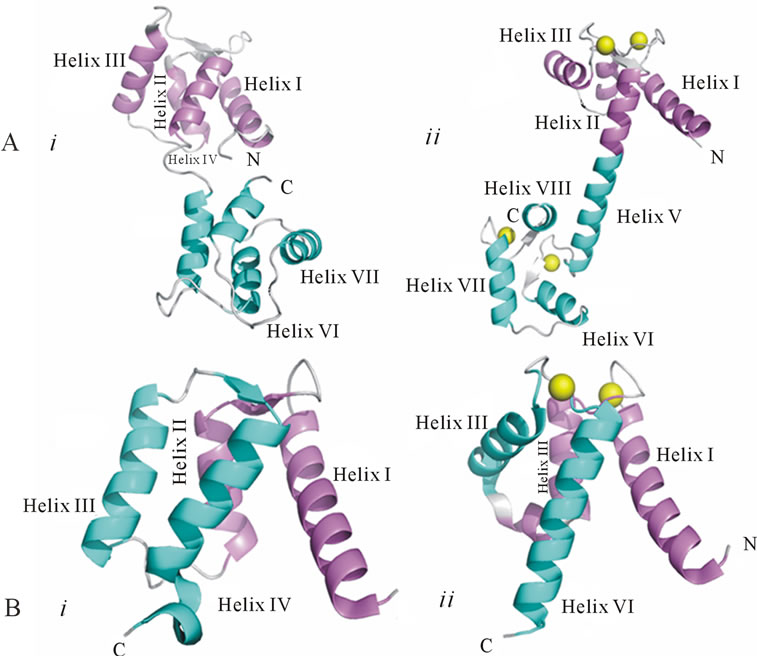
(A) i,apo-CaM; ii,Ca2+-CaM (B) i,apo-S100A6; ii,Ca2+-S100A6。在图中N端的螺旋用紫色标注,C端的螺旋用青色标注,钙离子用黄色标注,非螺旋结构用灰色标注。
Figure 4. Ca2+-induced conformational changes
图4. 钙离子诱导的构象变化
结构域协同变化相比,S100蛋白构象变化属于独立的螺旋的运动。Recoverin和NCS亚家族蛋白质,Ca2+结合引起结构域的转动,这种转动使得埋藏于蛋白质内部的肉豆蔻酰暴露,蛋白质转移到质膜[39]。对于抗药蛋白(sorcin)和其他含有5个EF-hand结构的亚家族成员而言,尽管Ca2+结合对EF-hand结构域本身没有什么影响,但是由于螺旋相互间作用力的改变,C端EF-hand结构域与N端EF-hand结构域的作用力减弱,导致大量疏水的靶蛋白结合位点暴露[40]。
3.2. 缓冲蛋白对钙离子的响应
缓冲蛋白作为钙离子浓度的调节器,其功能主要受以下几个方面影响:a) 细胞质中缓冲蛋白分子的浓度;b) 对钙离子或者其他可能的金属离子如镁离子的亲和性;c) 结合和释放钙离子的动态性;d) 缓冲蛋白自身在细胞内部的流动性。由于许多实际的原因,常常采用Ks(缓冲剂结合的钙离子的浓度变化与游离钙离子浓度的变化的比值)作为衡量许多内生缓冲剂的缓冲能力,特别是当对一种细胞内的钙离子结合蛋白的性质了解不是很清楚的情况下,如运动神经元的缓冲能力很低,Ks < 50;而典型的神经元细胞的Ks在60~200之间;Purkinje细胞由于calbindin D28k和PV的大量表达,Ks的值非常高,约在900~2000之间[4]。
PV是EF-hand蛋白家族中为数不多的含有3对EF-hand结构,但只有2对具有结合离子功能的缓冲蛋白。N端结构域的AB位点虽然不能结合钙离子,但对于维持整个PV分子的稳定起了很重要的作用。CD和EF位点对钙离子具有很高的亲和性,对镁离子的亲和性中等,也就是说CD和EF位点是Ca2+/Mg2+混合结合位点。当细胞处于静息状态时,细胞内镁离子的浓度达到0.5~1 mM[41],80%~90%的PV的Ca2+/Mg2+混合结合位点都被镁离子占据,也就是说,在钙离子结合上去之前,镁离子先要解离。正因为这个原因,PV的钙离子结合率主要由速度相对缓慢的镁离子解离率决定,同时,PV结合钙离子的动态性与缓冲速度很慢的合成缓冲剂EGTA非常相似,因此PV也是一个缓冲速度缓慢的缓冲剂。作为钙离子缓冲剂的另外一个重要参数是它们在细胞内的流动性。PV在水溶液(~140 um2/s)中的扩散系数要比在Purkinje神经元树突(~40 um2/s)中的大得多;在同样的Purkinje神经元其他的细胞结构中,PV的扩散速度降到约11 um2/s[42]。然而当把PV的流动性和一个分子量相似、不会和细胞内其他物质的组分发生特异性反应的右旋糖苷的流动性比较后发现这两个分子流动性下降的趋势是一样的。因此,在不同的细胞组分中PV的流动性不同是由于细胞质的粘度的差异。最近几年,随着越来越多的不同物种和形式的PV的在水溶液中的结构得到解析,研究发现PV虽然内部灵活性较CaM还是比较低,但有些形式的PV还是会发生钙离子诱导的构象变化[43]。以βPV为例,当钙离子从βPV中移除以后,CD位点中的两个螺旋、疏水核心、AB和CD-EF结构域之间的界面都会发生重排,这个发现表明也许βPV也具备信号蛋白的功能[44]。
Calbindin D28k也含有6个EF-hand结构,其中EF2被认为丧失了结合离子的能力,而EF6对钙离子的亲和性太低[45]。EF1、EF3、EF4、EF5位点由于对镁离子的亲和力很低(0.15 M KCl中对镁离子的亲和常数为2.5 × 103 M−1),对钙离子具有高/中等的亲和性(整个分子对钙离子的平均亲和常数为1.4 × 106 M−1),因此被认为是钙离子专一性结合位点[46]。一直以来,Calbindin D28k都被认为是一种典型的缓冲蛋白,也有许多的证据证明在细胞内Calbindin D28k能够作为钙离子的缓冲剂发挥作用,如增加细胞内Calbindin D28k的水平会降低细胞内钙离子的增长速度;将表达Calbindin D28k的基因突变后,钙离子的瞬变增加等。尽管目前大部分的报道针对的是Calbindin D28k对钙离子的缓冲作用,但仍有许多的证据表明Calbindin D28k也能作为信号蛋白发挥作用,如光谱研究结果显示,结合钙离子后,Calbindin D28k的构象发生了变化;在肾脏中,Calbindin D28k能够结合TRPV5并且调节它的活性;另外,作为信号蛋白,对钙离子的选择必须是镁离子的1000倍以上,Calbindin D28k也符合这个条件[46]。但是,如果Calbindin D28k同时也属于信号蛋白,那么它的作用机理是什么呢?2006年鼠的Ca2+-Calbindin D28k的晶体结构得到解析[24],结果表明整个蛋白是由6个不同的EF-hand结构域折叠组成的单一球体,而不是像CaM一样含有两个独立的结构域。同时,尽管Calbindin D28k会发生钙离子诱导的构象变化,但是apo-Calbindin D28k和Ca2+-Calbindin D28k都有疏水区的暴露[47]。因此Calbindin D28k不可能采取和CaM一样只有在结合了钙离子以后才暴露疏水区和靶酶结合的作用模式。对于Ca2+-Calbindin D28k的作用机理,目前还没有定论,还需要进一步的研究。
除了calbindin D28k、PV外,其他许多缓冲蛋白如calbindin D9K、Calretinin等发现都能够发生微小的钙离子诱导的构象变化,具有一些信号蛋白的特征。事实上,在所有EF-hand蛋白中,Ca2+结合和因此发生的构象变化都是由连接两个EF-hand结构域中Ca2+结合环的中间结构决定的,这个中间结构称为“EFβ-scaffold”[10]。EFβ-scaffold能够提供一个多功能的铰链,使得一些键能够发生转动从而引起Ca2+诱导的构象变化,也就是说EF β-scaffold扭矩的灵活性导致了EF-hand构象的多样性。同时,随着研究的深入,许多信号蛋白在细胞内浓度足够高时,也会参与钙离子浓度的调节,信号蛋白与缓冲蛋白两者的界限越来越模糊。
4. EF-Hand蛋白与其他金属离子的作用
除了Ca2+外,与EF-hand蛋白结合并调节其与Ca2+结合的生理功能相关的金属离子还包括Mg2+、Zn2+和Cu2+。与细胞内的Ca2+浓度从静息状态的10−7 M到激活状态的10−5 M较大波动不同[48,49],Mg2+的浓度相对恒定,变化范围一般在0.5~1.0 mM。很多EF-hand蛋白Ca2+结合位点对Mg2+具有亚毫克分子量级的离解常数,因此,在没有Cu2+的竞争的情况下可与Mg2+结合。目前在蛋白质数据库仅有三种Mg2+结合的EF-hand蛋白:小清蛋白(PV),Calbindin D9k(S100G)和扇贝肌球蛋白调节轻链(RLC)。在S100G中,Mg2+只和C端的S100特征EF环结合。EF环呈伸展构象,与没有结合Mg2+的构象类似。Glu12由于距离太远其侧链不能与Mg配位,取而代之的是一个水分子[50],两侧的螺旋部分与Ca2结合构象相比结合更加紧密。Mg2+与PV的环结合时,Glu12的侧链旋转120˚并且只提供一个氧原子与Mg2+配位。Mg2+能通过多种途径影响EF-hand蛋白功能。首先Mg2+和Ca2+对EF-hand蛋白结合位点具有竞争性,因而使得目标酶的激活需要更高的Ca2+浓度;其次,有研究表明Mg2+和Ca2+具有负变构效应[51,52];另外,Mg2+还可以通过与各种不同目标分子作用进而影响EF-hand蛋白的功能,例如最近Persechini的研究显示CaM的每一个结构域的不同构象对应一个特异的、不同的功能[53],Mg2+可能通过调节Ca2+对每个结构域的响应而更复杂的影响Ca2+信号途径。
Zn2+和Cu2+与S100家族的一些蛋白结合[54],其结合位点位于S100两个单体的界面之间[55,56]。由于配体和配位构象均不同,Zn2+、Cu2+与Ca2+和EF-hand蛋白结合不存在竞争关系。许多S100家族蛋白对Zn2+具有很高的亲和力,通过与Zn2+结合精细地调节蛋白的结构和功能[54,57]。S100B和S100A12结合Zn2+后可以提高它们对Ca2+的亲和力,将Zn2+和Ca2+信号通路联系起来[58]。Zn2+的浓度在细胞外可以达到微摩尔到毫摩尔级水平,参与S100分泌蛋白低聚物的形成和细胞外功能[59,60]。S100B是人大脑中含量最丰富的蛋白之一,它可以与Cu2+结合并有效隔绝Cu2+,抑制Cu2+引起的细胞损害,对神经起保护作用[61]。
一些重金属离子结合也可以与EF-hand蛋白结合,引起对Ca2+信号通路的干扰,引起中毒和致病。在分子水平,Pb2+能特异性作用于Ca2+电压门控通道,和TnC[62],并且可以在低浓度刺激CaM信号通路[63]。其他一些金属离子例如Ba2+、Sr2+、Hg2+、Cd2+和大部分镧系元素对包括CaM在内的许多Ca2+结合蛋白同样具有亲和力。最新的对Pb2+、Ba2+和Sr2+与痢疾变形虫的Ca2+结合蛋白-1(EhCaBP1)的晶体学和热力学研究显示其EF-hand结构域可以与几个重金属离子结合并且具有相似的亲和力,EhCaBP1对Pb2+有五个结合位点,对Ba2+和Sr2+有四个结合位点[64]。
5. EF-Hand蛋白功能与疾病
EF-hand蛋白对神经系统、免疫系统、生殖系统、运动系统等都发挥了重要的作用,几乎涉及到细胞内所有的功能调节。许多科学家认为EF-hand蛋白介导的调控异常是导致许多人类疾病的诱因。如CaM依赖的蛋白激酶对免疫系统,神经系统的调节和精子的形成都有重要的影响,当鼠的CaMKIV突变以后,表现出从骨质疏松到男性不育等一系列人类疾病的显著特征[65];Calbindin D28k在老年人以及神经退行性疾病人群中显著减低,老龄老鼠模拟试验显示,Calbindin D28k在整个脑部,其mRNA水平的表达降低了60%~80%;脊髓运动神经元由于缺乏PV和Calbindin D28k的表达而对钙离子介导的损伤非常敏感[66]。另外,许多其他的EF-hand蛋白如:Calcineurin、S100家族、NCS家族等对许多人类疾病的致病机理也密切相关。
5.1. 钙调磷酸化酶
钙调磷酸化酶(Calcineurin, CN)也属于EF-hand家族中的一员,由分子量较大的催化亚基CNA和分子量较小的调节亚基CNB组成,是目前唯一所知依赖钙和钙调蛋白的蛋白磷酸酶。当钙离子结合后,CNB的构象发生变化导致CNA暴露CAM结合位点,Ca2+/CaM的结合进一步使得自抑制结构域从催化结构域移开,CN被激活。CN对钙离子具有很高的亲和性,纳摩尔级浓度的钙离子就能将CN激活。CN在脑内的含量极高,因此许多脑内功能都与之有关,特别是对于神经突触的可塑性和记忆有重要作用。大脑中调节大部分兴奋性突触传导的谷氨酸AMPA受体(AMPA-R)的845位丝氨酸的脱磷酸化会降低神经传导的强度,这可能是长期抑郁现象(Long-term depression, LTD)的原因[6]。除此以外,CN通过脱磷酸化活化T细胞核因子(Nuclear factor of activated T cell, NFAT)使之活化而移位进入核内,然后参与IL-2和其他一些因子的转录过程。CN-NFAT的信号故障能够造成一系列的疾病,如心脏肥大、免疫功能失调、骨质疏松症、老年痴呆、唐氏综合症和癌症等。
5.2. S100蛋白家族
S100蛋白共有21个成员,只在脊柱动物中表达,是EF-hand信号蛋白家族中最大的亚家族。除了S100G(Calbindin D9k)属于缓冲蛋白,所有其他的成员都通过钙离子依赖的形式调节受体蛋白的活性。编码S100A1-S100A16蛋白的基因位于很容易发生染色体重排的1q21染色体上,显示S100家族蛋白和新陈代谢及肿瘤的形成相关[58,67,68]。除S100G(Calbindin D9k)是单体构型外,其他S100蛋白组织形式为紧密的同源二聚体[69],每个亚基由C端“经典的”EF-hand结构和N特有的“假的”EF-hand结构组成。结合钙离子以后,C端发生一个大的构象变化,暴露出与靶蛋白作用的疏水表面。S100的受体蛋白包括酶和非酶蛋白:S100B和S100A1调节涉及能量代谢和信号传导的蛋白酶;S100A8和S100A9调节某些激酶,S100A10调节PLA2,S100A11调节ATPase;S100蛋白家族中的25个成员中的13个与细胞骨架蛋白相互作用,进而调节它们的聚集或者磷酸化状态;有些S100蛋白和转录因子相互作用。S100蛋白功能的多样性与它们在何种类型的细胞内表达、浓度、细胞内的定位、和靶蛋白的协同表达、与不同金属离子结合的性质(Ca2+,Zn2+和Cu2+)、形成同源、异源、低聚物的能力等息息相关。S100蛋白还可以被运输排放到细胞外,激活细胞表面受体[70]。晚期糖化物终极受体(RAGE)是细胞表面受体之一,在糖尿病患者中具有异常高的含量。许多S100蛋白可以和RAGE作用,包括S100A1、S100A2、S100A4、S100A5、S100A6、100A7、S100A8/A9、S100A11、S100A12和S100B等[71],通过RAGE信号通路引起细胞因子和趋化因子的细胞外活性。S100-S100A8/S100A9异二聚体被称作钙网蛋白(CP),在某些白血球细胞中被大量表达。CP直接和=炎症及先天性免疫应答相关,在类风湿性关节炎、慢性支气管炎,AIDS、糖尿病和癌症患者中含量较高[72]。研究证实,在金黄色酿脓葡萄球菌引起的脓肿中,CP可以通过螯合Zn2+和Mn2+引起有效地抗菌活性[73]。最新研究发现S100A8/S100A9通过一个金属离子介导的淀粉蛋白寡聚化和纤维化过程参与淀粉样蛋白的形成[74]。由于S100蛋白家族和人类疾病如,癌症、神经退行性疾病、心肌疾病等的密切联系,近年来它们受到越来越多的关注,S100蛋白对于疾病诊断和作为潜在的药物靶点的价值也日益增加[68]。
5.3. NCS蛋白家族
NCS蛋白家族根据氨基酸序列分为五个亚家族,分别命名为A-E。尽管它们也含有4个EF-hand结构,但CaM的同源性非常低,EF1由于EF环氨基酸的非活性替换丧失了结合钙离子的功能。NCS蛋白家族的结构非常相似,与结合了钙离子的CAM哑铃状的构象不同,它们在结合了钙离子以后结构更加紧密、趋于球形。所有哺乳动物的的NCS A-D亚家族蛋白的N端都豆蔻酰化,有些NCS蛋白如recoverin和NCS B亚家族蛋白只有结合了钙离子以后才暴露出肉豆蔻酰基团。NCS具有一些独特的功能,可以通过十四酰基团锚泊在膜表面[75]。NCS蛋白主要在神经元细胞、心脏细胞和视网膜感光细胞中表达,参与调节神经元和视网膜感光器内许多细胞进程。Recoverin和GCAPs只在视网膜内有表达,这两个亚家族蛋白对光适应有特有的调节作用;KChIPs亚家族的所有成员都能与DNA中的下游调节元件(downstream regulator element, DRE)序列结合并抑制他们的转录。NCS-1被认为是NCS蛋白家族的原始蛋白之一,在不同的物种中序列具有高度保守性。与CaM相比,NCS-1对Ca2+具有更高的亲和力,可以对更低浓度的Ca2+做出响应。NCS-1参与调解神经信号传导,轴突的分支,与学习记忆有关的树突可塑构型。NCS-1能与多巴胺D2受体直接作用并抑制受体由于内化而产生的脱敏反应,NCS-1表达水平的波动可能会引起多巴胺系统活动异常,导致一些精神性和神经性疾病的发生。NCS-1可能与自闭症有关,一个NCS-1的错义突变(R102Q)在一个自闭症患者中被发现[75]。NCS-1的表达水平降低会引起精神病[76],而NCS-1表达水平的升高会导致心率失常[77]。
6. 展望
EF-hand蛋白参与了细胞的多种生理、病理的调控,多年来引起了极大的关注并进行了大量的研究。迄今为止,在人体内共发现242个EF-hand蛋白,同时,从其他物种中确定的EF-hand蛋白也已超过600个,研究领域囊括了EF-hand蛋白的结构与功能、定位、动力学、热力学、致病机理等内容。但是,大量的研究都集中在模型蛋白如CaM、TnC和Calbindin D28k等,其他EF-hand蛋白相对来说获得的关注非常少;另一方面,许多数据还不完善,只有非常少的结合和未结合钙离子形式的EF-hand蛋白高分辨率的X射线晶体结构得到了解析,这对于进一步研究它们的功能和致病机理造成了阻碍。因此,EF-hand蛋白家族还有许多广阔的未知领域等待我们去发掘,要阐明EF-hand蛋白家族的调节机制依旧任重道远。
参考文献(References)
[1] R. H. Kretsinger, C. E. Nockolds. Carp muscle calcium-binding protein II. Structure determination and general description. Journal of Biological Chemistry, 1973, 248(9): 3313-3326.
[2] E. S. Lander, L. M. Linton, B. Birren, C. Nusbaum, et al. Initial sequencing and analysis of the human genome. Nature, 2001, 409(6822): 860-921.
[3] E. Carafoli, L. Santella, D. Branca, et al. Generation control and processing of cellular calcium signals. Critical Reviews in Biochemistry and Molecular Biology, 2001, 36(2): 107-260.
[4] B. Chwaller. The continuing disappearance of “pure” Ca2+ buffers. Cellular and Molecular Life Sciences, 2009, 66(2): 275-300.
[5] A. C. Silva, F. C. Reinach. Calcium binding induces conformational changes in muscle regulatory proteins. Trends in Biochemical Sciences, 1991, 16(2): 53-57.
[6] K. Baumgartel, I. M. Mansuy. Neural functions of calcineurin in synaptic plasticity and memory. Learning and Memory, 2012, 19(9): 375-384.
[7] M. Yanez, J. Gil-Longo and M. Campos-Toimil. Calcium binding proteins. Advances in Experimental Medicine and Biology, 2012, 740: 461-482.
[8] J. L. Gifford, M. P. Walsh and H. J. Vogel. Structures and metalion-binding properties of the Ca2+-binding helix-loop-helix EF-hand motifs. Biochemical Journal, 2007, 405(2): 199-221.
[9] A. C. da Silva, J. Kendrick-Jones and F. C. Reinach. Determinants of Ion Specificity on EF-hands Sites: Conversion of the Ca2+/Mg2+ site of smooth muscle myosin regulatory light chain into a Ca(2+)-specific site. Journal of Biological Chemistry, 1995, 270(12): 6773-6778.
[10] Z. Grabarek. Structural basis for diversity of the EF-hand calciumbinding proteins. Journal of Molecular Biology, 2006, 395(3): 509-525.
[11] N. C. Strynadka, M. N. James. Crystal structures of the helixloop-hlelix calcium-binding proteins. Annual Review of Biochemistry, 1989, 58: 951-998.
[12] S. Vijay-Kumar, W. J. Cook. Structure of a sarcoplasmic calcium-binding protein from Nereis diversicolor refined at 2.0 A resolution. Journal of Molecular Biology, 1992, 224(2): 413-426.
[13] W. J. Cook, L. C. Jeffrey, J. A. Cox, et al. Structure of a sarcoplasmic calcium-binding protein from amphioxus refined at 2.4 A resolution. Jo-urnal of Molecular Biology, 1993, 229(2): 461- 471.
[14] R. E. Reid. Synthetic fragments of calmodulin calcium-binding site III. A test of the acid pair hypothesis. Journal of Molecular Biology, 1990, 265(11): 5971-5976.
[15] G. S. Shaw, R. S. Hodges and B. D. Sykes. Calcium-induced peptide association to form an intact protein domain: 1H NMR structural evidence. Science, 1990, 249(4966): 280-283.
[16] S. Linse, E. Thulin and P. Sellers. Disulfide bonds in homoand heter-odimers of EF-hand subdomains of calbindin D9k: Stability, calcium binding, and NMR studies. Protein Science, 1993, 2(6): 985-1000.
[17] I. Durussel, Y. Luan-Rilliet, T. Petrova, et al. Cation binding and conformation of tryptic fragments of Nereis sarcoplasmic calcium-binding protein: Calcium-induced homoand heterodimerization. Biochemistry, 1993, 32(9): 2394-2400.
[18] R. E. Reid, J. Gariepy, A. K. Saund and R. S. Hodges. Calciuminduced protein folding. Structure-affinity relationships in synthetic analogs of the helix-loop-helix calcium binding unit. Journal of Biological Chemistry, 1981, 256(6): 2742-2751.
[19] M. M. Lopez, D. H. Chin, R. L. Baldwin, et al. The enthalpy of the alanine peptide helix measured by isothermal titration calorimetry using metal-binding to induce helix formation. Proceedings of the National Academy of Sciences, 2002, 99(3): 1298- 1302.
[20] Y. S. Babu, J. S. Sack, T. J. Greenhough, et al. Three-dimensional structure of calmodulin. Nature, 1985, 315(6014): 37-40.
[21] O. Herzberg, M. N. James. Refined crystal structure of troponin C from turkey skeletal muscle at 2.0 A resolution. Journal of Molecular Biology, 1988, 203(3): 761-779.
[22] J. B. Ames, K. B. Hendricks, T. Strahl, et al. Structure and calcium-binding properties of Frq1, a novel calcium sensor in the yeast Saccharomyces cerevisiae. Biochemistry, 2000, 39(40): 12149-12161.
[23] G. Rabah, R. Popescu, J. A. Cox, et al. Solution structure and internal dynamics of NSCP, a compact calcium-binding protein. FEBS Journal, 2005, 272(8): 2022-2036.
[24] D. J. Kojetin, R. A. Venters, D. R. Kordys, et al. Structure, binding interface and hydrophobic transitions of Ca2+-loaded calbindin-D (28K). Nature Structural & Molecular Biology, 2006, 13(7): 641-647.
[25] S. Brunet, T. Scheuer, R. Klevit, et al. Modulation of Ca V1.2 channels by Mg2+ acting at an EF-hand motif in the COOH-terminal domain. The Journal of General Physiology, 2005, 126(4): 311-323.
[26] E. Babini, I. Bertini, F. Capozzi, et al. Principal component analysis of the conformational freedom within the EF-hand superfamily. Journal of Proteome Research, 2005, 4(6): 1961-1971.
[27] J. A. Cox, I. Durussel, D. J. Scott, et al. Remodeling of the AB site of rat parvalbumin and oncomodulin into a canonical EFhand. European Journal of Biochemistry, 1999, 264(3): 790-799.
[28] O. Herzberg, M. N. James. Structure of the calcium regulatory muscle protein troponin-C at 2.8 A resolution. Nature, 1985, 313(6004): 653-659.
[29] O. Herzberg, J. Moult and M. N. James. A model for the Ca2+- induced conformational transition of troponin C. A trigger for muscle contraction. Journal of Biological Chemistry, 1986, 261 (6): 2638-2644.
[30] D, Chin, A, R. Means. Calmodulin: A prototypical calcium sensor. Trends in Cell Biology, 2000, 10: 322-328.
[31] H. Kuboniwa, N. Tjandra, S. Grzesiek, et al. Solution structure of calcium-free calmodulin. Nature. Nature Structural & Molecular Biology, 1995, 2: 768-776.
[32] M. Zhang, T. Tanaka and M. Ikura. Calcium-induced conformational transition revealed by the solution structure of apo-calmodulin. Nature Structural & Molecular Biology, 1995, 2(9): 758- 767.
[33] N. Al-Shanti, C. E. Stewart. Ca2+/calmodulin-dependent transcriptional pathways: Potential editors of skeletal muscle growth and development. Biological reviews of the Cambridge Philosophical Society, 2009, 84(4): 637-652.
[34] M. B. Swindells, M. Ikura. Preformation of the semi-open conformation by the apo-calmodulin C-terminal domain and implications for binding IQ-motifs. Nature Structural & Molecular Biology, 1996, 3(6): 501-504.
[35] B. R. Sorensen, L. A. Faga, R. Hultman, et al. An interdomain linker increases the thermostability and decreases the calcium affinity of the calmodulin N-domain. Biochemistry, 2002, 41(1): 15-20.
[36] N. J. Anthis, M. Doucleff and G. M. Clore. Transient, sparsely populated compact states of apoand alcium-loaded calmodulin probed by paramagnetic relaxation enhancement: Interplay of conformational selection and induced fit. Journal of the American Chemical Society, 2011, 133(46): 18966-18974.
[37] L. R. Otterbein, J. Kordowska, C. Witte-Hoffmann, et al. Crystal structures of S100A6 in the Ca(2+)-free and Ca(2+)-bound states: The calcium sensor mechanism of S100 proteins revealed at atomic resolution. Structure, 2002, 10(4): 557-567.
[38] A. C. Drohat, N. Tjandra, D. M. Baldisseri, et al. The use of dipolar couplings for determining the solution structure of rat apo-S100B (beta-beta). Protein Science, 1999, 8(4): 800-809.
[39] K. L. Yap, J. B. Ames, M. B. Swindells, et al. Diversity of conformational states and changes within the EF-hand protein superfamily. Proteins, 1999, 37(3): 499-507.
[40] G. Colotti, C. Zamparelli, D. Verzili, et al. The W105G and W99G sorcin mutants demonstrate the role of the D helix in the Ca2+-dependent interaction with annexin VII and the cardiac ryanodine receptor. Biochemistry, 2006, 45(41): 12519-12529.
[41] M. Watanabe, M. Konishi. Intracellular calibration of the fluorescent Mg2+ indicator furaptra in rat ventricular myocytes. Pflugers Arch, 2001, 442(1): 35-40.
[42] H. Schmidt, O. Arendt, E. B. Brown, et al. Parvalbumin is freely mobile in axons, somata and nuclei of cerebellar Purkinje neurones. Journal of Neurochemistry, 2007, 100(3): 727-735.
[43] E. Babini, I. Bertini, F. Capozzi, et al. Solution structure of human beta-parvalbumin and structural comparison with its paralog alpha-par-valbumin and with their rat orthologs. Biochemistry, 2004, 43(51): 16076-16085.
[44] M. T. Henzl, J. J. Tanner. Solution structure of Ca2+-free rat betaparvalbumin (oncomodulin). Protein Science, 2007, 16(9): 1914- 1926.
[45] T. Cedervall, I. Andre, C. Selah, et al. Calbindin D28k EF-hand ligand binding and oligomerization: Four high-affinity sites-three modes of action. Biochemistry, 2005, 44(41): 13522-13532.
[46] T. Berggard, S. Miron, P. Onnerfjord, et al. Calbindin D28k exhibits properties characteristic of a Ca2+ sensor. Journal of Biological Chemistry, 2002, 277(19): 16662-16672.
[47] T. Berggard, M. Silow, E. Thulin, et al. Ca(2+)- and H(+)-dependent conformational changes of calbindin D(28k). Biocheistry, 2000, 39(23): 6864-6873.
[48] M. J. Berridge, B. D. Bootman and P Lipp. Calcium—A life and death signal. Nature, 1998, 395: 645-648.
[49] M. J. Berridge, P. Lipp and M. D. Bootman. The versatility and universality of calcium signalling. Nature Reviews Molecular Cell Biology, 2000, 1: 11-21.
[50] M. Andersson, M. A. Malmendal, S. Linse, et al. Structural basis for the negative allostery between Ca2+- and Mg2+-binding in the intracellular Ca2+-receptor calbindin D9k. Protein Science, 1997, 6: 1139-1147.
[51] R. D. Shannon. Revised effective ionic radii and systematic studies of interatomic distances in halides and chalcogenides. Acta Crystallographica Section A, 1976, 32: 751-767.
[52] M. Tanokura, K. Yamada. Calorimetric study of Caand Mgbinding by calmodulin. The Journal of Biochemistry (Tokyo), 1983, 94: 607-609.
[53] Z. Grabarek. Insights into modulation of calcium signaling by magnesium in calmodulin, troponin C and related EF-hand proteins. Biochimica et Biophysica Acta, 2011, 1813: 913-921.
[54] D. J. Black, J. Leonard and A. Persechini. Biphasic Ca-dependent switching in a calmodulin-IQ domain complex. Biochemistry, 2006, 45: 6987-6995.
[55] O. V. Moroz, A. A. Antson, S. J. Grist, et al. Structure of the human S100A12-copper complex: Implications for host parasite defence. Acta Crystallographica Section D, 2003, 59: 859-867.
[56] O. V. Moroz, E. V. Blagova, A. J. Wilkinson, et al. The crystal structures of human S100A12 in apo-form and in complex with zinc: New insights into S100A12 oligomerisation. Journal of Molecular Biology, 2009, 391: 536-551.
[57] H. M. Botelho, M. Koch, G. Fritz, et al. Metal ions modulate the folding and stability of the tumor suppressor protein S100A2. FEBS Journal, 2009, 276: 1776-1786.
[58] M. C. Schaub, C. W. Heizmann. Calcium, troponin, calmodulin, S100 proteins: From myocardial basics to new therapeutic strategies. Biochemical and Biophysical Research Communications, 2008, 369: 247-264
[59] N. Leukert, T. Vogl, K. Strupat, et al. Calcium-dependent tetramer formation of S100A8 and S100A9 is essential for biological activity. Journal of Molecular Biology, 2006, 359: 961-972.
[60] T. Ostendorp, E. Leclerc, A. Galichet, et al. Structural and functional insights into RAGE activation by multimeric S100B, EMBO Journal, 2007, 26: 3868-3878.
[61] T. Nishikawa, I. S. Lee, N. Shiraishi, et al. Identification of S100B proteinas copper-binding protein and its suppression of copperinduced cell damage. Journal of Biological Chemistry, 1997, 272: 23037-23041.
[62] S. H. Chao, C. H. Bu and W. Y. Cheung. Activation of troponin C by Cd2+ and Pb2+. Archives of Toxicology, 1990, 64: 490-496.
[63] J. M. Aramini, T. Hiraoki, M. R. Grace, T. W. Swaddle, et al. NMR and stopped-flow studies of metal ion binding to alphalactalbumins. Biochim Biophys Acta, 1996, 1293: 72-82.
[64] S. Kumar, et al. Flexibility of EF-hand motifs: Structural and thermodynamic studies of calcium binding protein-1 from entamoeba histolytica with Pb2+, Ba2+, and Sr2+. BMC Biophysics, 2012, 5: 15.
[65] C. B. Klee, A. R. Means. Keeping up with calcium: Conference on calcium-binding proteins and calcium function in health and disease. EMBO Reports, 2002, 3(9): 823-827.
[66] Y. Zhu. Roles of calcium binding protein in neural system diseases. Chinese Journal of Biochemistry and Molecular Biology, 2008, 24(5): 413-418.
[67] I. Marenholz, R. C. Lovering and C. W. Heizmann. An update of the S100 nomenclature. Biochimica et Biophysica Acta, 2006, 1763: 1282-1283.
[68] C. W. Heizmann, G. E. Ackermann and A. Galichet. Pathologies involving the S100 proteins and RAGE. Subcellular Biochemistry, 2007, 45: 93-138.
[69] N. J. Skelton, A. M. Kordel, et al. Signal transduction versus buffering activity in Ca2+-binding proteins. Nature Structural Biology, 1994, 1: 239-245.
[70] R. Donato. Intracellular and extracellular roles of S100 proteins. Microscopy Research and Technique, 2003, 60(6): 540-551.
[71] Nacken W,Kerkhoff C.The hetero-oligomeric complex of the S100A8/S100A9 protein is extremely protease resistant. FEBS Letter,2007,581:5127-5130.
[72] C. Gebhardt, J. Nemeth and P. Angel. S100A8 and S100A9 in inflammation and cancer. Biochemical Pharmacology, 2006, 72: 1622-1631.
[73] B. D. Corbin, E. H. Seeley, A. Raab, et al. Celation and inhibition of bacterial growth in tissue abscesses. Science, 2008, 319: 962-965.
[74] K. Yanamandra, O. Alexeyev, V. Zamotin, V. Srivastava, et al. Amyloid frmation by the po-inflammatory S100A8/A9 poteins in the aeing postate. PLoS ONE, 2009, 4: e5562.
[75] M. T. Handley, L. Y. Lian, L. P. Haynes and R. D. Burgoyne. Structural and functional deficits in a neuronal calcium sensor-1 mutant identifiedi nacase of autisticspectrum disorder. PLoS ONE, 2010, 5: e10534.
[76] W. Boehmerle, K. Zhang, M. Sivula, et al. Chronic exposure to paclitaxel diminishes phosphoinositide signaling by cal-painmediated neuronal calcium sensor-1 degradation. Proceedings of the National Academy of Sciences, 2007, 104: 11103-11108.
[77] K. Zhang, F M. Heidrich, B. DeGray, et al. Paclitaxel accelerates spontaneous calcium oscillations in cardiomyocytes by interacting with NCS-1 and the InsP3R. Journal of Molecular Cell Biology Cardiol, 2010, 49: 829-835.
NOTES
*国家自然基金资助项目(No. 21275032,30970631),上海市重点学科资助项目(B109)。
#对本工作同等贡献。
†通讯作者。