Biophysics
Vol.06 No.02(2018), Article ID:24889,24
pages
10.12677/BIPHY.2018.62002
The Dynamical Modeling Studies of the Quorum Sensing Mechanism in Bacteria
Xiaocui Zhang1, Shiyang Huang2, Xiang Li1 ,Yanping Liu1, Yuning Wu1, Jianwei Shuai1*,Zhaoshou Wang2*
1Department of Physics, College of Physical Science and Technology, Xiamen University, Xiamen Fujian
2Institute of Biochemical Engineering, Department of Chemical and Biochemical Engineering, College of Chemistry and Chemical Engineering, Xiamen University, Xiamen Fujian

Received: Apr. 25th, 2018; accepted: May 9th, 2018; published: May 16th, 2018

ABSTRACT
Quorum sensing is a signal exchange between bacteria, which can regulate many important biological functions of bacteria by monitoring their population density. At present researchers study the quorum sensing mechanisms mainly with experiment or mathematical modeling. The quorum sensing signaling pathways of bacteria are extremely complex, but theoretically we can focus on the signaling network modeling of key proteins to quantitatively discuss the dynamical mechanisms of bacterial quorum sensing. This paper reviews the research progress of bacterial quorum sensing network modeling both in the wild bacteria and artificial synthesis bacteria, including Gramnegative bacteria, Grampositive bacteria and the bacteria which both have Gramnegative and Grampositive quorum sensing systems. The various dynamical modeling methods of the quorum sensing systems and their regulation mechanism are discussed in details. Open questions for future modeling are also suggested and prospected.
Keywords:Quorum Sensing, Signal Molecule, Signal Network, Gene Regulation, Bacteria, Dynamic Modeling
细菌群体感应机制的动力学建模研究
张晓翠1,黄世阳2,李翔1,刘艳平1,吴宇宁1,帅建伟1*,王兆守2*
1厦门大学,物理科学与技术学院,物理系,福建 厦门
2厦门大学,化学化工学院,化学工程与生物工程系,生物化工研究所,福建 厦门
收稿日期:2018年4月25日;录用日期:2018年5月9日;发布日期:2018年5月16日

摘 要
群体感应是细菌之间的信号交流,可以通过监控自身群体密度来调控细菌许多重要生物学功能。目前人们主要通过实验和数学建模两种方法来研究群体感应机制,细菌的群体感应信号通路虽然非常复杂,但我们可以通过对关键蛋白的信号网络进行建模,定量研究细菌群体感应核心信号通路的动力学调控机理。本文总结介绍了野生细菌中和人工合成细菌中的群体感应信号网络系统模型,其中野生细菌群体中的群体感应系统包括革兰氏阴性菌、革兰氏阳性菌、革兰氏阳性菌和阴性菌共有的群体感应系统三类,我们分别详细讨论了几种具有代表性的细菌中群体感应系统的各种信号网络动力学建模方法及其动力学调控研究进展,也对未来进一步的建模研究进行了展望。
关键词 :群体感应,信号分子,信号网络,基因调控,细菌,动力学建模

Copyright © 2018 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 细菌的群体感应
二十世纪七十年代,人们在研究费氏弧菌(Vibrio fischeri)和哈氏弧菌(Vibrio harveyi)生物发光现象时发现了群体感应 [1] 。费氏弧菌生长于夏威夷鱿鱼的发光器官中,当细菌达到一定的密度后,就会诱导发光基因的表达从而发光。细菌的生物发光为鱿鱼提供光源以躲避敌害,保护自身 [2] 。细菌也以此来获得合适的栖息场所。细菌群体感应的研究已成为国内外研究的热点,国内的科学家也开始重视这一研究并参与其中。
细菌在生长环境中能够感知与其共同生长的其他细菌所分泌的一种小分子量的化学信号分子的浓度变化,从而促进细菌个体间相互交流,协调群体行为,该现象称为群体感应 [3] 。这些信号分子由细菌分泌并能调节自身生物行为,所以叫做自诱导因子(Autoinducers, AIs)。它的浓度会随着细菌数的增长而增长,一旦自诱导因子的浓度达到一定的阈值,它会激活或抑制一些目的基因的表达,从而调控细菌的群体生物行为 [4] 。群体感应参与调控细菌的多种生物行为,如生物发光、细胞膜的形成、细胞分化、胞外多糖的形成、运动性、抗生素的生成等 [5] 。
群体感应系统介导细菌生物膜的形成主要在膜的生长期和散播期发挥作用:生长期是指细菌的不可逆粘附与积累时期,这一阶段的特点是由于胞外粘性基质的分泌,菌体大量粘附聚集,信号分子感知细胞密度,达到阈值后引起细胞内特定RNA转录,促进生物膜的成熟 [6] ;散播期则是在生物膜成熟后,这一阶段由于生物膜内部细菌的代谢废物只能通过间质水通道排除,大量粘附聚集的细菌导致有限的空间内代谢废物的积累,出现种内或种间竞争。
1.1. 革兰氏阴性菌的群体感应系统
以脂肪类衍生物作为信号分子的群体感应系统,主要存在于革兰氏阴性菌中,它的信号分子大部分属于N-酰化高丝氨酸内酯(N-acylated homoserine lactones, AHLs)类 [7] 。在费氏弧菌的群体感应中主要有两种组分参与,LuxI蛋白和LuxR蛋白。LuxI蛋白是自诱导因子AHL的催化合成酶 [8] [9] ,LuxR蛋白是细胞质内的自诱导信号分子AHL的感受因子,也是一种DNA结合转录激活原件。AHL可以自由进出细胞内外 [10] ,且其浓度随着细胞密度的增加而积累。当这种信号分子的浓度积累到一定阈值时就与LuxR蛋白结合,结合后的复合物能激活荧光素酶基因转录,从而使其发光 [11] 。费氏弧菌的LuxI/LuxR调节模式被视为革兰氏阴性细菌群体感应调节系统的经典模式。
在几乎所有已知的革兰氏阴性细菌的群体感应系统中发现四个共同点 [2] 。首先,在这样的系统中的信号分子大部分属于N-酰化高丝氨酸内酯类,它们能够通过细胞膜自由扩散 [12] [13] [14] [15] 。第二,信号分子绑定在特定的受体上驻留在膜内或细胞质中。第三,群体感应通常会改变几十到几百个基因,支持各种生物过程 [17] [18] 。第四,诱导因子驱动的群体感应信号网络将进一步刺激激活自诱导因子的合成增加,从而建立了一个正反馈回路来促进群体中的同步基因表达,这一过程被称为自身诱导 [19] [20] 。
图1是革兰氏阴性菌中LuxI/LuxR型群体感应系统,该类型的群体感应系统涉及LuxI蛋白、LuxR蛋白、信号分子AHL。信号分子AHL是在酶LuxI蛋白的催化作用下合成的,它的浓度随着细胞密度的增加而增加,合成的信号分子AHL绑定到LuxR蛋白上,形成的复合体诱导目的基因的表达。图1(a)是细胞密度比较低时,细胞内外信号分子的浓度都很低,激活的LuxR蛋白的浓度也很低。图1(b)是细胞密度比较高时,信号分子绑定到LuxR蛋白来激活LuxR蛋白,从而诱导下游目的基因的表达。
1.2. 革兰氏阳性菌的群体感应系统
以短肽类为信号分子的群体感应系统,主要存在于革兰氏阳性菌中,它的群体感应信号分子是自诱导短肽(Autoinducing peptide, AIP) [21] 分子,通常由5~17个氨基酸组成,而氨基酸的侧链通常含有修饰性基团,如异戊烯基基团(芽孢杆菌属)、硫内酯环(葡萄球菌属) [22] 。当AIP在细胞中产生并积累到一定浓度后,可以作为信号分子被菌体上的AIP识别系统识别并产生相互作用。该识别系统主要起作用的是组氨酸激酶双元信号转导系统的受体分子 [23] [24] [25] 。AIP与受体的结合可以激活该双元系统的激酶活性,使受体发生磷酸化,并将磷酸化的受体传递给细胞质中的调节子。经磷酸化的调节子能够激活相关基因的转录表达,刺激产生更多的AIP分子,从而产生群体感应 [26] 。
图2是革兰氏阳性菌寡肽介导的群体感应系统,它的群体感应信号分子是由肽前体基因的诱导物诱导生成,修饰后由ABC转运蛋白输出到细胞外的。信号分子在细胞外积累并由H物质识别,H物质通过结合CSP自磷酸化。然后H通过将磷酸集团转移到它的同源响应调节子D上来进一步转移信号,结合了磷酸集团的D可以诱导和增强目的基因的表达。
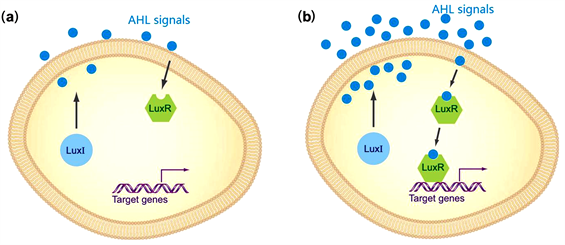
Figure 1. The LuxI/LuxR quorum sensing system of Gramnegative bacteria [16]
图1. 革兰氏阴性菌中LuxI/LuxR型群体感应系统 [16]

Figure 2. Oligopeptide mediated quorum sensing system in Grampositive bacteria [27]
图2. 革兰氏阳性菌寡肽介导的群体感应系统 [27]
不同革兰氏阳性菌的信号肽的结构各不相同 [25] ,而且,AIP的化学结构与受体结合效率密切相关。通常大多数革兰氏阳性菌,还有部分革兰氏阴性菌利用该系统进行种内之间的联系 [16] 。
1.3. 革兰氏阳性菌和阴性菌共有的群体感应系统
20世纪90年代,在多个革兰氏阴性菌中发现了另外一套群体感应系统,它的群体感应自诱导因子由LuxS蛋白形成,称为自诱导因子2 (Autoinducer 2,AI-2),其主要成分为呋喃酮酰硼酸二酯(Furanosyl borate diester) [28] [29] 。经磷酸化的调节子能够激活相关基因的转录,它可以参与革兰氏阳性和阴性菌的群体感应 [23] ,一般认为这种信号分子是种间细胞交流的通用信号分子 [8] [30] 。如在哈氏弧菌中,它的群体感应系统产生AHL类信号分子,但对它的检测却是一种类似革兰氏阳性细菌的双组份磷酸传递系统 [28] 。目前发现它还产生另外一种信号分子AI-2,AI-2分子的受体是LuxP蛋白 [31] 。LuxP-AI-2复合物结合到另一种蛋白LuxQ,LuxQ蛋白包含一个传感器激酶区和反应调节区 [32] [33] [34] [35] 。当细菌密度低并且缺乏AI-2时,在LuxU中间蛋白的作用下,LuxQ将LuxO磷酸化。然后,磷酸化的LuxO激活了抑制荧光素酶操纵子转录的抑制蛋白的转录 [22] [36] [37] 。当细菌密度过高时,AI-2的出现,促使LuxQ磷酸化酶活性降低,从而使LuxO失活,这样导致LuxR介导荧光素酶的转录 [38] [39] [40] [41] 。
图3是哈氏弧菌中的混合群体感应系统。它的信号分子由LuxS生成,生成的信号分子输出到细胞外并在细胞外积累。积累的信号分子由转运蛋白LuxP转运到细胞内,结合了信号分子的LuxP将LuxQ磷酸化。磷酸化的LuxQ又将磷酸集团转移给LuxO,结合了磷酸集团的LuxO诱导目的基因的表达。
1.4. 其他群体感应系统
已经大量研究的细菌群体感应系统中的信号分子包括AHL、AIP、AI-2。除了这三种信号分子,细菌之间还可以利用其他信号分子来感知细胞浓度变化及周围环境的改变,调控相关基因的表达及生理活动,更好地适应环境的改变。

Figure 3. The hybrid quorum sensing system circuit of V. harveyi [27]
图3. 哈氏弧菌中的混合群体感应系统 [27]
目前,研究发现病原微生物铜绿假单胞菌(Pseudomonas aeruginosa)还能够产生第四种信号分子喹诺酮类(Quinolone, POS)。间接地参与细菌种群之间的基因调控和生理活动 [4] 。此外,革兰氏阴性菌中还有一类特殊的信号分子环二肽。它像一般的信号分子一样,可以和调控蛋白结合形成具有调控细菌基因表达的二聚体。还有,土壤中的革兰氏阳性菌链霉菌(Streptomycetes)可以利用r-丁酸内酯介导的群体感应系统调控细胞的形态分化过程。
2. 人工合成细菌的群体感应研究
细菌群体感应最早是在海洋细菌中发现的,所以研究海洋细菌中的群体感应系统机制对研究细菌群体感应系统具有非常重要的作用。但是直接研究海洋细菌中的群体感应机制操作起来并不方便,于是人们人工合成了海洋细菌中的群体感应系统并将其植入大肠杆菌(Escherichia coli)细胞内,因为大肠杆菌细胞自身并不含海洋细菌中的群体感应系统,且大肠杆菌的培养简单、便捷,人们通过人工合成细菌中的群体感应系统来更便捷、深入地研究海洋细菌中的群体感应系统。
2.1. 合成生物学的概念
1980年,德国科学家芭芭拉.荷本(Barbara Hobom)第一次开始用合成生物学这个学术名词来表述基因重组技术 [42] 。在工程学思想的指导下,合成生物学旨在利用测序技术、生物工程技术、化学合成技术、计算机建模及模拟技术等,从头设计并构建新的生物组件、设备和系统,或对现有的、天然的生物系统进行重新设计和改造,达到利用工程化的生物系统或生物模块来处理信息、制造材料、提供食物、操作化合物、生产能源、保持和增强人类健康及改善环境等目的 [42] [43] [44] 。传统的生物学是通过解剖生命体以研究其内在的构造,而合成生物学的研究方法完全是相反的,它是从最基本的要素开始逐步构建生物体的零部件直至人工生命系统 [45] 。
从内容上来说,虽然合成生物学与转基因生物技术存在一定的重叠,但合成生物学要比转基因生物技术更加复杂与宽泛。转基因生物技术是把一个物种的基因延续、改变并转移到另一物种体内 [46] [47] ,而合成生物学的目标是通过自然与合成遗传物质的某些具体组分的组合来设计、研制和制造功能生物体,而其最终目标是完全独立、可自我繁殖、可表现稳定功能的生物部件、体系以及人造细胞和生物个体 [16] 。
目前,合成生物学的研究主要集中在三方面:1) 生物元件标准化及生物模块的设计与构建;2) 最小基因组研究;3) 基因组的设计、合成与组装。
合成生物学尚处于初级发展阶段,还面临一些挑战:1) 还未了解清楚合成生物学必需的分子器件,如基因启动子、报告基因、阻遏蛋白等的功能和特性,使其利用存在瓶颈;2) 合成生物学研究的技术发展还不够成熟,研究进展缓慢。
合成生物学的发展引发的主要问题体现在:合成生物学是否挑战了传统上的生命观念,合成生物学能否得到伦理上的辩护,现有的评估和监管制度能否解决合成生物学释放到环境中的产物所引发的生物安全和生物防护问题等等。
2.2. 人工合成细菌的群体感应系统实验研究介绍
You等 [48] 在2004年发表在Nature期刊上的文章重新构建了两个完全不同的基因片段,并把它们植入大肠杆菌细胞内,通过实验及模型证明了这两个片段可以互相耦合,并通过细胞交流的方式调控群体密度。
2006年,Li等 [49] 人工合成了大肠杆菌中AI-2群体信号分子的合成网络,通过哈氏弧菌中群体感应信号分子的模块来研究大肠杆菌中的AI-2群体信号分子的合成路径。得出了以下结论:1) 当细胞生长在葡萄糖存在的环境中,mRNA的转录和蛋白水平上AI-2的合成、Pfs和LuxS都不能显著提升AI-2的浓度;2) 在葡萄糖的存在下,这种合成途径伴随着代谢通量的增加,只是部分地解释了这种差异。他们预测,高通量的替代途径或其他生物步骤存在于AI-2的合成中。这些结果也得到了实验数据的验证。
2010年,Marguet等 [19] 构建了一个基因回路,它会引起细菌种群密度随时间的变化。基因回路含有两个模块,第一个模块赋予细胞杀死自身的作用,第二个模块是密度传感。他们的实验表明在含有合成自杀回路的大肠杆菌群体中产生的振荡不需要群体感应基因(luxR和luxI),也不需要已知的PLuxI启动子中的调控元件。相反,振荡很可能是由于密度依赖质粒扩增建立的群体水平负反馈。
2013年,Saeidi等 [50] 人工合成了铜绿假单胞菌中的群体感应体系,并将其植入到大肠杆菌体内,通过测群体荧光强度来测量群体密度,他们的实验数据验证了他们建立的模型的正确性。
2013年,Daniel等 [51] 合成的模拟基因回路利用反馈来实现对数线性传感、比率和幂的计算。他们的回路可以被用来实现高阶函数,可以用复杂的生化模型和简单的数学函数描述。
2014年,Wang等 [52] 人工构建了LuxI/LuxR群体感应系统,并将其植入到大肠杆菌体内,通过调控细胞群体密度来研究LuxI/LuxR群体感应系统,对一系列标准化细胞浓度控制元件的工作性能进行了考察。实验结果表明,含有RBS0.07群体感应通路的工程菌能够对细胞浓度进行自我调整,相对于未含有该通路的菌株,该通路延长了菌体生长的稳定期。通过对含有不同效率RBS细胞浓度控制元件的菌株稳定期活菌数进行测定发现:RBS效率越高其稳定期活菌数越低。
2015年,Chen等 [22] 描述了一种由两类不同的细胞组成的合成微生物集团:“活化剂”菌株和“阻遏物”菌株。这些菌株产生两个正交的细胞信号分子,在两个菌株的合成回路内调节基因表达。这两种菌株只有在一起培养时才会产生明显的种群水平振荡。两种应变回路的某些网络拓扑结构比其他回路更好地保持鲁棒性。通过多个合作菌株的基因工程来规划种群水平动态的能力,为多种细胞类型中人工合成组织和器官的设计指明了方向。
2016年,Huang等 [53] 构建了一个信号网络来调控细菌的存活。工程菌在医学和环境方面有很大的应用潜力。实现工程菌的应用需要工程行为的可控性和工程系统的可伸缩性。在此,他们提出了一种平台技术,即微生物群,其采用空间布局来控制工程菌的生长动态。他们构建了几种合成基因回路来调控在大肠杆菌中的集体生存:只有在足够高的种群中,工程菌才能存活。他们将工程菌群封装成空心水凝胶微胶囊,作为微生物群的基本单元,从而实现种群的空间布局。当这些细菌被可渗透膜包裹时,能感应到膜内的环境并作出相应的反应。由于其较高的密度,渗透膜包裹的细胞将存活下来。然而,那些从渗透膜中逃脱的细菌,由于密度的降低,将会被杀死。他们证明了这个设计概念是模块化的,易于推广。他们的工作为混合生物材料系统的工程集成和可编程控制奠定了基础。
合成生物学的出现为人们研究细菌群体感应的机制提供了更便捷、直观的方法,人们可以更准确、深入地研究细菌群体感应。
3. 群体感应研究中的数学模型
为了研究细菌中的群体感应机制,人们对细菌中的群体感应体系建立模型,运用数学的方法来分析研究细菌群体感应机制。本文概述了野生细菌中的群体感应系统模型及人工合成的细菌群体感应系统模型,其中野生细菌中的群体感应系统模型又分革兰氏阴性菌、革兰氏阳性菌、革兰氏阳性菌和阴性菌共有的群体感应系统三类,分别详细讨论了几种具有代表性的细菌中的群体感应系统模型。
3.1. 革兰氏阴性菌中的群体感应模型
革兰氏阴性菌包括许多种细菌,例如铜绿假单胞菌、苜蓿根瘤菌(Sinorhizobium meliloti)、恶臭假单胞菌(Pseudomonas putida)、豆科植物根瘤菌(Rhizobium leguminosarum)、根癌土壤杆菌(Agrobacterium tumefaciens)等,下面我们选取一些典型的革兰氏阴性菌作为代表来分别介绍它们的群体感应体系。
3.1.1. Dockery模型
2001年,基于已知的生物化学中对信号机制极其重要的自诱导因子的调节,Dockery等 [54] 针对铜绿假单胞菌中的群体感应系统建立了一个数学模型。Dockery等的模型中只考虑Las系统,信号网络见图4。

Figure 4. Schematic diagram showing the gene regulation for the las and rhl systems in P. aeruginosa [54]
图4. 铜绿假单胞菌中rhl基因调控系统示意图 [54]
铜绿假单胞菌主要有两个信号传导系统,即LasR-LasI和RhlR-RhlI。lasR是编码AI合成酶的基因,后者合成AI分子N-3-氧化癸酰-L-高丝氨酸内酯(3-oxo-C12-HSL)。lasR编码转录活化因子R蛋白(LasR),后者的N端结合信号物质AI,而C端则与DNA结合,AI复合物可以调节铜绿假单胞菌中lasR、lasA、aprA和toxA等毒力基因的表达,从而影响其致病力,也可以调节lasI基因的表达,来形成一种自我反馈。该复合物还调控rsaL基因的表达,其产物可以抑制lasI基因的表达,对数量感知系统是一种负向调节。
在RhlR-RhlI系统中,rhlR编码转录活化因子RhlR,rhlI则编码AI合成酶,合成信号物质N-丁基-L-高丝氨酸内酯(C4-HSL),RhlR-C4-HSL复合物可以调节rhlAB、lasB、aprA稳定期δ因子、Rpos以及次级代谢产物绿脓菌素和氰化物的产生。
Dockery等建立了一个偏微分方程来研究细胞外空间的自诱导因子的非均匀分布,
(3.1)
其中方程(3.1)中的因子ρ和另一个因子1 − ρ是区分内部空间和外部空间体积不同的必要参考因子,A是细胞生成自诱导因子的浓度,E细胞外的自诱导因子的浓度, 是细胞外自诱导因子的降解速率。Dockery等假设细胞占据的一维区域0< x < L,在细胞区域的边界x = L,他们假设有大量自诱导因子转移到流体中。边界条件方程中的α是一个正参数。
通过这个模型,Dockery等提出群体感应是通过两个稳定解之间的生化开关工作的,一个是低水平的自诱导因子,一个是高水平的自诱导因子。
3.1.2. Chen模型
2004年,Chen等 [55] 以rhlI缺失突变株为研究对象,研究了自诱导因子PAI2对铜绿假单胞菌产生鼠李糖脂(RL)的影响。他们建立了一个模型来描述由rhl群体感应系统调控的RL生成动力学:首先,PAI2与RhlR蛋白结合;其次,激活的复合物RhlR:PAI2触发编码鼠李糖基转移酶的rhlAB操纵子的转录(和表达);最后,该酶催化RL的合成。该模型较好地描述了实验结果。模型的预测如下:在加入PAI2后,诱导酶的合成速率最高。随着自诱导剂的降解,诱导酶的合成速率随时间的延长而减小。然而,酶的浓度仍在不断增加,直到在自体酶耗尽时到达最大值。PAI2的添加浓度越高,酶的初始合成速率越高,诱导的合成时间也越长。随着酶浓度的增加,RL的产率也随之增加,导致RL浓度的加速上升。当PAI2耗尽时,RL浓度的增加呈线性增长。根据所得到的最佳拟合模型参数也得出了以下结论:对于更加复杂的细胞内RhlR蛋白,需要1.61 μM PAI2,即野生型PAO1固定相培养中PAI2浓度的一半左右。另一方面,以其最大速率的一半激活鼠李糖基转移酶合成将需要39%的RLR与PAI2的结合。培养基中的最大RL产率为0.042 g/L h,完全诱导培养至少需要1.61 h才能将该酶合成到产生RL所需的水平。
3.1.3. Melke模型
2010年,Melke等用生长的微小菌落的细胞模型在单细胞水平上来研究群体感应机制 [56] 。他们发现菌落确实有一个细胞密度依赖的行为,并且高度依赖于局部细胞群集和群体进化空间的几何形状。他们分析了信号网络的两个正反馈回路,找到多稳区、展示群体感应机制取决于不同的模型参数。该信号网络如图5。

Figure 5. The quorum sensing network in V. fischeri [56]
图5. 费氏弧菌中的群体感应信号网络 [56]
在该信号网络中,LuxI蛋白合成的信号分子可以穿透细胞膜,通过结合到LuxR蛋白上来激活LuxR蛋白,激活的LuxR蛋白可以形成复合体来诱导信号分子和LuxR蛋白的合成。
细胞之间信号交流的模型如下:
根据Fick定律,用一个有限差分形式的正态扩散方程对细胞外介质区域的扩散进行了模拟,给出了一个元素i的导数:
其中
(3.2)
其中A代表自诱导因子,可以渗出细菌细胞外。Ae是细菌中心附近元素浓度。 、 是细菌和元素的体积,a是细菌的表面积。N(i)代表元素i的附近元素,aij是元素i和j之间的区域,dij是两个元素之间的距离。 代表自诱导因子A的流量, 是流进细胞内的自诱导因子A, 是流出细胞外的自诱导因子A。Melke等的模型仅从LuxR、AHL、LuxR-AHL来建立数学模型研究群体感应机制。
Melke等发现,在细菌本身产生自诱导因子的自然环境中,相比于引入外部诱导因子的环境的交换能力,会导致更多的参数约束。以细胞为基础的模型也允许调查混合细胞群,在混合细胞群体中不产生诱导因子的细胞更具有适应性,但是在竞争中仍不能完全超越产生诱导因子的细胞。
3.1.4. Mcintosh模型
2013年,Mcintosh等 [57] 基于微分方程系统,对苜蓿根瘤菌中的群体感应建立了一个最小的数学模型,并对其做了随机模拟,随机模型中复合体ExpR/AHL的量不同于确定性模型,通过与确定性模型的对比表明该体系对于ExpR的总量是非常敏感的。该群体感应的参与因子有ExpR,SinR,SinI和该系统的信号分子AHL,信号网络图见图6。
根据微生物实验,该数学模型如下:
(3.3)
其中方程(3.3)中的 是细胞i内的SinR的数量, 是细胞i中的SinI的数量, 是细胞i中的ExpR/AHL复合物的数量, 是细胞i内的AHL的数量, 是培养基中的AHL的数量,B是群体的细胞总数。因为他们假设ExpR不被降解,所以有一个常数 ,ExpR的分子数为 , (i = 2~4)、 (i = 1~2)、 (i = 1~2)、 (i = 2~4) 是速率常数。
Mcintosh等指出群体感应系统经历三个阶段:在细菌密度比较低时,所有产生的AHL迅速离开细胞;在中等细胞密度时,AHL的浓度增加的很缓慢,AHL的浓度随着细胞密度的增加而积累,AHL还可以被运回细胞去调节自身的生成等从而形成一个正反馈回路;如果细胞数相当多,AHL的生成就会关闭。
3.1.5. Barbarossa模型
2016年,Barbarossa 等 [58] 针对连续培养基中的恶臭假单胞菌中的群体感应系统提出了一种简单的时滞微分方程(Delay differential equations,简称DDES),它是由于信号分子降解酶的作用产生了一个负反馈,从而有延迟而产生的时滞。这个群体感应系统包含一个正反馈和一(延迟)的负反馈机制。他们的研究结果展示了一些时滞微分方程解的基本性质,例如存在性、唯一性和非负性。最后一个性质对生物中的数学模型非常重要,在建立时滞微分方程时经常违背。研究了解的定性行为,特别是定态及其稳定性,结果表明,对于参数值的选择,系统对时滞给出稳定切换;另一方面,当时滞设为零时,可能会出现关于一个负反馈参数的霍普夫分歧。模型参数与实验数据吻合,表明时滞系统足以解释和预测生物观测值。

Figure 6. The quorum sensing network in S. meliloti [57]
图6. 苜蓿根瘤菌中的群体感应信号网络 [57]
3.1.6. Marenda模型
考虑到边界条件,2016年,Marenda等 [59] 建立了数学模型来模拟在开放的边界扩展系统中细菌群体感应对细胞密度的调控。完整的群体感应系统模型包括信号分子动力学、群体感应的激活和着色分子的生成和动力学,分别介绍如下:
信号分子动力学:豆科植物根瘤菌在密度为 时,在每个细胞中以速率常数 生成信号分子AHL,均匀覆盖悬浮的根癌土壤杆菌传感器的密度为 时可以检测到信号分子的存在。在时间t,距离底座距离为x处的信号分子的浓度C遵循下面的方程
(3.4)
Ds是琼脂内信号分子的扩散常数,ke是细胞外环境中的化学降解速率, 是生物降解速率,假设这些参数对于生产和报告菌株是一样的。为了满足边界条件,他们假设
(3.5)
群体感应的激活:信号分子绑定到启动子上,可以激活群体感应启动子,从而诱导感应菌中β半乳糖苷的生成。这个过程可以用一个简单的化学反应: 来模拟。这意味着平衡时激活细菌细胞所占的比例 可以用希尔方程表示:
(3.6)
其中m是协同性参数,C*是群体感应的阈值,他们希望小时间间隔 内产生的酶量 等价于启动子的启动能力,在他们的假设下:通过最大的启动能力、最大启动子活性A,它和希尔方程成比例 [49] 。
着色分子的生成和动力学:β半乳糖苷酶(E)分解X-gal分子(S)来生成观测的绿色分子(P)。Marenda等用下面的酶促反应来模拟这个过程。
(3.7)
在同样的边界条件下,S和P像AHL一样遵从扩散法则,但是以不同的扩散常数Dx扩散,这些分子浓度随时间的变化可用如下方程描述:
(3.8)
方程(3.8)通过有限差分方法数值求解 [30] 。其中, 、 、 、 种物质的浓度, 、 、 都是速率常数。
在Marenda等的工作中,他们展示了另一个方面的依赖性,即远离开放边界的系统空间扩展。这里提出的模型强调了信号扩散和信号退化在调整空间扩展对群体感应激活的影响中的关键作用。
3.2. 革兰氏阳性菌中的群体感应模型
3.2.1. Karlsson模型
为了更好地理解肺炎双球菌(Streptococcus pneumoniae)中的群体感应系统,2006年,Karlsson等 [60] 针对图7中的肺炎双球菌中群体感应调节ComABCDE的通路建立了一个数学模型。
群体感应信号分子是一个称为CSP (能力刺激肽)的十七肽,是由ComC通过分裂生成并由转运蛋白ComAB 输出到细胞外的。CSP在细胞外积累并由组氨酸激酶ComD识别,ComD通过结合CSP自磷酸化。然后ComD通过将磷酸集团转移到它的同源响应调节子ComE上来进一步转移信号。结合了磷酸集团的ComE可以诱导和增强其同源调节子ComE作为转录激活因子促进comAB、comCDE和 comX的表达 [61] 。一组具有特定功能的基因的转录翻译取决于ComE的激活。在图中,信号流用实线表示,虚线表示信号间的影响。
(3.9)
其中方程(3.9)中的X是各种物质的总量, 、 、 、 、 都是速率常数, 是最大的细胞合成速率, 是细胞体积, 是局部细胞密度, 是磷酸化速率。
通过分析模型,Karlsson等认为关闭感受态可能发生在comCDE操纵子上的转录水平上。由于引入一个假定的ComX依赖性抑制因子,从而抑制comCDE和comX的表达,建立的数学模型证明感受态是呈波浪形出现的 [3] 。实验研究也证实了肺炎双球菌间歇培养中的感受态周期的出现。
虽然对肺炎双球菌中群体感应的研究已经有很多年了,但是其中的负反馈调节一直没有发现明确的机制。在该篇文章中,Karlsson等不仅研究了诱发感受态的群体感应,而且还指出了该系统中突然关闭的过程中可能的信号分子机制,他们认为肺炎双球菌中感受态系统中下降调节在转录水平上是由一个假定的抑制因子抑制comCDE和comX的表达。从而导致感受态成波浪形出现。
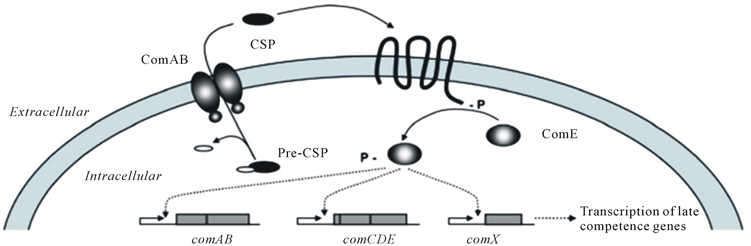
Figure 7. Illustration of the quorum sensing regulated ComABCDE pathway in S. pneumonia [60]
图7. 肺炎双球菌中群体感应调节ComABCDE的通路 [60]
3.2.2. Koerber模型
为了研究金黄色葡萄球菌(Staphylococcus aureus)毒力因子的产生机理,Koerber等建立了确定性和随机性模型 [62] 。图8是金黄色葡萄球菌中的群体感应原理图:
AgrD通过AgrB的处理生成AIP,生成的AIP可以被分泌到细胞外。AgrC是AIP的接受体,并使AgrA磷酸化,然后与SarA作用促进启动子P2和P3调节ageBDCA和RNAIII的表达 [63] 。
为了描述一个细菌从内涵体的逃逸过程,Koerber等假设是由于agr群体感应调节系统控制的细菌酶(蛋白酶和脂肪酶)破坏了内涵体的体壁 [57] 。Koerber等的模型把agr网络的细节简化成封装的“黑盒子”模型,假定其中所有的内在遗传机制是非常迅速的。基于该模型,Koerber等能够确定封闭形式渐近和数值解法在生物学相关的细菌的上下游调节速率的渐近极限,它们的速率都要小于1。
3.3. 革兰氏阳性菌和阴性菌的共有的群体感应模型
哈氏弧菌中的群体感应系统信号网络如图9所示:哈氏弧菌分泌和检测三种不同的信号分子的密度,它们分别是CAI-1,HAI-1和AI-2。细菌密度低时,磷酸转移网络转移磷酸集团到LuxU蛋白,然后LuxU蛋白把磷酸集团转移给LuxO,磷酸化的LuxO促进Qrrs sRNAs的生成。sRNAs负责LuxR转录后的表达。在高细菌密度时,磷酸集团在网络中流向减少sRNAs生成的方向,结果产生高浓度的LuxR蛋白,激活发光。哈氏弧菌中的AI-2信号分子是在LuxS蛋白的参与下生成的,细胞外的AI-2和跨膜的LuxP蛋白结合,传导信号给LuxQ双组分激酶/磷酸酶。最终启动lux操纵子产生荧光。
3.3.1. Banik模型
2009年,Banik等 [64] 建立了一个简单的模型来研究信号分子在群体感应中传导的通路,分析了控制系统反应的关键的无量纲参数。这些参数是利用不同突变株的发光表型的实验数据来估计的。相应的模型预测与不作为确定参数输入的实验结果相一致。此外,提出的框架可以预测发光表型的变化和网络结构的扰动。对于信号分子绑定到它的同源受体上的过程,一般用如下动力学反应方程来表示

Figure 8. A schematic of the proposed agr system in S. aureus [62]
图8. 金黄色葡萄球菌中agr系统的原理图 [62]

Figure 9. The V. harveyi quorum sensing gene network [64]
图9. 哈氏弧菌中的群体感应基因网络 [64]
(3.10)
其中 是激酶模式下的传感器蛋白, 是磷酸酶模式下的传感器蛋白, 是自诱导因子。考虑到结合的协同效应,Banik等认为激酶/磷酸酶分数是
(3.11)
其中的 、 分别表示激酶模式下和磷酸酶模式下的传感器蛋白的浓度, 是一些参考浓度。其中
(3.12)
希尔系数n = 1时对应反应方程(3.10),较高的n值对应于激酶到磷酸酶的更尖锐的转换。 是绑定常数,[AIi]对应于群体密度, 是传感器激酶强度参数, 、 是比例参数。
3.3.2. 其他模型
此外,Hunter [65] [66] [67] 等的工作用大量数据更详细的支撑了V. harveyi中的sRNA循环模型。Hunter等通过对比分析V. harveyi和V. cholerae中的群体感应机制,便于人们更好的理解V. harveyi中的群体感应调节机制。Fenley等也是通过对比V. harveyi和V. cholerae中的群体感应机制,来研究V. harveyi中的群体感应调节机制。Long [68] ,Teng [69] [70] 等在单个细胞水平上研究了V. harveyi中的群体感应调节机制。
3.4. 人工构建的群体感应系统模型
为了更方便地研究细菌群体感应机制,研究人员人工构建了典型的几种细菌中的群体感应体系,并把它植入便于研究且易得的大肠杆菌细胞中去更加深入地研究细菌群体感应机制。由于自诱导因子AHL是整个群体感应系统中最重要的组分,考虑到模型中对自诱导因子AHL考虑的精细程度,我们依次介绍了一些数学模型,这些数学模型从只考虑AHL到考虑细胞内外的AHL,再到考虑细胞膜上的AHL,由简到精地对群体感应体系中的AHL进行建模研究。
3.4.1. Nilsson模型
1983年,Lee等从基因层面利用分子机理模型分析了生长速率对重组大肠杆菌种群生产力的影响 [71] 。2001年,Nilsson等 [72] 建立了一个数学模型来模拟细胞内和膜上的信号分子AHL的浓度随时间的变化。他们的模型如下:
(3.13)
是细胞内AHL的总量, 是细胞膜内AHL的总量。 代表所有细胞的总体积, 表示AHL的净产率, 表示以时间以t为函数的细胞数, 表示AHL扩散出细胞内的净扩散速率, 表示AHL扩散出细菌细胞膜外的净扩散速率。K (0 < K)对应于细菌达到渐近细胞密度时的细菌密度(例如“固定相”)的密度 , 是种群中细菌的初始密度,而 (0 < ) 是内在增长率, 是一个种群参数,因此是整个细菌种群中细胞分裂率的平均值。 是通透性常数, 是单个细菌的表面积, 是细胞膜内AHL的浓度, 是细菌内的AHL的浓度, 是细胞膜的总体积, 是细菌的总体积, 是细胞内的总体积, 是决定细胞密度的一个参数。
AHL浓度随时间的改变可以反映群体生长速率、AHL扩散速率和自身诱导速率。Nilsson等模拟的结果显示单个细菌内的信号分子浓度在细菌生长初始阶段会成倍增加,接着达到一个稳定的平衡期,然后会有一个下降阶段,接着达到第二个稳定平衡期。开始细胞内部高浓度的AHL积累是由于信号分子扩散到细胞外的速率低,细菌生长速率低,自诱导因子分泌速率高。相反的,平衡稳定期时,细菌生长速率高,自诱导因子分泌速率低,信号分子扩散到细胞外的速率高。这些结果表明AHL介导的表达可以在相对较低的细胞密度和低水平的外部环境中的AHL浓度下发生。
3.4.2. You模型
2003年,Ojalvo等 [73] 利用群体感应耦合模拟了一种合成的多细胞时钟。Marguet等 [74] 用数学模型分析了怎么通过扩散损耗减少群体感应里的噪声。
You等 [48] 在2004年发表在Nature期刊上的文章通过模型证明了luxI/luxR 基因与目的基因这两个片段可以互相耦合,并通过细胞交流的方式调控群体密度。You等合成的群体感应网络见图10,模型如下:
(3.14)
(3.15)
(3.16)
这些方程可以模拟图10中回路的功能,公式(3.14)代表细胞数N的生长和死亡,公式(3.15)和公式(3.16)分别表示致死蛋白E和自诱导因子A的生成和降解。他们假设:(3.14)如果没植入的基因片段,细胞密度的生长会有一个固有的生长速率k(h−1)和环境的最大承载能力Nm (h−1),它们可以通过实验测得;2) 对于受植入基因片段调控的细胞生长,细胞的死亡以速率常数d (nM−1∙h−1)正比于细胞内致死蛋白的浓度(E,nM);3) E的生成速率与自诱导因子A的浓度(A,nM)成比例,速率常数为kE (h−1);4) 信号分子的生成速率与细胞数成比例,速率常数为vA (nM∙ml∙h−1);5) dE (h−1)和dA (h−1)分别代表致死蛋白和自诱导因子A的降解速率常数。
这篇文章预测并验证了以下结论:
1) 作者构建的系统在合适的参数值下可以达到稳定的菌群密度。
2) 在植入合成基因片段的体系中,致死蛋白的生成速率受信号分子合成的制约。
3) 稳定时的菌体密度和信号分子的降解速率常数成比例。
4) 当环境中的pH改变时,细胞内的致死蛋白的量保持稳定。

Figure 10. A population-control circuit programmes population dynamics by broadcasting, sensing and regulating the cell density using cell-cell communication and negative feedback [48]
图10. 通过细胞间交流、负反馈回路感应和传播来调控细胞密度的群体调控网络 [48]
所有细胞成分都表现出细胞内噪声是由于单个分子的随机产生和消亡以及环境扰动引起的细胞外噪声而产生的。基因调控,尤其是转录调控、选择性剪接、翻译、扩散和化学修饰反应,都是随机涨落的过程。这样的随机噪声不仅会影响整个系统的动态,也可以被活的有机体利用以积极促进某些功能,如:促进合作行为和沟通。
3.4.3. Chen模型
Chen等 [75] 在2005年提供了一个通用的模型和分析工具,用来分析细胞内和细胞外的随机波动对多系统的合作行为。他们建模时都有考虑细胞外环境中的信号分子,分析了信号在细胞间传递的机制。下面我们以Chen等的模型详细介绍建立模型研究群体感应机制时怎么考虑信号分子从细胞内流出细胞外的过程。
Chen等的工作将信号分子的扩散也考虑进模型中,进一步分析了群体感应机制的作用机理,他们用合成基因网络构成的多系统来证明噪声、延迟和耦合对集体动力学的影响。这些结果对理解噪音在合作动力学中的重要作用奠定了理论和定量基础,例如细胞间的交流。他们构建的信号网络见图11。
该模型考虑如下反应:
(3.17)
其中AI2和LuxR2分别代表AI和LuxR的二聚体,AL和ALD分别代表AI2- LuxR2和AI2-LuxR2- DNA的复合体。反应方程(3.17)表示AI的生成、蛋白的多聚化和多聚体绑定到DNA的过程。

Figure 11. A two-gene model of a gene regulatory network [75]
图11. 一个双基因模型的基因调控网络 [75]
(3.18)
(3.19)
(3.20)
反应方程(3.18)涉及到单个细胞中的转录、翻译和降解。在细胞群体中,AI可以自由进出细胞间,对于扩散过程,可以用时间延迟来近似模拟。这样的过程可以用反应方程(3.19)来表示,其中d是细胞和环境间的扩散速率或耦合系数,Y是环境中的AI。 , ,其中 、 分别是单个细胞的体积和总的环境体积,A是阿伏伽德罗常数。细胞外的噪音对AI扩散的影响可以用反应方程(3.20)描述。 是噪音强度。Chen等用这个模型解释了噪声可以诱发多细胞体系的合作行为。
3.4.4. Li模型
2006年,Li等 [49] 为了研究大肠杆菌中的AI-2信号通路,建立了一个随机模型。对于生物系统,若具有非常低的物种浓度(或数量)和缓慢的反应速率,由于固有的随机事件,随机波动可能存在。反过来也可以产生显著的系统行为变异,特别是随机建模时。由于AI-2的浓度非常低,所以Li等建立了一个随机模型来研究AI-2信号通路。
Li等通过研究AI-2的合成、提取和调控,提出了一些可检测的假设,得出了以下结论:1) 当细胞生长在葡萄糖存在的环境中,mRNA的转录和蛋白水平上AI-2的合成、Pfs和LuxS都不能显著提升AI-2的浓度;2) 在葡萄糖的存在下,这种合成途径伴随着代谢通量的增加,只是部分地解释了这种差异。他们预测,高通量的替代途径或其他生物步骤存在于AI-2的合成中;3) 实验结果验证了这一假说。这项工作证实了基于系统的随机模型可以和细胞生理学联系起来。Tian等也有用随机模型来研究群体感应 [76] 。
3.4.5. Saeidi模型
2013年,Saeidi等 [50] 人工合成了一个群体感应系统,针对人工合成的群体感应体系来研究铜绿假单胞菌中的群体感应机制,他们的模型从基因层面上更加详细地对细菌群体感应机制进行了模拟。下面我们以Saeidi等的模型为例详细讲解从基因层面建模研究群体感应的过程,他们人工构建的网络如图12所示。
图12阐明了触发作为报告蛋白的荧光蛋白的生成步骤,他们建立了如下方程来描述群体感应体系:
(3.21)
(3.22)
方程(3.21)和方程(3.22)描述了mRNA和LasR蛋白的生成,他们假设mRNA和LasR蛋白的生成遵循质量作用定律,k1和k2分别代表它们的生成速率常数,y1和y2分别代表它们的降解速率常数, 、 分别表示这些物质的浓度,接下来所有方程中这样的表达方式都表示该物质的浓度。
(3.23)

Figure 12. A schematic of the quorum sensing system modeled [50]
图12. 模拟的群体感应体系的原理图 [50]
(3.24)
(3.25)
方程(3.23)代表复合体LasR_AHL的生成,复合体的生成速率和LasR、AHL的浓度成比例,其中k3表示结合速率常数,k4表示分离速率常数。方程(3.24)代表AHL浓度的变化,方程(3.25)表示复合体LasR_AHL绑定到启动子pLuxR上的过程,其中k5、k6分别代表结合速率常数和分离速率常数,A.pLuxR代表结合了复合体的启动子pLuxR。
(3.26)
(3.27)
(3.28)
方程(3.26)描述了荧光蛋白的生成,他们假设AHL的浓度和启动子pLuxR相比微不足道,而且,因为他们用的质粒是高复制率的质粒,因此,他们认为A.pLuxR的浓度主要取决于AHL的浓度。
在方程(3.26)中,T代表当输出达到最大输出的63.2%时的时间常数,GFPmax是不同AHL浓度下GFP的最大浓度(平衡时GFP的量)。实验结果显示1/T和GFPmax随AHL浓度的变化遵循希尔方程,因此,方程(3.27)和(3.28)表示1/T和GFPmax的浓度随AHL浓度的变化,方程(3.27)和(3.28)是A.pLuxR的函数方程。
Saeidi等的敏感性分析模拟结果表明k11、k12是体系中最敏感的两个参数,k7、k8是对达到平衡所需时间影响最大的两个参数。他们的模型可以用来预测目前实验条件(例如温度是37℃)下GFP的产量,未来,这个模型也可以延伸去捕获不同实验条件下的其他特征。这个模型可以为将来CAD(Computer Aided Design,计算机辅助设计)在合成生物学设计中生物部分提供有用的信息。
4. 总结与展望
本文介绍了革兰氏阴性菌中的群体感应系统、革兰氏阳性菌中的群体感应系统和革兰氏阴性菌和革兰氏阳性菌共有的群体感应系统以及合成生物学及人工合成群体感应实验的研究,总结了野生群体感应建模和合成群体感应建模,野生群体感应建模又从革兰氏阴性菌、革兰氏阳性菌和革兰氏阴性和革兰氏阳性菌共有的群体感应建模三种类型来分析群体感应系统。人们在建立群体感应模型来研究群体感应系统时,从单一的只考虑群体感应系统中的几个重要组分建模到构建一个信号网络来建模;从不考虑信号分子进出细胞外到考虑信号分子在细胞间的流动;从不考虑噪音到研究空间扩散、边界效应在群体感应中的影响来研究群体感应系统。随着人们对群体感应系统研究的深入,构建的群体感应网络考虑了各个方面的因素,更加接近于真实细胞中的群体感应系统从而更加准确地对群体感应系统进行研究。这些理论研究加深了我们对群体感应机制的了解,为进一步研究群体感应机制及其在生产生活中的应用提供了理论依据。
当然,目前人们在研究群体感应建模时在仍存在以下一些问题和不足之处:
1) 构建的信号通路可能还不够完善,群体感应机制中更加细分的许多过程还不明确,群体感应系统中涉及到的重要蛋白组分在细胞中也可能参与到其他生命活动中。随着人们对群体感应系统研究的深入,如果能将更多的生命活动的信号网络和群体感应系统的信号网络联系在一起,逐渐完善细胞生命活动的信号网络,那么人们对群体感应系统的研究乃至细胞整个生命活动的研究都会更加深入和细致,便于人们对生物体生命活动进行进一步地了解。
2) 现在人们构建的群体感应模型中虽然有些是从基因层面开始构建的,但是具体的诱导剂作用到基因上的启动子,启动子启动基因的转录翻译这些过程人们都还不是很明确,在构建模型时一般都只省略概括为一到两个方程,这样的模型并没有将生物体真实的生命活动完全还原出来,对细菌群体感应系统及生物体的真实生命活动过程的研究都是不足的,在今后的研究中,构建数学模型时,这些过程都要更加详细地表示出来,更加真实地还原生命活动。
3) 目前人们在研究群体感应系统时构建的数学模型对细胞的死亡过程考虑的还不是很全面,对于细胞生长后期,培养体系中营养物质缺乏时,细胞由于代谢副产物的积累,具体是哪些副产物导致细胞死亡的机制不是特别明晰,因此在建立数学模型时不能很好地遵循真实生命活动来建模,今后人们在研究群体感应系统建模时要更加明确细胞生长后期细胞自身的死亡机制,更接近于真实生命活动来建立数学模型。
4) 现在人们在研究细菌群体感应系统建模时建立的数学模型一般都是简单的常微分方程,而且忽略了许多生命过程中的噪声及空间扩散、边界效应的影响,这样建立的数学模型可能并不能真实地反应生命活动,因此在今后建立数学模型时,要将每个生命活动的机制都研究清楚,有些不能忽略的噪声、信号分子之间的相互作用、空间扩散、边界效应等都要考虑在内,在更接近真实情况下准确地建立数学模型。遇到具有非常低浓度(或数量)的物种的遗传调控网络和缓慢的反应速率,由于固有的随机事件,随机波动可能存在,这种情况下可以建立随机模型。
用数学模型去解释实验上难以阐明的问题,用实验数据去验证数学模型提出的预测,实验与理论紧密结合,才能更好的研究群体感应机制及生物体其他生命活动,以便人们更好的利用群体感应机制,造福人类社会。
致谢
本课题获得如下基金项目支持:国家自然科学基金资助项目(批准号:11675134),国家111项目(批准号:b16029),国家海洋局海洋生物遗传资源重点实验室开放研究基金(批准号:HY201505)和国家博士后科学基金(批准号:2016m602071)。
文章引用
张晓翠,黄世阳,李 翔,刘艳平,吴宇宁,帅建伟,王兆守. 细菌群体感应机制的动力学建模研究
The Dynamical Modeling Studies of the Quorum Sensing Mechanism in Bacteria[J]. 生物物理学, 2018, 06(02): 7-30. https://doi.org/10.12677/BIPHY.2018.62002
参考文献
- 1. Parsek, M.L. Val, D., Hanzelka, B., Cronan, J. and Greenberg, E.P. (1999) Acyl Homoserine-Lactone Quorum-Sensing Signal Generation. Proceedings of the National Academy of Sciences of the United States of America, 96, 8789-8793. https://doi.org/10.1073/pnas.96.8.4360
- 2. Dandekar, A.A., Chugani, S. and Greenberg, E.P. (2012) Bacterial Quorum Sensing and Metabolic Incentives to Cooperate. Science, 338, 264-266. https://doi.org/10.1126/science.1227289
- 3. Li, Y.H., Tang, N., Aspiras, M.B., Lau, P.C.Y., Lee, J.H., Ellen, R.P. and Cvitkovitch, D.G. (2002) A Quorum-Sensing Signaling System Essential for Genetic Competence in Streptococcus mutans Is Involved in Biofilm Formation. Journal of Bacteriology, 184, 2699-2708. https://doi.org/10.1128/JB.184.10.2699-2708.2002
- 4. Diggle, S.P., Griffin, A.S., Campbell, G.S. and West, S.A. (2007) Cooperation and Conflict in Quorum-Sensing Bacterial Populations. Nature, 450, 411-414. https://doi.org/10.1038/nature06279
- 5. Hooshangi, S. and Bentley, W.E. (2008) From Unicellular Properties to Multicellular Behavior: Bacteria Quorum Sensing Circuitry and Applications. Current Opinion in Biotechnology, 19, 550-555. https://doi.org/10.1016/j.copbio.2008.10.007
- 6. Stoodley, P., Sauer, K., Davies, D.G. and Costerton, J.W. (2002) Biofilms as Complex Differentiated Communities. Annual Review of Microbiology, 56, 187-209. https://doi.org/10.1146/annurev.micro.56.012302.160705
- 7. Hagen, S.J., Son, M., Weiss, J.T. and Young, J.H. (2010) Bacterium in a Box: Sensing of Quorum and Environment by the LuxI/LuxR Gene Regulatory Circuit. Journal of Biological Physics, 36, 317-327. https://doi.org/10.1007/s10867-010-9186-4
- 8. Ng, W.L. and Bassler, B.L. (2009) Bacterial Quorum-Sensing Network Architectures. Annual Review of Genetics, 43, 197-222. https://doi.org/10.1146/annurev-genet-102108-134304
- 9. Tsai, C.S. and Winans, S.C. (2010) LuxR-Type Quo-rum-Sensing Regulators That Are Detached from Common Scents. Molecular Microbiology, 77, 1072-1082. https://doi.org/10.1111/j.1365-2958.2010.07279.x
- 10. Daniels, R., Vanderleyden, J. and Michiels, J. (2004) Quorum Sensing and Swarming Migration in Bacteria. FEMS Microbiology Reviews, 28, 261-289. https://doi.org/10.1016/j.femsre.2003.09.004
- 11. Hao, Y., Winans, S.C., Glick, B.R. and Charles, T.C. (2010) Identification and Characterization of New LuxR/LuxI-Type Quorum Sensing Systems from Metagenomic Libraries. Environmental Microbiology, 12, 105-117. https://doi.org/10.1111/j.1462-2920.2009.02049.x
- 12. Kambam, P.K., Sayut, D.J., Niu, Y., Eriksen, D.T. and Sun, L. (2008) Directed Evolution of LuxI for Enhanced OHHL Production. Biotechnology and Bioengineering, 101, 263-272. https://doi.org/10.1002/bit.21901
- 13. Rampioni, G., Leoni, L. and Williams, P. (2014) The Art of Antibacterial Warfare: Deception through Interference with Quorum Sensing-Mediated Communication. Bioorganic Chemistry, 55, 60-68. https://doi.org/10.1016/j.bioorg.2014.04.005
- 14. Steindler, L. and Venturi, V. (2007) Detection of Quorum-Sensing N-Acyl Homoserine Lactone Signal Molecules by Bacterial Biosensors. FEMS Microbiology Letters, 266, 1-9. https://doi.org/10.1111/j.1574-6968.2006.00501.x
- 15. Li, Z. and Nair, S.K. (2012) Quorum Sensing: How Bacteria Can Coordinate Activity and Synchronize Their Response to External Signals? Protein Science, 21, 1403-1417. https://doi.org/10.1002/pro.2132
- 16. Jayaraman, A. and Wood, T.K. (2008) Bacterial Quorum Sensing: Signals, Circuits, and Implications for Biofilms and Disease. Annual Review of Biomedical Engineering, 10, 145-167. https://doi.org/10.1146/annurev.bioeng.10.061807.160536
- 17. Bjarnsholt, T., Jensen, P.O., Jakobsen, T.H., Phipps, R., Nielsen, A.K., Rybtke, M.T., Tolker-Nielsen, T., Givskov, M., Hoiby, N. and Ciofu, O., The Scandinavian Cystic Fibrosis Study Consortium (2010) Quorum Sensing and Virulence of Pseudomonas aeruginosa during Lung Infection of Cystic Fibrosis Patients. PLoS ONE, 5, e10115. https://doi.org/10.1371/journal.pone.0010115
- 18. Whitehead, N.A., Barnard, A.M.L., Slater, H., Simpson, N.J.L. and Salmond, G.P.C. (2001) Quorum-Sensing in Gram-Negative Bacteria. FEMS Microbiology Reviews, 25, 365-404.
- 19. Perez, P.D. and Hagen, S.J. (2010) Heterogeneous Response to a Quorum-Sensing Signal in the Luminescence of Individual Vibrio fischeri. PLoS ONE, 5, e15473. https://doi.org/10.1371/journal.pone.0015473
- 20. Scott, S.R. and Hasty, J. (2016) Quorum Sensing Communication Modules for Microbial Consortia. ACS Synthetic Biology, 5, 969-977. https://doi.org/10.1021/acssynbio.5b00286
- 21. Marchand, N. and Collins, C.H. (2016) Synthetic Quorum Sensing and Cell-Cell Communication in Gram-Positive Bacillus megaterium. ACS Synthetic Biology, 5, 597-606. https://doi.org/10.1021/acssynbio.5b00099
- 22. Davis, R.M., Muller, R.Y. and Haynes, K.A. (2015) Can the Natural Diversity of Quorum-Sensing Advance Synthetic Biology? Frontiers in Bioengineering and Biotechnology, 3, 30.
- 23. Annous, B.A., Fratamico, P.M. and Smith, J.L. (2009) Scientific Status Summary. Journal of Food Science, 74, R24-R37. https://doi.org/10.1111/j.1750-3841.2008.01022.x
- 24. Antunes, L.C., Ferreira, R.B., Buckner, M.M. and Finlay, B.B. (2010) Quorum Sensing in Bacterial Virulence. Microbiology, 156, 2271-2282. https://doi.org/10.1099/mic.0.038794-0
- 25. Rutherford, S.T. and Bassler, B.L. (2012) Bacterial Quorum Sensing: Its Role in Virulence and Possibilities for Its Control. Cold Spring Harbor Perspectives in Medicine, 2, 705-709. https://doi.org/10.1101/cshperspect.a012427
- 26. Deep, A., Chaudhary, U. and Gupta, V. (2011) Quorum Sensing and Bacterial Pathogenicity: From Molecules to Disease. Journal of Laboratory Physicians, 3, 4-11. https://doi.org/10.4103/0974-2727.78553
- 27. Novick, R.P. (2003) Autoinduction and Signal Transduction in the Regulation of Staphylococcal Virulence. Molecular Microbiology, 48, 1429-1449. https://doi.org/10.1046/j.1365-2958.2003.03526.x
- 28. Defoirdt, T., Miyamoto, C.M., Wood, T.K., Meighen, E.A., Sorgeloos, P., Verstraete, W. and Bossier, P. (2007) The Natural Furanone (5Z)-4-Bromo-5-(Bromomethylene)-3-Butyl-2(5H)-Furanone Disrupts Quorum Sensing-Regulated Gene Expression in Vibrio harveyi by Decreasing the DNA-Binding Activity of the Transcriptional Regulator Protein LuxR. Environmental Microbiology, 9, 2486-2495. https://doi.org/10.1111/j.1462-2920.2007.01367.x
- 29. Fernandes, R., Roy, V., Wu, H.C. and Bentley, W.E. (2010) Engineered Biological Nanofactories Trigger Quorum Sensing Response in Targeted Bacteria. Nature Nanotechnology, 5, 213-217. https://doi.org/10.1038/nnano.2009.457
- 30. Abubakar, B.M. (2014) The Significant Role of Quorum Sensing in the Control of Vibrio cholerae Virulence. Advances in Bioscience and Bioengineering, 2, 1.
- 31. Camilli, A. and Bassler, B.L. (2006) Bacterial Small-Molecule Signaling Pathways. Science, 311, 1113-1116. https://doi.org/10.1126/science.1121357
- 32. Ahlgren, N.A., Harwood, C.S., Schaefer, A.L., Giraud, E. and Greenberg, E.P. (2011) Aryl-Homoserine Lactone Quorum Sensing in Stem-Nodulating Photosynthetic Bradyrhizobia. Proceedings of the National Academy of Sciences of the United States of America, 108, 7183-7188. https://doi.org/10.1073/pnas.1103821108
- 33. Venturi, V., Rampioni, G., Pongor, S. and Leoni, L. (2011) The Virtue of Temperance: Built-In Negative Regulators of Quorum Sensing in Pseudomonas. Molecular Microbiology, 82, 1060-1070. https://doi.org/10.1111/j.1365-2958.2011.07890.x
- 34. Xavier, K.B. and Bassler, B.L. (2005) Interference with AI-2-Mediated Bacterial Cell-Cell Communication. Nature, 437, 750-753. https://doi.org/10.1038/nature03960
- 35. Swords, W.E. (2012) Quorum Signaling and Sensing by Nontypeable Haemophilus influenzae. Frontiers in Cellular and Infection Microbiology, 2, 100. https://doi.org/10.3389/fcimb.2012.00100
- 36. Papenfort, K. and Bassler, B.L. (2016) Quorum Sensing Sig-nal-Response Systems in Gram-Negative Bacteria. Nature Reviews Microbiology, 14, 576-588. https://doi.org/10.1038/nrmicro.2016.89
- 37. von Bodman, S.B., Willey, J.M. and Diggle, S.P. (2008) Cell-Cell Communication in Bacteria: United We Stand. Journal of Bacteriology, 190, 4377-4391. https://doi.org/10.1128/JB.00486-08
- 38. Collins, C.H., Leadbetter, J.R. and Arnold, F.H. (2006) Dual Selection Enhances the Signaling Specificity of a Variant of the Quorum-Sensing Transcriptional Activator LuxR. Nature Biotechnology, 24, 708-712. https://doi.org/10.1038/nbt1209
- 39. Neiditch, M.B., Federle, M.J., Miller, S.T., Bassler, B.L. and Hughson, F.M. (2005) Regulation of LuxPQ Receptor Activity by the Quorum-Sensing Signal Autoinducer-2. Molecular Cell, 18, 507-518.
- 40. Pappas, K.M., Weingart, C.L. and Winans, S.C. (2004) Chemical Communication in Proteobacteria: Biochemical and Structural Studies of Signal Synthases and Receptors Required for Intercellular Signalling. Molecular Microbiology, 53, 755-769. https://doi.org/10.1111/j.1365-2958.2004.04212.x
- 41. Rutherford, S.T., van Kessel, J.C., Shao, Y. and Bassler, B.L. (2011) Apha and LuxR/HapR Reciprocally Control Quorum Sensing in Vibrios. Genes & Development, 25, 397-408. https://doi.org/10.1101/gad.2015011
- 42. Heinemann, M. and Panke, S. (2006) Synthetic Biology—Putting Engineering into Biology. Bioinformatics, 22, 2790-2799. https://doi.org/10.1093/bioinformatics/btl469
- 43. Leonard, E., Nielsen, D., Solomon, K. and Prather, K.J. (2008) Engineering Microbes with Synthetic Biology Frameworks. Trends in Biotechnology, 26, 674-681.
- 44. Swami, M. (2009) Synthetic Biology: Avoiding the Chop. Nature Reviews Genetics, 10, 660-660. https://doi.org/10.1038/nrg2671
- 45. Bedau, M.A., Parke, E.C., Tangen, U. and Hantsche-Tangen, B. (2009) Social and Ethical Checkpoints for Bottom-Up Synthetic Biology, or Protocells. Systems and Synthetic Biology, 3, 65-75. https://doi.org/10.1007/s11693-009-9039-2
- 46. Danino, T., Mondragon-Palomino, O., Tsimring, L. and Hasty, J. (2010) A Synchronized Quorum of Genetic Clocks. Nature, 463, 326-330. https://doi.org/10.1038/nature08753
- 47. Haseltine, E.L. and Arnold, F.H. (2008) Implications of Rewiring Bacterial Quorum Sensing. Applied and Environmental Microbiology, 74, 437-445. https://doi.org/10.1128/AEM.01688-07
- 48. Webster, S.M., Del Camino, D., Dekker, J.P. and Yellen, G. (2004) Intracellular Gate Opening in Shaker K+ Channels Defined by High-Affinity Metal Bridges. Nature, 428, 864-868. https://doi.org/10.1038/nature02468
- 49. Li, J., Wang, L., Hashimoto, Y., Tsao, C.Y., Wood, T.K., Valdes, J.J., Zafiriou, E. and Bentley, W.E. (2006) A Stochastic Model of Escherichia coli AI-2 Quorum Signal Circuit Reveals Alternative Synthesis Pathways. Molecular Systems Biology, 2, 67. https://doi.org/10.1038/msb4100107
- 50. Saeidi, N., Arshath, M., Chang, M.W. and Poh, C.L. (2013) Characterization of a Quorum Sensing Device for Synthetic Biology Design: Experimental and Modeling Validation. Chemical Engineering Science, 103, 91-99. https://doi.org/10.1016/j.ces.2012.12.016
- 51. Daniel, R., Rubens, J.R., Sarpeshkar, R. and Lu, T.K. (2013) Synthetic Analog Computation in Living Cells. Nature, 497, 619-623. https://doi.org/10.1038/nature12148
- 52. Wang, Z., Wu, X., Peng, J., Hu, Y., Fang, B. and Huang, S. (2014) Artificially Constructed Quorum-Sensing Circuits Are Used for Subtle Control of Bacterial Population Density. PLoS ONE, 9, e104578. https://doi.org/10.1371/journal.pone.0104578
- 53. Huang, S., Lee, A.J., Tsoi, R., Wu, F., Zhang, Y., Leong, K.W. and You, L. (2016) Coupling Spatial Segregation with Synthetic Circuits to Control Bacterial Survival. Molecular Systems Biology, 12, 859. https://doi.org/10.15252/msb.20156567
- 54. Dockery, J.D. and Keener, J.P. (2001) A Mathematical Model for Quorum Sensing in Pseudomonas aeruginosa. Bulletin of Mathematical Biology, 63, 95-116. https://doi.org/10.1006/bulm.2000.0205
- 55. Chen, F., Chen, C.-C., Riadi, L. and Ju, L.-K. (2004) Modeling rhl Quorum-Sensing Regulation on Rhamnolipid Production by Pseudomonas aeruginosa. Biotechnology Progress, 20, 1325-1331. https://doi.org/10.1021/bp049928b
- 56. Melke, P., Sahlin, P., Levchenko, A. and Jonsson, H. (2010) A Cell-Based Model for Quorum Sensing in Heterogeneous Bacterial Colonies. PLoS Computational Biology, 6, e1000819. https://doi.org/10.1371/journal.pcbi.1000819
- 57. McIntosh, M., Czuppon, P., Best, K., Becker, A. and Pfaffelhuber, P. (2013) Modeling Quorum Sensing in Sinorhizobium meliloti. International Journal of Biomathematics and Biostatistics, 2, 59-74.
- 58. Barbarossa, M. and Kuttler, C. (2016) Mathematical Modeling of Bacteria Communication in Continuous Cultures. Applied Sciences, 6, 149. https://doi.org/10.3390/app6050149
- 59. Marenda, M., Zanardo, M., Trovato, A., Seno, F. and Squartini, A. (2016) Modeling Quorum Sensing Trade-Offs between Bacterial Cell Density and System Extension from Open Boundaries. Scientific Reports, 6, Article No. 39142. https://doi.org/10.1038/srep39142
- 60. Karlsson, D., Karlsson, S., Gustafsson, E., Normark, B.H. and Nilsson, P. (2007) Modeling the Regulation of the Competence-Evoking Quorum Sensing Network in Streptococcus pneumoniae. Biosystems, 90, 211-223. https://doi.org/10.1016/j.biosystems.2006.08.005
- 61. Voloshin, S.A. and Kaprelyants, A.S. (2004) Cell-Cell Interactions in Bacterial Populations. Biochemistry (Moscow), 69, 1268-1275. https://doi.org/10.1007/s10541-005-0072-9
- 62. Koerber, A.J., King, J.R. and Williams, P. (2005) Deterministic and Stochastic Modelling of Endosome Escape by Staphylococcus aureus: “Quorum” Sensing by a Single Bacterium. Journal of Mathematical Biology, 50, 440-488. https://doi.org/10.1007/s00285-004-0296-0
- 63. West, S.A., Winzer, K., Gardner, A. and Diggle, S.P. (2012) Quorum Sensing and the Confusion about Diffusion. Trends in Microbiology, 20, 586-594.
- 64. Banik, S.K., Fenley, A.T. and Kulkarni, R.V. (2009) A Model for Signal Transduction during Quorum Sensing in Vibrio harveyi. Physical Biology, 6, Article ID: 046008. https://doi.org/10.1088/1478-3975/6/4/046008
- 65. Svenningsen, S.L., Waters, C.M. and Bassler, B.L. (2008) A Negative Feedback Loop Involving Small RNAs Accelerates Vibrio cholerae’s Transition out of Quorum-Sensing Mode. Genes & Development, 22, 226-238. https://doi.org/10.1101/gad.1629908
- 66. Hunter, G.A. and Keener, J.P. (2014) Mechanisms Underlying the Additive and Redundant Qrr Phenotypes in Vibrio harveyi and Vibrio cholerae. Journal of Theoretical Biology, 340, 38-49. https://doi.org/10.1016/j.jtbi.2013.08.034
- 67. Hunter, G.A., Vasquez, F.G. and Keener, J.P. (2013) A Mathematical Model and Quantitative Comparison of the Small RNA Circuit in the Vibrio harveyi and Vibrio cholerae Quorum Sensing Systems. Physical Biology, 10, Article ID: 046007. https://doi.org/10.1088/1478-3975/10/4/046007
- 68. Long, T., Tu, K.C., Wang, Y., Mehta, P., Ong, N.P., Bassler, B.L. and Wingreen, N.S. (2009) Quantifying the Integration of Quorum-Sensing Signals with Single-Cell Resolution. PLoS Biology, 7, e1000068. https://doi.org/10.1371/journal.pbio.1000068
- 69. Teng, S.W., Schaffer, J.N., Tu, K.C., Mehta, P., Lu, W., Ong, N.P., Bassler, B.L. and Wingreen, N.S. (2011) Active Regulation of Receptor Ratios Controls Integration of Quorum-Sensing Signals in Vibrio harveyi. Molecular Systems Biology, 7, 491. https://doi.org/10.1038/msb.2011.30
- 70. Teng, S.W., Wang, Y., Tu, K.C., Long, T., Mehta, P., Wingreen, N.S., Bassler, B.L. and Ong, N.P. (2010) Measurement of the Copy Number of the Master Quorum-Sensing Regulator of a Bacterial Cell. Biophysical Journal, 98, 2024-2031. https://doi.org/10.1016/j.bpj.2010.01.031
- 71. Lee, S.B. and Bailey, J.E. (2002) Analysis of Growth Rate Effects on Productivity of Recombinant Escherichia coli Populations Using Molecular Mechanism Models. Biotechnology and Bioengineering, 79, 550-557. https://doi.org/10.1002/bit.10439
- 72. Nilsson, P., Olofsson, A., Fagerlind, M., Fagerstrom, T., Rice, S., Kjelleberg, S. and Steinberg, P. (2001) Kinetics of the AHL Regulatory System in a Model Biofilm System: How Many Bacteria Constitute a “Quorum”? Journal of Molecular Biology, 309, 631-640. https://doi.org/10.1006/jmbi.2001.4697
- 73. Garcia-Ojalvo, J., Elowitz, M.B. and Strogatz, S.H. (2004) Modeling a Synthetic Multicellular Clock: Repressilators Coupled by Quorum Sensing. Proceedings of the National Academy of Sciences of the United States of America, 101, 10955-10960. https://doi.org/10.1073/pnas.0307095101
- 74. Tanouchi, Y., Tu, D., Kim, J. and You, L. (2008) Noise Reduction by Diffusional Dissipation in a Minimal Quorum Sensing Motif. PLoS Computational Biology, 4, e1000167. https://doi.org/10.1371/journal.pcbi.1000167
- 75. Chen, L., Wang, R., Zhou, T. and Aihara, K. (2005) Noise-Induced Cooperative Behavior in a Multicell System. Bioinformatics, 21, 2722-2729. https://doi.org/10.1093/bioinformatics/bti392
- 76. Tian, T. and Burrage, K. (2006) Stochastic Models for Regulatory Networks of the Genetic Toggle Switch. Proceedings of the National Academy of Sciences of the United States of America, 103, 8372-8377. https://doi.org/10.1073/pnas.0507818103
NOTES
*通讯作者。