Advances in Geosciences
Vol.2 No.3(2012), Article ID:2925,10 pages DOI:10.4236/AG.2012.23026
Advance in Research on Diversity of Marinephycovirus*
1State Key Lab of Marine Environmental Science, Xiamen University, Xiamen
2Key Lab of Ministry of Education for Coast and Wetland Ecosystems, Life Science Department, Xiamen University, Xiamen
Email: zhangran22105@163.com, #microzh@xmu.edu.cn
Received: Jun. 18th, 2012; revised: Jun. 27th, 2012; accepted: Jul. 6th, 2012
ABSTRACT:
Phycovirus, as an important member in the marine ecosystem, is playing a greatly prominent role in the microbial loop, biogeochemical cycling, community population and diversity. Because of the numerous advantages of using virus to regulate the red-tide and the water bloom, it has become a hot research orientation that people adopted algae virus to control the algae. Nowadays, there were already many algae virus that had been isolated and identified and scientists also have some research regarding to their practical use, finding that the effect that viruses have on the practical ecosysystem is extremelycomplex. So focus was put on the virus zoology. It is known that the genetic diversity of phycovirus is extremely high, butseeking the way to consider its worldwide distribution and some efficient ways to detect and analysis it has become a global research hotspot. This article introduces the recent research progress of algae virus related with the ecological distribution, geneticdistribution, correlative target genes and frequently-used analytical procedure and makes some prospects of phycovirus, hoping to provide some references for the research in the ecology of phycovirus and the biological control of red-tide.
Keywords: Diversity; Phycovirus; Biological Control of Red-Tide; Marine Environment
海洋藻类病毒多样性研究的现状与展望*
陈章然1,2,郑 伟1,2,郑天凌1,2#
1厦门大学近海海洋环境科学国家重点实验室,厦门
2厦门大学生命科学学院,滨海湿地生态系统教育部重点实验室,厦门
Email: zhangran22105@163.com, #microzh@xmu.edu.cn
摘 要:
作为海洋生态系统中的重要成员,藻类病毒在微生物环、生物地球化学循环、群落结构及多样性等方面起着重要作用。因为病毒在治理赤潮藻华问题方面具有诸多优势,所以利用藻类病毒治藻已成为一个热点的研究方向。目前已分离鉴定出许多种类的藻类病毒,并且人们对藻类病毒的实际应用也进行了一定的探讨,发现病毒在实际的生态系统中作用比较复杂。于是,针对藻类病毒生态的研究也越来越多。众所周知,藻类病毒的遗传多样性极其丰富,但其分布是否具有全球性,如何对其进行有效的检测和分析等都是国际上的研究热点。本综述就近年来国内外对藻类病毒研究的相关进展和本实验室开展的相关研究情况进行了概述,尤其对藻类病毒的生态分布、遗传多样性、研究中所涉及的靶基因及主要的分析方法进行了总结,提出海洋藻类病毒的研究展望。旨在为藻类病毒生态学、赤潮生物防治提供参考。
收稿日期:2012年6月18日;修回日期:2012年6月27日;录用日期:2012年7月6日
关键词:多样性;藻类病毒;海洋环境;赤潮生物防治
1. 引言
赤潮作为严重的全球性海洋灾害之一,已经引起世界各地的高度关注。近年来,探究有效调控赤潮生消的重要微生物因子,充分利用其与赤潮生物的互作特点与规律,开发和利用源自赤潮频发海域的特殊功效微生物从而进行赤潮的有效治理将可能成为解决赤潮灾害这一问题的关键。众多研究表明,细菌、放线菌[1,2]等各种微生物可以通过直接接触杀藻或间接的分泌杀藻活性物质如蛋白[3]等来专一性地抑制藻类的生长甚至裂解藻细胞,但是藻类病毒因为满足“高效、易得、无毒、低廉”的杀藻要求而成为当前研究的一个热点。目前为止,研究者已分离藻类病毒上百种,但考虑到实际环境的复杂多变及对生物群落功能的潜在影响,将藻类病毒投放于有害藻华爆发区的安全性亟待考察。所以,必须对病毒–藻–微生物–环境因素之间的相互关系进行更为深入的研究,从而确定病毒的分布与生态地位。
藻类病毒可分为噬藻体和藻病毒,前者主要是感染蓝藻,后者主要是感染真核藻类。水体生态系统中藻类病毒极其丰富(可达106~107/mL),它被认为是水体微生物群落中重要的活性成分[4,5],同时它还能影响生物地球化学循环和微生物碳泵效率等[4](如图1)。其研究对于了解病毒分类、病毒与宿主之间的相互关系和病毒在生态系统中的地位等具有深远的意义[6]。本文综述了海洋藻类病毒的研究进展,着重从生态分布、遗传类型等方面分析并论述了藻类病毒多样性在生态系统中的作用,同时对当今藻类病毒多样性主要的研究方法予以归纳,旨在为藻类病毒生态学、赤潮的病毒防治提供参考。
2. 生态分布多样性
2.1. 近海海域藻类病毒多样性
近年来,随着工农业生产的发展,近海水域富营养化程度日趋严重,由浮游植物引发的赤潮频发,并造成了严重的生态灾害及经济损失,受到国内外学者的广泛关注。这一生态系统中,海洋病毒占有重要的生态位,对其生态分布及多样性的相关研究逐渐增多。
1996年Chen等[7]利用RFLP技术对墨西哥湾近海海域病毒多样性进行了调查研究,得到了5个系统分类单元(OTUs),均属于藻DNA病毒科,其中4个属于寄生藻病毒属,而另1个OTU在寄生藻病毒属和金藻病毒属间形成1个单独的分支。2001年Hodson等人[8]对佐治亚近海感染海洋聚球藻的噬藻体的分布

Figure 1. Viruses can affect biogeochemical cycling and the efficiency of the biological pump[4]
图1. 病毒影响地球化学循环和生物泵效率[4]
和多样性进行了调查。研究发现,随着盐度增加,噬藻体丰度上升,并且80%为肌尾噬藻体,它们的形态多样,基因型也非常丰富。2009年Chen等人[9]利用病毒DNA聚合酶基因序列对Chesapeake Bay冬夏两季蓝细菌短尾噬菌体的多样性和动态组成也进行了相当详尽的阐述,均揭示出近海海域藻类病毒种类及丰度的多样性特征。
近年来国内也在相关方面做了大量的研究,如白晓歌等人[10]开展了冬季和春季长江口及其近海水域浮游病毒丰度的分析,结果显示,夏季浮游病毒丰度和异养细菌丰度是显著相关的,而冬季两者的昼夜变化并不完全一致;夏季浮游病毒丰度与盐度成负相关性,冬季浮游病毒丰度与盐度没有明显的相关性。肖天等人[11]利用荧光显微镜技术对胶州湾浮游病毒的分布情况进行了研究,结果显示浮游病毒的丰度在0.48 × 107~22.78 × 107/mL,从湾内至湾口至湾外呈递减的趋势,垂直分布变化不明显,且其丰度与异养细菌和叶绿素相关,与温度和盐度则无明显相关性。
本实验室也从90年代开始致力于海洋病毒生态和病毒调控赤潮的相关研究,在病毒生态方面,王斐等[12]总结了海洋病毒在微生物食物环中所扮演的角色。杨小茹等[13]评析了海洋病毒与赤潮生物的关系,探讨了利用病毒防治赤潮的可能性及有效性。王慧等[14]讨论了海洋病毒在调节海洋生态系统中的群落结构、种群数量、物质循环、生物间遗传物质的转移及气候变化等方面所起的调控作用。在病毒调控赤潮方面,蔡雯蔚等人[15]自厦门海域分离出一株能够裂解杀藻菌SP48的噬菌体øZCW1(如图2和3),并探讨了塔玛亚历山大藻–病毒øZCW1–细菌SP48三者共培养下的相互关系,研究发现病毒可以通过裂解杀藻菌来间接调控赤潮藻的生长,这是首次发现病毒具有间接影响赤潮形成和消亡过程的证据,也为人们更加全面的认识赤潮生消机理提供了一个很好的模型。
2.2. 远海海域藻类病毒多样性
虽然远海受人类活动的影响相对较小,但是随着人类航海运输、石油勘探等活动引发的一些环境问题,远海深海考察也逐渐进入人们的研究视野。据估计,从深海到近海海域,病毒含量范围是从3 × 106/mL到108/mL。病毒的生物量如此之大,它的种类、数量

Figure 2. Plaque formed after 24 h infection by SP48
图2. 感染杀藻菌SP48 24 h后形成的噬菌斑
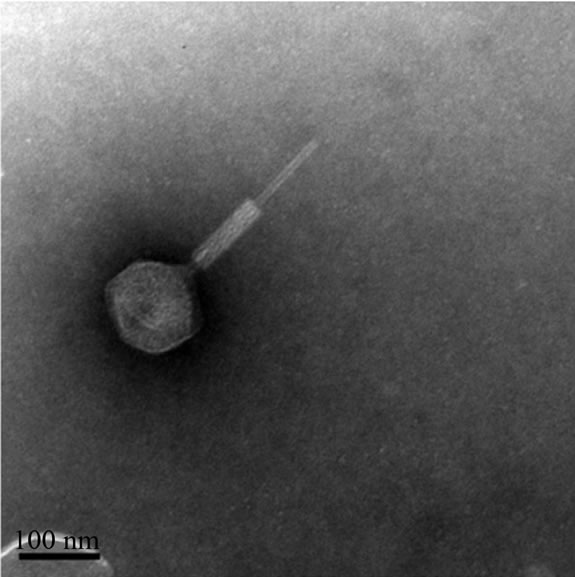
Figure 3. Characteristics of the phage øZCW1 under TEM observation
图3. 噬菌体øZCW1电镜下的形态特征
及群落演替变化势必也会影响到整个海洋的物质能量循环。
在20世纪90年代,Bratbak、Noble、Fuhrman和Marie等人分别利用电镜、荧光显微镜和流式细胞仪来监测海洋中病毒的数量[16-19]。而随着一些病毒被分离鉴定并测序,研究者们设计出一些病毒扩增引物,结合其他分子技术,越来越多的不同种类的病毒多样性信息被揭示。大量的研究表明,聚球藻病毒广泛存在于大洋环境中[8,20,21]。Suttle等[22]对太平洋地区噬菌体多样性进行研究,发现了一些新的病毒序列,并揭示了不同水体环境中病毒参与水平基因转移的现象。黄思军等人[23]利用病毒DNA聚合酶基因序列对世界多处采集病毒样品进行扩增,揭示了全球海洋中蓝细菌短尾噬菌体的广泛分布的特点。国外关于远海病毒多样性的研究较多,并且取得了一定的发展。国内相关的研究则处于起步阶段,但随着国家在海洋研究方面投入更多,远洋科考次数也越来越多,并且随着研究技术和手段的不断进步,人们对藻类病毒的多样性及分离研究也更加深入。
2.3. 极地藻类病毒多样性
极地环境恶劣,样品不容易获取,此环境下的微生物具有很多独特生理特性[24]。因此极地环境中的病毒也会表现出不同于近海、远海海域中的病毒的特征。
最初,Bratbak等[16]科学家利用透射电镜对极地环境下的病毒颗粒进行了简单计数。Kepner等人[25]于1998年对采集于南极洲的水样进行了形态观察,根据形态特征发现它们主要是感染藻类和原生动物的双链DNA病毒,这是第一例在冰雪覆盖的北极地区观察到病毒颗粒。Wilson等人[26]对南极洲的Signy岛处的病毒颗粒用透射电镜观察,他发现病毒颗粒广泛存在、形态多样、浓度高(4.9 × 106/mL~3.1 × 107/mL)且宿主范围广。他们还观察到一个头部为370 × 330 nm,尾部为1.3 μm的异常大的病毒形态。尽管研究者们也做了些关于病毒对极地动植物和微生物群落结构影响的研究,但是针对病毒的生态作用的研究还比较少[27]。
随着分子生物学及生物信息学的快速发展,以对极地环境中病毒的目的基因进行扩增或使用宏基因组的方法探索病毒多样性特征,不仅获得了病毒种类丰度信息,还得到了部分病毒–细菌相互作用的线索。如Laybourn等[28]对南极的一个咸湖里的病毒动态学特征及溶源性进行了调查分析。作者发现,病毒粒子浓度大,达到1.26 × 108/mL,病毒/宿主比率为70.6,溶源性比例高,此研究中病毒的高溶源性和高病毒、宿主比率是以往极地环境病毒研究中最高的。由此看来,极地环境下病毒与一般环境病毒相比在性质上表现出很大的不同。
人们对不同生境中病毒的多态性分布的研究,已经取得了一定的成果。但是由于不同种病毒核酸序列差别大,保守的通用引物现在还未发现,所以我们现在所了解的多样性信息仍然只是一小部分。
3. 遗传类型多样性
3.1. 藻病毒
藻病毒,是指感染真核藻类的病毒[29-40]。大部分归属于藻类DNA病毒科(Phycodnacirida)和海洋RNA病毒科(Marnaviridae),除此之外,还有一些未分类的藻病毒。藻类DNA病毒科是双链DNA病毒,基因组大小在60~560 kb[29,37,41-46]。小球藻病毒PBCV-1是研究较深入、较清楚的真核藻类DNA病毒,于是在研究真核藻类病毒多样性、结构及与宿主作用关系时,它常常作为代表病毒株。
PBCV-1全基因组长度为333 kbp,G + C含量为40%,其全部核苷酸序列于1996年由Li和Lu等定序[47]。随着研究的深入,越来越多的基因功能得到注释。受此研究的启发,研究者对其它分离到的藻病毒也进行全基因组测序,并通过比较它们的序列信息,发现基因组中存在着某段保守序列。于是人们推测这种保守序列可能是进化的结果,Chen等人[48]基于感染内共生小球藻的病毒PBCV-1、NY-2A和感染鞭毛藻的病毒MpV-SP1共同保守序列设计出引物对AVS1/ AVS2,这开启了研究藻类DNA病毒科病毒的遗传分子多样性的时代[10,49-61]。随后,Schroeder等[62]发现球石藻病毒的主要衣壳蛋白(major capsid protein,MCP)基因具有保守性,可用作靶标基因研究球石藻病毒属的多样性。关于MCP基因研究藻类病毒多样性的研究也有较多,研究者们针对不同水体、不同季节、不同深度,从局部到世界范围内均有大量的研究,发现了很多新的病毒序列[49,51,53,55-58,63-68],不仅揭示了真核藻类病毒的多样性特征[4,57,61,69],还部分揭示了藻病毒与宿主之间进行水平基因转移的证据,并指出病毒参与的水平基因转移可能是造成生物多样性的原因[58]。本实验室也开展了相关研究,如蔡雯蔚等利用AVS1/ AVS2引物对厦门近海真核藻类病毒多样性进行了研究,发现主要类群为Ostreococcuslucimarinus藻病毒。藻类病毒的分离方面,黄丽萍利用实验室的无菌化藻种三角褐指藻从厦门海域分离出特殊溶藻微生物PTV1(图4),陈章然利用无菌化的自养小球藻分离藻病毒并进行了初步的研究工作(图5)。藻的空斑图像如图4、5。
3.2. 噬藻体
蓝藻病毒同噬菌体相似,因此通常称蓝藻病毒为“噬藻体”(Cyanophage),其dsDNA基因组大小约30~60 kb。噬藻体在分类学上划在噬菌体一类,共有3个科:肌病毒科(Myoviridae)、短尾病毒科(Podoviridae)
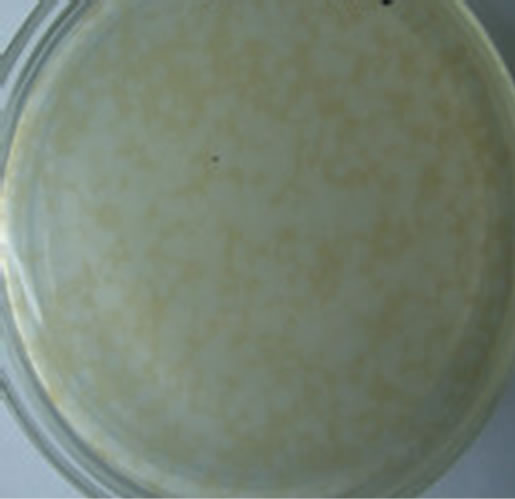
Figure 4. PTV1 plaque formed in the Phaeodactylumtriconutum plate
图4. PTV1在三角褐指藻平板上形成的藻空斑

Figure 5. Plaque formed in the Chlorella qutotropica plate
图5. 在自养小球藻平板上形成的藻空斑
和长尾病毒科(Siphoviridae),分别对应于T4、T7和lambda为代表的噬菌体科[70]。
目前研究最多的是聚球藻病毒和原绿球藻病毒[71],但有关噬藻体遗传多样性的资料还不是很多,主要因为噬藻体是高度差异性的病毒类群,缺乏广泛的遗传标记。直到2002年5月发表了第一株噬藻体“P60”的基因组全序列的文章,基因组全长47,827 bp,序列分析发现P60的DNA复制系统由DNA引发酶、DNA解旋酶和DNA聚合酶组成[70-74]。研究者们现已从海洋中分离了多株聚球藻病毒[75],它们在3个病毒科中都有分布,但是遗传学差异很大,短尾病毒科噬藻体的绝大多数是裂解性的,肌病毒科和长尾病毒科的噬藻体则大多是溶源性的。
随着研究的深入,研究者发现肌尾病毒科噬藻体的结构蛋白基因具有相当强的保守性,因此国内外利用该基因[76-79]对多种不同环境条件下的噬藻体序列扩增,取得一系列成果。如Chen等[76]发现河口与开放海域间噬藻体种类组成及结构完全不同,同一位点处不同深度差异也很大。人们最初的研究主要是针对海水,随后Suttle等[22]发现淡水环境中噬藻体的多样性也十分丰富,亲缘关系较近的宿主和感染它们的病毒是广泛分布的,并且其多样性可能是因为不同环境下噬藻体间发生了水平基因转移。黄思军等人[23]根据噬藻体DNA聚合酶基因序列设计引物对来自北大西洋、赤道太平洋、南太平洋与中国南海取来的海水进行噬藻体序列扩增,发现噬藻体广泛分布于广大海域,并且他们在所有的样品中都检测到蓝细菌短尾噬菌体subclusterⅧ类群的序列,暗示了它是一个广泛分布的基因型。以上情况表明噬藻体的分布具有全球性,生物多样性相当丰富。
4. 藻类病毒分子遗传标记
藻类病毒是高度差异性病毒类群,因此寻找应用性好的遗传标记成为近年来噬藻体分子生物学研究的关键[80]。
4.1. DNA聚合酶基因
许多学者研究DNA聚合酶的结构和功能以及编码基因,发现在DNA聚合酶中有高度保守的胺基酸序列。由于DNA聚合酶的必需性和保守性,它可作为研究生物体间演化关系的分子标记[74]。基于保守胺基酸序列,Chen等设计兼并引物通过巢式PCR扩增藻病毒聚合酶基因,并结合RFLP和DGGE对藻病毒进行了遗传多样性的分析,AVS1/AVS2[43,81]引物对现已广泛用于真核藻类病毒基因扩增。用DNA聚合酶基因检测藻病毒多样性的方法同样也适用于噬菌体和噬藻体[82]。虽然用聚合酶进行分类会排除不编码聚合酶的病毒,在确定病毒间进化关系上存在不足,但是相对如今的研究水平,在病毒的分类方面仍可以广泛应用。
4.2. 结构蛋白基因g20
1998年Fuller等[83]发现遗传起源截然不同的肌病毒科噬藻体S-PM2、S-BnM1和S-WHM1共有1个保守基因,此基因能编码病毒结构蛋白,与噬菌体T4的衣壳组装蛋白编码序列极其相似。研究发现这些序列具有高度的保守性,故可用作研究噬藻体遗传多样性的分子标记。
最早,研究者针对噬藻体中结构基因g20的保守序列设计兼并引物CPS1和CPS2,证明能特异性地扩增海洋噬藻体。在此基础上,又设计了几对引物用于噬藻体多样性的研究。如Chen等利用引物对CPS1/CPS2扩增出病毒组装蛋白g20的592 bp基因[76],Marston等[77]使用了四对引物对相同样品进行扩增,并进行了一定的比较(见表1)。在使用引物时,必须考虑到引物的特异性及可扩增长度,故研究者多采用CPS1/CPS8引物扩增噬菌体基因并研究相应序列的系统发育关系。
4.3. g91基因
Takashima和Yoshida等[84]根据感染有毒铜绿微囊藻的MaLMM01尾鞘蛋白编码基因g91设计实时定量PCR引物,对铜绿微囊藻水华发生前后水体中的MaLLM01进行定量分析,为淡水噬藻体研究及铜绿微囊藻水华的生态影响提供了新方法。通过此方法以日本某湖为研究对象。研究发现:当铜绿微囊藻数量下降时噬藻体的丰度明显上升,它们之间呈负相关。另外,数据也表明噬藻体动力学可能会影响产微囊藻毒素和不产微囊藻毒素的群落更替。
4.4. MazG基因
MazG是细菌中的焦磷核苷酸水解酶,起到调节营养压力应激的作用,是细胞程序性死亡的调控子[85,86]。Bryan等[87]根据MazG基因构建了噬藻体与其宿主的系统树,发现两者并没有归在同一序列簇中,推测噬藻体的MazG基因可能不是在其宿主中获得的,海洋噬藻体可能通过水平基因转移的方式进行了强烈的MazG基因交换。Goldsmith等人利用新的编码焦磷酸水解酶的标记基因phoH研究海洋噬菌体的多样性,发现phoH基因存在于所研究的6个位点,并且序列多样性丰富。大多数序列属于未可培养的。不同深度不同位点有着独特的phoH组成[88]。很多相关研究都揭示了该基因在水平基因转移中所起的作用[89,90]。
4.5. 光合基因psbA和psbD
psbD、psbA基因编码光系统Ⅱ(PSⅡ)反应中心蛋白D1和D2蛋白,研究发现它们具有一定的保守性,而蓝藻是最为丰富和常见的、能够进行光合作用的物种,因此光合基因可作为蓝藻株系之间的鉴定依据。众多研究结果表明在海洋聚球藻和绿球藻的噬藻体中都发现了这两个基因,之后在许多噬藻体中均有发现。噬藻体的光合作用基因能够在感染宿主的过程中表达,增加宿主的光合作用,增加宿主的光合作用能力,同时增加病毒释放量,提高自身生存率。这种机

Table 1. PCR primer for the research of cyanophage diversity
表1. 用于研究噬藻体多样性的PCR引物
制增加了病毒的适应性[91-93]。在培养研究的基础上,众多科学家展开了自然群体中病毒光合基因多样性的探索。2008年报道了高纬度冷水中病毒光合基因的多样性。研究发现分离到的病毒多数含有psbA基因,有的不含有psbD基因[93]。同年,Chenard[94]利用psbA基因探讨了海水和淡水中噬藻体的序列多样性,结果表明同一区域的病毒psbA序列都会形成不同的分支,这为探究水生环境下噬藻体的多样性和进化史提供了另外的基因标记。
5. 主要分析技术
对海洋藻类病毒多样性的分析研究主要是结合传统的病毒分离纯化技术和现今的常用分子生物学技术,并且随着高通量测序技术的快速发展,获得的信息量逐渐增大。当然研究者主要还是以PCR为核心技术,结合DGGE、PFGE、RFLP等高灵敏度的检测方法加以检测,或是利用核酸分子杂交、DNA微阵列和宏基因组学等手段进行研究。
5.1. PCR扩增藻类病毒保守序列
PCR技术在藻类病毒检测中显示了巨大的潜力及广阔的应用前景,现已用于藻类病毒种类分布、分子进化图谱及病毒宿主进化关系的研究中。PCR技术中的关键就是标记基因,而上文中已经提到几种常用的藻类病毒扩增引物,并且随着研究的深入和藻类病毒基因库的不断丰富,将会获得更多的高专一性、高特异性的引物序列,利用PCR技术对藻类病毒进行研究的方法将日趋完善。通过此方法,人们已经获得了一些以前用常规方法所不能得到的病毒种群结构的相关数据,并且不需要了解宿主相关的信息,不需对宿主和病毒进行纯化培养,且灵敏度高、准确性好。缺点是对引物的依赖性高,仅能从宏观上了解病毒的组成,而不能获得病毒的纯培养。
5.2. 脉冲场凝胶电泳(PFGE)
脉冲电场凝胶电泳是一项全基因组指纹图谱分析技术,是80年代初期发展起来的,该技术靠有规律的改变电场方向而使大分子DNA得以分离。能根据可识别条带揭示海洋藻类病毒基因组的大小、丰度及病毒与环境因素之间的相互关系。这一独具高分辨力使脉冲场电泳技术的应用范围几乎涉及所有的生物基因组研究。目前,已能分辨高达60,000 kb乃至100,000 kb以上的DNA片段。最近,PFGE被应用于水环境中病毒群落的动力学研究。如Sandaa等[95]利用PFGE技术调查了挪威近海病毒群落的季节性变化,结果表明该海域病毒多样性十分丰富,表现出动态的季节性变化,有29种不同大小的病毒种类,基因组大小范围是在26 kb~500 kb。运用脉冲场电泳不仅可以研究病毒的基因组大小、不同基因组大小的病毒丰度,还能对不同基因组大小的浮游病毒与环境因素间的相互关系进行研究。例如,Holmfeldt等[96]对Bothnian湾和Bothnian海的理化和生物因素的相关性进行了研究,均揭示出病毒种群动态与环境参数有关。PFGE是一种快速检测病毒种群的方法,但运用PFGE检测病毒基因组多样性存在明显的缺点:PFGE适于研究病毒群落的整体结构,而不适用于特定病毒类群的研究;PFGE不能有效的将具有相似大小基因组的病毒分离开来,低估了病毒多样性[74]。
5.3. 限制性片段长度多态性(RFLP)
RFLP技术的主要原理是利用不同DNA片段中特定限制性内切酶酶切位点的不同产生不同的限制性片段,从而将其分开。现在它已广泛应用于细菌、真核藻和病毒群落结构的研究。S. Jiang等人[97]研究了靠近南加利福尼亚某片海域的噬藻体的多样性及垂直分布特征,发现此区域的病毒基因组大小在40 kb~200 kb之间,并且病毒多样性远比其宿主的多样性要丰富。Jameson等运用RFLP技术检测了北–南大西洋横断面处的蓝细菌肌病毒的种群多样性,得到630个g20基因克隆,建库分析发现了许多新的g20分枝[98]。更有研究显示通过此技术发现了水生系统中病毒参与的水平基因转移现象[99],而病毒介导的水平基因转移是造成群落多样性的一个重要原因。RFLP技术快捷、敏感、可重复性强,是检测微生物群落结构和功能的常用方法。而以上研究表明RFLP筛选法是一种鉴定未知病毒的实用方法。
5.4. 变性梯度凝胶电泳(DGGE)
变性梯度凝胶电泳技术,主要原理是利用梯度变性胶来分离不同的DNA片段。DGGE一般是结合PCR同时使用,首先采用PCR方法利用特征引物对藻类病毒的目的基因进行扩增,然后利用DGGE把不同的碱基组成的序列分开,按照条带位置的不同割取相应条带测序,进而测定藻类病毒的分布的多样性和动态变化。虽然DGGE使用广泛,能快速的检测环境中微生物的多样性,但是仍有缺陷。如DGGE分离片段数不能代表实际扩增到的靶序列;差异性很高的序列可能会迁移到相同位置,而实际上几乎相同的序列却可能在胶上相差很远,可能是因为引物是兼并引物,使同一模板产生多重PCR产物。所以在使用DGGE研究病毒多样性时最好采用特异引物。
5.5. 核酸分子杂交技术
核酸分子杂交技术是基于核酸链间互补碱基对的特异性结合,测定核酸碱基顺序同源性的一种现代技术。近年来也有不少研究者利用此方法检测藻类病毒。杂交的双方是待测核酸序列及探针,探针必须是已知的病毒序列,待测序列可以是克隆的基因也可以是基因组。这种方法得到的病毒多样性信息比较少,因为数据库里的病毒序列信息毕竟有限,在设计探针的时候难免会有不全,检测的结果也只是已知的病毒序列,得不到新的基因。但这种方法准确性非常高,甚至可以检测到同源基因的差异性。
5.6. DNA微阵列
DNA微阵列也称为DNA芯片(DNA chip)它是采用原位合成或直接点样的方法将大量DNA片段或寡核苷酸片段以预先设计的方式排列在载体上形成微矩阵,待检测样品用荧光分子标记后,与微阵列杂交,通过荧光扫描及计算机分析即可获得样品中大量的基因序列及表达信息,以达到快速、高效、高通量的分析生物信息的目的。而DNA芯片应用于病毒多样性研究则要从球石藻病毒的全基因组测序开始,Allen等[100]以全基因组信息为基础设计探针,从基因组水平上去评估球石藻病毒属的多样性,也从转录水平调查球石藻病毒的感染过程。然后他们利用、标记基因、宿主范围和微阵列从基因型到表型上去探讨球石藻病毒的株间多样性,通过微阵列结果发现以前认为是相同的两个病毒株实际上完全不同[101]。在以往的研究中,研究者只是研究病毒的单个基因,在确定属内病毒间的遗传距离时受到限制,而基因芯片技术却可以分析病毒属内的多样性。DNA芯片技术可以检测到同源基因的差异性,精度更高。但是它和核酸序列杂交技术一样,必须用已知病毒基因座探针,也只是验证下目的序列是否存在,得不到新的基因。
5.7. 宏基因组学
宏基因组学就是直接将特定环境中的全部DNA,克隆到可培养的宿主细胞中,构建宏基因组文库,从所获得的重组克隆子中筛选活性物质和相关基因,避开了环境微生物分离培养的难题。宏基因组学揭示了惊人的遗传多样性,尤其是在海洋环境病毒群落多样性的研究中。例如,Ngando等[102]利用宏基因组技术对Senegal的一个超盐湖进行了病毒——宿主多样性研究,发现了许多的序列是现有数据库中没有的,尤其是在极端地热环境下发现古细菌病毒序列。宏基因组文库技术可以避免PCR偏好性所导致的误差,但PCR文库可矫正构建宏基因组文库的基因遗漏现象,为了减少单一技术造成的误差,将宏基因组文库与PCR文库结合起来进行分析将会更准确地反映不同生境病毒的多样性。
6. 研究展望
藻类病毒是普遍存在于水生系统中,作为藻类的致死剂、基因信息的载体及群落结构的调节者,势必会引起更多生态学者和生物工作者的关注。但是因为不断发现海水中存在非常丰富的藻类病毒,所以很多的关于藻类病毒存在、丰度、生态作用的研究工作都是在海洋环境中,淡水藻类病毒的研究显得十分欠缺。另外,病毒遗传多样性的研究是以数据库中大量的相关遗传信息为背景和基础的,而目前已知的藻病毒、噬藻体等的全基因组序列还比较少。要获得病毒的全基因组序列,必须利用传统的单株病毒分离纯化方法,而目前水体中很多的藻类还不能进行体外培养,所以很难在体外建立比较稳定的针对不同病毒的培养系统,这是限制藻类病毒研究的因素之一。
我国在藻类病毒方面的研究还处于发展阶段,真核藻类病毒方面除了小球藻病毒等少数病毒的分离工作外,未见更多的报道。随着我国经济的发展,人类活动增加,水体富营养化程度和赤潮水华现象日益严重,赤潮生物防治工作十分紧迫,而藻类病毒的基础研究在我国还相当薄弱,需要研究人员不断努力,把我国藻类病毒学研究逐步提高到国际水平。我国海水、淡水资源丰富,这为我们分离出一批具有很强杀藻效果的藻类病毒提供了很好的物质基础。另外关于藻类病毒的生态应用也基本上停留在理论水平上,本实验室也有关于藻类病毒的杀藻模拟方面的研究工作,如蔡雯蔚发现了一株名为øZCW1的病毒,它能通过调控杀藻菌的生长来达到间接调控赤潮藻塔玛亚历山大藻的生长。当然藻类病毒应用于环境中需要我们对病毒——宿主体系相互作用模式有着非常充分的了解。
结合上述研究现状和发展趋势,笔者认为,在未来的关于海洋藻类病毒的研究过程中,人们应当从以下几个方面作为着眼点:1) 分离出更多的纯系藻类病毒并测序,获得尽可能多的病毒序列信息;2) 加强藻类病毒生理学、基因组学的研究,充分了解它们的生理特征和遗传信息,研究不同宿主范围、生理及生态型病毒在环境中自然分布;3) 藻类病毒和宿主藻稳定感染体系的系统研究。目前大部分的研究都是以病毒为单一研究对象,而忽略了病毒在实际生态环境中受理化因素和其他生物因素的影响。所以必须从整体出发研究藻类病毒的丰度、多样性以及感染规律;4) 着重于对富营养化水体中藻类数量变化的控制机制的研究,将藻类病毒作为防治有害藻华有效生物调控因子加以利用;5) 开展藻类病毒的基因组学及蛋白组学研究,了解病毒感染的分子基础,为藻类病毒是否能够大规模应用于实际环境中的生态安全性提供参考。
参考文献 (References)
[1] B. X. Wang, X. R. Yang, Y. Y. Zhou, J. L. Lv, J. Q. Su, Y. Tian, J. Zhang, G. H. Lin and T. L. Zheng. An algicidal protein produced by bacterium isolated from the Donghai Sea, China. Harmful Algae, 2012, 13: 83-88
[2] 郑天凌, 吕静琳, 周艳艳等. 海洋有害赤潮调控功能菌的发现与研究[J]. 厦门大学学报: 自然科学版, 2011, 50(2): 445- 454.
[3] B. X. Wang, X. R. Yang, J. L. Lu and T. L. Zheng. A marine bacterium producing protein with algicidal activity against Alexandrium tamarense. Harmful Algae, 2012, 13: 83-88.
[4] C. A. Suttle. Viruses in the sea. Nature, 2005, 437(7057): 356- 361.
[5] C. A. Suttl. Marine viruses—Major players in the global ecosystem. Nature Reviews Microbiology, 2007, 5(10): 801-812.
[6] 李洪波, 肖天, 林凤翱. 海洋浮游病毒的研究方法[J]. 海洋科学, 2010, 34(9): 97-101.
[7] F. Chen, C. A. Suttle and S. M. Short. Genetic diversity in marine algal virus communities as revealed by sequence analysis of DNA polymerase genes. Applied and Environmental Microbiology, 1996, 62(8): 2869-2874.
[8] J. Lu, F. Chen and R. E. Hodson. Distribution, isolation, host specificity, and diversity of cyanophages infecting marine Synechococcus spp. in river estuaries. Applied and Environmental Microbiology, 2001, 67(7): 3285-3290.
[9] F. Chen, K. Wang, S. Huang, et al. Diverse and dynamic populations of cyanobacterial podoviruses in the Chesapeake Bay unveiled through DNA polymerase gene sequences. Environmental Microbiology, 2009, 11(11): 2884-2892.
[10] 白晓歌, 汪珉, 马晶晶等. 冬季和春季长江口及其近海水域浮游病毒丰度的分析[J]. 海洋与湖沼, 2007, 38(4): 367-372.
[11] 赵苑, 武洪庆, 李洪波等. 胶州湾浮游病毒的分布研究[J]. 海洋科学, 2010, 34(7): 86-92.
[12] 王斐, 郑天凌, 洪华生. 海洋病毒在微生物食物环中的重要作用[J]. 海洋科学, 1998, 4: 41-43.
[13] 杨小茹, 郑天凌. 海洋病毒——一种新的、潜力巨大的赤潮防治工具[J]. 应用与环境生物学报, 2005, 11(5): 651-656.
[14] 王慧, 柏仕杰, 郑天凌. 海洋病毒——海洋生态系统结构与功能的重要调控者[J]. 微生物学报, 2009, 5: 552-560.
[15] W. W. Cai, et al. Influence of a bacteriophage on the population dynamics of toxic dinoflagellates by lysis of algicidal bacteria. Applied and Environmental Microbiology, 2011, 77(21): 7837- 7840.
[16] K. Børsheim, G. Bratbak and M. Heldal. Enumeration and biomass estimation of planktonic bacteria and viruses by transmission electron microscopy. Applied and Environmental Microbiology, 1990, 56(2): 352-356.
[17] R. T. Noble, J. A. Fuhrman. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquatic Microbial Ecology, 1998, 14(2): 113-118.
[18] D. Marie, C. P. D. Brussaard, R. Thyrhaug, et al. Enumeration of marine viruses in culture and natural samples by flow cytometry. Applied and Environmental Microbiology, 1999, 65(1): 45-52.
[19] 肖天, 陈騳, 张虹. 用DAPI染色检出对虾肝胰腺细小样病毒[J]. 海洋科学, 1999, 2: 2-3.
[20] C. A. Suttle. Marine cyanophages infecting oceanic and coastal strains of synechococcus: Abundance, morphology, cross-infectivity and growth characteristics. Marine Ecology-Progress Series, 1993, 92: 99-109.
[21] 蔡海元, 焦念志. 聚球藻病毒研究进展[J]. 地球科学进展, 2010, 10: 1031-1039.
[22] C. M. Short, C. A. Suttle. Nearly identical bacteriophage structural gene sequences are widely distributed in both marine and freshwater environments. Applied and Environmental Microbiology, 2005, 71(1): 480-486.
[23] S. Huang, S. W. Wilhelm, N. Jiao, et al. Ubiquitous cyanobacterial podoviruses in the global oceans unveiled through viral DNA polymerase gene sequences. The ISME Journal, 2010, 4(10): 1243- 1251.
[24] 曾胤新, 郑天凌. 极地环境污染物的微生物降解及其生物修复研究进展[J]. 极地研究, 2008, 1: 64-71.
[25] R. L. Kepner Jr., R. A. Wharton Jr. and C. A. Suttle. Viruses in Antarctic lakes. Limnology and Oceanography, 1998, 43(7): 1754- 1761.
[26] W. H. Wilson, D. Lane, D. A. Pearce, et al. Transmission electron microscope analysis of virus-like particles in the freshwater lakes of Signy Island, Antarctica. Polar Biology, 2000, 23(9): 657- 660.
[27] D. A. Pearce, W. H. Wilson. Viruses in Antarctic ecosystems. Antarctic Science, 2003, 15(3): 319-331.
[28] J. Laybourn-Parry, W. A. Marshall and N. J. Madan. Viral dynamics and patterns of lysogeny in saline Antarctic lakes. Polar Biology, 2007, 30(3): 351-358.
[29] S. M. Short. The ecology of viruses that infect eukaryotic algae. Environmental Microbiology, 2012.
[30] 张远征, 王苏燕, 盛刚等. 小球藻病毒的分离[J]. 微生物学报, 1996, 1: 67-68.
[31] L. V. Tchistyakova, A. S. Karpov and A. V. Goodkov. Experimentally induced endocytobiosis of Amoeba amazonas with different Chlorella vulgaris strains: Fine structural investigations. Endocytobiosis and Cell Research, 1997, 12(1-2): 57-63.
[32] H. Hosoya, N. Nishihara, S. Horiike, et al. Cloning and characterization of endosymbiotic algae isolated from Paramecium bursuria. Protoplasma, 1998, 203(1-2): 91-99.
[33] C. P. Queimalinos, B. E. Modenutti and E. G. Balseiro. Symbiotic association of the ciliate Ophrydium naumanni with Chlorella causing a deep chlorophyll a maximum in an oligotrophic South Andes Lake. Journal of Plankton Research, 1999, 21(1): 167-178.
[34] L. P. Villarreal, V. R. DeFilippis. A hypothesis for DNA viruses as the origin of eukaryotic replication proteins. Journal of Virology, 2000, 74(15): 7079-7084.
[35] M. Kalafatic, G. Kovacevic, N. Ljubesic, et al. Effects of ciprofloxacin on green hydra and endosymbiotic alga. Periodicum Biologorum, 2001, 103(3): 267-272.
[36] T. Kawano, T. Kadono, T. Kosaka, et al. Green paramecia as an evolutionary winner of oxidative symbiosis: A hypothesis and supportive data. Zeitschrift fur Naturforschung C—A Journal of Biosciences, 2004, 59(7-8): 538-542.
[37] M. Kang, D. D. Dunigan and J. L. Van Etten. Chlorovirus: A genus of Phycodnaviridae that infects certain chlorella—Like green algae. Molecular Plant Pathology, 2005, 6(3): 213-224.
[38] F. Wolfe-Simon, D. Grzebyk, O. Schofield, et al. The role and evolution of superoxide dismutases in algae. Journal of Phycology, 2005, 41(3): 453-465.
[39] B. P. Karajan, A. E. Vishnyakov, M. V. Tavrovskaya, et al. Infection of algae-free Climacostomum virens with symbiotic Chlorella sp. isolated from algae-containing C-virens. European Journal of Protistology, 2007, 43(2): 141-146.
[40] S. M. Short. The ecology of viruses that infect eukaryotic algae. Environmental Microbiology, 2012: 1-19.
[41] D. D. Dunigan, L. A. Fitzgerald and J. L. Van Etten. Phycodnaviruses: A peek at genetic diversity. Virus Research, 2006, 117 (1): 119-132.
[42] K. D. Weynberg, M. J. Allen, K. Ashelford, et al. From small hosts come big viruses: The complete genome of a second Ostreococcus tauri virus, OtV-1. Environmental Microbiology, 2009, 11(11): 2821-2839.
[43] L. Bellec, N. Grimsley and Y. Desdevises. Isolation of Prasinoviruses of the green unicellular algae Ostreococcus spp. on a worldwide geographical scale. Applied and Environmental Microbiology, 2010, 76(1): 96-101.
[44] M. J. L. Coolen. 7000 years of Emiliania huxleyi viruses in the Black Sea. Science, 2011, 333(6041): 451.
[45] A. R. Matteson. Quantification and ecological perspectives on cyanophage and aquatic viruses. University of Tennessee, 2011.
[46] J. L. Van Etten, D. D. Dunigan. Chloroviruses: Not your everyday plant virus. Trends in Plant Science, 2011, 17(1): 1-8.
[47] 韩继刚. 小球藻病毒的分离提纯及其几丁质酶基因的克隆[J]. 2000.
[48] F. Chen, C. A. Suttle. Amplification of DNA polymerase gene fragments from viruses infecting microalgae. Applied and Environmental Microbiology, 1995, 61(4): 1274-1278.
[49] A. I. Culley, A. S. Lang and C. A. Suttle. High diversity of unknown picorna-like viruses in the sea. Nature, 2003, 424(6952): 1054-1057.
[50] A. G. Jones. Sea turtles: Old viruses and new tricks. Current Biology, 2004, 14(19): R842-R843.
[51] R. Danovaro, C. Corinaldesi, A. Dell’Anno, et al. Viruses, prokaryotes and DNA in the sediments of a deep-hypersaline anoxic basin (DHAB) of the Mediterranean Sea. Environmental Microbiology, 2005, 7(4): 586-592.
[52] L. E. Wells, J. W. Deming. Modelled and measured dynamics of viruses in Arctic winter sea-ice brines. Environmental Microbiology, 2006. 8(6): 1115-1121.
[53] M. Breitbart, L. R. Thompson, C. A. Suttle, et al. Exploring the vast diversity of marine viruses. Oceanography, 2007, 20(2): 135- 139.
[54] 白晓歌. 北黄海浮游病毒的丰度变化及与微微型浮游植物、异养细菌相关性研究[D]. 中国海洋大学, 2007.
[55] A. I. Culley, B. F. Asuncion and G. F. Steward. Detection of inteins among diverse DNA polymerase genes of uncultivated members of the Phycodnaviridae. The ISME Journal, 2008, 3(4): 409-418.
[56] K. Nagasaki. Dinoflagellates, diatoms, and their viruses. The Journal of Microbiology, 2008, 46(3): 235-243.
[57] S. M. Short, C. M. Short. Diversity of algal viruses in various North American freshwater environments. Aquatic Microbial Ecology, 2008. 51(1): 13-21.
[58] J. L. Clasen, C. A. Suttle. Identification of freshwater Phycodnaviridae and their potential phytoplankton hosts, using DNA pol sequence fragments and a genetic-distance analysis. Applied and Environmental Microbiology, 2009, 75(4): 991.
[59] Y. Park, K. Lee, Y. S. Lee, et al. Detection of diverse marine algal viruses in the South Sea regions of Korea by PCR amplification of the DNA polymerase and major capsid protein genes. Virus Research, 2011, 159(1): 1-82.
[60] S. M. Short, O. Rusanova and M. A. Staniewski. Novel phycodnavirus genes amplified from Canadian freshwater environments. Aquatic Microbial Ecology, 2011, 63(1): 61-67.
[61] J. Moss, M. J. Butler IV, D. C. Behringer, et al. Genetic diversity of the Caribbean spiny lobster virus, Panulirus argus virus 1 (PaV1), and the discovery of PaV1 in lobster postlarvae. Aquatic Biology, 2012, 14(3): 223-232.
[62] D. Schroeder, J. Oke, G. Malin, et al. Coccolithovirus (Phycodnaviridae): Characterisation of a new large dsDNA algal virus that infects Emiliana huxleyi. Archives of Virology, 2002, 147(9): 1685-1698.
[63] S. Oshima, J. Hata, C. Segawa, et al. A method for direct DNA amplification of uncharacterized DNA viruses and for development of a viral polymerase chain reaction assay: Application to the red sea bream iridovirus. Analytical Biochemistry, 1996, 242(1): 15-19.
[64] R. Danovaro, A. Dell’Anno, A. Trucco, et al. Determination of virus abundance in marine sediments. Applied and Environmental Microbiology, 2001, 67(3): 1384-1387.
[65] 李娟, 病毒: 海洋生态动力学和疾病学研究的创新点[J]. 海洋湖沼通报, 2005, 2: 79-87.
[66] 刘艳鸣. 武汉东湖浮游病毒的丰度及多样性[J]. 水生生物学报, 2005, 29(1): 1-6.
[67] 刘艳鸣. 东湖浮游病毒形态多样性、时空分布及基因组大小测定[D]. 中国科学院研究生院水生生物研究所, 2005.
[68] M. J. Allen. Aquatic virus diversity accessed through omic techniques: A route map to function. Current Opinion in Microbiology, 2008: 226-232.
[69] C. Winter, M. G. Weinbauer. Randomly amplified polymorphic DNA reveals tight links between viruses and microbes in the bathypelagic zone of the Northwestern Mediterranean Sea. Applied and Environmental Microbiology, 2010, 76(20): 6724-6732.
[70] 赵以军, 裴达, 石正丽等. 藻类病毒研究40年[J]. 华中师范大学学报(自然科学版), 2003, 37(3): 399-405.
[71] 王淼星, 向安, 魏大巧等. 噬藻体研究进展[J]. 生物技术, 2009, 6: 89-92.
[72] 赵以军, 石正丽, 黄国锦等. 蓝藻病毒(噬藻体)的研究进展[J]. 中国病毒学, 1999, 14(2): 100-105.
[73] 张奇亚. 浮游病毒[J]. 水生生物学报, 2002, 26(6): 691-696.
[74] 汪珉. 浮游病毒遗传多样性的研究进展[J]. 中国海洋大学学报(自然科学版), 2009, 39(6): 1224-1232.
[75] J. Lu, F. Chen and R. E. Hodson. Distribution, isolation, host specificity, and diversity of cyanophages infecting marine Synechococcus spp. in river estuaries. Applied and Environmental Microbiology, 2001, 67(7): 3285-3290.
[76] Y. Zhong, F. Chen, S. W. Wilhelm, et al. Phylogenetic diversity of marine cyanophage isolates and natural virus communities as revealed by sequences of viral capsid assembly protein gene g20. Applied and Environmental Microbiology, 2002, 68(4): 1576- 1584.
[77] M. F. Marston, J. L. Sallee. Genetic diversity and temporal variation in the cyanophage community infecting marine Synechococcus species in Rhode Island’s coastal waters. Applied and Environmental Microbiology, 2003, 69(8): 4639-4647.
[78] U. Dorigo, S. Jacquet and J. F. Humbert. Cyanophage diversity, inferred from g20 gene analyses, in the largest natural lake in France, Lake Bourget. Applied and Environmental Microbiology, 2004, 70(2): 1017-1022.
[79] K. Wang, F. Chen. Genetic diversity and population dynamics of cyanophage communities in the Chesapeake Bay. Aquatic Microbial Ecology, 2004, 34(2): 105-116.
[80] D. Q. Wei, L. Liu, A. Xiang and X. S. Xia. Molecular biological studies on cyanophages. Biotechnology Bulletin, 2009, 5: 31-38.
[81] T. Kawano, K. Irie, S. Furukawa, et al. A green paramecium strain with abnormal growth of symbiotic algae. Zeitschrift fur Naturforschung Section C—A Journal of Biosciences, 2010, 65(11-12): 681-687.
[82] S. Short, C. Suttle. Use of the polymerase chain reaction and denaturing gradient gel electrophoresis to study diversity in natural virus communities. Hydrobiologia, 1999, 401: 19-33.
[83] Y. Zhong. Phylogenetic diveristy of marine cyanophages as revealed by sequences of viral capsid assembly protein gene g20. Applied and Environmental Microbiology, 2002, 68(4): 1576- 1584.
[84] M. Yoshida, T. Yoshida, Y. Yoshida-Takashima, et al. Real-time PCR detection of host-mediated cyanophage gene transcripts during infection of a natural Microcystis aeruginosa population. Microbes and Environments, 2010, 25(3): 211-215.
[85] I. Drobnak, A. Korenčič, R. Loris, et al. Energetics of MazG unfolding in correlation with its structural features. Journal of Molecular Biology, 2009, 392(1): 63-74.
[86] L. Lu, Q. Sun, X. Fan, et al. Mycobacterial MazG is a novel NTP pyrophosphohydrolase involved in oxidative stress response. Journal of Biological Chemistry, 2010, 285(36): 28076-28085.
[87] M. J. Bryan, N. J. Burroughs, E. M. Spence, et al. Evidence for the intense exchange of MazG in marine cyanophages by horizontal gene transfer. PloS One, 2008, 3(4): e2048.
[88] D. B. Goldsmith, G. Crosti, B. Dwivedi, et al. Development of phoH as a novel signature gene for assessing marine phage diversity. Applied and Environmental Microbiology, 2011, 77(21): 7730-7739.
[89] A. M. D. Gonçalves, D. de Sanctis and S. M. McSweeney. Structural and functional insights into DR2231 protein, the MazGlike nucleoside triphosphate pyrophosphohydrolase from Deinococcus radiodurans. Journal of Biological Chemistry, 2011, 286(35): 30691-30705.
[90] J. C. I. Espinoza, M. B. Sullivan. Phylogenomics of T4 cyanophages: Lateral gene transfer in the “core” and origins of host genes. Environmental Microbiology, 2012, 14(8): 2113-2126.
[91] D. Lindell, J. D. Jaffe, Z. I. Johnson, et al. Photosynthesis genes in marine viruses yield proteins during host infection. Nature, 2005, 438(7064): 86-89.
[92] M. B. Sullivan, D. Lindell, J. A. Lee, et al. Prevalence and evolution of core photosystem II genes in marine cyanobacterial viruses and their hosts. PLoS Biology, 2006, 4(8): e234.
[93] R. A. Sandaa, M. Clokie and N. H. Mann. Photosynthetic genes in viral populations with a large genomic size range from Norwegian coastal waters. FEMS Microbiology Ecology, 2008, 63(1): 2-11.
[94] C. Chenard, C. A. Suttle. Phylogenetic diversity of sequences of cyanophage photosynthetic gene psbA in marine and freshwaters. Applied and Environmental Microbiology, 2008, 74(17): 5317- 5324.
[95] R. A. Sandaa, A. Larsen. Seasonal variations in virus-host populations in Norwegian coastal waters: Focusing on the cyanophage community infecting marine Synechococcus spp. Applied and Environmental Microbiology, 2006, 72(7): 4610-4618.
[96] K. Holmfeldt, J. Titelman and L. Riemann. Virus production and lysate recycling in different sub-basins of the Northern Baltic Sea. Microbial Ecology, 2010, 60(3): 572-580.
[97] S. Jiang, W. Fu, W. Chu, et al. The vertical distribution and diversity of marine bacteriophage at a station off Southern California. Microbial Ecology, 2003, 45(4): 399-410.
[98] J. M. Labonté, K. E. Reid and C. A. Suttle. Phylogenetic analysis indicates evolutionary diversity and environmental segregation of marine podovirus DNA polymerase gene sequences. Applied and Environmental Microbiology, 2009, 75(11): 3634-3640.
[99] P. A. Sobecky, T. H. Hazen. Horizontal gene transfer and mobile genetic elements in marine systems. Methods in Molecular Biology, 2009, 532: 435-453.
[100] M. J. Allen, W. H. Wilson. The coccolithovirus microarray: An array of uses. Briefings in Functional Genomics & Proteomics, 2006, 5(4): 273-279.
[101] M. J. Allen, J. Martinez-Martinez, D. C. Schroeder, et al. Use of microarrays to assess viral diversity: From genotype to phenotype. Environmental Microbiology, 2007, 9(4): 971-982.
[102] T. Sime-Ngando, S. Lucas, A. Robin, et al. Diversity of virushost systems in hypersaline Lake Retba, Senegal. Environmental Microbiology, 2011, 13(8): 1956-1972.
NOTES
*基金项目:国家自然科学基金重点项目、面上项目(40930847,40876061),福建省自然科学基金项目(2012J01150),国家级“海洋生物地球化学机制与过程”创新团队项目(41121091)和海洋公益性行业科研专项经费资助(201305022)。
#通讯作者。