Hans Journal of Computational Biology
Vol.04 No.04(2014), Article ID:14825,14
pages
10.12677/HJCB.2014.44008
Bioinformatics Analysis of Diguanylate Cyclases and c-di-GMP-Specific Phosphodiesterases from Xanthomonas Species
Xia Zou, Liangbo Huang, Jin He*
State Key Laboratory of Agricultural Microbiology, College of Life Science and Technology, Huazhong Agricultural University, Wuhan Hubei
Email: *hejin@mail.hzau.edu.cn
Received: Jan. 23rd, 2015; accepted: Feb. 4th, 2015; published: Feb. 11th, 2015
Copyright © 2014 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



ABSTRACT
The genus Xanthomonas currently comprises 27 species that collectively cause serious diseases in a wide variety of economically important crops and horticultural plants. The bacterial signal transduction mechanism and the bacterium-host plant interaction have been hot issues for microbiologists, plant pathologists and botanists. c-di-GMP is an ubiquitous bacterial second messenger. It participates in the regulation of many bacterial physiological processes, such as adhesion, EPS synthesis, biofilm formation, motility and virulence, etc. The c-di-GMP turnover is controlled by two functionally opposite enzymes: diguanylate cyclase containing GG(D/E)EF domain and c-di- GMP-specific phosphodiesterase containing EAL or HD-GYP domain. In this study, we performed bioinformatics analysis on the proteins containing GG(D/E)EF, EAL and HD-GYP domians from 15 Xanthomonas strains with complete genome sequences. The distribution and phylogenetic tree analysis revealed that the evolutionary differences of these proteins were related to their hosts; COG and GO analysis demonstrated that these functional proteins were mainly signal transduction related proteins, involved in gene transcription and bacterial motility. These results established solid foundation for the construction of the regulation network mediated by c-di-GMP and for the discovery of new drug targets for Xanthomonas control.
Keywords:Xanthomonas, Second Messenger Molecule, c-di-GMP, Diguanylate Cyclase, Phosphodiesterase, Bioinformatics

黄单胞菌中c-di-GMP二鸟苷酸环化酶和磷酸二酯酶的生物信息学分析
邹霞,黄良博,何进*
华中农业大学生命科学技术学院,农业微生物学国家重点实验室,湖北 武汉
Email: *hejin@mail.hzau.edu.cn
收稿日期:2015年1月23日;录用日期:2015年2月4日;发布日期:2015年2月11日

摘 要
黄单胞菌是一类重要的作物病原菌,其信号转导机制及其与寄主植物的相互作用一直是微生物学、植物病理学及植物学工作者研究的热点问题。c-di-GMP是一种广泛存在于细菌中的第二信使分子,参与细菌的粘附、胞外多糖的合成、生物膜形成、运动性及毒力等许多细菌生理过程的调节。c-di-GMP主要由含有GG(D/E)EF结构域的二鸟苷酸环化酶合成,由含有EAL或HD-GYP结构域的磷酸二酯酶降解,其在胞内的浓度受到二鸟苷酸环化酶与c-di-GMP特异性磷酸二酯酶的协同调节。本文对黄单胞菌属中十五株具有完整基因组序列的菌株中含有这三类结构域的蛋白进行了包括分布规律、进化树、COG及GO分析在内的生物信息学分析,揭示了GG(D/E)EF、EAL与HD-GYP结构域蛋白在黄单胞菌属中的进化差异及其与侵染对象的关联性;研究结果也表明这些结构域蛋白主要为信号转导蛋白,参与基因转录和细胞运动。这些结果为构建c-di-GMP介导的调控网络和寻找新的控制黄单胞菌的药物靶标奠定了良好的基础。
关键词 :黄单胞菌,第二信使信号分子,c-di-GMP,二鸟苷酸环化酶,磷酸二酯酶,生物信息学

1. 引言
黄单胞菌(Xanthomonas)属于变形菌门,假单胞菌科。细胞直杆状,革兰氏阴性,以单极生鞭毛运动,无芽胞和荚膜。菌落一般为黄色,光滑或粘液状。黄单胞菌属目前包括27个种,能侵染大约400多种植物,引起病害。这些植物主要是重要的农作物与园艺作物,如水稻、油菜、柑桔、番茄等[1] 。病害症状多为叶枯、叶斑、溃疡等(表1)。致病种与致病变种具备高度的宿主特异性,比如多种蔬菜、油菜等十字花科植物叶片感染野油菜黄单胞菌野油菜致病变种后发生黑腐病(black rot)导致严重减产[2] ;同时,一个种也能有多个致病变种,导致不同的病害,比如水稻感染水稻黄单胞菌不同致病变种后分别发生水稻白叶枯病(bacterial blight) [3] 和水稻细菌性条斑病(bacterial leaf streak) [4] 。因此,深入了解其致病机理对黄单胞菌的防治具有重要的意义。
细菌中的第二信使分子c-di-GMP是1987年Benziman课题组在研究葡萄糖木质素醋酸菌(Acetobacter xylinum)的纤维素生物合成途径时首先发现的[5] 。1998年Tal等人克隆了c-di-GMP的合成酶与降解酶基因[6] ,自此拉开了c-di-GMP研究的大幕。细菌体内c-di-GMP的水平是由其合成酶与降解酶协同调节的[7] 。含GG(D/E)EF结构域的二鸟苷酸环化酶(diguanylate cyclase, DGC)是c-di-GMP的合成酶,DGC能够利用两分子的GTP合成一分子c-di-GMP(图1);具有EAL结构域的磷酸二酯酶(phosphodiesterase, PDE)
Table 1. Pathogenicity of crop pathogens Xanthomonas species
表1. 黄单胞菌对作物的致病性

Figure 1. Synthesis and degradation of the c-di-GMP messenger molecule
图1. c-di-GMP信使分子的合成和降解
能够将c-di-GMP水解为pGpG,而包含HD-GYP结构域的PDE能够将c-di-GMP进一步水解为两分子GMP。涉及命名的GG(D/E)EF、EAL和HD-GYP系这些结构域活性位点的保守氨基酸残基[15] -[17] 。此外,新鉴定的枯草芽胞杆菌YybT蛋白包含DHH/DHHA1结构域,能将c-di-GMP水解为pGpG [18] 。
c-di-GMP能够调节细菌的多种生理功能,包括运动性、毒力因子产生、细胞分化、生物膜形成等,尤其在细菌的生物膜形成和致病因子产生方面起到至关重要的调节作用[7] 。近年来的研究表明c-di-GMP直接或间接参与黄单胞菌的致病途径[19] -[23] 。
随着微生物基因组大规模测序的展开,研究者可以通过基因组注释直接获得DGC和PDE,但部分注释为DGC和PDE的蛋白中并不含有保守的GG(D/E)EF或EAL结构域。因此,我们利用HMMER来进行同源蛋白搜索,以获取含有GG(D/E)EF、EAL和HD-GYP结构域的蛋白。HMMER系基于隐马尔科夫模型,给替换、插入、删除突变设置了一个位置特异性评分系统的查询文件。与BLAST及FASTA等传统序列比对和同源搜索工具相比,HMMER利用的数学模型能够更加准确地发现亲缘关系较远的同源体。
直系同源蛋白质聚类(Clusters of orthologous groups of proteins, COG)分析法由Tatusov等[24] 提出,即利用不同种族的蛋白成对相似聚类法将它们划分成各种直系同源簇,可以用同一簇中的功能已知的蛋白来注释功能未知的蛋白,从而达到对功能未知蛋白进行注释的目的。GO (Gene ontology)是一套国际标准化的基因功能描述的分类系统[25] 。GO分为三大类:Biological process、Molecular function和Cellular component,分别用来描述基因编码的产物所参与的生物过程、所具有的分子功能及所处的细胞环境。蛋白质或者编码基因可以通过ID对应或序列注释的方法找到与之对应的GO号,而GO号可以对应到不同层次的GO term来进行功能分类或细胞定位。
NCBI上目前公布的全基因组测序完成的黄单胞菌属共十五株,本文以这十五株黄单胞菌属菌株为对象,具体分析了这些菌株细胞内含有GG(D/E)EF、EAL及HD-GYP结构域的相关蛋白的分布及进化关系,并进行了COG功能注释和GO功能分类,以期为更好地研究水稻黄单胞菌中c-di-GMP的调控网络,为寻找防治黄单胞菌的药物靶标奠定基础。
2. 材料与方法
2.1. 序列来源
选取如下十五株全基因组测序完成的黄单胞菌属菌株为对象进行分析。全基因组序列来自GenBank (http://www.ncbi.nlm.nih.gov/genome)。部分菌株的基因组注释来自华中农业大学DIGAP数据库 (http://ibi.hzau.edu.cn/digap/phytopathogens.php)。菌株全称、简称及NCBI登入号(GenBank accession)见表2。十五株黄单胞菌中包括水稻黄单胞菌四株(两株XOO(其中一株的基因与编码蛋白带有下划线)、一株XOC和一株PXO),野油菜黄单胞菌五株(XC、XCC、xccb100、XCV、XCR),地毯草黄单胞菌三株(XAC、XAC29、XACM),以及柑桔黄单胞菌(XCAW)、白纹黄单胞菌(XALc)与褐色黄单胞菌(XFF4834R)各一株。
2.2. 序列的搜索获取
先在NCBI后台ftp://ftp.ncbi.nlm.nih.gov/genomes/Bacteria/下载十五株全基因组测序完成的黄单胞菌属菌株蛋白序列,在Pfam数据库上分别搜索GG(D/E)EF、EAL及HD-GYP蛋白家族(分别对应PF00990、
Table 2. Proteins containing GG(D/E)EF, EAL and HD-GYP domains in Xanthomonas
表2. 黄单胞菌中c-di-GMP二鸟苷酸环化酶与磷酸二酯酶编码基因数目统计
PF00563和PF11871),在Alignment中下载比对好的Stockholm格式文件。利用HMMER程序分别在Pfam库中比对搜索对应结构域的序列,设定E-value为1e-3,得到目的蛋白。Interpro是一个集成了包括Pfam、TIGRFAM、SMART、PANTHER等数据库在内的蛋白质家族、结构域和功能位点的非冗余数据库,我们将得到的蛋白利用Interpro的注释软件InterProScan5进行注释,得出格式为xml的比对结果,编写Perl脚本程序提取信息以检验确认。GG(D/E)EF和EAL结构域蛋白随后用Perl脚本提取。
2.3. 多重序列比对及进化树构建
蛋白质多重序列比对采用ClustalW软件[26] 进行,将大片段的缺失插入和一些模糊不清的联配位置从联配结果中去除。进化树利用同源蛋白非同义突变的Neighbor-Joining方法分析,采用ClustalW和Mega (Molecular Evolutionary Genetics Analysis) Vision 6软件[27] 进行构建,采用引导程序方法(Boot Straping)检验蛋白树的稳健性(1000次重复),设置Cut-off Value for Consensus Tree为50%,选择泊松修正(Poisson Correction)模型。
2.4. 共线性分析
四株水稻黄单胞菌的共线性分析使用MCScanX软件[28] 完成,主要分为两步,第一步:使用基因组的蛋白序列进行blastp比对,比完之后直接作为这个软件的输入文件运行分析,第二步:在对应关系基础上搜索共线性。
2.5. COG功能分析
COG注释主要通过如下步骤获得:以myva文件建立蛋白质数据库,利用ncbi-BLAST-2.2.30+中的blastp程序进行本地BLAST,得出格式为xml的比对结果。编写Perl脚本程序,利用whog文件提取COG编号对应的功能分类编号,结合fun.txt文件来进行COG分类注释。
2.6. GO功能分类
GO功能分类采用Blast2GO软件[29] 进行,首先分别将这些蛋白进行Nr注释,然后进行GO映射,将结果上传至WEGO [30] 进行绘图分析。
3. 结果与分析
3.1. 黄单胞菌中含有GG(D/E)EF、EAL及HD-GYP结构域蛋白编码基因的数量
对选取的十五株黄单胞菌株按照其侵染对象归为几类:水稻黄单胞菌四株(XOO、XOO、XOC、PXO),野油菜黄单胞菌野油菜致病变种三株(XC、XCC、xccb100),地毯草黄单胞菌柑桔类致病变种(XAC、XAC29、XACM)及柑桔黄单胞柑桔亚种(XCAW)共四株,以及辣椒/番茄斑点病原菌野油菜黄单胞菌番茄致病变种(XCV)、甘蓝黑腐病原菌野油菜黄单胞菌甘蓝致病变种(XCR)、甘蔗病原菌白纹黄单胞菌(XALc)、菜豆细菌性疫病病原菌褐色黄单胞菌褐色亚种(XFF4834R)各一株。
利用全基因组序列进行生物信息学分析,发现203个含有GG(D/E)EF结构域的蛋白质中只有64个为GGDEF结构域,占约1/3,另外139个为GGEEF结构域。统计出的GG(D/E)EF、EAL及HD-GYP结构域蛋白的编码基因情况见表2。从该表2中可以看到:各菌株含有的编码EAL及HD-GYP结构域蛋白的基因数目非常接近,相对来说编码GG(D/E)EF结构域蛋白的基因数目波动较大,每株菌GG(D/E)EF结构域编码蛋白的数量在8~17之间、EAL结构域蛋白编码基因的数量在10~13之间、HD-GYP的数量在1~3之间。
各菌株基因组中含有GGDEF和EAL双结构域蛋白的数量不多,但却在c-di-GMP的浓度调控方面起着很重要的作用[7] 。经过多序列比对,发现除XALc_1672蛋白外,剩下的23个双结构域蛋白的序列相似性总体较高,并且这类蛋白的活性位点均为GGDEF而非GGEEF,见图2。
从图2可以看到:左上部分的13个蛋白相似性非常高,而下部分10个蛋白相似性也非常高,说明这类蛋白在黄单胞菌属中进化很保守。分析EAL结构域时发现这23个蛋白的EAL结构域位点却不像其GGDEF结构域一样保守,多序列比对可以将这十个蛋白再聚为两类:见图2右下部分。
需要指出的是,这种含有双结构域的蛋白通常是其中一个结构域相对保守并具有相应的酶活性,而另外一个结构域则逐渐退化进而丧失其酶活性。一般来说,绝大多数含有双结构域的蛋白发挥的是降解酶的作用。如新月柄杆菌(Caulobacter crescentus)中含有GGDEF-EAL双结构域的蛋白CC3396,其GGDEF结构域中的Gly退化成了Glu,因而并不具备c-di-GMP合成酶的活性,但它却能够与GTP高度结合从而激活EAL结构域,使其发挥降解酶的活性[17] 。当然,其中也有少数蛋白同时具有DGC和PDE两种酶的活性,如具有双结构域的副溶血性弧菌(Vibrio parahaemolyticus)的ScrC蛋白。ScrC主要发挥DGC合成酶活性,当ScrC与ScrA、ScrB同时存在时,又能降解细胞内c-di-GMP,从而精细地调节细胞内c-di-GMP的水平[31] 。还有研究表明双结构域中某个结构域已经退化,成为c-di-GMP的受体[32] [33] 。另外,十五株黄单胞菌中均不存在GG(D/E)EF与HD-GYP双结构域的蛋白和EAL与HD-GYP双结构域的蛋白。
Cui与He受PleD模型[34] 的启发,认为一个可能的模型是当信号感受结构域受到信号分子刺激或被磷酸化时,双结构域蛋白以二聚体形式激活GG(D/E)EF结构域的DGC活性;而当环境信号改变或去磷酸化时,双结构域蛋白则以单体形式激活EAL结构域的PDE活性[35] 。
3.2. 黄单胞菌中含有GG(D/E)EF、EAL及HD-GYP结构域蛋白的进化图
为了更好地了解相关蛋白的保守性,我们分别构建了不同菌株中含有GG(D/E)EF、EAL和HD-GYP结构域蛋白的进化树,如图3~5。
图3为含有GG(D/E)EF结构域蛋白的进化树。以得到100% Bootstrap支持的蛋白为例(以阴影标出),XAC1345、XAC29_06785、XACM_1328三个蛋白分别存在于三株地毯草黄单胞菌柑桔类致病变种中,XOO1879、PXO_01741、XOO_1775 (XOO后带有下划线的代表Xanthomonas oryzae pv. oryzae MAFF311018菌株中基因编码的蛋白质,以下同)、XOC_3158分别存在于四株水稻黄单胞菌致病变种中,

Figure 2. Multiple sequence alignment of dual-domain proteins containing GGDEF and EAL domains
图2. GGDEF和EAL双结构域蛋白多序列比对
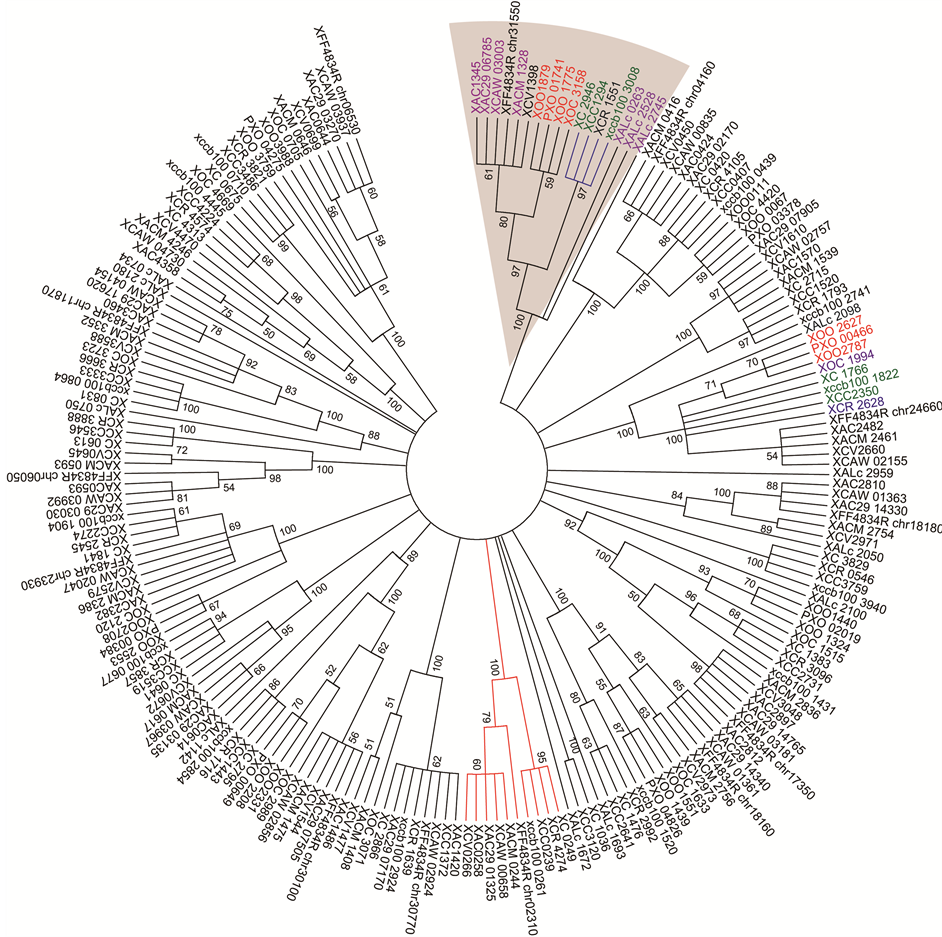
Figure 3. Phylogeny and evolution studies of proteins containing GG(D/E)EF domains in 15 Xanthomonas strains
图3. 十五株黄单胞菌中含有GG(D/E)EF结构域蛋白的进化树分析
XC_2946、XCC1294、xccb100_3008分别存在于三株野油菜黄单胞菌野油菜致病变种中,XALc_0263、XALc_2528、XALc_2745均存在于甘蔗病原菌白纹黄单胞菌中,说明一些含有GG(D/E)EF结构域蛋白在种内进化差异比种间进化差异小,进化比较保守。该结论进一步在蛋白XOO_2627、PXO_00466、XOO2787、XOC_1994(以红色标示)被分在同一分支中得到体现。以得到100% Bootstrap支持的蛋白XC_0249、XCR_4274、XCC0239、xccb100_0261、XFF4834R_chr02310、XACM_0244、XCAW_00658、XAC29_01325、XAC0258、XCV0266为例(见进化树的红色分支),这十个蛋白分别存在于三株野油菜黄单胞菌野油菜致病变种(XC、XCC、xccb100),三株地毯草黄单胞菌柑桔类致病变种(XAC、XAC29、XACM)、柑桔黄单胞柑桔亚种(XCAW)以及辣椒/番茄斑点病原菌野油菜黄单胞菌番茄致病变种(XCV)、甘蓝黑腐病原菌野油菜黄单胞菌甘蓝致病变种(XCR)、菜豆细菌性疫病病原菌褐色黄单胞菌褐色亚种(XFF4834R),而四株水稻黄
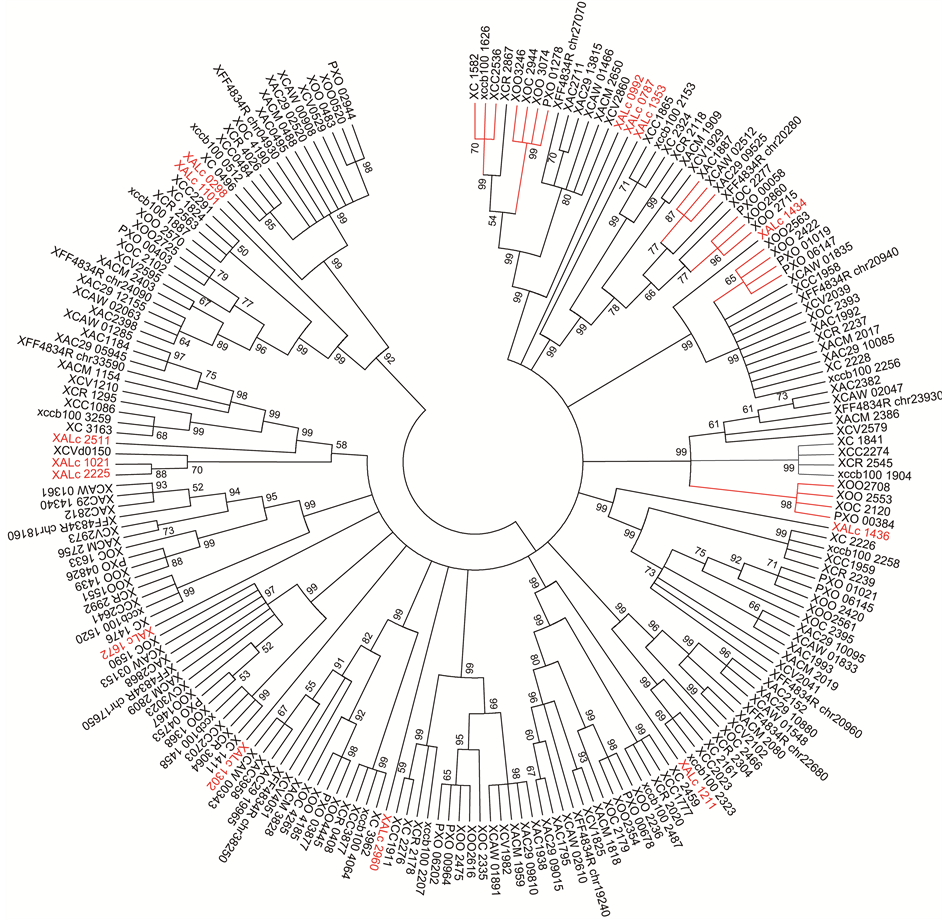
Figure 4. Phylogeny and evolution studies of proteins containing EAL domains in 15 Xanthomonas strains
图4. 十五株黄单胞菌中含有EAL结构域蛋白的进化树分析
单胞菌致病变种中均没有聚类到该蛋白,说明在进化过程中,水稻黄单胞菌种该蛋白发生了较大变异,而其他菌株中该蛋白较保守。另外,一株病原菌一般在一个分支上只有一个蛋白,但甘蔗病原菌白纹黄单胞菌中的XALc_0263、XALc_2528、XALc_2745是特例,可能是进化过程中基因组重排所致。
EAL结构域蛋白进化树也可以得到类似的规律(图4)。从进化树上看,侵染相同作物的菌株几乎都归为一类(以红色分支为例),且普遍得到较高的Bootstrap支持,说明EAL结构域蛋白比GG(D/E)EF蛋白更加保守。但令人意外的是,甘蔗病原菌白纹黄单胞菌中的十四个蛋白(进化树上以红色标示)大多无法和其他菌株聚为一类,这可能与该菌株偏爱含糖量较高的侵染对象甘蔗有关,这一特性可能导致其与其他种进化关系较远。
HD-GYP结构域蛋白数量相对较少(图5),水稻黄单胞菌XOC中含有四个此类蛋白,从进化树中可

Figure 5. Phylogeny and evolution studies of proteins containing HD- GYP domains in 15 Xanthomonas strains
图5. 十五株黄单胞菌中含有HD-GYP结构域蛋白的进化树分析
以看到,除XC2333蛋白外,这些蛋白明显聚为两类,说明含有该结构域的蛋白进化非常保守。
从这三个进化图中可以看到:按照侵染对象划分的菌株大部分都分别聚类在一起,这说明大部分含有这三个结构域的蛋白进化趋于保守。但也存在多个进化关系较远的蛋白,这可能与病原菌的习性多样性和侵染对象的不同相关。不同的GG(D/E)EF、EAL和HD-GYP蛋白可能位于细胞中不同的“小室”中[7] ,从而更精确控制c-di-GMP浓度,提高菌体对信号感应的精确性,这种情况是细菌不断适应环境变化的体现。
3.3. 四株水稻黄单胞菌的共线性分析
以四株水稻黄单胞菌为例,对其全基因组蛋白进行共线性分析,从图6可以看出:四株菌株的共线性程度都很高,而左上菌株Xanthomonas oryzae pv. oryzae MAFF311018和左下菌株Xanthomonas oryzae pv. oryzae KACC 10331共线性程度更好,该结果也解释了这两株菌株中GG(D/E)EF、EAL和HD-GYP结构域蛋白数量高度一致以及进化树中多被归为一类的原因。
3.4. 黄单胞菌中含有GG(D/E)EF、EAL及HD-GYP结构域蛋白的COG功能分析
COG功能注释分析主要有如下作用:1) 通过已知蛋白对未知序列进行功能注释;2) 通过查看指定的COG编号对应的蛋白是否存在及具体数目,从而能推导特定的代谢途径是否存在;3) 每个COG编号是一类蛋白,将查询序列和比对上的COG编号的蛋白进行多序列比对,能确定保守位点,分析其进化关系。
将这些含有GG(D/E)EF、EAL和HD-GYP结构域的蛋白进行COG功能分析,得到的结果见图7。
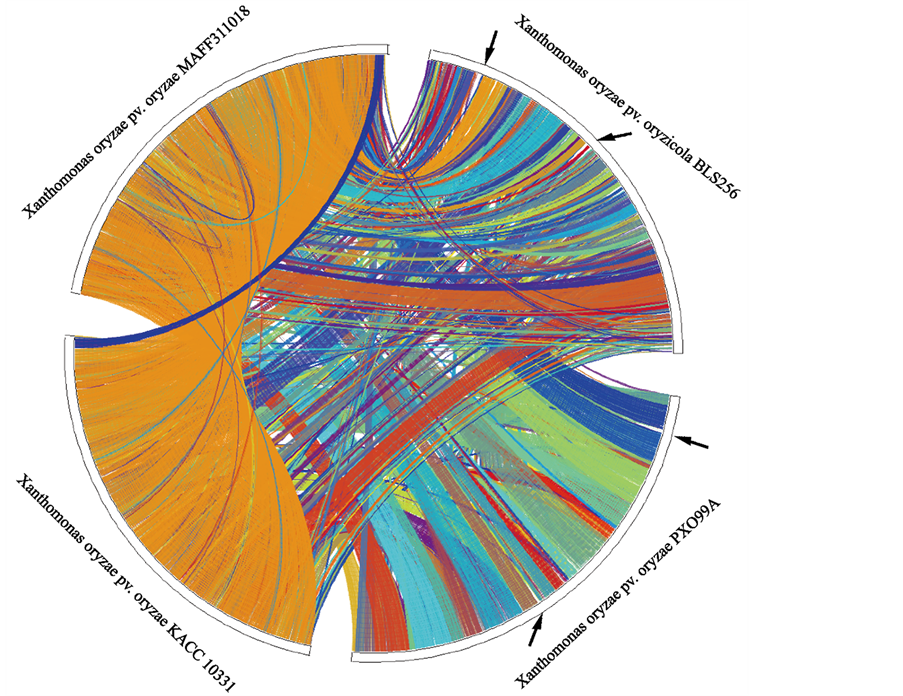
Figure 6. Collinearity analysis of four Xanthomonas oryzaes trains
图6. 四株水稻黄单胞菌的共线性分析
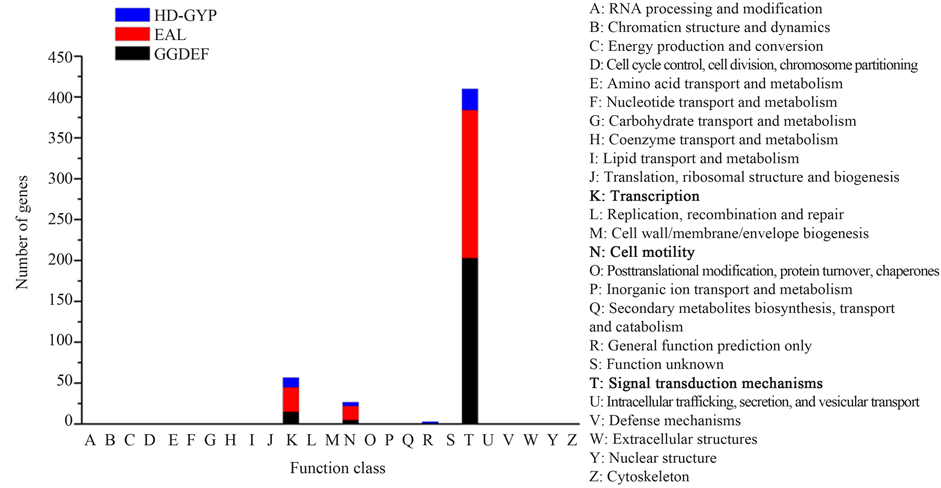
Figure 7. COG analysisof proteins containing GG(D/E)EF, EAL and HD-GYP domains in 15 Xanthomonas strains
图7. 十五株黄单胞菌中含有GG(D/E)EF、EAL和HD-GYP结构域蛋白COG功能分析
从图中可以看到:绝大多数蛋白都被注释为信号转导蛋白,从另一方面也说明该类蛋白功能保守。还有部分蛋白参与转录和细胞运动,这与已经报道的c-di-GMP参与转录调节和细菌运动性相一致[23] [36] 。
3.5. 黄单胞菌中含有GG(D/E)EF、EAL及HD-GYP结构域蛋白的GO注释分析
对含有GG(D/E)EF、EAL及HD-GYP结构域的蛋白进行GO注释分析,结果见图8。被注释至Cellular component上的蛋白均含有跨膜结构域以及HMAP linker结构域,Marcos等通过研究GGDEF和EAL双结构域的蛋白LapD的晶体结构,发现HAMP通过形成S螺旋,抑制c-di-GMP与EAL结构域结合[37] 。被注释至binding的蛋白绝大多数都含有GAF结构域,GAF结构域是一个真核和原核生物中都普遍存在的典型信号感受结构域,能够结合cAMP和cGMP [38] ,GAF能够感受氧化压力和营养胁迫的信号分子MetO [39] 和NO分子[40] 。值得注意的是,NO在真核宿主体内介导免疫系统的抗菌活性[41] 。可以看出,GAF结构域多样性信号配体既包括了真核细胞中的信号分子也包括了细菌胞内的压力胁迫信号分子。因此,病原菌中包含GAF结构域的蛋白质很有可能在胁迫应答以及与宿主相互作用中发挥潜在作用。常和GG(D/E)EF、EAL和HD-GYP一起出现的信号感受结构域还有PAS、REC等[7] 。被注释至Biological process上的蛋白表明这些蛋白主要起着信号传导的作用,如参与双组分信号系统等,该结果与COG功能分析的结果一致。
4. 讨论
c-di-GMP是目前研究最为深入的核苷类第二信使,在细菌代谢调节中居于中心调节子的地位[42] 。
目前已经发现,c-di-GMP可以调节细菌多种多样的生理活动。包括酶活性的变构调节;生物膜相关

Figure 8. GO analysis of proteins containing GG(D/E)EF, EAL and HD-GYP domains in 15 Xanthomonas strains
图8. 十五株黄单胞菌中含有GG(D/E)EF、EAL和HD-GYP结构域蛋白GO功能分析
的胞外多糖、粘附因子等的合成与降解;通过调节鞭毛的合成与组装来调控细菌的运动状态;影响长期存活及压力响应;调节病原菌的毒力因子的表达、宿主菌的免疫系统;影响细胞周期、分化与形态;调节周质空间的蛋白酶活性、RNA加工过程等[7] [42] ,这些调节是与其受体直接相关的。已有的研究表明,c-di-GMP具有多种类型的受体(见图9),主要包括含PilZ结构域的蛋白(如YcgR) [43] [44] 、转录因子(如WspR) [45] 、TCS中的组氨酸激酶(histidine kinase) (如SgmT) [46] 、多核苷酸磷酸化酶(PNPase) [47] 、核酸开关(riboswitch) [48] 、具有I位点的GG(D/E)EF结构域蛋白(如PleD) [34] 以及退化的GG(D/E)EF和EAL结构域蛋白(如LapD)等[49] 。这些受体中既有蛋白质也有RNA,既有各种催化活性的酶也有调控类蛋白。黄单胞菌属中目前发现的c-di-GMP的受体主要有:转录因子Clp [50] 、退化的GGDEF-EAL双结构域蛋白Filp [17] 、PilZ结构域蛋白XC1028 [51] 以及新发现的YajQ蛋白[23] 。
黄单胞菌属作为一类重要的作物病原菌,给人们造成很大的经济损失,第二信使分子c-di-GMP与该属的侵染系统——III型分泌系统(type III protein secretion system, TTSS)以及群体感应(quorum sensing, QS)系统密切相关[52] 。TTSS和QS系统又与c-di-GMP介导的信号转导系统在诸多功能上存在着交叉,如菌体的运动性、生物膜的形成以及毒力因子的产生等。因此,研究c-di-GMP的代谢网络和调控网络意义重大。
本文对十五株全基因组测序完成的黄单胞菌属菌株中含有这三个结构域的蛋白进行了比对搜索,发现许多Pfam上注释出来的含有GG(D/E)EF、EAL或HD-GYP结构域的蛋白并不包含保守结构域,耻垢分枝杆菌(Mycobacterium smegmatis MC2 155)中得MSMEG_2774即属于这类,该蛋白并不含有保守的GG(D/E)EF结构域,我们通过体外酶活性验证,发现该蛋白没有合成c-di-GMP的活性,也不能和c-di-GMP结合。我们利用HMMER对Pfam数据库中注释出的含有GG(D/E)EF、EAL或HD-GYP的蛋白重新进行了比对搜索,然后提取含有GG(D/E)EF、EAL、HD-GYP残基的蛋白进行了包括进化树、COG分析及GO分析在内的生物信息学分析。
总体来说,选取的十五株黄单胞菌中GG(D/E)EF、EAL或HD-GYP结构域的蛋白数目波动不大,四株水稻黄单胞菌中的GG(D/E)EF结构域蛋白略少于其他菌株。XOC菌株与其他三株水稻黄单胞菌差异较大,通过基因组蛋白共线性分析,发现该菌株基因组蛋白共线性程度略低于其他三株,该现象也侧面说明GG(D/E)EF、EAL或HD-GYP结构域的蛋白进化过程整体趋于保守。203个GG(D/E)EF结构域蛋白中,只有64个含有GGDEF结构域,另外约2/3的蛋白含有GGEEF结构域。通过进化树可以看出GG(D/E)EF结构域蛋白、EAL结构域蛋白和HD-GYP结构域蛋白进化都比较保守。但是从进化树上可以看到这些蛋白中也有部分变异较大,可能是病原菌针对不同的侵染对象长期选择进化的结果:在同种菌体中定位不同的DGC、PDE或在不同种菌体中定位相同的DGC、PDE长期感受不同的外界信号刺激,
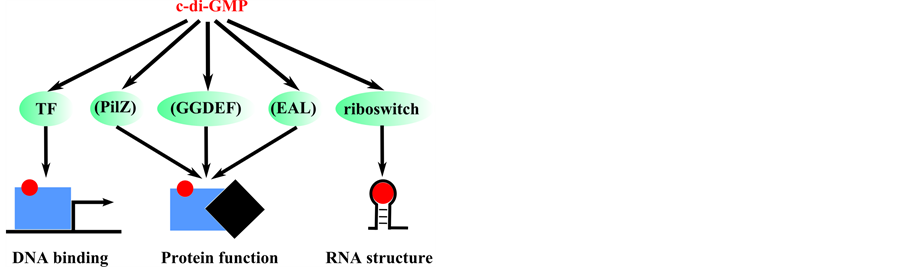
Figure 9. The c-di-GMP effectors
图9. 目前已经发现的c-di-GMP的受体
发生自然选择所致。另外,结核分枝杆菌(M. tuberculosis)、牛分枝杆菌(M. bovis)、炭疽杆菌(Bacillus anthraci)等人与动物病原菌只含有少数几个GG(D/E)EF、EAL和HD-GYP结构域蛋白,远少于黄单胞菌属等与植物相互作用的细菌,这可能与植物性病原菌生存条件更复杂、响应的信号更加多元相关。
随着研究的深入,人们发现GG(D/E)EF、EAL和HD-GYP结构域蛋白可能不仅仅只行使合成酶或降解酶作用。野油菜黄单胞菌中含有HD-GYP结构域的蛋白RpfG和组氨酸激酶RpfC组成一对双组分系统,使毒力增强,胞外酶和生物膜的产生增加[15] [53] [54] 。Xac中含HD-GYP结构域的RpfG蛋白能够与DGC发生直接相互作用,从而调节DGC的功能[55] 。Ryan等研究发现RpfG能够和含有GG(D/E)EF结构域的蛋白结合,通过PilZ蛋白XC2249来控制菌体运动[56] 。所以,含有这三个结构域蛋白目前研究得并不充分,除了行使其本身的酶的功能,可能也同时参与多种多样的生理调节,而且这些合成酶、降解酶与其他信号调节的相互作用及其与致病性等的关系均有待于进一步研究与挖掘。
总之,c-di-GMP作为细菌中广泛存在的第二信使分子,在病原菌行使致病的调控网络中占有非常重要的地位。通过深入研究,可为构建c-di-GMP介导的调控网络和寻找新的防治黄单胞菌的药物靶标奠定良好的基础。
致谢
本文受到国家自然科学基金(31070065)与973项目(2010CB126105)的资助。
文章引用
邹 霞,黄良博,何 进, (2014) 黄单胞菌中c-di-GMP二鸟苷酸环化酶和磷酸二酯酶的生物信息学分析
Bioinformatics Analysis of Diguanylate Cyclases and c-di-GMP-Specific Phosphodiesterases from Xanthomonas Species. 计算生物学,04,68-82. doi: 10.12677/HJCB.2014.44008
参考文献 (References)
- 1. Ryan, R.P., Vorhölter, F.J., Potnis, N., et al. (2011) Pathogenomics of Xanthomonas: Understanding bacterium-plant interactions. Nature Reviews Microbiology, 9, 344-355.
- 2. Qian, W., Jia, Y., Ren, S.X., et al. (2005) Comparative and functional genomic analyses of the pathogenicity of phytopathogen Xanthomonas campestris pv. campestris. Genome Research, 15, 757-767.
- 3. Lee, B.M., Park, Y.J., Park, D.S., et al. (2005) The genome sequence of Xanthomonas oryzae pathovar oryzae KACC10331, the bacterial blight pathogen of rice. Nucleic Acids Research, 33, 577-586.
- 4. Bogdanove, A.J., Koebnik, R., Lu, H., et al. (2011) Two new complete genome sequences offer insight into host and tissue specificity of plant pathogenic Xanthomonas spp. Journal of Bacteriology, 193, 5450-5464.
- 5. Weinhouse, H., Sapir, S., Amikam, D., Shilo, Y., Volman, G., Ohana, P. and Benziman, M. (1997) c-di-GMP-binding protein, a new factor regulating cellulose synthesis in Acetobacter xylinum. FEBS Letters, 416, 207-211.
- 6. Tal, R., Wong, H.C., Calhoon, R., Gelfand, D., Fear, A.L., Volman, G., et al. (1998) Three cdg operons control cellular turnover of cyclic di-GMP in Acetobacter xylinum: Genetic organization and occurrence of conserved domains in isoenzymes. Journal of Bacteriology, 180, 4416-4425.
- 7. Römling, U., Galperin, M.Y. and Gomelsky, M. (2013) Cyclic di-GMP: The first 25 years of a universal bacterial second messenger. Microbiology and Molecular Biology Reviews, 77, 1-52.
- 8. Thieme, F., Koebnik, R., Bekel, T., et al. (2005) Insights into genome plasticity and pathogenicity of the plant pathogenic bacterium Xanthomonas campestris pv. vesicatoria revealed by the complete ge-nome sequence. Journal of Bacteriology, 187, 7254-7266.
- 9. Bogdanove, A.J., Koebnik, R., Lu, H., et al. (2011) Two new complete genome sequences offer insight into host and tissue specificity of plant pathogenic Xanthomonas spp. Journal of Bacteriology, 193, 5450-5464.
- 10. da Silva, A.C.R., Ferro, J.A., Reinach, F.C., et al. (2002) Comparison of the genomes of two Xanthomonas pathogens with differing host specificities. Nature, 417, 459-463.
- 11. Jalan, N., Aritua, V., Kumar, D., et al. (2011) Comparative genomic analysis of Xanthomonas axonopodis pv. citrumelo F1, which causes citrus bacterial spot disease, and related strains provides insights into virulence and host specificity. Journal of Bacteriology, 193, 6342-6357.
- 12. Jalan, N., Kumar, D., Yu, F., et al. (2013) Complete genome sequence of Xanthomonas citri subsp. citri strain AW12879, a restricted-host-range citrus canker-causing bacterium. Genome Announcements, 1, e00235-13.
- 13. Pieretti, I., Royer, M., Barbe, V., et al. (2009) The complete genome sequence of Xanthomonas albilineans provides new insights into the reductive genome evolution of the xylem-limited Xanthomo-nadaceae. BMC Genomics, 10, 616.
- 14. Darrasse, A., Carrère, S., Barbe, V., Boureau, T., Arrieta-Ortiz, M.L., Bon-neau, S., et al. (2013) Genome sequence of Xanthomonas fuscans subsp. fuscans strain 4834-R reveals that flagellar motility is not a general feature of xanthomonads. BMC Genomics, 14, 761.
- 15. Ryan, R.P., Fouhy, Y., Lucey, J.F., Crossman, L.C., Spiro, S., He, Y.W., et al. (2006) Cell-cell signaling in Xanthomonas campestris involves an HD-GYP domain protein that functions in cyclic di-GMP turnover. Proceedings of the National Academy of Sciences of the United States of America, 103, 6712-6717.
- 16. Galperin, M.Y. (2004) Bacterial signal transduction network in a genomic perspective. Environmental Microbiology, 6, 552-567.
- 17. Christen, M., Christen, B., Folcher, M., Schauerte, A. and Jenal, U. (2005) Identification and characterization of a cyclic di-GMP-specific phosphodiesterase and its allosteric control by GTP. Journal of Biological Chemistry, 280, 30829- 30837.
- 18. Rao, F., See, R.Y., Zhang, D., Toh, D.C., Ji, Q. and Liang, Z.X. (2010) YybT is a signaling protein that contains a cyclic dinucleotide phosphodiesterase domain and a GGDEF domain with ATPase activity. Journal of Biological Chemistry, 285, 473-482.
- 19. Yang, F., Tian, F., Li, X., Fan, S., Chen, H.M., Wu, M.S., et al. (2014) The degenerate EAL-GGDEF domain protein Filp functions as a cyclic di-GMP receptor and specifically interacts with the PilZ-domain protein PXO_02715 to regulate virulence in Xanthomonas oryzae pv. oryzae. Molecular Plant-Microbe Interactions, 27, 578-589.
- 20. Yang, F., Tian, F., Sun, L., Chen, H.M., Wu, M.S., Yang, C.H. and He, C.Y. (2012) A novel two-component system PdeK/PdeR regulates c-di-GMP turnover and virulence of Xanthomonas oryzae pv. oryzae. Molecular Plant-Microbe Interactions, 25, 1361-1369.
- 21. Ryan, R.P. (2013) Cyclic di-GMP signalling and the regulation of bacterial virulence. Microbiology, 159, 1286-1297.
- 22. Ryan, R.P. and Dow, J.M. (2010) Intermolecular interactions between HD-GYP and GGDEF domain proteins mediate virulence-related signal transduction in Xanthomonas campestris. Virulence, 1, 404-408.
- 23. An, S., Caly, D.L., McCarthy, Y., Murdoch, S.L., Ward, J., Febrer, M., et al. (2014) Novel cyclic di-GMP effectors of the YajQ protein family control bacterial virulence. PLoS Pathogens, 10, e1004429.
- 24. Tatusov, R.L., Koonin, E.V. and Lipman, D.J. (1997) A genomic perspective on protein families. Science, 278, 631- 637.
- 25. Ashburner, M., Ball, C.A., Blake, J.A., Botstein, D., Butler, H., Cherry, J.M., et al. (2000) Gene Ontology: Tool for the unification of biology. Nature Genetics, 25, 25-29.
- 26. Thompson, J.D., Gibson, T. and Higgins, D.G. (2002) Multiple sequence alignment using ClustalW and ClustalX. Current Protocols in Bioinformatics, Unit 2.3, 1-22.
- 27. Tamura, K., Stecher, G., Peterson, D., Filipski, A. and Kumar, S. (2013) MEGA6: Molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 30, 2725-2729.
- 28. Wang, Y., Tang, H., DeBarry, J.D., Tan, X., Li, J.P., Wang, X.Y., et al. (2012) MCScanX: A toolkit for detection and evolutionary analysis of gene synteny and collinearity. Nucleic Acids Research, 40, e49.
- 29. Conesa, A., Götz, S., García-Gómez, J.M., Terol, J., Talón, M. and Robles, M. (2005) Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics, 21, 3674-3676.
- 30. Ye, J., Fang, L., Zheng, H., Zhang, Y., Chen, J., Zhang, Z.J., et al. (2006) WEGO: A web tool for plotting GO annotations. Nucleic Acids Research, 34, W293-W297.
- 31. Ferreira, R.B., Antunes, L.C., Greenberg, E.P. and McCarter, L.L. (2008) Vibrio parahaemolyticus ScrC modulates cyclic dimeric GMP regulation of gene expression relevant to growth on surfaces. Journal of Bacteriology, 190, 851- 860.
- 32. Navarro, M.V.A.S., De, N., Bae, N., Wang, Q. and Sondermann, H. (2009) Structural analysis of the GGDEF-EAL domain-containing c-di-GMP receptor FimX. Structure, 17, 1104-1116.
- 33. Jenal, U. and Malone, J. (2006) Mechanisms of cyclic-di-GMP signaling in bacteria. Annual Review of Genetics, 40, 385-407.
- 34. Chan, C., Paul, R., Samoray, D., Amiot, N.C., Giese, B., Jenal, U. and Schirmer, T. (2004) Structural basis of activity and allosteric control of diguanylate cyclase. Proceedings of the National Academy of Sciences of the United States of America, 101, 17084-17089.
- 35. Cui, T. and He, Z.G. (2012) C-di-GMP signaling and implications for pathogenesis of My-cobacterium tuberculosis. Chinese Science Bulletin, 57, 4387-4393.
- 36. Povolotsky, T.L. and Hengge, R. (2012) “Life-style” control networks in Escherichia coli: Signaling by the second messenger c-di-GMP. Journal of Biotech-nology, 160, 10-16.
- 37. Navarro, M.V.A.S., Newell, P.D., Krasteva, P.V., Chatterjee, D., Madden, D.R., O’Toole, G.A., et al. (2011) Structural basis for c-di-GMP-mediated inside-out signaling controlling periplasmic proteolysis. PLoS Biology, 9, e1000588.
- 38. Hurley, J.H. (2003) GAF domains: Cyclic nucleotides come full circle. Science Signaling, 2003, pe1.
- 39. Lin, Z., Johnson, L.C., Weissbach, H., Brot, N., Lively, M.O. and Lowther, W.T. (2007) Free methionine-(R)-sulfoxide reductase from Escherichia coli reveals a new GAF domain function. Proceedings of the National Academy of Sciences of the United States of America, 104, 9597-9602.
- 40. Sardiwal, S., Kendall, S.L., Movahedzadeh, F., Rison, S.C.G., Stoker, N.G. and Djordjevic, S. (2005) A GAF domain in the Hypoxia/NO-inducible Mycobacterium tuberculosis DosS protein binds haem. Journal of Molecular Biology, 353, 929-936.
- 41. Boscá, L., Zeini, M., Través, P.G. and Hortelano, S. (2005) Nitric oxide and cell viability in inflammatory cells: A role for NO in macrophage function and fate. Toxicology, 208, 249-258.
- 42. Kalia, D., Merey, G., Nakayama, S., Zheng, Y., Zhou, J., Luo, Y.L., et al. (2013) Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews, 42, 305- 341.
- 43. Amikam, D. and Galperin, M.Y. (2006) PilZ domain is part of the bacterial c-di-GMP binding protein. Bioinformatics, 22, 3-6.
- 44. Ryjenkov, D.A., Simm, R., Römling, U. and Gomelsky, M. (2006) The PilZ domain is a receptor for the second messenger c-di-GMP: The PilZ domain protein YcgR controls motility in enterobacteria. Journal of Biological Chemistry, 281, 30310-30314.
- 45. De, N., Pirruccello, M., Krasteva, P.V., Bae, N., Raghavan, R.V. and Sondermann, H. (2008) Phosphorylation-inde- pendent regulation of the diguanylate cyclase WspR. PLoS Biology, 6, e67.
- 46. Petters, T., Zhang, X., Nesper, J., Treuner-Lange, A., Gomez-Santos, N., Hoppert, M., et al. (2012) The orphan histidine protein kinase SgmT is a c-di-GMP receptor and regulates composition of the extracellular matrix together with the orphan DNA binding response regulator DigR in Myxococcus xanthus. Molecular Microbiology, 84, 147-165.
- 47. Tuckerman, J.R., Gonzalez, G. and Gilles-Gonzalez, M.A. (2011) Cyclic di-GMP activation of polynucleotide phosphorylase signal-dependent RNA processing. Journal of Molecular Biology, 407, 633-639.
- 48. Sudarsan, N., Lee, E.R. and Weinberg, Z. (2008) Riboswitches in eubacteria sense the second messenger cyclic di-GMP. Science, 321, 411-413.
- 49. Breaker, R.R. (2011) Prospects for riboswitch discovery and analysis. Molecular Cell, 43, 867-879.
- 50. Chin, K.H., Lee, Y.C., Tu, Z.L., Chen, C.H., Tseng, Y.H., Yang, J.M., et al. (2010) The cAMP receptor-like protein CLP is a novel c-di-GMP receptor linking cell-cell signaling to virulence gene expression in Xanthomonas campestris. Journal of Molecular Biology, 396, 646-662.
- 51. Li, T.N., Chin, K.H., Liu, J.H., Wang, A.H.J. and Chou, S.H. (2009) XC1028 from Xanthomonas campestris adopts a PilZ domain-like structure without ac-di-GMP switch. Proteins: Structure, Function, and Bioinformatics, 75, 282-288.
- 52. He, Y.W., Ng, A.Y., Xu, M., Lin, K., Wang, L.H., Dong, Y.H. and Zhang, L.H. (2007) Xanthomonas campestris cell-cell communication involves a putative nucleotide receptor protein Clp and a hierarchical signalling network. Molecular Microbiology, 64, 281-292.
- 53. Dow, J.M., Fouhy, Y., Lucey, J.F. and Ryan, R.P. (2006) The HD-GYP domain, cyclic di-GMP signaling, and bacterial virulence to plants. Molecular Plant-Microbe Interactions, 19, 1378-1384.
- 54. Slater, H., Alvarez-Morales, A., Barber, C.E., Daniels, M.J. and Dow, J.M. (2000) A two-component system involving an HD-GYP domain protein links cell-cell signalling to pathogenicity gene expression in Xanthomonas campestris. Molecular Microbiology, 38, 986-1003.
- 55. Andrade, M.O., Alegria, M.C., Guzzo, C.R., Docena, C., Rosa, M.C.P., Ramos, C.H.I. and Farah, C.S. (2006) The HD-GYP domain of RpfG mediates a direct linkage between the RpfG quorum-sensing pathway and a subset of diguanylate cyclase proteins in the phytopathogen Xanthomonas axonopodis pv. citri. Molecular Microbiology, 62, 537-551.
- 56. Ryan, R.P., McCarthy, Y., Kiely, P.A., O’Connor, R., Farah, C.S., Armitage, J.P. and Dow, J.M. (2012) Dynamic complex formation between HD-GYP, GGDEF and PilZ domain proteins regulates motility in Xanthomonas campestris. Molecular Microbiology, 86, 557-567.
