Hans Journal of Biomedicine
Vol.
12
No.
01
(
2022
), Article ID:
47601
,
8
pages
10.12677/HJBM.2022.121001
多片段拼接法克隆人表皮生长因子受体2基因
陈婉煜,刘君怡,李国庆,刘媛媛,陈华波*
湖北文理学院,基础医学院,湖北 襄阳
收稿日期:2021年10月16日;录用日期:2021年12月23日;发布日期:2021年12月30日

摘要
鉴于从cDNA库中克隆长基因较困难的现实,采取分步扩增与DNA组装相结合的方式,提出一种长基因克隆解决方案。以克隆人表皮生长因子受体2基因为例,先分别扩增出该基因的三个片段F1、F2和F3。通过重叠延伸PCR融合前两个片段得到F12,再利用双片段连接法将F12与F3一起连接插入载体以构建完整基因。从双片段连接物中筛选出上百个菌落,经检测和序列测定,得到一个序列完全准确的克隆。对于难以直接克隆的长基因,可先扩增其各个片段,再将它们拼接成完整基因。其中双片段连接法可突破严格的酶切位点要求对单片段顺序连接的限制,再辅以重叠延伸PCR融合法,即可实现多个基因片段的正确拼接。
关键词
基因克隆,重叠延伸PCR,双片段连接法,人表皮生长因子受体2基因

Cloning of Human Epidermal Growth Factor Receptor 2 Gene by Multi Fragment Assembly
Wanyu Chen, Junyi Liu, Guoqing Li, Yuanyuan Liu, Huabo Chen*
Basic Medical College, Hubei University of Arts and Science, Xiangyang Hubei
Received: Oct. 16th, 2021; accepted: Dec. 23rd, 2021; published: Dec. 30th, 2021

ABSTRACT
It was difficult to clone long genes from cDNA library due to a series of factors, such as mRNA instability and the low efficiency of reverse transcription. Combining partial amplification with DNA assembly, an optimal solution was proposed for cloning lone gene that was difficult to amplify from cDNA directly. Taking the human epidermal growth factor receptor 2 (HER2) gene as an example, three fragments of HER2 were amplified firstly. F1 and F2 were fused into F12 by overlap extension PCR, and then enzyme cleaved F12 and F3 were linked together into vector, so as to recombine the whole HER2 gene. Hundreds of colonies were screened out from double fragments ligation and one positive clone matching completely to HER2 sequence was detected. For long gene that was difficult to clone directly, it was feasible to amplify its segments and then assemble them into a complete one. Double fragments ligation breaking the rigorous demand of restriction sites of step by step ligation, in the assistance of overlap extension PCR, could join DNA fragments into a complete gene.
Keywords:Gene Cloning, Overlap Extension PCR, Double Fragments Ligation, HER2

Copyright © 2022 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
从cDNA库中克隆真核基因,再将其导入细胞内表达,是研究其功能与调节机制的常用做法 [1]。然而科研工作者都有类似经验,即长度较小的基因相对容易克隆,而长度较大的基因难以直接克隆。除了受基因表达丰度影响,还可能存在以下几种原因:一是RNA酶广泛存在于实验环境中,且极难灭活 [2]。尽管会以焦碳酸二乙酯(Diethyl pyrocarbonate, DEPC)处理实验器材,但仍难以完全避免mRNA在提取、纯化及保存过程中的降解,其中尤以长链mRNA稳定性较差。其二是受逆转录酶连续合成能力制约,长链mRNA的逆转录产物含量更低;去除RNase H活性可适当增加逆转录酶稳定性,但仍无法保证获得长基因的完整cDNA [3] [4]。此外,mRNA的机械性损伤、引物长度、反应条件和RNA酶抑制剂等因素都对逆转录结果有所影响,导致长链cDNA产量偏低 [5]。尽管如此,从cDNA库中扩增基因的部分片段相对容易,特别是以随机引物逆转录而来的cDNA库,往往能扩增出目标基因的多个片段。若能得到涵盖基因全部编码序列的多个DNA片段,再将其拼接起来即可得到完整基因。
限制性酶切–连接与重叠延伸PCR (overlapping extension PCR, OE-PCR)是常用的DNA拼接方法。这两种方法皆技术成熟,操作相对简单,但各有利弊。单片段顺序连接受制于严格的酶切位点要求,但双片段连接法可拓展其适用范围 [6] [7]。OE-PCR融合成功率一般随着上、下游DNA片段长度增加而降低,但可以作为酶切–连接法的补充。结合具体情况灵活选择两种方法及其组合,就能将DNA片段拼接成完整基因。
人表皮生长因子受体2基因(human epidermal growth factor receptor 2 gene, HER2)又称c-ERBB2或neu,是重要的癌基因,其表达水平可作为乳腺癌治疗的预后判断指标 [8] [9]。HER2基因编码序列全长3768 bp (GenBank: NM_004448),编码1255个氨基酸的跨膜受体蛋白 [10]。本研究以克隆HER2基因为例,对常规DNA拼接方法进行优化,以期提供一种长基因克隆解决方案。
2. 材料与方法
2.1. 材料
DH5α感受态大肠杆菌(CB101)购自天根生化科技(北京)有限公司;pcDNA3.1质粒(V800-20)购自美国Thermo Fisher公司。
EasyPure RNA Kit、EasyScript First-Strand cDNA Synthesis SuperMix和2×EasyTaq PCR SuperMix (北京全式金生物技术有限公司);TaKaRa LA PCR Kit Ver.2.1、DNA Ligation Kit Ver.2.1 (Takara公司,日本);限制性内切酶EcoRI、HindIII、NdeI及XhoI (NEB公司,英国);DNA回收试剂盒和质粒小量抽提试剂盒(杭州博日科技有限公司)。
2.2. 引物设计与合成
引物采用Primer Premier 5.0软件设计,由北京奥科鼎盛生物科技有限公司合成,经PAGE纯化。引物序列见表1。
Table 1. Primers used for PCR
表1. PCR引物序列
2.3. 细胞培养及cDNA制备
MCF-7细胞生长在含10%胎牛血清,100 U/mL青霉素,100 µg/mL链霉素的DMEM培养基中,培养条件为5% CO2,37℃。将对数生长期细胞以2 × 104细胞/cm2密度接种到
2.4. 基因克隆
第一轮PCR:25 μL 2×GC buffer,7 μL dNTP,0.5 μL primer 1/0.5 μL primer 2,0.5 μL TaKaRa
2.5. 阳性克隆的筛选与序列测定
从过夜平板上随机挑取数个菌落,置于4 mL氨苄青霉素-LB培养基中继续培养6 h。采用2×EasyTaq PCR SuperMix,直接以菌液为模板进行PCR检测 [13]。对阳性克隆取3 ml菌液小量提取质粒进行序列测定,DNA测序由北京奥科鼎盛生物科技有限公司完成。采用DNAMAN 4.0软件进行限制性酶切分析与序列比对。
3. 结果与分析
3.1. 从cDNA库中扩增HER2基因的三个片段
欲克隆HER2基因,考虑到该基因较长,可能不易从细胞总cDNA中扩增出基因全长。对HER2基因作限制性酶切分析,发现其中含一个KpnI位点(位于基因下游),两个BglII位点(皆位于基因上游)及五个BamHI位点。这些酶切位点的分布特征使其不宜用于酶切–连接法拼接基因片段。基因中另有两个EcoRI位点和一个NdeI位点,尤其是1301位EcoRI位点和2340位NdeI位点将HER2基因分成三个大小适中的片段F1 (1-1301)、F2 (1302-2340)与F3 (2341-end) (图1)。根据酶切分析结果设计相应引物先扩增HER2基因及其片段。

Figure 1. Restriction map of HER2 gene and primer matching sites
图1. HER2基因酶切图谱及引物匹配位点
利用寡聚脱氧胸苷(Oligo dT)或随机引物获取MCF-7细胞总cDNA为PCR模板,分别以S0/A1, S1/A2, S2/A0为引物扩增HER2基因F1, F2, F3片段。经多次调整PCR反应条件,Oligo dT组只扩增出F3片段;随机引物组顺利扩增出全部三个基因片段;而以S0/A2, S1/A0, S0/A0为引物始终未能扩增出相应的F12, F23片段及全长基因(图2)。以上结果与长基因难以直接克隆的经验预期相符。
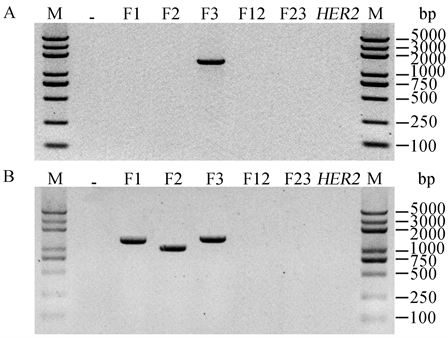
Figure 2. Amplification of HER2 gene fragments. The cDNA was obtained by reverse transcription with oligo Dt (A) or random primers (B), and then used as PCR template
图2. HER2基因片段的扩增。以Oligo dT (A)或随机引物(B)逆转录获得的cDNA为模板,通过PCR扩增HER2基因片段
3.2. 完整HER2基因的克隆
为将上述HER2基因三片段拼接成完整基因,先尝试以OE-PCR法。将F1与F2组合重叠延伸扩增出F12片段,F2与F3组合扩增出F23片段,但F1、F2与F3三者组合未能扩增出HER2全长基因(图3)。以回收的F12片段组合F3或以F23片段组合F1亦未能扩增出HER2全长基因(结果未展示),推测可能是长链DNA片段干扰了重叠延伸效率所致。
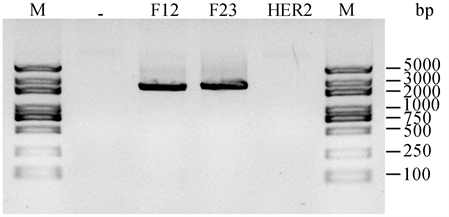
Figure 3. Fusion of partial HER2 gene fragments by OE-PCR
图3. OE-PCR法融合HER2基因部分片段
由于F3片段内含有EcoRI位点,故F23片段无法与F1通过EcoRI位点实现酶切–连接;而F12片段与F3则可通过NdeⅠ酶切后实现连接(图1)。pcDNA3.1质粒载体多克隆位点(Multiple Cloning Site, MCS)内无NdeI位点,故无法采用分步连接法,两片段同时与载体连接成为可选方案。理论上,F1、F2、F3三片段也可直接通过酶切–连接法一次性拼接成完整基因。将F1、F2、F3、F12及pcDNA3.1质粒载体作双酶切(图4),续以双片段连接(F12: HindIII/NdeI + F3: NdeI/ XhoI + Vector: HindIII/XhoI)或三片段连接(F1: HindIII/EcoRI + F2: EcoRI/NdeI + F3: NdeI/XhoI + Vector: HindIII/XhoI),对比检测两种方案的可行性。

Figure 4. Restriction enzyme digestion of HER2 gene fragments and vector. F1H/E indicated that the F1 fragment was digested by HindIII/EcoRI, and so on. VH/X indicated that pcDNA3.1 vector was digested by HindIII/XhoI
图4. HER2基因片段及载体的酶切。F1H/E表示F1片段以HindIII/EcoRI双酶切,余者类似;VH/X表示pcDNA3.1载体以HindIII/XhoI双酶切
3.3. 目标克隆的筛选与鉴定
连接产物转化感受态大肠杆菌DH5α后经氨苄青霉素-LB平板筛选,双片段连接组得到上百个菌落,而三片段连接组仅有十余个菌落(图5(A)、图5(B)),提示三片段连接效率较低。分别从两个平板上挑取数个菌落进行PCR检测。方案一选用匹配pcDNA3.1载体MCS上游的T7与A1引物组合,阳性结果表示该克隆含pcDNA3.1质粒载体,且含有HER2基因前半部分。A平板检测得到#1、#3、#4、#5四个阳性克隆。方案二选用S2与匹配pcDNA3.1载体MCS下游的BGH引物组合,阳性结果表示该克隆含pcDNA3.1质粒载体,且含有HER2基因后半部分。检测结果与方案一一致(图5(C))。上述两种方案均未从B平板上检测到阳性菌落(结果未展示)。选用T7与BGH引物组合,从B平板中检测出两个疑是菌落,但产物大小仅约250 bp (图5(D)),推测这两个克隆仅含pcDNA3.1空载体。
为进一步确认检测结果的准确性,对选自A平板的四个阳性克隆作序列测定。结果显示其中#1、#4、#5克隆各有数个点突变,#3克隆序列完全准确(图5(E))。说明成功克隆了HER2基因,并已将其插入pcDNA3.1载体。
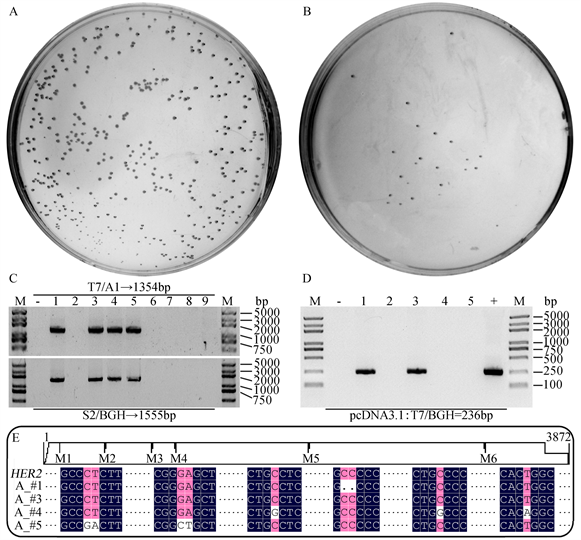
Figure 5. Detection of positive colonies. (A/B) The colonies on the plate of double fragments/tri-fragments ligation. (C) The PCR detection results of random colonies on A plate. The primers and expected length product were shown in the figure and ddH2O acted as negative control. (D) The PCR detection results of random colonies on B plate. pcDNA3.1 plasmid acted as positive control. (E) Multiple alignments between four positive plasmids with HER2 sequence. Upper half show there were total 6 mutation points and their critical sequences were shown at the lower half
图5. 目标克隆的筛选与检测结果。(A/B) 双片段/三片段连接组平板菌落。(C) A平板菌落的PCR检测结果。引物及产物预期长度见图示,以ddH2O做负对照。(D) B平板菌落的PCR检测结果,以pcDNA3.1空质粒做正对照。(E) 阳性克隆与HER2基因序列比对结果。上半部分显示共有6个突变位点,下半部分为上述6个突变位点附近碱基序列
4. 讨论
虽然全基因合成可以节省基因克隆时间,但是长基因合成的价格昂贵且工作周期较长。显然,克隆基因部分片段较基因全长更容易。以HER2基因为例,虽未能直接扩增出全长基因,但顺利得到了该基因的三个片段。单独使用OE-PCR融合法或酶切–连接法都未能将上述基因片段拼接成完整基因,但两种方法互相取长补短,成功实现了基因片段拼接而得到完整HER2基因(图6)。
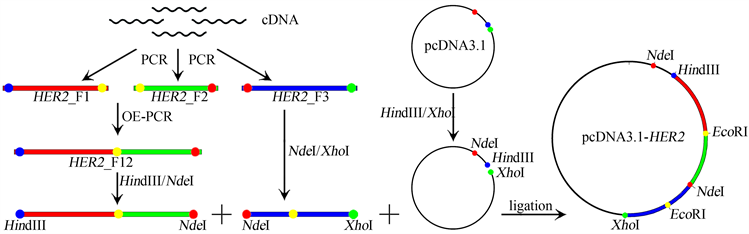
Figure 6. The procedure diagram of HER2 gene cloning
图6. HER2基因克隆过程示意图
某些非典型酶切连接技术和同源重组技术在此并不适用。依赖于同尾酶的BioBrick技术需添加酶切位点并在DNA连接处形成“疤痕” [14] [15],显然不适合此处基因片段的精确拼接。依赖于IIs型限制性内切酶的Golden gate克隆技术要求识别位点不能存在于所连接的DNA片段内部,否则需对相关位点进行沉默突变 [16] [17]。同源重组对于超长DNA片段的组装有明显优势,但是对DNA拼接的精确控制性稍差 [18]。从理论上分析,Gibson组装也适用于本例基因片段的拼接 [19]。该技术目前主要应用于大片段、多片段组装,如病毒载体的改造和重构等 [20] [21],对其拼接效率、影响因素和技术细节还缺乏清晰认识。
双片段连接法可突破严格的酶切位点要求对单片段顺序连接的限制。本例中载体MCS内并无NdeI位点,反倒是其他位置另有一个干扰性NdeI位点(图1),因此两个基因片段无法顺序连接插入载体,双片段连接法则不受影响。相对常规单片段连接,双片段连接时得到的转化克隆数较少,且阳性率略低 [22]。增加同时连接的基因片段数会进一步降低正确连接概率,以致难以筛选出阳性克隆。OE-PCR的影响因素似乎更加难以琢磨。上、下游片段长度、浓度、重叠区域长度及序列特征等因素,以及PCR退火温度、缓冲液成分等反应条件都在一定程度上影响其结果。两种DNA拼接方法的具体组合方案要结合实际情况和实验室条件进行针对性设计。
基金项目
襄阳市医疗卫生领域科技计划项目(2020YL37);湖北文理学院大学生创新创业训练计划项目(X202110519011)资助。
文章引用
陈婉煜,刘君怡,李国庆,刘媛媛,陈华波. 多片段拼接法克隆人表皮生长因子受体2基因
Cloning of Human Epidermal Growth Factor Receptor 2 Gene by Multi Fragment Assembly[J]. 生物医学, 2022, 12(01): 1-8. https://doi.org/10.12677/HJBM.2022.121001
参考文献
- 1. Church, D.M. and Buckler, A.J. (1999) Gene Identification by Exon Amplification. Methods in Enzymology, 303, 83-99. https://doi.org/10.1016/S0076-6879(99)03008-6
- 2. 陈悦康, 李活, 王飞, 等. 核酸筛查实验室RNA酶污染防止对策探讨[J]. 中国自然医学杂志, 2007, 9(2): 137-138.
- 3. Yasukawa, K., Mizuno, M., Konishi, A., et al. (2010) Increase in Thermal Stability of Moloney Murine Leukaemia Virus Reverse Transcriptase by Site-Directed Mutagenesis. Journal of Biotechnology, 150, 299-306. https://doi.org/10.1016/j.jbiotec.2010.09.961
- 4. Patterson, J.T., Nickens, D.G. and Burke, D.H. (2006) HIV-1 Reverse Transcriptase Pausing at Bulky 2’ Adducts Is Relieved by Deletion of the RNase H Domain. RNA Biology, 3, 163-169. https://doi.org/10.4161/rna.3.4.4020
- 5. 颜冰. 长链逆转录PCR的研究进展[J]. 微生物学免疫学进展, 1998(4): 81-84.
- 6. 梁明星, 马梦琪, 程盈盈, 等. 部分扩增结合双片段连接法克隆系列截短基因突变体[J]. 生物技术进展, 2021, 11(1): 111-117.
- 7. 肖娟, 马梦琪, 梁明星, 等. 部分扩增与双片段连接相结合制作长基因定点突变[J]. 生物工程学报, 2020, 36(6): 1232-1240.
- 8. Moasser, M.M. (2007) Targeting the Function of the HER2 Oncogene in Human Cancer Therapeutics. Oncogene, 26, 6577-6592. https://doi.org/10.1038/sj.onc.1210478
- 9. Magnifico, A., Albano, L., Campaner, S., et al. (2007) Protein Kinase Calpha Determines HER2 Fate in Breast Carcinoma Cells with HER2 Protein Overexpression without Gene Amplification. Cancer Research, 67, 5308-5317. https://doi.org/10.1158/0008-5472.CAN-06-3936
- 10. Bragin, P.E., Mineev, K.S., Bocharova, O.V., et al. (2016) HER2 Transmembrane Domain Dimerization Coupled with Self-Association of Membrane-Embedded Cytoplasmic Juxtamembrane Regions. Journal of Molecular Biology, 428, 52-61. https://doi.org/10.1016/j.jmb.2015.11.007
- 11. Han, X., Wang, R., Zhou, Y., et al. (2018) Mapping the Mouse Cell Atlas by Microwell-Seq. Cell, 172, 1091-1107. https://doi.org/10.1016/j.cell.2018.02.001
- 12. Li, P., Du, J., Goodier, J.L., et al. (2017) Aicardi-Goutieres Syndrome Protein TREX1 Suppresses L1 and Maintains Genome Integrity through Exonuclease-Independent ORF1p Depletion. Nucleic Acids Research, 45, 4619-4631. https://doi.org/10.1093/nar/gkx178
- 13. 杨林, 王柳月, 李慧美, 等. 改进的多片段重叠延伸PCR制作基因多位点突变[J]. 中国生物工程杂志, 2019, 39(8): 52-58.
- 14. Tavassoli, A., Soleymani, S., Haghparast, A., et al. (2020) Reverse Genetics Assembly of Newcastle Disease Virus Genome Template Using Asis-Sal-Pac BioBrick Strategy. Biological Procedures Online, 22, 9. https://doi.org/10.1186/s12575-020-00119-3
- 15. Rokke, G., Korvald, E., Pahr, J., et al. (2014) BioBrick Assembly Standards and Techniques and Associated Software Tools. Methods in Molecular Biology, 1116, 1-24. https://doi.org/10.1007/978-1-62703-764-8_1
- 16. HamediRad, M., Weisberg, S., Chao, R., et al. (2019) Highly Efficient Single-Pot Scarless Golden Gate Assembly. ACS Synthetic Biology, 8, 1047-1054. https://doi.org/10.1021/acssynbio.8b00480
- 17. Chiasson, D., Gimenez-Oya, V., Bircheneder, M., et al. (2019) A Unified Multi-Kingdom Golden Gate Cloning Platform. Scientific Reports, 9, Article No. 10131. https://doi.org/10.1038/s41598-019-46171-2
- 18. 倪现朴, 季州翔, 夏焕章. 同源重组方法在制备DNA大片段中的应用[J]. 沈阳药科大学学报, 2014, 31(8): 663-668.
- 19. Gibson, D.G., Young, L., Chuang, R.Y., et al. (2009) Enzymatic Assembly of DNA Molecules Up to Several Hundred Kilobases. Nature Methods, 6, 343-345. https://doi.org/10.1038/nmeth.1318
- 20. 邹小辉, 夏百成, 郭小娟, 等. 利用Gibson DNA组装技术构建人5型腺病毒感染性克隆[J]. 病毒学报, 2018, 34(3): 349-355.
- 21. Li, Z.N., Jelkmann, W., Sun, P.P., et al. (2020) Construction of Full-Length Infectious cDNA Clones of Apple Stem Grooving Virus Using Gibson Assembly Method. Virus Research, 276, Article ID: 197790. https://doi.org/10.1016/j.virusres.2019.197790
- 22. 王柳月, 李慧美, 马梦琪, 等. 利用旁侧引物提高重叠延伸PCR定点突变效率[J]. 生物技术通报, 2019, 12(35): 10-16.
NOTES
*通讯作者。
