Hans Journal of Agricultural Sciences
Vol.08 No.06(2018), Article ID:25648,17
pages
10.12677/HJAS.2018.86100
Diospyros oleifera and D. deyangensis Are Revealed as the Closest Relatives to D. kaki by E3 Ubiquitin-Protein Ligase UPL3 DNA Sequences
Wenqing Li1*, Yong Yang2*, Xiaoman Xie1, Yizeng Lu1, Qing Chang3, Xiaobai Jin4, Zhili Suo5#
1Shandong Provincial Center of Forest Tree Germplasm Resources, Jinan Shandong
2College of Horticulture, Northwest A & F University, Yangling Shaanxi
3Xinjiang Institute of Ecology and Geography, Chinese Academy of Sciences, Urumqi Xinjiang
4Beijing Botanical Garden, Institute of Botany, Chinese Academy of Sciences, Beijing
5State Key Laboratory of Systematic and Evolutionary Botany, Institute of Botany, Chinese Academy of Sciences, Beijing

Received: Jun. 4th, 2018; accepted: Jun. 20th, 2018; published: Jun. 28th, 2018

ABSTRACT
Diospyros of the family Ebenaceae includes more than 400 species in the world. Fruits of persimmon plants are well-known traditional food. In the past decades, persimmon germplasm resources are threatened due to urbanization and environmental destruction. There is an urgent need to make an evaluation on the genetic diversity of Diospyros for conservation and utilization. However, due to the shortage and limited sensitivity of morphological and DNA markers, development of molecular identification tools for the Diospyros plants is still a global challenge. In this study, a new DNA marker from the E3 ubiquitin-protein ligase UPL3 gene region was found to be sensitive for discriminating Diospyros plants. Diospyros deyangensis and D. oleifera were revealed as the closest relatives to D. kaki by the E3 ubiquitin-protein ligase UPL3 DNA sequences. Our results would be of significant value for understanding the origin of cultivated persimmon cultivars and for future researches on global genetic diversity assessment and phylogeny of Diospyros plants.
Keywords:Diospyros, E3 Ubiquitin-Protein Ligase, DNA Marker, Cultivar, Origin, Genetic Diversity
E3泛素–蛋白连接酶UPL3 DNA序列揭示德阳柿和油柿为栽培柿的最近缘物种
李文清1*,杨勇2*,解孝满1,鲁仪增1,常青3,靳晓白4,索志立5#
1山东省林木种质资源中心,山东 济南
2西北农林科技大学园艺学院,陕西 杨凌
3中国科学院新疆生态与地理研究所,新疆 乌鲁木齐
4中国科学院北京植物园,北京
5中国科学院植物研究所系统与进化植物学国家重点实验室,北京
收稿日期:2018年6月4日;录用日期:2018年6月20日;发布日期:2018年6月28日

摘 要
柿树的果实是著名传统食物。在过去几十年,由于城市化和环境破坏,柿树的种质资源受到威胁。柿科的柿属植物,在全世界约有400多种。为了保护和利用,急需评价柿属植物的遗传多样性。然而,由于形态和DNA标记数量少,灵敏度有限,柿属植物的鉴定仍然存在困难,开发分子鉴定方法仍然是全球的挑战。本文中,利用E3泛素–蛋白连接酶UPL3 DNA区域研发出新的DNA序列标记,可用于柿属植物分类。该DNA序列标记揭示,德阳柿和油柿是栽培柿的最近缘物种。对于深入理解栽培柿的起源、研究全球柿属植物的遗传多样性以及系统发育关系具有重要意义。
关键词 :柿属植物,E3泛素–蛋白连接酶,DNA序列标记,栽培品种,起源,遗传多样性

Copyright © 2018 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
柿属植物广泛分布于热带和温带,全世界约有400种。该属的栽培柿品种的果实,在中国、韩国和日本自古以来是食物来源。其树皮、叶、木材和种子也作为药材。目前,中国约有1000多个栽培柿品种 [1] [2] [3] 。
柿(Diospyros kaki)是果树产业发展的最佳选择之一。在中国,不同地区有当地的品种。甜柿品种和抗病品种是新品种培育的目标。通常,近缘物种具有优良的遗传背景,是重要的育种亲本来源,也是研究栽培柿起源的重要材料 [3] 。柿D. kaki的起源、柿属植物的精确鉴定及遗传关系一直是研究的重点和热点 [1] [3] - [15] 。
过去几十年来,由于人类活动,如城市化、畜牧业、农业、矿业以及火灾等影响,柿的种质资源受到威胁 [3] [4] [5] 。柿属植物种质多样性的评价和保护急需深入研究。
柿属植物的形态特征,例如,果实形态、颜色、品质以及花部特征,已有较详细的调查记载 [1] [3] [6] [7] [8] [9] 。根据染色体数目,柿属植物分为4个倍性类型,二倍体(2n = 2x = 30)、四倍体(2n = 4x = 60)、六倍体(2n = 6x = 90)及九倍体(2n = 9x = 135) [7] 。
在柿属植物的分子鉴定研究方面,开发了一些叶绿体DNA标记(如,rbcL、matK、trnH-psbA)和细胞核DNA标记(例如,核糖体DNA内转录间隔区,internal transcribed spacer of ribosomal DNA,ITS) [16] [17] [18] [19] [20] 。近年来,叶绿体基因组信息也开始应用于柿属植物的遗传分析 [21] [22] 。然而,上述DNA分析对于近缘种和品种的鉴定,分辩能力有限 [16] - [23] 。有待开发更高效的DNA序列标记,来揭示柿属植物的遗传多样性和遗传关系。
本课题组研究发现,利用泛素连接酶基因序列开发的分子标记,在胡桃科(Juglandaceae)植物的种、变种及品种水平上具有分辨力 [16] 。泛素连接酶基因是泛素–蛋白酶系统的相关基因的一类,在植物界广泛存在,属于进化速率较高的基因组区域之一 [16] [21] [22] [24] [25] 。
“泛素–蛋白酶体系统”的作用是降解蛋白质,对于维持细胞的自动调节与稳恒性必不可少,存在于已知的所有真核生物中 [24] 。待降解的蛋白质需要泛素化和靶定,通过如下3种酶的参与来实现:即泛素活化酶E1 (ubiquitin activating enzyme E1)、泛素交联酶E2 (ubiquitin conjugating enzyme E2)以及泛素连接酶E3 (the ubiquitin ligase E3)。加了泛素标记的需要降解的蛋白质(底物蛋白质)只有移动到桶状的蛋白酶的腔体内,才能最终被分解为较小的多肽或其它具有生物活性的小分子,以便重复利用。Aaron Ciechanover、Avram Hershko和Irwin Rose因发现特定的蛋白质在被降解之前会被泛素标记,获得了2004年诺贝尔化学奖 [24] 。在植物中,不仅是蛋白质降解过程,而且包括DNA修复过程、细胞周期调控、细胞生长、免疫系统功能、激素介导的信号传递等许多细胞功能方面,泛素都具有重要作用。近年来,据报道泛素连接酶E3,能够提供用于系统发育分析的有价值的信息 [24] 。
本研究的目的在于:1) 为柿属植物鉴定开发新的DNA序列标记;2) 评价泛素连接酶基因序列在柿属植物遗传分析方面的应用价值。
2. 材料与方法
2.1. 取样和DNA提取
柿属植物的供试样品(野生种和品种)见表1,包含了中国国内可采集到的全部野生种。2016年在北京植物园以及陕西省杨凌的西北农林科技大学管理的国家柿种质资源圃(北纬34˚16'56.24'',东经108˚04'27.95'',海拔420 m)采集嫩叶,利用变色硅胶快速干燥备用。利用天根生物技术有限公司的基因组DNA提取试剂盒(DP305)提取基因组DNA。
2.2. PCR扩增与测序
新设计开发出的引物DUB_5498F (5’-ACTTAGGGTAGGGAGGTTGC-3’)和DUB_6322R (5’-GGATAGCCAGGAAGGGTGA-3’)用于扩增柿样品的E3泛素-蛋白连接酶UPL3基因区域。Taq DNA聚合酶和PCR (Polymerase Chain Reaction,聚合酶链式反应)缓冲液(TaKaRa Code: DR100B)来自宝生物技术有限公司(TaKaRa Biotechnology Co. Ltd.) (中国大连)。根据宝生物技术有限公司的实验指南(protocol of TaKaRa Code: DR100B)进行PCR扩增。PCR程序为:94℃预变性4.5 min;94℃变性55 s,引物的退火温度57.2℃ 15 s,72℃延伸1.2 min,34个循环;最后,72℃延伸10 min。
利用热循环仪(Applied Biosystems VeritiTM 96-Well Thermal Cycler, Model#: 9902, made in Singapore)扩增目标区域的DNA序列。PCR产物送到上海美吉生物医药公司(Shanghai Majorbio Bio-Pharm Technology Co., Ltd. (Beijing)),利用同样的引物,在3730xl DNA analyzer (Applied Biosystems, 115 Foster City, CA, USA)测序仪上进行测序。根据BigDye® Terminator v1.1 Cycle Sequencing Kit (Applied Biosystems, Foster City, CA, USA)实验指南做测序反应。测序反应条件为:96℃预变性1 min;96℃变性10 s,引物的退火温度50℃ 5 s,60℃延伸4 min,25个循环;最后,在4℃保存。获得了高质量的序列。Gen Bank的序列号为MF156269-MF156286,见表1。
2.3. 数据分析
利用Sequencher (v.4.6)软件,手工校对DNA序列和排序。利用Mega6.0软件比对 [26] 。比对序列数据的长度为696 bp,包含18条泛素连接酶基因序列。利用Mega6.0软件,采用Tamura 3-parameter参数,重复抽样1000次,构建邻接树状图(Neighbor-joining, NJ)。
3. 结果与分析
3.1. 柿属植物样品的鉴定
在696 bp长的比对序列数据中,包含了被扩增的核基因组的泛素连接酶基因区域的全部可变碱基位点。可变碱基位点的位置编号如表2、图1、图3所示。根据Suo et al. (2012, 2015, 2016)报道的方法 [16] [17] [18] ,选择关键的单核苷酸多态位点构建简化的核苷酸分子式(表2),用于进一步编制分子分类检索表(图1)。利用泛素连接酶基因序列,成功检测到柿属植物的种间、品种间以及种内个体间的遗传差异(表2、图1、图2以及图3)。
Table 1. Diospyros accessions used in this study
表1. 供试的柿属植物样品
#引自中国西南地区,栽培于中国科学院北京植物园展览温室。其它样品来自陕西省杨凌的国家柿种质资源圃。
3.2 遗传关系
E3泛素–蛋白连接酶UPL3DNA序列揭示,18个样品属于7个谱系(图2)。谱系I包括台湾乌木Diospyros discolor,在遗传关系上,是距离柿D. kaki最远的谱系。谱系II包括老鸦柿和乌柿,均为雌雄异株,与柿属的其它谱系的特征相比,具有显著狭小的叶片,与形态分类结果一致(图2) [1] 。谱系III (金枣柿)、谱系V (油柿)以及谱系VI (德阳柿)各含有1个种。谱系IV包含浙江柿、君迁子和美洲柿三个种,浙江柿和君迁子共同构成的一支,与美洲柿互为姐妹群。
在本研究中,油柿(Lineage V)和德阳柿(Lineage VI)被识别为柿D. kaki (LineageVII)的最近缘的物种。柿D. kaki所属的谱系用3个品种代表,分别为‘怀胎柿’和‘安溪油柿’ (完全涩柿,pollination-constant and astringent,PCA)以及‘甜宝盖’ (完全甜柿,pollination-constant and non-astringent,PCNA)。
4. 讨论
DNA条形码策略成本较低,是植物分类的发展方向。然而,推荐使用的4个DNA标记(rbcL、matK、trnH-psbA和ITS)仅能够满足种级或以上水平的植物鉴定,对于近缘种和/或品种,没有分辨能力 [4] [5] [10] [13] - [22] 。寻找种内水平的植物鉴定的理想解决方案一直是巨大的挑战。
近年来,据报道泛素连接酶E3存在几种类型:环指型E3 (RING finger-containing E3s) (RBR和TRIM家族)、cullin E3复合体、Ubox型E3以及HECT型E3 [24] 。HECT蛋白的结构域足够长(约350个氨基酸),能够提供用于系统发育分析的有价值的信息 [24] 。在胡桃科,来自泛素–蛋白酶系统的E3泛素连接
Table 2. Molecular formula constructed using 16 single nucleotide polymorphic base sites from the ubiquitin ligase gene region for characterizing molecularly the eighteen Diospyros accessions
表2. 利用来自泛素连接酶基因区域的16个单核苷酸多态位点构建的分子式可以区分柿属18个样品

Figure 1. Molecular taxonomic key to the eighteen Diospyros accessions used in this study
图1. 供试的柿属18个样品的分子分类检索表
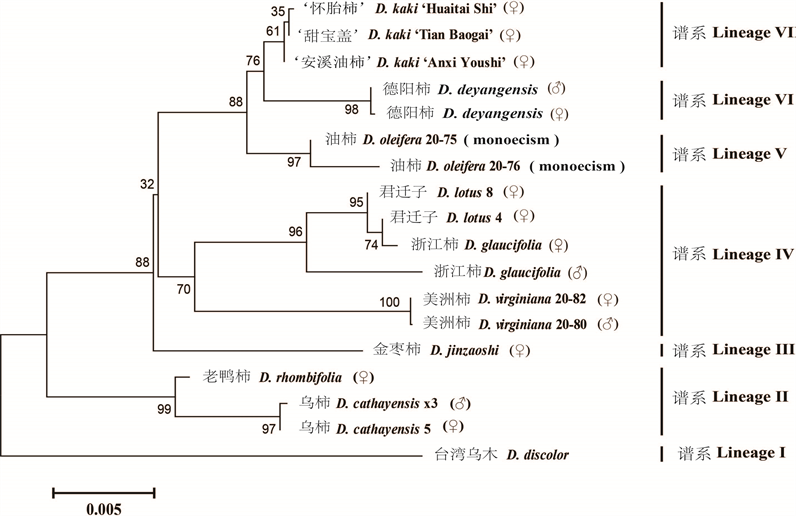
Figure 2. Genetic relationship of the eighteen Diospyros accessions based on the DNA sequences from the E3 ubiquitin-protein ligase UPL3 gene region showing 7 lineages
图2. 基于E3泛素-蛋白连接酶UPL3 DNA序列揭示的柿属18个样品7谱系间的遗传关系
酶基因区域的DNA标记,表现出可以用于种/变种/品种水平的植物鉴定 [16] 。在紫薇属(Lagerstroemia L.),来自泛素–蛋白酶系统的染色体重塑基因区域的DNA标记,在紫薇品种水平上具有分辨力 [17] 。在本研究中,来自泛素–蛋白酶系统的E3泛素–蛋白连接酶UPL3基因序列,表现为可用于种级/种内水平上进行柿属植物的分类。与泛素–蛋白酶系统相关的DNA序列,在开发高灵敏度的DNA序列标记方面有价值。泛素–蛋白酶系统在真核生物中广泛存在,与泛素–蛋白酶系统相关的DNA序列在开发DNA分子标记方面潜力巨大。我们的实验显示,E3泛素–蛋白连接酶基因区域,含有阶段性分布的保守区,在保守区之间,存在进化速率较快的DNA序列,通过开发引物,可以获得灵敏度较高的用于植物分类的DNA序列。有待针对不同的植物科属开展研究,积累数据,总结深层次的规律。柿D. kaki的起源及柿属内物种间的系统发育关系一直是学术界关注的重要问题之一。根据形态和细胞学证据,Ng (1978) [27] 提出油柿Diospyros oleifera是柿D. kaki的亲本之一。根据叶绿体DNA的限制性片段长度多态性,Yenomeri et al. (1998) [28] 认为美洲柿和柿D. kaki具有较为近缘的关系。然而,Choi et al. (2003) [12] 则认为,与柿属的其它物种相比,美洲柿与柿D. kaki的关系可能较远。
我们获得的DNA证据支持先前的分类提案,根据形态和分子特征以及染色体数目,德阳柿和金枣柿应该被提升为种的等级 [7] [8] 。
基于E3泛素–蛋白连接酶UPL3基因序列的遗传关系树状图,德阳柿和油柿位于最靠近栽培柿的位









Figure 3. The 696 bp length sequence alignment generated from ubiquitin ligase gene region of the eighteen Diospyros accessions with thirty-five parsimony informative sites (*) highlighted. M = A/C, W = A/T, K = G/T, Y = T/C, R = A/G, S = G/C, D = G/A/T (i.e., heterozygous base site)
图3. 来自18个柿属植物样品的泛素连接酶基因区域的696 bp长的比对序列,强调了35个简约信息位点(*)。 M = A/C,W = A/T,K = G/T,Y = T/C,R = A/G, S = G/C,D = G/A/T (为杂合碱基位点)
置。值得关注的是,柿D. kaki的染色体数目(2n = 6x = 90),相当于德阳柿(2n = 4x = 60)和油柿(2n = 2x = 30)的染色体数目之和 [7] 。油柿自然分布于中国长江以南,适应性好,耐湿热环境。德阳柿自然分布于中国的四川省。二者的自然分布有一定的重叠 [1] [3] [7] 。它们对于理解柿D. kaki的起源,具有重要价值。
本研究中,代表柿D. kaki谱系的3个品种,分别为‘怀胎柿’、‘甜宝盖’和‘安溪油柿’。在中国,不同地区通常有适应当地环境条件的不同栽培品种。‘怀胎柿’为陕西省的传统品种,脱涩后味道甜美。‘甜宝盖’是湖北省罗田县的传统品种,果实味甜,可以直接食用。‘安溪油柿’为福建省的著名地方品种,通常用来制柿饼。
我们的结果显示,老鸦柿和乌柿在遗传上与栽培柿的关系较远,与形态分类结果一致 [1] [3] [7] 。构建的系统发育关系仅部分地与ITS或matK序列的分析结果一致 [8] 。将来,有必要扩大取样量,深入研究君迁子和浙江柿之间的物种界限。浙江柿和君迁子原产于中国,美洲柿原产于美洲。对这3个种以及这些野生种与柿D. kaki之间的关系的深入研究,有可能增进理解柿属植物的生物地理和进化。
基金项目
山东省农业良种工程重大课题“林木种质资源收集保护与评价”(鲁科字[2014] 96号),合作项目(70009C1036),中国国家自然科学基金项目(No. 31770744), 国家林木种质资源共享服务平台项目(2005DKA21003),农业部物种品种资源保护项目(111721301354052032)以及国家农作物种质资源共享服务平台柿资源子平台项目(NICGR2017-50)。
致谢
感谢王建平老师、王仁梓老师、张会金老师在材料采集期间的指导和帮助。
文章引用
李文清,杨 勇,解孝满,鲁仪增,常 青,靳晓白,索志立. E3泛素–蛋白连接酶UPL3 DNA序列揭示德阳柿和油柿为栽培柿的最近缘物种
Diospyros oleifera and D. deyangensis Are Revealed as the Closest Relatives to D. kaki by E3 Ubiquitin-Protein Ligase UPL3 DNA Sequences[J]. 农业科学, 2018, 08(06): 657-673. https://doi.org/10.12677/HJAS.2018.86100
参考文献
- 1. Lee, S., Michael, G.G. and Frank, W. (1996) Ebenaceae. In: Wu, Z.Y., Raven, P.H. and Hong, D.Y., Eds., Flora of China, Science Press/Botanical Garden Press, Beijing/Missouri/St. Louis, 215-234.
- 2. Angiosperm Phylogeny Group (2016) An Update of the Angiosperm Phylogeny Group Classification for the Orders and Families of Flowering Plants: APG IV. Botanical Journal of the Linnean Society, 181, 1-20. https://doi.org/10.1111/boj.12385
- 3. Wang, R.Z., Yang, Y. and Li, G.C. (1997) Chinese Persimmon Germplasm Resources. Acta Horticulturae, 436, 43-50.
- 4. Duangjai, S.,Wallnöffer, B., Samuel, R., et al. (2006) Generic Delimita-tion and Relationships in Ebenaceae sensu lato: Evidence from Six Plastid DNA Regions. American Journal of Botany, 93, 1808-1827. https://doi.org/10.3732/ajb.93.12.1808
- 5. Duangjai, S., Samuel, R., Munzinger, J., et al. (2009) A Multi-Locus Plastid Phylogenetic Analysis of the Pantropical Genus Diospyros (Ebenaceae), with an Emphasis on the Radiation and Biogeographic Origins of the New Caledonian Endemic Species. Molecular Phylogenetics and Evolution, 52, 602-620. https://doi.org/10.1016/j.ympev.2009.04.021
- 6. 张永芳, 胡超琼, 杨勇, 等. 柿属8种植物花粉形态观察[J]. 园艺学报, 2016, 43(6): 1167-1174.
- 7. Zhang, Y.F., Yang, Y., Guo, J., et al. (2016) Taxonomic Status of Deyangshi Based on Chromosome Number and SRAP Markers. Scientia Horticulturae, 207, 57-64. https://doi.org/10.1016/j.scienta.2016.05.006
- 8. Tang, D.L., Hu, Y., Zhang, Q.L., et al. (2014) Discriminant Analysis of “Jinzaoshi” from Persimmon (Diospyros kaki Thunb.; Ebenaceae): A Comparative Study Conducted Based on Morphological as Well as ITS and matK Sequence Analyses. Scientia Horticulturae, 168, 168-174. https://doi.org/10.1016/j.scienta.2014.01.033
- 9. Mallavadhani, U.V., Panda, A.K. and Rao, Y.R. (1998) Phar-macology and Chemotaxonomy of Diospyros. Phytochemistry, 49, 901-951. https://doi.org/10.1016/S0031-9422(97)01020-0
- 10. Yang, Y., Jing, Z.B., Ruan, X.F., et al. (2015) Development of Simple Sequence Repeat Markers in Persimmon (Diospyros L.) and Their Potential Use in Related Species. Genetics and Molecular Research, 14, 609-618. https://doi.org/10.4238/2015.January.30.2
- 11. Yang, Y., Yang, T.T. and Jing, Z.B. (2015) Genetic Diversity and Taxonomic Studies of Date Plum (Diospyros lotus L.) Using Morphological Traits and SCoT Markers. Biochemical Systematics and Ecology, 61, 253-259. https://doi.org/10.1016/j.bse.2015.06.008
- 12. Choi, Y.A., Tao, R., Yonemori, K., et al. (2003) Genomic Distribu-tion of Three Repetitive DNAs in Cultivated hexaploid Diospyrosspp. (D. kaki and D. virginiana) and Their Wild Rela-tives. Genes & Genetic Systems, 78, 301-308.
- 13. 耿攀, 阮小凤, 杨勇, 等. 柿属植物种质资源遗传多样性的SSR分析[J]. 西北农林科技大学学报: 自然科学版, 2010, 38(12): 190-196. https://doi.org/10.13207/j.cnki.jnwafu.2010.12.021
- 14. Jing, Z.B., Ruan, X.F., Wang, R.Z., et al. (2013) Genetic Diversity and Relationships between and within Persimmon (Diospyros L.) Wild Species and Cultivated Varieties by SRAP Markers. Plant Systematics and Evolution, 299, 1485-1492. https://doi.org/10.1007/s00606-013-0810-1
- 15. Liang, J.J., Liang, Y.Q. and Fu, J.M. (2014) Genetic Relation-ships of Diospyros kaki and Related Diospyros Species Using Chloroplast DNA PCR-RFLP Markers. Acta Horticul-turae Sinica, 2474-2480.
- 16. Suo, Z.L., Chen, L.N., Pei, D., et al. (2015) A New Nuclear DNA Marker from Ubiquitin Ligase Gene Region for Genetic Diversity Detection of Walnut Germplasm Resources. Biotechnology Reports, 5, 40-45. https://doi.org/10.1016/j.btre.2014.11.003
- 17. Suo, Z.L., Li, W.Y., Jin, X.B., et al. (2016) A New Nuclear DNA Marker Revealing Both Microsatellite Variations and Single Nucleotide Polymorphic Loci: A Case Study on Classifica-tion of Cultivars in Lagerstroemia indica L. Journal of Microbial & Biochemical Technology, 8, 266-271. https://doi.org/10.4172/1948-5948.1000296
- 18. Suo, Z.L., Zhang, C.H., Zheng, Y.D., et al. (2012) Revealing Genetic Diversity of Tree Peonies at Micro-Evolution Level with Hyper-Variable Chloroplast Markers and Floral Traits. Plant Cell Reports, 31, 2199-2213. https://doi.org/10.1007/s00299-012-1330-0
- 19. Xu, C., Dong, W.P., Li, W.Q., et al. (2017) Comparative Analysis of Six Lagerstroemia Complete Chloroplast Genomes. Frontiers in Plant Science, 8, 15. https://doi.org/10.3389/fpls.2017.00015
- 20. Dong, W.P., Xu, C., Li, D.L., et al. (2016) Comparative Analysis of the Complete Chloroplast Genome Sequences in Psammophytic Haloxylon Species (Amaranthaceae). PeerJ, 4, e2699. https://doi.org/10.7717/peerj.2699
- 21. Fu, J.M., Liu, H.M., Hu, J.J., et al. (2016) Five Complete Chloroplast Ge-nome Sequences from Diospyros: Genome Organization and Comparative Analysis. PLoS ONE, 11, e0159566. https://doi.org/10.1371/journal.pone.0159566
- 22. Dong, W.P., Liu, J., Yu, J., et al. (2012) Highly Variable Chlo-roplast Markers for Evaluating Plant Phylogeny at Low Taxonomic Levels and for DNA Barcoding. PLoS ONE, 7, e35071.
- 23. Dong, W.P., Chao, X., Li, W.Q., et al. (2017) Phylogenetic Resolution in Juglans Based on Complete Chloroplast Genomes and Nuclear DNA Sequences. Frontiers in Plant Science, 8, 1148. https://doi.org/10.3389/fpls.2017.01148
- 24. Ganoth, A., Tsfadia, Y. and Wiener, R. (2013) Ubiquitin: Molecular Modeling and Simulations. Journal of Molecular Graphics and Modelling, 46, 29-40. https://doi.org/10.1016/j.jmgm.2013.09.006
- 25. Marin, I. (2013) Evolution of Plant Hect Ubiquitin Ligases. PLoS ONE, 8, e68536. https://doi.org/10.1371/journal.pone.0068536
- 26. Tamura, K., Stecher, G., Peterson, D., et al. (2013) MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0. Molecular Biology and Evolution, 30, 2725-2729. https://doi.org/10.1093/molbev/mst197
- 27. Ng, F.S.P. (1978) Diospyros roxburghii and the Origin of Diospyros kaki. The Malaysian Forester, 41, 43-50.
- 28. Yonemori, K., Kanzaki, S., Parfitt, D.E., et al. (1998) Phylogenetic Rela-tionship of Diospyros kaki (Persimmon) to Diospyros spp. (Ebenaceae) of Thailand and Four Temperate Zone Diospyros spp. from an Analysis of RFLP Variation in Amplified cpDNA. Genome, 41, 173-182. https://doi.org/10.1139/g97-106
NOTES
*该作者做出了同等重要的贡献。
#通讯作者。
