Hans Journal of Computational Biology
Vol.06 No.01(2016), Article ID:17198,8
pages
10.12677/HJCB.2016.61003
Characterization Analysis of Codon Usage of Glutenin Subunits Genes in Wheat
Xingqin Cao1, Yanping Ren2, Jianbing Qin3*
1College of Computer Science, Yangtze University, Jingzhou Hubei
2College of Agriculture, Xinjiang Agricultural University, Urumqi Xinjiang
3College of Life Science, Yangtze University, Jingzhou Hubei

Received: Mar. 7th, 2016; accepted: Mar. 21st, 2016; published: Mar. 24th, 2016
Copyright © 2016 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



ABSTRACT
The codon characters of 13 high molecular weight glutenin subunits genes from wheat were analyzed by software CodonW1.4.2 and SPSS16.0. The results showed that ENC of HMW-GS genes ranges between 40 and 50, but 1Dy10 gene’ is less than 40; GC3 contents are between 0.38 and 0.45; 1Bx14 gene prefers to use A or T at the end of codon, and the remaining 12 genes prefer to use G or C ending codons; the Euclidean distance coefficient is minimum between the alleles. The results indicated that the wheat HMW-GS genes have strong codon usage bias; the codon usage is similar; there are bias codons for each amino acid. Considering the codon usage differences, except 1By9 and 1Dy10, the difference of codon use frequency is minimum between x- or y-type subunit genes of glutenin in each chromosome group. The results have certain significance in the efficient use of HMW-GS genetic resources and the cloning of new LMW-GS genes.
Keywords:Wheat, HMW-GS, Codon Bias

小麦麦谷蛋白亚基基因密码子用法特性分析
曹兴芹1,任燕萍2,覃建兵3*
1长江大学计算机学院,湖北 荆州
2新疆农业大学农学院,新疆 乌鲁木齐
3长江大学生命科学学院,湖北 荆州

收稿日期:2016年3月7日;录用日期:2016年3月21日;发布日期:2016年3月24日

摘 要
该文运用CodonW1.4.2和SPSS16.0软件对13个来源于小麦麦谷蛋白亚基基因的密码子用法特性进行分析。结果显示,除1Dy10基因ENC值小于40外,其余亚基基因ENC值均介于40~50之间;GC3含量均在0.38~0.45之间;1Bx14基因偏向于使用A或T结尾的密码子,其余12个亚基基因偏向于使用G或C结尾的密码子;1Dy10与其余12个亚基基因间距离系数较大,然而各组染色体麦谷蛋白x-型或y-型亚基基因间距离系数较小。结果表明,小麦麦谷蛋白亚基基因具有较强的密码子使用偏爱性,各基因密码子用法上具有相似性,每一种氨基酸均存在1~2个偏爱密码子。在密码子用法差异上表现为,除1By9和1Dy10之外,无论是A染色体组、B染色体组,还是D染色体组,各组染色体上麦谷蛋白x-型或y-型亚基基因间密码子使用频率差异最小。这一分析结果对小麦HMW-GS功能基因资源的高效利用和LMW-GS新基因的克隆具有一定的指导意义。
关键词 :小麦,麦谷蛋白亚基,密码子偏爱性

1. 引言
某一物种或某一类基因普遍存在同义密码子非均衡使用的现象,即密码子偏好性,其中通常倾向于使用的一种或几种特定的同义密码子被称为最优密码子 [1] 。大肠杆菌 [2] 、细菌 [3] 、古菌 [4] 、拟南芥 [5] 、玉米 [6] 、水稻 [7] 、籼稻 [8] 、杨树 [9] 、鱼 [10] 、牦牛 [11] 等不同物种的某一类功能基因在密码子使用上存在着明显的偏性,而且某些特定基因 [12] 密码子使用频率与其功能在一定的范围内呈现正相关趋势。然而,有关小麦(Triticum aestivum)麦谷蛋白亚基基因密码子用法特征分析还少见报道,也值得进一步探究。
小麦是国内外最重要的粮食作物之一。我国面包小麦严重缺乏,面包粉消费几乎完全依赖进口。目前,应用麦谷蛋白亚基组成和表达去改良小麦面包烘烤品质研究仍是该领域研究的焦点问题 [13] [14] 。因此,深入探究小麦麦谷蛋白亚基基因密码子使用特性、不同小麦麦谷蛋白亚基基因密码子用法的差异,为进一步深入开展小麦高分子量麦谷蛋白亚基(high molecular weight glutenin subunit, HMW-GS)基因功能研究提供基础数据,同时也为相对滞后的小麦低分子量麦谷蛋白亚基(low molecular weight glutenin subunit, LMW-GS)基因的克隆、表达、结构和功能研究提供科学的理论数据参考。
因此,该文拟以NCBI数据库中提供的小麦麦谷蛋白亚基基因完整的全长编码区序列(coding DNA sequence,简称CDS)为对象,运用CodonW1.4.2软件分析麦谷蛋白亚基基因的密码子使用特性,并结合SPSS16.0软件使用聚类分析的方法比较不同麦谷蛋白亚基基因密码子用法的差异,希望能够为小麦HMW-GS功能基因资源的高效利用和LMW-GS新基因的克隆提供参考。
2. 数据和分析方法
2.1. 小麦麦谷蛋白亚基基因密码子使用特性分析
该文下载的小麦麦谷蛋白亚基基因完整的CDS序列来源于GenBank,其在NCBI数据库(http://www.ncbi.nlm.nih.gov/)的登录号见表1。
Table 1. The accession number of HMW-GS genes in wheat
表1. 小麦HMW-GS基因序列登录号
运用密码子分析软件CodonW1.4.2,计算以下衡量同义密码子使用偏性的主要参数:有效密码子数(ENC)、全长基因GC含量(GC)、密码子第三位GC含量(GC3)、同义密码子相对使用度(RSCU)。
2.2. 基于密码子使用偏好性的聚类
利用SPSS16.0软件对13个来源于小麦麦谷蛋白亚基基因进行基于密码子使用偏性的聚类分析。在对基因密码子使用概率分析时,将每一条基因作为一个对象,同义密码子的RSCU值作为变量,采用59个同义密码子的RSCU值对其密码子使用偏性进行分析。基因间的距离规定为同义密码子相对使用度的欧拉平方距离 [15] 。构建距离系数矩阵,并进行基于该系数的聚类分析。
3. 结果与分析
3.1. 麦谷蛋白亚基基因同义密码子使用偏性分析
来源于小麦的13个麦谷蛋白亚基基因同义密码子使用偏性主要参数ENC、GC、GC3计算结果见表2。ENC值能反映密码子家族中同义密码子偏爱程度。其范围在20~61之间,越靠近20偏性越强。研究结果显示,1Ax1、1Ax2*、1Bx14、1Bx23、1By9、1By15、1By16、1Dx1.5、1Dx2、1Dx5、1Dy11、1Dy12*基因的ENC值均介于40~50之间,表明它们的密码子使用偏爱性中等;而1Dy10基因的ENC值为36.76,表明该基因具有较强的密码子使用偏爱性;进一步分析小麦的麦谷蛋白亚基基因GC含量发现,不同麦谷蛋白亚基基因GC含量较高,均在0.5以上;从密码子第三位GC含量来看,各亚基基因GC3含量在0.38~0.45之间,没有太大差异。由此可见,不同的小麦麦谷蛋白亚基基因的密码子用法上具有相似性。
3.2. 麦谷蛋白亚基基因同义密码子使用频率分析
运用CodonW1.4.2软件计算并分析不同的小麦麦谷蛋白亚基基因密码子使用情况,RSCU值去除了氨基酸组成对密码子使用的影响,且直观地反映了密码子使用的偏爱性。如某一密码子的RSCU值等于1,则表明该密码子的使用没有偏爱;RSCU值大于1,表明该密码子的使用频率相对较高;RSCU值小
Table 2. The ENC values and content of GC for glutenin subunits gene in wheat
表2. 不同麦谷蛋白亚基基因的有效密码子数和GC含量
于1,表明该密码子的使用频率低。研究结果显示,1Ax1基因有23个RSCU值大于1的密码子,1Ax2*有22个,1Bx23有24个,1By9有28个,1By15有27个,1By16有27个,1Dx1.5有23个,1Dx2有24个,1Dx5有23个,1Dy10有21个,1Dy11有25个,1Dy12*有24个,且这些密码子绝大部分以G或C碱基结尾;而1Bx14基因有23个RSCU值大于1的密码子,且大部分以A或T碱基结尾(表3)。由此可见,小麦麦谷蛋白亚基基因偏爱G或C结尾的密码子。
另外,根据同义密码子相对使用度可以看出,在小麦麦谷蛋白亚基中,编码Ala的偏爱密码子为GCA,编码Cys的偏爱密码子为TGC,编码Asp的偏爱密码子为GAC,编码Glu的偏爱密码子为GAG,编码Phe的偏爱密码子为TTT,编码Gly的偏爱密码子为GGA/GGG,编码His的偏爱密码子为GAC,编码Ile的偏爱密码子为ATA,编码Lys的偏爱密码子为AAG,编码Leu的偏爱密码子为CTA/CTC,编码Asn的偏爱密码子为AAC,编码Pro的偏爱密码子为CCA,编码Gln的偏爱密码子为CAA,编码Arg的偏爱密码子为AGG,编码Ser的偏爱密码子为TCA/TCT,编码Thr的偏爱密码子为ACT,编码Val的偏爱密码子为GTC/GTG,编码Tyr的偏爱密码子为TCA。由此可见,不同的小麦麦谷蛋白亚基基因表达,每一种氨基酸均存在1~2个偏爱密码子。
3.3. 不同麦谷蛋白亚基基因密码子使用频率的差异
基于表3中各密码子的RSCU值,利用SPSS计算不同麦谷蛋白亚基基因之间密码子使用偏性的欧式距离系数(表4),并进行基于该系数的聚类分析(图1)。各基因间的距离系数越大,则表达它们在密码子使用上的差异越大。由表4可以看出,1Dy10与1Dx1.5、1Dx2和1Dx5基因间距离系数最大,和1Ax1、1Ax2*、1Bx14、1Bx23、1By9、1Dy11和1Dy12*基因间距离系数次之,而各组染色体上麦谷蛋白x-型或y-型亚基基因间距离系数较小。研究表明,1Dy10与其他麦谷蛋白亚基基因的密码子使用差异较大,而各组染色体上麦谷蛋白x-型或y-型亚基基因之间密码子使用差异较小。
基于距离系数的聚类分析可知,1Dx1.5、1Dx2和1Dx5基因之间密码子用法差异相对较小,成为一
Table 3. The relative synonymous codon usage of glutenin subunits genes in wheat
表3. 不同的小麦麦谷蛋白亚基基因同义密码子的相对使用度
续表
注:大于1的RSCU值用黑体字表示。
亚类;1Bx14和1Bx23成为一亚类;1Ax1和1Ax2*成为一亚类;1By15和1By16成为一亚类;1Dy11和1Dy12*成为一亚类;最后再与密码子用法差异最大的1Dy10成为一类。这一聚类分析结果同样表明,除1By9和1Dy10之外,无论是A染色体组、B染色体组,还是D染色体组,各组染色体上麦谷蛋白x-型或y-型亚基基因间密码子使用频率差异最小。
4. 讨论
该文对13个小麦麦谷蛋白亚基基因密码子的用法分析发现,麦谷蛋白亚基基因的GC含量高。进一步分析发现,该类基因每个氨基酸在同义密码子的使用上都存在明显的偏爱性,大部分麦谷蛋白亚基基因主要偏爱于使用G或C碱基结尾的密码子,少量使用A或T碱基结尾的密码子。该类基因与前人报道的单子叶植物基因组密码子偏爱特性趋势一致 [16] 。
密码子使用偏性产生的生物学基础目前还不是很清楚。近几年研究中,通过对模式生物的密码子用法研究,表明同义密码子使用偏性主要由突变偏性、翻译选择或两者共同作用产生 [17] 。不同物种或同一物种的不同基因对密码子的用法特性有所不同,这种密码子使用偏爱性差异大小与物种的亲缘关系远近有关,亲缘关系越远,密码子偏爱性差异越大,越近则差异越小 [18] 。该文研究结果表明,在小麦麦谷蛋
Table 4. Coefficient of squared Euclidean distance of codon usage bias between samples of glutenin subunits gene
表4. 各麦谷蛋白亚基基因间相对密码子使用度的欧氏距离系数
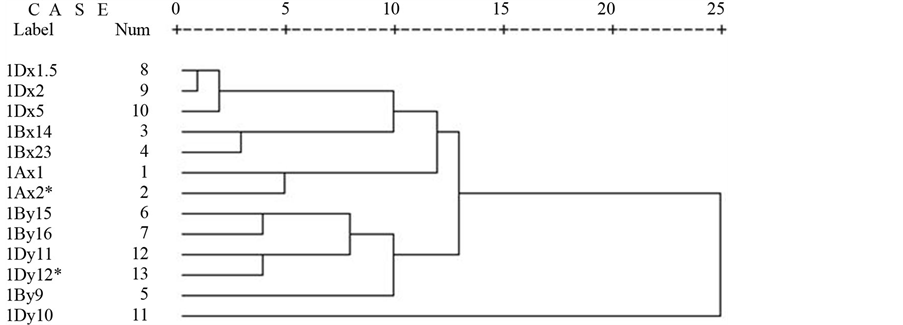
Figure 1. Cluster analysis dendrogram of RSCU for glutenin subunits genes
图1. 基于麦谷蛋白亚基基因相对密码子使用度的聚类图
白亚基基因之间,1Dy10与其他麦谷蛋白亚基基因的密码子使用差异较大,然而无论是A染色体组、B染色体组,还是D染色体组,各组染色体上麦谷蛋白x-型或y-型亚基基因之间密码子使用差异较小;除1By9和1Dy10之外,各组染色体麦谷蛋白x-型或y-型亚基基因间密码子使用频率差异最小。
因此,基于目前编码区密码子使用偏好性和使用信息的聚类分析,可为进一步深入研究小麦HMW-GS基因功能提供基础数据,同时也为相对滞后的小麦LMW-GS基因的克隆、表达、结构和功能研究提供科学的理论数据参考。
文章引用
曹兴芹,任燕萍,覃建兵. 小麦麦谷蛋白亚基基因密码子用法特性分析
Characterization Analysis of Codon Usage of Glutenin Subunits Genes in Wheat[J]. 计算生物学, 2016, 06(01): 18-25. http://dx.doi.org/10.12677/HJCB.2016.61003
参考文献 (References)
- 1. 郭秀丽, 王玉, 杨路成, 等. 茶树CBF1基因密码子使用特性分析[J]. 遗传, 2012, 34(12): 1614-1623.
- 2. 王侃, 刘次全, 曹槐, 等. 大肠杆菌mRNA编码区长度、形成二级结构倾向与密码子偏好性的关系[J]. 微生物学报, 2006, 46(6): 895-899.
- 3. 钟东, 赵贵军, 刘宇虎, 等. 62种细菌基因组中密码子碱基位置上碱基分布的差异性及相关性[J]. 中国微生态学杂志, 2003, 15(3): 134-136.
- 4. Wu, H.N., Da, Y., Wei, J.W., et al. (2007) Synonymous Codon Usage in Methanosarcinamazei str. Goe1 and Other Euryarchaeota Microorganisms. Journal of Southeast University, l23, 289-293.
- 5. 范三红, 郭蔼光, 单丽伟, 等. 拟南芥基因密码子偏爱性分析[J]. 生物化学与生物物理进展, 2003, 30(2): 221- 225.
- 6. 赵耀, 刘汉梅, 顾勇, 等. 玉米waxy基因密码子偏好性分析[J]. 玉米科学, 2008, 16(2): 16-21.
- 7. 张晓峰, 薛庆中. 水稻和拟南芥NBS-LRR基因家族同义密码子使用偏好的比较[J]. 作物学报, 2005, 31(5): 596- 602.
- 8. 刘庆坡, 谭军, 薛庆中. 籼稻品种93-11同义密码子的使用偏性[J]. 遗传学报, 2003, 30(4): 335-340.
- 9. 单文娟, 周猛, 卫巍, 等. 杨树部分基因起始密码子AUG侧翼序列特征分析[J]. 南京林业大学学报(自然科学版), 2008, 32(5): 61-64.
- 10. 龙华, 王祖荣. 15种鱼转铁蛋白cDNA序列密码子使用比较分析[J]. 南方水产, 2005, 1(1): 6-10.
- 11. 赵胜, 张琴, 廖伟璇, 等. 41条牦牛CDS序列的密码子偏好性分析[J]. 西南民族大学学报, 2005, 31(5): 755-760.
- 12. 刘华贵, 王小花, 刘玉方, 等. 五个鸡群中催乳素基因密码子使用频率与产蛋量的关系分析[J]. 生物化学与生物物理进展, 2007, 34(10): 1101-1106.
- 13. 武茹, 高德荣, 别同德, 等. 转基因小麦1Dx5基因沉默的遗传表达和品质效应分析[J]. 麦类作物学报, 2010, 30(6): 991-996.
- 14. 祝长青, 赵惠新, 李艳红, 等. 转基因小麦后代外源目的基因的遗传分析[J]. 干旱区研究, 2008, 25(6): 808-811.
- 15. Das, S., Paul, S. and Dutta, C. (2006) Synonymous Codon Usage in Adenoviruses: Influence of Mutation, Selection and Protein Hydropathy. Virus Research, 117, 227-236. http://dx.doi.org/10.1016/j.virusres.2005.10.007
- 16. 刘汉梅, 赵耀, 顾勇, 等. 几种植物waxy基因的密码子用法特性分析[J]. 核农学报, 2010, 24(3): 476-481.
- 17. Liu, Q., Feng, Y., Zhao, X., et al. (2004) Synonymous Codon Usage Bias in Oryza sativa. Plant Science, 167, 101-105. http://dx.doi.org/10.1016/j.plantsci.2004.03.003
- 18. 吴宪明, 吴松锋, 任大明. 密码子偏性的分析方法及相关研究进展[J]. 遗传, 2007, 29(4): 420-426.
