Botanical Research
Vol.05 No.02(2016), Article ID:17244,11
pages
10.12677/BR.2016.52009
Screening Telomere Binding Proteins from Ginkgo biloba L. Using Yeast One-Hybrid Library
Luyao Jiang, Lihong Li, Xiaoyun Yao, Qiang Zhang, Jingyi Han, Ying Wang, Hui Li, Hai Lu, Di Liu
College of Biological Sciences and Biotechnology, Beijing Forestry University, Beijing

Received: Mar. 5th, 2016; accepted: Mar. 22nd, 2016; published: Mar. 29th, 2016
Copyright © 2016 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



ABSTRACT
Objective: To provide a powerful experimental basis for the research of telomere binding proteins and telomeres in woody plants especially in Ginkgo, we investigated the telomere-binding proteins that bind with telomere sequence in ginkgo. Methods: Using Ginkgo leaves as experimental material, we obtained some gene sequences encoding telomere-binding proteins through yeast one-hybrid library screening technology, then verified their binding specificity by GFP yeast one- hybrid experiments. Results: We did not get any desired DNA sequences using bait vector contain telomere specificity sequence (TTTAGGG)3. However, we obtained 52 DNA sequences using bait carrier contain telomere specificity sequence (TTTAGGG)5 successfully. Our results suggested that the telomere specificity sequence (TTTAGGG)3 might be too short to binding any proteins. After removing repeat sequences, we found that 10 genes were encoded by these 52 DNA sequences through sequences analysis. One gene among them was confirmed that could bind with Ginkgo telomere specificity sequence. Through GFP yeast one-hybrid technology. Conclusion: Our study established a screening method to investigate telomere binding proteins in Ginkgo through yeast one-hybrid library screening and GFP yeast one-hybrid technology, and obtained a telomere binding protein which binding with Ginkgo telomere specificity sequence specifically.
Keywords:Ginkgo biloba L., Telomere Binding Protein, Yeast One-Hybrid Library, Yeast One-Hybrid

利用酵母单杂交文库技术筛选银杏端粒结合蛋白
蒋璐瑶,李丽红,要笑云,张强,撖静宜,王莹,李慧,陆海,刘頔
北京林业大学生物科学与技术学院,北京

收稿日期:2016年3月5日;录用日期:2016年3月22日;发布日期:2016年3月29日

摘 要
目的:为了获得与银杏端粒结合序列相结合的端粒结合蛋白,为木本植物端粒结合蛋白的研究提供实验依据,从而丰富对银杏端粒的研究。方法:以银杏叶片为实验材料,利用酵母单杂交文库筛选获得可能为银杏端粒结合蛋白的基因片段,并通过GFP酵母单杂交实验验证获得基因片段与端粒序列的结合特异性。结果:使用端粒特异性结合序列(TTTAGGG)3的诱饵载体没有获得合适大小的扩增片段,而使用端粒特异性结合序列(TTTAGGG)5的诱饵载体成功获得52个扩增片段,这说明端粒DNA序列(TTTAGGG)3过短,不利用蛋白质的结合。比较这些扩增片段序列并去除相同序列,结果显示这52个扩增片段分别属于10个基因片段。对这10个基因片段进行进一步序列分析,并使用酵母单杂交技术验证,结果显示其中一个基因能够与银杏端粒序列特异性结合。结论:本研究通过酵母单杂交文库筛选,GFP酵母单杂交验证等实验方法建立了银杏端粒结合蛋白的筛选方法,并成功获得了一个和银杏端粒重复序列特异性结合的端粒结合蛋白。
关键词 :银杏,端粒结合蛋白,酵母单杂交文库,酵母单杂交

1. 引言
【研究意义】银杏(Ginkgo biloba L.)是现存最古老的种子植物,被称为植物中的活化石,在遗传学上有重要的地位 [1] 。端粒是位于真核生物染色体末端的特殊DNA-蛋白复合结构,具有保护染色体的作用。其中端粒结合蛋白作为端粒的重要组成部分,在维持端粒结构、调控端粒长度和端粒保护功能方面起十分重要的作用。在木本植物端粒相关蛋白的研究还很空缺 [2] ,只在少部分木本植物中如毛果杨(Populus trichocarpa Torr. & Gray)中克隆得到一些可能的端粒结合蛋白,关于这些可能的端粒结合蛋白的作用机制少见报道。银杏作为我国特有的珍稀名贵树种,其分子生物学、发育生物学及遗传学等领域的研究价值日益重要 [3] 。【前人研究进展】从模式生物拟南芥中首次克隆得到植物端粒序列TTTAGGG。目前已发现的植物端粒双链结合蛋白有水稻中的OsRTBP1 [4] ,拟南芥中的AtTRP1、AtTRB1、AtTRB2 [5] ,玉米中的Smh1等 [6] ,单链结合蛋白有烟草中的NtGTBP1等 [7] 。【本研究切入点】近几年,酵母单杂交系统被广泛用于模式植物转录因子的克隆研究 [8] [9] 。杏作为一种长寿树种,在我国各地普遍存在几百年至数千年的银杏树。这暗示了银杏 能存在特殊的端粒保护结构。因此深入比较研究银杏端粒结合蛋白对端粒的保护机制,在植物端粒生物学研究领域意义重大。【拟解决的关键问题】本研究以银杏为材料,尝试使用酵母单杂交文库筛选端粒结合蛋白,并进一步利用酵母单杂交技术,验证其结合特异性。
2. 材料与方法
2.1. 试验材料与试剂
试验材料为银杏叶片,取自北京林业大学校园内。筛库用酵母单杂交体系Matchmaker Gold Yeast One-Hybrid Library Screening System、Aureo-basidin A(AbA)、Matchmaker Insert Check PCR Mix 1、Matchmaker Insert Check PCR Mix 2均购自Clontech公司;GFP酵母单杂交体系The Grow’n’Glow GFP One-Hybrid Kit购自Mo Bi Tec公司;Easyspin Plus植物RNA快速提取试剂盒购自北京艾德莱生物科技有限公司;mRNA分离试剂盒PolyATtract mRNA Isolation Systems购自Promega公司;反转录试剂盒FastQuant RT Kit购自天根生化科技(北京)有限公司;高保真DNA聚合酶TransStart® FastPfu DNA Polymerase、E. coil DH5а感受态细胞均购自北京全式金生物技术有限公司;限制性内切酶均购自NEB公司;DNA序列合成由生工生物工程股份有限公司完成;DNA测序由北京六合华大基因科技股份有限公司完成。
2.2. 银杏酵母单杂交体系建立
2.2.1. 诱饵酵母载体pBait-AbAi构建
为了提高端粒序列与端粒结合蛋白的识别率和结合率,合成端粒序列(TTTAGGG)的3次和5次串联重复序列与诱饵载体pAbAi进行连接。由于合成片段过短和载体连接效率较低,本研究以载体pBI121为模板,利用PCR扩增其部分GUS基因片段,延长与诱饵载体的连接序列,从而提高连接成功率。根据GUS基因序列设计上、下游引物(表1)。反应体系25 μL,包括上、下游引物(10 μmol/L) 1 μL、pBI121载体(20 ng/μL)稀释100倍取1 μL、2 × Taq Mix 12.5 μL、ddH2O 9.5 μL。扩增程序:94℃ 5 min,94℃ 30 s,55℃ 30 s,72℃ 1 min,30个循环,72℃延伸10 min。经1%琼脂糖凝胶电泳检测回收纯化与载体pMD-18T连接,16℃连接3 h,转化大肠杆菌DH5α,用含氨苄西林的LB固体培养基筛选阳性克隆,并测序鉴定。载体pAbAi和测序正确的T载体同时用限制性内切酶Hind Ⅲ和XhoⅠ进行双酶切,酶切产物胶回收纯化进行连接,转化大肠杆菌DH5α。阳性克隆单菌落提取质粒,并以限制性内切酶KpnⅠ进行酶切,去除GUS基因片段,剩余片段16℃自连接过夜转化大肠杆菌DH5α。阳性克隆单菌落测序鉴定。
2.2.2. 诱饵载体转化酵母获得诱饵酵母
pAbAi载体(图1(a))含有尿嘧啶报告基因URA3和AUR1-C,其中AUR1-C基因使载体对金担子素A(AbA)具有抗性,在AUR1-C上游的多克隆位点插入诱饵序列,如有顺式作用元件与诱饵序列结合,激活AUR1-C基因表达,诱饵酵母便可在含有AbA培养基上生长,故可利用AbA筛选获得阳性酵母单菌落。
游离的pAbAi载体不能在酵母细胞中表达,只有经限制性酶切位点BstB I或Bbs I线性化的pAbAi载体与Y1HGold酵母基因ura3-52位点进行同源重组,整合到酵母基因组中,才能在酵母中稳定表达。同时使酵母细胞具有编码Ura的能力,可用SD-Ura筛选培养基筛选阳性诱饵酵母单菌落。
参照Yeastmaker Yeast Transformation System 2说明制备酵母菌Y1HGold感受态细胞。用经BstBⅠ酶切线性化的pBait-AbAi 1μg转化Y1HGold感受态细胞,涂布于SD/-Ura固体培养基上,30℃培育3 d,挑取直径在2~3 mm单菌落,利用Matchmaker Insert Check PCR Mix 1进行菌落PCR鉴定,如获得长度为1.35 kb加上三次或五次端粒重复序列产物,则说明诱饵载体正确整合到酵母基因中。
2.2.3. 测定报告基因本底表达水平
为了排除酵母内源转录因子与目的顺式元件相互作用的干扰,需要在筛库之前确定AbA最低使用浓度。挑取在SD/-Ura固体培养基上正常生长的诱饵酵母,悬浮在0.9% NaCl中,将OD600调整至0.002并分别涂布在含0、100、300、500、800、1000和1500 ng/mL AbA的SD/-Ura固体培养基上,根据酵母生长情况,确定筛库所需AbA的最低浓度。
2.2.4. cDNA文库合成
采用TRIzol法提取银杏叶片总RNA。使用PolyATtract mRNA Isolation Systems试剂盒分离出mRNA。以0.5 μg mRNA为模板,采用SMART技术合成单链cDNA,并利用LD-PCR扩增技术获得双链cDNA。
Table 1. PCR primer sequences used for amplifying GUS fragments
表1. 扩增GUS基因片段的引物序列


 (a) (b) (c)
(a) (b) (c)
Figure 1. The structure chart of pAbAi, pGNG2 and pJG4-5 vector
图1. pAbAi、pGNG2和pJG4-5载体示意图
取7 μL扩增产物用1%琼脂糖凝胶电泳检测,其余使用CHROMA SPINTE-400树脂层析柱(Clontech公司)纯化。
2.2.5. 酵母单杂交文库构建及初步筛选
将20 μL经过纯化的双链cDNA(2~5 μg)和3 μg经Sma I线性化的pGADT7-Rec载体混合,参照Yeastmaker Yeast Transformation System 2方法共转化诱饵酵母感受态细胞。将酵母细胞悬浮液分别稀释至1/10、1/100、1/1000和1/10000,各取100 μL分别涂布在SD/-leu和SD/-leu/AbA固体培养基上,以便计算建库的总克隆数,总克隆数小于1.0 × 106会降低结果的可信度。将剩余的菌液涂布在SD/-leu/AbA固体培养基上,30℃培育3 d,挑取直径在2~3 mm单菌落,利用Matchmaker Insert Check PCR Mix 2进行菌落PCR鉴定,对cDNA插入片段进行测序和初步的鉴定和分析。
2.3. GFP酵母单杂交鉴定
2.3.1. 诱饵载体pGNG2-bait构建
为了保持研究一致性,仍设计端粒序列(TTTAGGG) 5次串联重复序列与诱饵载体pGNG2 (图1(b))进行连接,方法同pBait-AbAi载体的构建,限制性酶切位点为Not I、Spe I和Nru I。构建好的载体测序鉴定。
2.3.2. 诱饵酵母构建
诱饵载体pGNG2含有选择标记URA3和报告基因GFPuv,其中URA3能使酵母细胞在SD/-Ura培养基上生长。GAL1,10启动子启动GFPuv基因,在GAL1,10启动子上游的多克隆位点插入诱饵序列,如待鉴定序列所表达蛋白能正确结合诱饵序列,则能使GFPuv基因表达,酵母细胞在紫外(360~400 nm)下发绿色荧光。故可以通过酵母细胞是否发绿色荧光鉴定待测序列和端粒重复序列的结合特异性。
采用LiAc法制备酵母菌EGY48感受态细胞,将构建好的pGNG2-bait诱饵载体转化到EGY48酵母细胞中,涂布在SD/-Ura(Glu)固体培养基上,30℃培养3 d,挑取单克隆提取质粒,通过PCR鉴定并测序。
2.3.3. 表达载体pJG4-5构建
载体pJG4-5 (图1(c))含有GAL1诱导型启动子,控制融合蛋白的表达,在含有半乳糖的培养基中正常表达,抑制其在含葡萄糖的培养基上的表达。载体上的TRP1标记基因使酵母细胞能在缺少色氨酸培养基上生长。
在待鉴定序列首尾两端分别添加限制性酶切位点EcoR I和Xho I,并与pMD18-T载体连接,转化大肠杆菌DH5α,阳性克隆单菌落测序鉴定。构建好的T载体与载体pJG4-5同时用EcoR I和Xho I进行双酶切,1%琼脂糖凝胶电泳检测并回收纯化,回收产物连接,转化大肠杆菌DH5α,双酶切鉴定。
2.3.4. 构建GFP酵母单杂交体系
采用LiAc法制备诱饵酵母感受态细胞。将构建好的含有待鉴定序列的表达载体pJG4-5转化到诱饵酵母细胞中,涂布在SD/-TRP-URA(GAL/RAF)固体培养基上,30℃培养3 d。挑取单菌落置于荧光显微镜下观察,发绿色荧光即可认为待测序列能与端粒序列(TTTAGGG)特异性结合。
3. 结果与分析
3.1. 构建酵母单杂交文库
3.1.1. 诱饵载体pBait-pAbAi构建
在本实验室课题组的前期研究中已鉴定银杏端粒序列为拟南芥型TTTAGGG序列。为了增加可能的端粒结合蛋白与端粒的结合能力,在构建诱饵载体pBait-pAbAi时,分别使用了两个特异性结合序列(TTTAGGG)3和(TTTAGGG)5构建两个诱饵载体pBait-pAbAi。此外,由于特异性结合序列片段较小,为了增加构建成功率,使用了GUS基因片段作为引导序列(图2)。
以载体pBI121为模板,表一的序列为引物扩增GUS基因片段,获得GUS片段加端粒序列(TTTAGGG)三次及五次重复序列的长度约640 bp的条带(图3(a)),与载体pMD-18T连接后测序鉴定正确。
以Hind III和Xho I分别酶切质粒pMD-18T-(TTTAGGG)3/5-GUS和载体pAbAi,分别得到长度约为640 bp和5000 bp的片段,进行连接,转化到大肠杆菌DH5α,Hind III和Xho I双酶切鉴定(图3(b))。
使用限制性内切酶Kpn I对质粒pAbAi-(TTTAGGG)3/5-GUS进行酶切,获得约5000 bp和600 bp两个片段,对长度约5000 bp的片段利用T4连接酶进行自连。经测序鉴定,(TTTAGGG)3/5序列成功连接到载体pAbAi上。
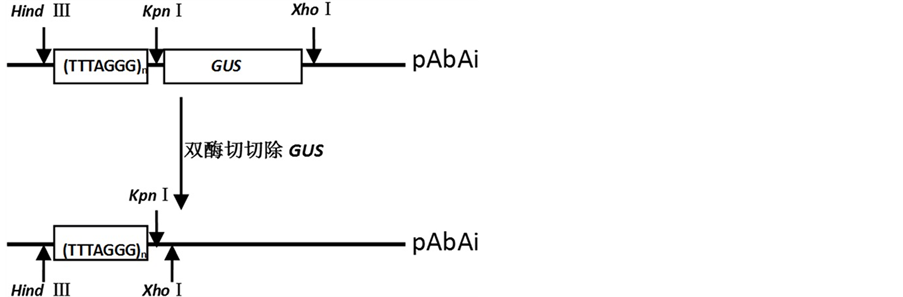
Figure 2. Construction of target-reporter pBait-pAbAi
图2. 诱饵载体pBait-pAbAi构建示意图
3.1.2. 诱饵载体转化酵母Y1HGold
经Bst b I线性化的两个pBait-pAbAi转化酵母感受态细胞,涂布于SD/-Ura固体培养基上,30℃培养3 d,挑取2~3 mm单菌落进行PCR检测。获得长度约1.4 kb的片段(图3(c)),说明诱饵载体成功转入酵母Y1HGold中。
同时将经过0.9% NaCl稀释的酵母菌液涂布于含AbA梯度浓度的SD/-Ura培养基上,当AbA浓度达到1000 ng/mL时完全抑制了酵母单菌落生长,如图4所示,确定本研究所需AbA浓度为1000 ng/mL。
3.1.3. 银杏酵母单杂交文库构建
提取银杏RNA并进行1%琼脂糖凝胶电泳检测,由图5(a)可知,其中28S和18S条带比例约为2:1,总RNA浓度为1200 ng/μL,OD260/280为1.92,说明RNA纯度质量好,可以用于构建文库。


 (a) (b) (c)A: (TTTAGGG)3/5-GUS片段PCR扩增电泳检测图, M:DM 2000 Plus DNA Marker;1-2: (TTTAGGG)3-GUS片段电泳结果;3-4:(TTTAGGG)5-GUS片段电泳结果;B:质粒pMD-18T-(TTTAGGG)5-GUS和载体pAbAi双酶切电泳图, M:DL 15000 DNA Marker;1: 质粒pMD-18T-(TTTAGGG)5-GUS;2: pMD-18T-(TTTAGGG)5-GUS双酶切;3:载体pAbAi;4: pAbAi双酶切线性化;C: 诱饵酵母菌落PCR电泳检测图, M:DL2000 DNA Marker;1:诱饵酵母菌落PCR扩增结果
(a) (b) (c)A: (TTTAGGG)3/5-GUS片段PCR扩增电泳检测图, M:DM 2000 Plus DNA Marker;1-2: (TTTAGGG)3-GUS片段电泳结果;3-4:(TTTAGGG)5-GUS片段电泳结果;B:质粒pMD-18T-(TTTAGGG)5-GUS和载体pAbAi双酶切电泳图, M:DL 15000 DNA Marker;1: 质粒pMD-18T-(TTTAGGG)5-GUS;2: pMD-18T-(TTTAGGG)5-GUS双酶切;3:载体pAbAi;4: pAbAi双酶切线性化;C: 诱饵酵母菌落PCR电泳检测图, M:DL2000 DNA Marker;1:诱饵酵母菌落PCR扩增结果
Figure 3. PCR detection results of vector construction in yeast one-hybrid library
图3. 酵母单杂交文库载体构建电泳检测图
 (a)(b)A:不含AbA的SD/-Ura培养基上诱饵酵母生长情况;B:AbA浓度为1000 ng/mL的SD/-Ura培养基完全抑制诱饵酵母生长
(a)(b)A:不含AbA的SD/-Ura培养基上诱饵酵母生长情况;B:AbA浓度为1000 ng/mL的SD/-Ura培养基完全抑制诱饵酵母生长
Figure 4. The minimal inhibitory concentration of Aureobasidin A
图4. AbA最低使用浓度筛选结果

 (a)(b)A:银杏总RNA电泳检测结果;B:酵母单杂交文库筛选片段电泳检测结果,M:DL15000 DNA Marker;1-18:酵母单菌落PCR电泳结果
(a)(b)A:银杏总RNA电泳检测结果;B:酵母单杂交文库筛选片段电泳检测结果,M:DL15000 DNA Marker;1-18:酵母单菌落PCR电泳结果
Figure 5. Total RNA detection result of Ginkgo biloba L. and PCR detection results of yeast one-hybrid library
图5. 银杏总RNA及酵母单杂交文库筛选片段电泳检测图
总RNA利用磁珠分离出mRNA,浓度为20 ng/μL,OD260/280为1.90。以3 μL mRNA为模板,通过反转录及LD-PCR获得双链cDNA,最后通过CHROMASPINTE-400树脂层析柱进行纯化。
cDNA文库与经Sma I线性化的pGADT7-Rec载体共转化诱饵酵母感受态细胞。将经过转化的酵母细胞稀释1000倍后涂布在SD/-Leu培养基上,共长出20个单菌落,经过计算可知总克隆数即转化效率为3.0 × 106,大于构建文库最低限度1.0 × 106。转化效率 = (菌落个数/涂布菌液体积) × 稀释倍数 × 总体积。
剩余酵母转化产物在SD/-Leu/AbA (1000 ng/mL)培养基上30℃培养3 d,挑取单克隆进行菌液PCR检测(图5(b)),并对其中大于250 bp的扩增片段进行测序。
3.1.4. 可能的银杏端粒结合蛋白的鉴定
结果显示,使用端粒特异性结合序列(TTTAGGG)3的诱饵载体没有能够获得合适大小的扩增片段,显示该端粒特异性结合序列难以有效结合端粒结合蛋白。而使用端粒特异性结合序列(TTTAGGG)5的诱饵载体成功获得52个扩增片段,显示该端粒特异性结合序列能够有效结合端粒结合蛋白。
对52个扩增片段的进行测序,并进行序列比对,去除序列相同的结果,得到10个cDNA序列可能为银杏端粒结合蛋白基因序列片段。进一步利用NCBI的BLAST分析,其中有3个序列显示为功能未知蛋白,7个为无对应序列,这可能是由于缺乏有效的银杏基因组序列的结果。进一步通过DNAMAN等软件分析,这10个序列中有7个序列中存在较多的终止子,不能翻译成相对应的蛋白。说明这7个序列可能是银杏cDNA文库构建过程中的干扰序列。该结果在已报道的酵母单杂交文库筛选技术研究中也时有报道,显示该技术存在一定的假阳性现象。其余3个序列为端粒结合蛋白的相应序列的可能性较大。为了方便区分,暂以A-C来标明该3个基因片段(图6)。由于酵母单杂交文库不能有效鉴定蛋白质与DNA的结合的一一对应关系,为了进一步这3个序列是否独立与端粒序列特异性结合,研究组进一步使用酵母单杂交技术进行分析。
3.2. GFP酵母单杂交鉴定
3.2.1. 诱饵载体pGNG2-bait构建
诱饵载体pGNG2-bait构建方法同诱饵载体pBait-pAbAi的构建,此处不再详述。仅将限制性酶切位点改为Not I、Spe I和Nru I。以序列5’-GCCCAATACGCAAACCGCCT-3’为引物,测序鉴定。
3.2.2. 诱饵载体转化酵母EGY48
将构建好的诱饵载体pGNG2-bait转化到酵母EGY48感受态细胞中,涂布在SD/-URA(Glu)培养基上,30℃培养3 d。挑取单克隆置于SD/-URA(Glu)液体培养基中震荡培养,提取质粒,转化大肠杆菌DH5α,在含氨苄西林的LB培养基上生长,根据挑取阳性单菌落提取质粒电泳检测验证诱饵载体已成功转化入酵母EGY48中。
3.2.3. 表达载体A/B/C-pJG4-5构建
A、B、C三个序列使用DNAMAN软件分析确定碱基翻译序列,分别在上下游添加限制性酶切位点EcoR I和Xho I设计引物,以载体A/B/C-pGADT7-Rec为模板,通过PCR扩增获得长度约1000bp的条带,与载体pMD-18T连接。测序鉴定。
以EcoR I和Xho I酶切A/B/C-pMD-18T与表达载体pJG4-5,获得长度约1000 bp和6500 bp的条带,利用T4连接酶连接。转化大肠杆菌DH5α,EcoR I和Xho I双酶切鉴定(图7)。
3.2.4. GFP酵母单杂交体系建立
将构建好的A/B/C-pJG4-5载体转化到诱饵酵母细胞中,并涂布于SD-TRP-URA(GAL/RAF)固体培养基,30℃培养3 d。挑取单菌落用0.9% NaCl稀释,通过在荧光显微镜下观察,其中A-pJG4-5转化的酵母细胞发绿色荧光(图8),其余未见绿色荧光。说明A序列可以与银杏端粒序列(TTTAGGG)特异性结合。



Figure 6. The nucleotide sequences of (a) (b) (c) gene
图6. (a) (b) (c)基因序列片段


 (a) (b) (c)
(a) (b) (c)
Figure 7. Double enzyme digestion results of A/B/C-pJG4-5 expression vector
图7. 表达载体A/B/C-pJG4-5双酶切电泳检测图

 左图为蛋白与端粒序列特异性结合,细胞发绿色荧光;右图为阴性对照。
左图为蛋白与端粒序列特异性结合,细胞发绿色荧光;右图为阴性对照。
Figure 8. Fluorescent detection of GFP yeast one-hybrid
图8. GFP酵母单杂交荧光显示图
而B,C两个基因与银杏端粒序列(TTTAGGG)特异性结合能力较弱或不能独立与银杏端粒序列(TTTAGGG)特异性结合。
4. 讨论
端粒位于真核生物染色体末端,是一种具有保护染色体作用的特殊DNA-蛋白复合结构。端粒的功能在于防止染色体DNA降解、非正常重组和染色体缺失等,从而起到保护染色体基因完整性的功能,因此端粒功能的发挥为生命活动的正常运作提供了重要保障 [10] 。绝大多数真核生物的端粒DNA序列是由富含TG的重复序列组成,在脊椎动物中为TTAGGG,大多数植物中为TTTAGGG [11] 。
端粒结合蛋白不仅在维持端粒结构上非常重要,且在调控端粒长度和端粒保护功能方面起十分重要作用 [12] 。端粒结合蛋白主要分两类:一类是在哺乳动物中被称为端粒重复结合因子(TRF),其结合于端粒双链区域 [13] 。其中TRF1和TRF2均结合在端粒Myb区域,以二聚体形式存在 [14] 。TRF1控制端粒长度,TRF2参与维持染色体的稳定 [15] 。另一类蛋白以端粒保护蛋白(POT1)为代表,通过寡糖/寡核苷酸结合在端粒单链序列上 [16] 。
端粒在植物学领域的研究还比较缺少。通过凝胶迁移实验,首次在拟南芥中发现了端粒结合蛋白 [17] [18] 。在目前的研究中可以看出,对植物端粒及端粒结合蛋白的相关研究主要集中于草本模式植物,例如拟南芥、水稻、玉米等。对于木本植物端粒的相关研究还少有报道。
一般来说,在酵母单杂交文库筛选过程中,可能存在载体构建时因合成片段过短导致和载体连接效率较低的问题,本研究以载体pBI121为模板,利用PCR扩增其部分GUS基因片段,延长与诱饵载体的连接序列,从而提高连接成功率,后期再通过切除GUS基因片段,载体自我连接即可完成载体构建。目前酵母单杂交文库筛选一般使用植物总RNA来构建cDNA文库,但是存在大量的tRNA和rRNA会对筛选产生干扰,造成过多的假阳性产生。本研究采用磁珠法从总RNA中分离出mRNA来构建cDNA文库,可以有效降低假阳性率。在构建重组酵母时,应确保OD600在0.4~0.6之间,否则易造成转化效率低或者不能成功转化。为了排除酵母内源转录因子与目的顺式元件相互作用的干扰,在筛库之前须确定AbA的最低使用浓度。
总之,本研究通过酵母单杂交文库筛选,GFP酵母单杂交验证等实验方法建立了银杏端粒结合蛋白的筛选技术,并获得了一个和银杏端粒重复序列特异性结合的端粒结合蛋白。本研究丰富了对银杏端粒的研究,为木本植物端粒结合蛋白的研究提供了有力的实验依据。
5. 结论
本研究通过酵母单杂交文库筛选和GFP酵母单杂交等实验方法获得一个与银杏端粒重复序列(TTTAGGG)特异性结合的基因片段。
基金项目
国家自然科学基金项目(31370590, J1103516)。
文章引用
蒋璐瑶,李丽红,要笑云,张强,撖静宜,王莹,李慧,陆海,刘頔. 利用酵母单杂交文库技术筛选银杏端粒结合蛋白
Screening Telomere Binding Proteins from Ginkgo biloba L. Using Yeast One-Hybrid Library[J]. 植物学研究, 2016, 05(02): 55-65. http://dx.doi.org/10.12677/BR.2016.52009
参考文献 (References)
- 1. Jacobs, B.P. and Browner, W.S. (2000) Ginkgo biloba: A Living Fossil. American Journal of Medicine, 108, 341-342. http://dx.doi.org/10.1016/S0002-9343(00)00290-4
- 2. Shakirov, E.V., Song, X., Joseph, J.A., et al. (2009) POT1 Proteins in Green Algae and Land Plants: DNA-Binding Properties and Evidence of Co-Evolution with Telomeric DNA. Nucleic Acids Research, 37, 7455-7467. http://dx.doi.org/10.1093/nar/gkp785
- 3. 慕莹, 赵晓燕, 景丹龙. 银杏不同组织器官及愈伤组织培养中端粒酶活性测定[J]. 北京林业大学学报, 2014, 36(3): 95-99.
- 4. Yu, E.Y., Kim, S.E., Kim, J.H., et al. (2000) Se-quence-Specific DNA Recognition by the Myb-Like Domain of Plant Telomeric Protein RTBP1. The Journal of Bio-logical Chemistry, 275, 24208-24214. http://dx.doi.org/10.1074/jbc.M003250200
- 5. Hwang, M.G., Chung, I.K., Kang, B.G., et al. (2001) Sequence Specific Binding Protein 1 (AtTBP1). FEBS Letters, 503, 35-40. http://dx.doi.org/10.1016/S0014-5793(01)02685-0
- 6. Schrumpfova, P., Kuchar, M., Mikova, G., et al. (2004) Characterization of Two Arabidopsis thaliana Myb-Like Proteins Showing Affinity to Telomeric DNA Sequence. Ge-nome, 47, 316-324. http://dx.doi.org/10.1139/g03-136
- 7. Hirata, Y., Suzuki, C. and Sakai, S. (2004) Characte-rization and Gene Cloning of Telomere-Binding Protein from Tobacco BY-2 Cells. Plant Physiol Biochem, 42, 4-14. http://dx.doi.org/10.1016/j.plaphy.2003.10.002
- 8. 刘蕾, 胡旭东, 张强, 等. 毛白杨酵母单杂交体系的建立[J]. 南方农业学报, 2015, 46(3): 370-375.
- 9. 王琪, 朱延明, 王冬冬. 酵母单杂交系统在植物基因工程研究中的应用[J]. 北京林业大学学报, 2008, 30(1): 141- 147.
- 10. 朱雅新, 麻浩. 端粒和端粒酶的结构与功能及其应用[J]. 湖南农业大学学报, 2005, 31(1): 98-105.
- 11. Shakirov, E.V., Salzberg, S.L., Alam, M. and Shippen, D.E. (2008) Analysis of Carica papaya Telomeres and Telomere- Associated Proteins: Insights into the Evolution of Telomere Maintenance in Brassicales. Tropical Plant Biology, 1, 202-215. http://dx.doi.org/10.1007/s12042-008-9018-x
- 12. Kuchar, M. (2006) Plant Telomere-Binding Proteins. Biologia Plantarum, 50, 1-7. http://dx.doi.org/10.1007/s10535-005-0067-9
- 13. Sue, S.C., Hsiao, H.H., Chung, B.C., et al. (2006) Solution Structure of the Arabidopsis thaliana Telomeric Repeat- Binding Protein DNA Binding Domain: A New Fold with an Additional C-Terminal Helix. Journal of Molecular Biology, 356, 72-85. http://dx.doi.org/10.1016/j.jmb.2005.11.009
- 14. Bianchi, A., Smith, S., Chong, L., et al. (1997) TRF1 Is a Dimer and Bends Telomeric DNA. The EMBO Journal, 16, 1785-1794. http://dx.doi.org/10.1093/emboj/16.7.1785
- 15. Wei, C. and Price, M. (2003) Protecting the Terminus: t-Loops and Telomere End-Binding Proteins. Cellular and Molecular Life Sciences CMLS, 60, 2283-2294. http://dx.doi.org/10.1007/s00018-003-3244-z
- 16. Baumann, P. and Cech, T.R. (2001) Pot1, the Putative Telomere End-Binding Protein in Fission Yeast and Humans. Science, 292, 1171-1175. http://dx.doi.org/10.1126/science.1060036
- 17. Regad, F., Lebas, M. and Lescure, B. (1994) Interstitial Telomeric Repeats within the Arabidopsis thaliana Genome. Journal of Molecular Biology, 239, 163-169. http://dx.doi.org/10.1006/jmbi.1994.1360
- 18. Zentgraf, U. (1995) Telomere-Binding Proteins of Arabidopsis thaliana. Plant Molecular Biology, 27, 467-475. http://dx.doi.org/10.1007/BF00019314
