Open Journal of Nature Science
Vol.06 No.01(2018), Article ID:23362,7
pages
10.12677/OJNS.2018.61004
Identification Method of Two Oyster Species in the South China Sea
Yulan Zhou1, Shouquan Yan2, Lianying Jiang1, Yixia Yang1, Jiafeng Wang2*
1Clinical Medical Research Center, Affiliated Hospital of Guangdong Medical University, Zhanjiang Guangdong
2Stem Cell Research and Clinical Translation Center, Affiliated Hospital of Guangdong Medical University, Zhanjiang Guangdong

Received: Dec. 21st, 2017; accepted: Jan. 2nd, 2018; published: Jan. 9th, 2018
*通讯作者。

ABSTRACT
Two kinds of new oyster species (C. zhanjiangensis and C. dianbaiensis) were identified recently in the South China Sea. However, there was no identification method that could distinguish the two new oyster species except barcode gene sequencing. In this study, we aimed to develop a new identification method for the two new oyster species based on HRM. Amplifying the COI sequence by PCR, the HRM analysis was employed to distinguish the different species. One pair of HRM primers were screened out which could identify the two new oyster species accurately. Sanger sequencing certified that this method was accurate and simple which can be easily used in laboratories or aquaculture departments. This method is of great significance to further genetic study and aquaculture of oyster.
Keywords:C. zhanjiangensis, C. dianbaiensis, Species Identification, COI, HRM
中国南海两种新发现牡蛎物种鉴定方法
周玉兰1,闫守泉2,江连英1,杨漪霞1,王家丰2*
1广东医科大学附属医院临床医学研究中心,广东 湛江
2广东医科大学附属医院干细胞研发与临床转化中心,广东 湛江

收稿日期:2017年12月21日;录用日期:2018年1月2日;发布日期:2018年1月9日

摘 要
最近在我国南海沿岸一带发现两种新的牡蛎物种湛江牡蛎(C. Zhanjiangensis)和电白牡蛎(C. Dianbaiensis)。除了对条形码基因测序外,这两种牡蛎很难用以往的物种鉴定方法区分。本研究拟开发一种基于高分辨率熔解曲线(HRM)的新的区分这两种新牡蛎物种的方法。设计引物扩增该两种牡蛎条形码基因COI(细胞色素C氧化酶I基因)序列显著差异片段,并利用HRM技术对扩增片段进行精细区分从而区分物种信息。结果我们筛选到一组引物能够有效区分这两种牡蛎新物种。经验证该方法精确有效,简单易行,是一般实验室或水产生产单位易于掌握推广的方法。为进一步研究牡蛎物种遗传信息和牡蛎养殖具有重要意义。
关键词 :湛江牡蛎,电白牡蛎,物种鉴定,细胞色素C氧化酶I基因,HRM

Copyright © 2018 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
牡蛎(oyster)是重要的海水养殖资源,全世界每年牡蛎养殖量十分巨大,是产量最大的海洋水产养殖动物(FAO; http://www.fao.org),我国牡蛎的养殖量一直居于世界前列。牡蛎种质资源的多样性是保障养殖高产的重要生物资源。我国境内海域已逐渐发现并鉴定了多种牡蛎物种,不同物种间可能存在生殖隔离,优势养殖性状等现象 [1] [2] 。因此,物种鉴定是牡蛎基础研究,生产养殖的首要的基础步骤。目前在我国境内分布最为广泛的牡蛎物种,包括Crassostrea hongkongensis;Crassostrea ariakensis;Crassostrea angulata;Crassostrea sikamea;和Crassostrea gigas等已经有比较成熟的物种鉴定方法 [3] [4] 。最近中国科学院南海海洋研究所在中国南海地域发现两种新的牡蛎物种C. zhanjiangensis和C. dianbaiensis;目前除了条形码基因测序(包括COI基因(mitochondrial cytochrome c oxidase subunit I)和ITS基因(internal transcribed spacer)等)还没有相应的简单易行的鉴定区分方法 [5] [6] 。本研究拟结合条形码基因序列信息和高分辨率熔解曲线技术开发一种能够精确区分该两种新发现牡蛎物种的简单易行的方法。
2. 材料与方法
2.1. 实验样本材料
收集广东省湛江市,茂名市电白区以及海南省清澜港海域沿岸地区的野生牡蛎样本作为候选实验材料,样本采集地点以及数量见图1。
2.2. 样本DNA提取与物种确认
将采集到的样本全部取样闭壳肌利用无水乙醇保存,然后利用基因组DNA提取试剂盒提取基因组DNA,并利用凝胶电泳检测DNA质量。随机在上述3各地点各选取5个样本,利用COIL1490 (GGTCAACAAATCATAAAGATATTGG)和COIH2198(TAAACTTCAGGGTGACCAAAAAATCA)引物 [5] [6] 对COI基因进行PCR扩增后利用Sanger测序后与NCBI标准序列比对确认物种信息。在每个物种选取一个样本作为标准对照样本参与后续物种鉴定实验。
2.3. 高分辨率熔解曲线(HRM)引物设计与筛选
将两种牡蛎新物种C. zhanjiangensis和C. dianbaiensis的COI基因进行序列比对,筛选能够区分两
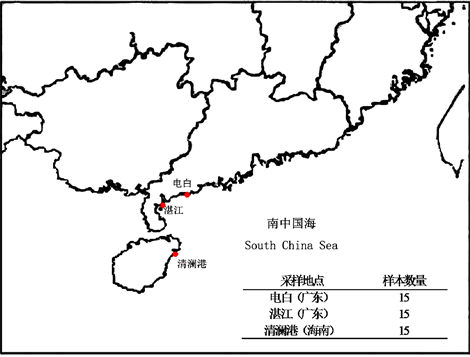
Figure 1. Location and quantity of oyster sample collection
图1. 牡蛎样本采集地点与数量
物种的序列片段,并针对其设计相关引物。引物设计满足以下要求:1) 扩增产物长度在40~150 nt之间;2) 扩增子尽量有多个碱基差异,G+C含量差异明显,以满足不同物种扩增子退火温度有明显差异;3.引物中也尽量避免多态性位点,以确保引物扩增效率和通用型。
利用基因组DNA为模板进行PCR扩增,优化退火温度和反应条件获取最优的扩增效率;然后利用聚丙烯酰氨凝胶电泳检测扩增产物,选取扩增效率好,电泳条带单一的引物进行下一步HRM分型实验。
2.4. HRM分型检测
以已知物种信息样本的基因组DNA为模板,利用上述筛选出的引物组合进行PCR扩增。同时添加已知物种信息的两个样本作为标准参照。然后在扩增产物中添加LCgreen饱和核酸染料,利用PCR仪进行95℃高温变性后冷却到室温。然后利用Lightscanner 96 (Idahol,USA)对PCR产物进行高分辨率熔解曲线检测,温度变化范围设定为40℃~98℃,温度变化速率为0.1℃每秒。根据荧光信号转化后的熔解曲线区分不同物种。
2.5. 准确性验证
以未知物种信息的样本基因组DNA为扩增模板,利用上述筛选的引物组合经过PCR扩增后利用Lightscanner 96分析熔解曲线。在HRM熔解曲线不同的牡蛎新物种样本中随机挑选3个样本,用上述COI基因扩增引物进行PCR扩增后进行Sanger测序,确认上述方法的准确性。
3. 实验结果
3.1. 引物设计与筛选
两种牡蛎新物种C. zhanjiangensis和C. dianbaiensis的COI基因序列信息比对后选取两物种序列碱基信息差异明显的片段设计引物。采用多条前后引物组合的方式对同一片段区域进行扩增,从而增加高扩增效率引物筛选几率。引物扩增区域与引物组合情况如图2。
根据上述COI基因序列设计引物共计5组(表1),经过筛选后有两组引物(ZD-1,ZD-3)扩增效率和特

Figure 2. Primers design. The sequence of COI genes of C. zhanjiangensis (upper) and C. dianbaiensis (lower) are blast. The boxes and different letter colors are represent different primers; the background letters show the difference base between the two species
图2. 引物设计示意图。上下比对的两条序列分别为C. zhanjiangensis和C. dianbaiensis部分COI基因序列。方框与颜色不同字体均表示不同引物,背景字体显示序列碱基差异
Table 1. Primer sequence information
表1. 引物序列信息
ZD-F为前引物,ZD-R为后引物,前后引物分别组成5组引物组合。
ZD-F: front primer; ZD-R: reverse primer. All the primers comprise 5 sets PCR primers.
异性符合HRM要求。
3.2. HRM检测
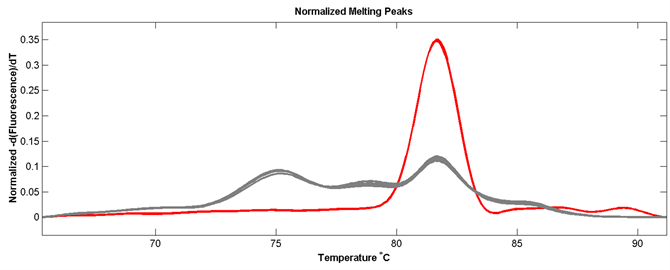
利用HRM分析上述两组引物,结果显示引物组合ZD-1的熔解曲线峰图复杂,不利于正确判读分型(图3(a))。而引物组合ZD-3的熔解曲线峰型简单,所有熔解曲线都汇集成2束,并且每束熔解曲线Tm差异明显,易于分型判读(图3(b))。
3.3. 测序验证
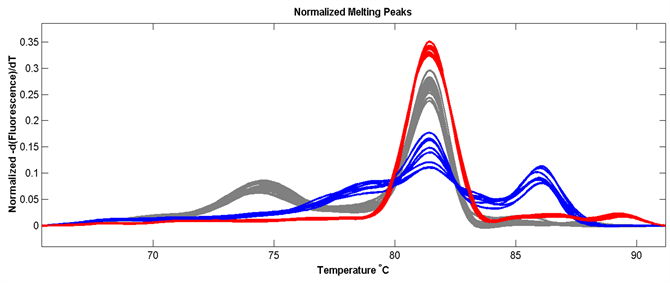
引物组合ZD-3扩增多个未知物种信息的牡蛎样本后呈现3种不同熔解曲线(图3(c))。在三种熔解曲线不同的样本中分别随机选取3个样本的PCR产物Sanger测序后进行序列比对。发现熔解曲线与标准样本聚为一束的样本跟标准样本物种信息相同,另外一束熔解曲线的样本(蓝色曲线)为我国南方较为普遍的香港巨牡蛎(C. hongkongensis)。
4. 讨论
物种鉴定是一项系统工程,也是各种生命科学分类、研究、生产的先决条件之一。随着科技进步,目前的物种鉴定一般都建立在分子生物学基础上,大多的鉴定方法都以稳定的遗传物质为主要的鉴定依据。COI基因因为广泛存在于大多数生命体中,并且序列信息在物种存在适度的变异,在个体间十分保
 (a)
(a)
 (b)
(b)
 (c)
(c)
Figure 3. Melting caves of COI sequence. (a) melting caves of ZD-1; (b) melting caves of ZD-3; (c) melting caves of ZD-3 with unknown species
图3. COI基因片段熔解曲线。(a) ZD-1熔解曲线;(b) ZD-3熔解曲线;(c) ZD-3扩增未知样本后熔解曲线
守,而在多种近缘物种的识别鉴定中被广泛采用做物种鉴定的条形码基因 [2] [7] [8] 。
牡蛎因为其外形受生活环境、空间的影响比较大,并且营养条件和水温等生活条件也能够影响其外形 [9] 。所以无论在基础科学研究还是在水产育种、养殖等过程中,物种鉴定都是首要步骤。以往在我国境内发现的最为普遍的五种牡蛎除了条形码基因测序以外已经有比较成熟的分型方法 [3] [4] 。而目前除了条形码基因测序外,尚没有方法快捷方便的分型鉴定最近新发现的两种牡蛎新物种C. zhanjiangensis和C. dianbaiensis。
HRM技术是以利用核酸序列差异引起双链结构的退火温度差异而区分不用序列片段为核心的核酸研究技术。因为其能够检测到单个核苷酸差异的精度水平而被开发成为多种针对核酸序列差异的研究技术,广泛应用于单核苷酸多态性(SNP)检测,扩增产物特异性检测,遗传鉴定等等方面。近年来发展了多种改进方法,大大拓展了HRM的精度和利用范围,也已在物种鉴定中得到广泛应用 [10] [11] [12] 。
本研究结合HRM技术和COI基因序列片段的差异区分这2种牡蛎新物种;筛选出一组引物组合,能够借助直观的熔解曲线准确快速的识别鉴定C. zhanjiangensis和C. dianbaiensis。该方法无需借助测序仪等大型仪器,仅需针对PCR扩增产物进行HRM分析即可完成C. zhanjiangensis和C. dianbaiensis的区分鉴定,可大大节省成本。而且该方法建立在COI序列差异基础上,可保障鉴定依据的准确性。此外,该方法借助能够区分单个核苷酸差异的HRM技术,保障了该方法的精确性,本实验中该方法对C. zhanjiangensis和C. dianbaiensis样本的区分准确率为100%。结合之前我国5种常见的牡蛎物种的鉴定方法 [3] [4] ,可以有效地区分鉴定我们境内目前发现的牡蛎物种。
5. 结论
本研究提出的基于HRM技术可区分鉴定C. zhanjiangensis和C. dianbaiensis的方法是一种精确、简单并适合中小型实验室和水产养殖公司等生产应用场所的牡蛎物种鉴定方法,对展开牡蛎分子遗传研究与助力水产养殖均有重要意义。
基金项目
本研究受到以下研究项目资助:国家自然科学基金(No. 81600445),广东省医学科研基金(No. A2016522),广东医科大学科研基金(2XJ14006, 2XJ14040),广东医学院附属医院博士科研基金(528B20150012)。
文章引用
周玉兰,闫守泉,江连英,杨漪霞,王家丰. 中国南海两种新发现牡蛎物种鉴定方法
Identification Method of Two Oyster Species in the South China Sea[J]. 自然科学, 2018, 06(01): 17-23. http://dx.doi.org/10.12677/OJNS.2018.61004
参考文献 (References)
- 1. 王海艳. 中国近海常见牡蛎分子系统演化和分类的研究[D]: [博士学位论文]. 北京: 中国科学院研究生院(海洋研究所), 2004.
- 2. 杜玄, 郭希明, 钱鲁闽. 福建沿海巨蛎属牡蛎的主要种类及其分布[J]. 应用海洋学学报, 2009, 28(3): 399-404.
- 3. Wang, H. and Guo, X. (2008) Identification of Crassostrea ariakensis and Related Oysters by Multiplex Species-Specific PCR. Journal of Shellfish Research, 27, 481-487. https://doi.org/10.2983/0730-8000(2008)27[481:IOCAAR]2.0.CO;2
- 4. Wang, J.F., Xu, F., Li, L., et al. (2014) A New Identification Method for Five Species of Oysters in Genus Crassostrea from China Based on High-Resolution Melting Analysis. Chinese Journal of Oceanology and Limnology, 32, 419-425.
- 5. Wu, X.Y., Xiao, S. and Yu, Z.N. (2013) Mitochondrial DNA and Morphological Identification, of Crassostrea zhanjiangensis sp. nov. (Bivalvia: Ostreidae): A New Species in Zhanjiang, China. Aquatic Living Resources, 26, 273-280. https://doi.org/10.1051/alr/2013065
- 6. Xia, J.J., Wu, X.Y., Xiao, S., et al. (2014) Mitochondrial DNA and morphological identification of a new cupped oyster species Crassostreadianbaiensis (Bivalvia: Ostreidae) in the South China Sea. Aquatic Living Resources, 27, 41-48. https://doi.org/10.1051/alr/2014004
- 7. 陈军, 李琪, 孔令锋, 等. 基于COI序列的DNA条形码在中国沿海缀锦蛤亚科贝类中的应用分析[J]. 动物学研究, 2010, 31(4): 345-352.
- 8. Dawnay, N., Ogden, R., Mcewing, R., et al. (2007) Validation of the Barcoding Gene COI for Use in Forensic Genetic Species Identification. Forensic Science International, 173, 1-6. https://doi.org/10.1016/j.forsciint.2006.09.013
- 9. 阙华勇, 刘晓, 王海艳, 等. 中国近海牡蛎系统分类研究的现状和对策[J]. 动物学杂志, 2003, 38(4): 110-113.
- 10. Simenc, J. and Potocnik, U. (2011) Rapid Differentiation of Bacterial Species by High Resolution Melting Curve Analysis. Prikladnaia Biokhimiia I Mikrobiologiia, 47, 256-263.
- 11. Gurtler, V., Grando, D., Mayall, B.C., et al. (2012) A Novel Method for Simultaneous Enterococcus Species Identification/Typing and van Genotyping by High Resolution Melt Analysis. Journal of Microbiological Methods, 90, 167-181. https://doi.org/10.1016/j.mimet.2012.05.002
- 12. Wongkamchai, S., Monkong, N., Mahannol, P., et al. (2013) Rapid Detection and Identification of Brugia malayi, B. pahangi, and Dirofilaria immitis by High-Resolution Melting Assay. Vector-Borne & Zoonotic Diseases, 13, 31-36. https://doi.org/10.1089/vbz.2012.0971
