Pharmacy Information
Vol.
10
No.
03
(
2021
), Article ID:
42396
,
10
pages
10.12677/PI.2021.103014
海洋曲霉属真菌天然产物及基因组挖掘技术的应用
杜新阳,李欢欢,祁江峰,陈超逸,王颖*
中国药科大学,江苏 南京

收稿日期:2021年4月17日;录用日期:2021年5月12日;发布日期:2021年5月19日

摘要
海洋环境具有高压、高盐、低营养、低光照、低氧的特征,因此相比于陆地来源的相同种属的真菌,海洋来源真菌具有更大的产生新型天然产物的能力。到目前为止,已经从海洋来源真菌当中分离得到了上万种化合物。曲霉属真菌作为目前研究最为深入的一个种属,深受研究人员的关注和重视。基因组挖掘技术的发展打破了传统化学分离手段的限制,为寻找新型天然产物提供了新的方法,已经被应用于海洋天然产物的发现当中。本篇文章对海洋来源曲霉属真菌天然产物进行了总结,并对基因组挖掘技术在海洋真菌天然产物发现中的应用进行了初步概括。基因组挖掘技术已经被应用于海洋真菌天然产物的开发当中,是海洋真菌及其天然产物开发的常用手段,加快了先导化合物和生物资源的研究效率。
关键词
海洋真菌,海洋曲霉,天然产物,基因组挖掘,生物合成基因簇
The Natural Products of Marine-Derived Aspergillus Fungi and Application of Genome Mining
Xinyang Du, Huanhuan Li, Jiangfeng Qi, Chaoyi Chen, Ying Wang*
China Pharmaceutical University, Nanjing Jiangsu

Received: Apr. 17th, 2021; accepted: May 12th, 2021; published: May 19th, 2021

ABSTRACT
The marine environment has the characteristics of high pressure, high salt, low nutrition, low light, and low oxygen. Therefore, compared with fungi of the same species from terrestrial sources, marine-derived fungi have a greater ability to produce new natural products. So far, tens of compounds have been isolated from marine-derived fungi. As the most researched species, Aspergillus has received much attention and researchers. The development of genome mining technology has broken the limitations of traditional chemical separation methods and provided a new method for finding new natural products. It has been applied to the discovery of marine natural products. This article summarizes the natural products of marine-derived Aspergillus fungi, and preliminarily summarizes the application of genome mining technology in the discovery of marine fungal natural products. Genome mining technology has been applied to the development of natural products of marine fungi. It is a common method for the development of marine fungi and their natural products, speeding up the research efficiency of lead compounds and biological resources.
Keywords:Marine Fungi, Marine-Derived Aspergillus Fungi, Natural Product, Genome Mining, Biosynthetic Gene Clusters

Copyright © 2021 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
海洋约占地球总表面积的70%,生物资源相当丰富,包括微生物资源、植物资源、动物资源等,其中约80%的生物资源还没有被开发 [1]。天然产物是生物活性物质的重要来源,是新药开发的重要资源。近年来,随着海洋采样技术、海洋生物技术、分子生物学技术的快速发展,海洋生物已经成为活性天然产物的重要来源 [2]。海洋生态系统与陆地生态系统不同,具有高压、高盐、低温、低氧、黑暗、寡营养等特征,这也是陆地来源与海洋来源的天然产物分子结构相差很大的主要原因 [3],同时这也使得从海洋中分离获得新结构且活性较好的天然产物成为可能,为新药开发提供了宝贵的资源 [4]。此外,海洋来源的天然产物陆续被开发为癌症等多种疾病的先导化合物 [5]。
海洋微生物能够产生结构新颖、化学特征独特、活性较好的天然产物 [6],在脂质降解、碳水化合物代谢、有机碳转换中扮演着重要的角色,在生物地球化学循环中发挥着至关重要的作用 [7],直接影响着海洋生态环境 [8]。海洋真菌不仅存在于深海热液口、冰川、海底火山口等各类海洋环境中,还附生在鱼类、藻类、珊瑚、红树林、海绵等海洋动植物中 [9]。研究发现,最初从海绵、珊瑚、软体动物等海洋动植物中分离得到的化合物并不是由生物体自身产生的,而是由其附生或者共生的微生物产生的 [10]。与海鞘、海绵、珊瑚等共生或者附生的真菌产生的化学物质不仅参与了真菌–微生物和宿主–真菌的相互作用,还对宿主的存活具有重要的作用 [11]。真菌作为海绵微生物群落的重要组成部分,因其次级代谢产物活性好、结构新颖而被视为海洋次级代谢产物的重要来源,主要包括曲霉属、青霉属、蓝状菌属等 [12]。
2. 海绵来源真菌活性代谢产物
曲霉属(Aspergillus)是典型的霉菌,次级代谢产物十分丰富,在医学、农业、工业等具有重要的价值 [13]。部分曲霉可以感染人类,引起曲霉病,例如烟曲霉 [14]。部分曲霉可以感染玉米等粮食作物,并产生霉菌毒素,例如黄曲霉、黑曲霉等。部分曲霉被用于化学产业和食品产业,例如酱油曲霉、米曲霉等 [15]。土曲霉能够合成多种天然产物,例如衣康酸和洛伐他汀,被广泛的应用于工业生产和医学,此外土曲霉也是一种典型的人类病原体 [16]。过去几十年中,从海绵及其附生真菌中分离得到了萜烯类、生物碱类、聚酮类、内酯类、肽类等多种结构的化合物 [17]。
2.1. 萜烯类
Takashi等人从海洋海绵中分离得到了倍半萜烯酚Dictyoceratin-A (1)和Dictyoceratin-C (2),见图1,可以降低DU145人源前列腺癌细胞HIF-1α的积累,与RNA聚合酶II相关蛋白3 (RPAP3)结合,抑制R2TP/PEDL/HSP90复合物的形成,对DU145细胞表现出明显的生长抑制作用,这是目前发现为数不多作用于RPAP3的化合物,具有被开发为癌症化疗药物的潜力 [18]。
Liu等人从亚得里亚海域海绵Suberites domuncula中分离得到一株Aspergillus ustus,并从其发酵产物中分离得到两种新型倍半萜化合物(3)和(4),见图1,对L5178Y,HeLa和PC12三种肿瘤细胞均表现出明显的抑制作用,最大抑制浓度(IC50)值在0.6至5.3 µg/mL [19]。
Sun等人从南海海绵Xestospongia testudinaria从分离得到一株曲霉属真菌,从其发酵产物中分离得到二酚二倍半萜类化合物(5),见图1对人肝癌细胞系HepG-2和人宫颈细胞系Caski表现出中等程度的抑制作用,最大抑制浓度(IC50)值的一半为9.31和12.40 µg/mL [20]。
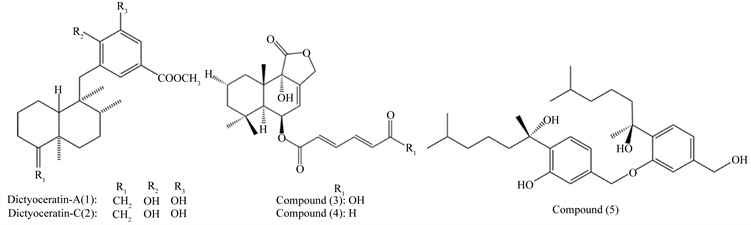
Figure 1. Terpenes from marine Aspergillus
图1. 海洋曲霉来源萜烯类化合物
2.2. 生物碱类
Liu等人从地中海海域海绵Suberites domuncula分离得到一株曲霉属真菌Aspergillus ustus,从其发酵产物中获得了两种新型吡咯烷生物碱类化合物(6)和(7),见图2,这两个化合物对L5178Y鼠淋巴瘤细胞具有一定的细胞毒性,IC50值约为10 mg/ml [21]。
Suradet Buttachon等人从泰国南部海域海绵Epipolasis sp中获得了曲霉属真菌Aspergillus candidus,通过对该菌株进行发酵,采用各种化学手段从其发酵产物中分离得到了新型生物碱类化合物preussin C (8),见图2,该化合物对S. aureus、MRSA、VRE、E. faecalis具有明显的抑制作用。此外,该化合物与粘菌素具有较强的协同作用 [22]。
Zhou等人从中国广东省附近海域来源海绵中分离得到一株曲霉属真菌Aspergillus sp,并从其发酵产物中分离到了新型吲哚生物碱类化合物miszrtine A (9),见图2,该化合物对LNCaP和HL60细胞系具有明显的抑制作用,IC50分别为4.9 µM和3.1 µM。此外,生物活性评价结果显示,吲哚氮是该化合物生物活性的重要官能团 [23]。

Figure 2. Alkaloids from marine Aspergillus
图2. 海洋曲霉来源生物碱类化合物
Takaaki Kubota等人从日本冲绳海域来源海绵中分离得到一株曲霉属真菌Aspergillus sp,并得到了两种新型溴酪氨酸生物碱类化合物tyrokeradine G (10)和tyrokeradine H (11),见图2,其中tyrokeradine G是首个报道的具备β-丙氨酸的溴酪氨酸生物碱,tyrokeradine H具有N-取代吡啶环的稀有溴酪氨酸生物碱,生物活性结果表明,这两个化合物具有较强的抗真菌活性 [24]。
2.3. 内酯类
Huang等人从某海域海绵Haliclona sp获得曲霉属真菌Aspergillus sp. LS45,从其代谢产物种分离得到了新型内酯类化合物aspergilactones A (12),见图3,该化合物对拟南芥的生长具有一定的抑制作用,结果表明,在浓度为100 µM时可以明显抑制拟南芥侧根的生长 [25]。
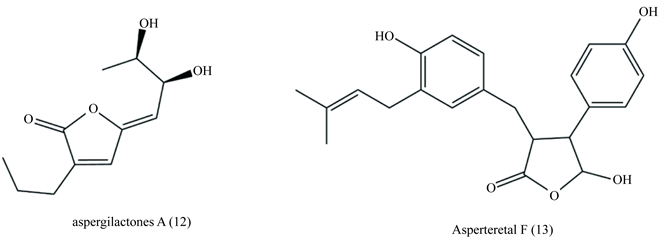
Figure 3. Lactone from marine Aspergillus
图3. 海洋曲霉来源内酯类化合物
Yang等人从中国南海分离得到一株曲霉属真菌Aspergillus terreus Y10,对该菌株进行液体发酵并从乙酸乙酯提取物中获得新型丁烯内酯类化合物Asperteretal F (13),见图3,该化合物对TNF-α具有一定的抑制作用,呈现出明显的剂量依赖性,IC50为7.6 µg/ml,具备开发为抗神经炎药物的潜力 [26]。
2.4. 肽类
Mokhlesi等人从印度尼西亚Ambon岛海域海绵Clathria basilana中分离得到了一种新型环半胱氨酸桥肽microcionamides C (14),见图4,该化合物对A2780人卵巢癌细胞、HL-60急髓白血病M3细胞株具有较强的细胞毒活性。此外该化合物还能够破坏细菌细胞膜电位,对S. aureus具有较强抑制作用 [27]。

Figure 4. Peptides from marine Aspergillus
图4. 海洋曲霉来源肽类化合物
2.5. 其他类
Kotoku等人从印尼海域海绵来源的Trichoderma真菌中分离得到新型二萘并呋喃类衍生物Xylarianaphthol-1 (15),见图5,该化合物能够促进MG63人骨头瘤细胞p21蛋白的表达,对MG63细胞的增殖具有明显的抑制作用,有望被开发成为癌症的化疗药物 [28]。
Xu等人从Plakortis属海绵中分离得到了一种聚酮类化合物Plakortide F acid (PFA) (16),见图5,该化合物对Candida albicans,Cryptococcus neoformans,Aspergillus fumigatus具有明显的抑制作用 [29]。
Gu等人从中国永兴岛海域来源海绵Phakellia fusca获得一株曲霉属真菌Aspergillus flocculosus 16D-1,从其代谢产物中分离得到一种新型甾体类化合物asperflosterol (17),见图5,该化合物可以明显抑制TNF-α的产生 [30]。
海洋真菌因其代谢产物强大的生物活性和药理活性而受到研究人员的广泛关注。海洋天然产物具有明显的免疫调节、抗氧化、抗肿瘤、抗寄生虫、抗病毒、抗菌等生物活性,是先导化合物的重要来源 [31]。曲霉属真菌产生的次级代谢产物结构复杂,通常具备多个手性中心,生物学活性较好,因而受到科学家们的重视 [19]。就已经发现的海洋真菌来源的天然产物来看,结构比较复杂,虽然具备一定的生物学活性,但是选择性不高,可以通过化学手段进行修饰,从而获得活性好、选择性高的先导化合物 [32]。对海洋真菌资源的开发和利用丰富了先导化合物库,增加了人们对海洋微生物资源的了解和认知,为生物医药产业的发展注入新的活力。
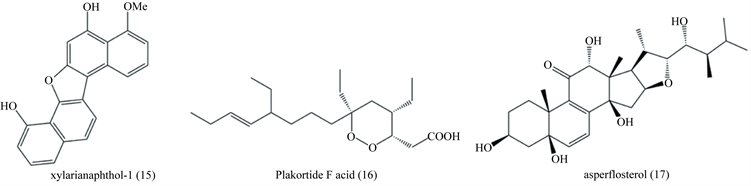
Figure 5. Other compounds from marine Aspergillus
图5. 其他类海洋曲霉来源化合物
3. 基因组挖掘与次级代谢产物发现
基因组挖掘是利用计算机分析,找到假定的次生代谢产物的生物合成基因,以及其合成产物的新兴的遗传方法或者化学方法 [33]。近年来,随着基因组测序技术的发展,加快了海洋生物基因组测序的步伐,为发现新型海洋天然产物提供了重要的支撑 [34]。研究发现,在人工培养的条件下,海洋真菌的大部分基因簇是处于“沉默”状态的,这主要是因为人工培养无法完全复制海洋环境,而海洋环境与陆地环境又有很大的差别 [35]。基因组挖掘技术不仅可以用于发现天然产物的生物合成基因簇,还能够准确的分析天然产物的来源,快速的找到具备合成新结构或者新活性的次生代谢产物的生物 [36]。在基因组数据的支持下,利用生物信息学技术找到天然产物的生物合成基因簇(BGC),预测其编码的化合物结构,从而发现新型衍生物 [37]。微生物基因组中存在大量的BGC,其数量远远多于常规发酵筛选得到的化合物,这些BGC是有功能的,在某种特定的生长条件下这些BGC会表达并且合成相应的产物 [38]。换言之,微生物具备合成不同天然产物的潜力,但是天然产物的表达与培养条件和环境条件密切相关,基因组挖掘技术可以打破这些条件的限制,同时检测多种生物活性天然产物 [39]。基因组挖掘技术已经逐渐被应用至海洋天然产物的发现。
3.1. 生物合成基因簇的发现与修饰
次生代谢产物的生物合成基因通常以生物合成基因簇(Biosynthetic gene clusters, BGC)的形式存在于基因组中 [40]。通过对基因组序列信息进行分析,可以找到天然产物的生物合成基因簇(BGCs),确定BGC在全基因组序列的分布。此外,这些BGC不仅包含合成天然产物的核心酶和定制酶,还包含调节其生物合成所需的小分子产物或者相关基因 [41]。因此,若要激活沉默的BGC,就需要对BGC进行编辑或者修饰。目前,研究人员普遍利用合成生物学或者分子遗传学手段对BGC进行编辑,例如大肠杆菌Red/ET重组、在酵母细胞内利用CRISPR/Cas9和TAR对启动子进行改造和修饰、在细胞外利用CRISPR/Cas9直接编辑基因等 [42]。
3.2. 靶向基因组挖掘
靶向基因组挖掘的主要任务是通过对基因组序列进行分析发现新的生物合成基因簇 [43]。BGCs编码的核心酶主要分为三类:非核糖体肽合成酶(Non-ribosomal peptide synthetase, NRPS)、聚酮合成酶(Polyketide synthase, PKS)、聚酮合成酶–非核糖体肽合成酶(Polyketide synthase-Nonribosomal peptide synthetase enzymes, PKS-NRPS) [44]。其中PKS-NRPS负责聚酮化合物–氨基酸化合物的合成,PKS负责聚酮化合物的合成,NRPS负责铁载体、抗生素等小分子 [45]。除了以上三种BGC,还包括indole、terpene等多种类型。在筛选新型天然产物的BGC的过程中,最关键的步骤是找到生物合成的核心基因或者与天然产物核心结构修饰相关的酶的基因。通常情况下,BGC位于基因组的特定位置,且其编码的酶参与多个化合物的合成 [46]。Kou-San Ju等人对278株不同来源的放线菌进行全基因组测序,利用生物信息学手段对基因组数据进行分析,发现了膦酸酯生物合成的新途径,其产物是新型膦酸肽argolaphos,该肽类化合物具有广谱抗菌作用 [47]。基因组挖掘技术可以找到在正常培养条件下不表达或者表达量很低的次级代谢产物生物合成基因簇。由于生长环境直接影响着基因簇的表达,且决定了代谢产物的分布,可以采用优化培养条件、核糖体工程学、异源表达等方法激活低表达或者不表达的基因簇,从而获得新结构的天然产物 [48]。Mo等人利用基因组挖掘技术,对不同生物的亚麻酸生物合成基因簇进行了分析,通过激活前体肽修饰酶得到了对微球菌具有明显抑制作用的新型Linaridin肽类化合物T2S [49]。
综上所述,基因组挖掘技术能够将天然产物的合成途径与结构紧密的联系起来,使天然产物的分离不再受生物活性筛选方法、培养条件、环境条件等诸多因素的限制,加快新型天然产物的发现速度,丰富天然产物数据库。
4. 总结
天然产物是生物活性化合物的重要来源,是开发人类疾病新药的关键资源。海洋天然产物具有多种生物活性,例如抗菌、抗肿瘤、抗病毒、抗寄生虫、抗氧化等生物活性,因此受到很多研究人员的关注。目前,已经有7种海洋药物被批准上市,12种海洋药物正在进行临床试验 [11]。至本世纪,科学家已经从海洋真菌中分离得到了上万余种具有生物活性的化合物。但是,单纯的依靠传统分离手段大大降低了天然产物的发现速度。基因组挖掘技术是根据化合物的生物合成途径和基因簇序列寻找新型天然产物的综合方针,降低了发现结构新颖、活性强大的先导化合物的难度。在对传统分离手段进行优化的基础上,利用生物信息学手段对基因组信息进行分析挖掘真菌基因簇资源,提高天然产物的发现效率。
致谢
感谢王颖副教授对本文的指导。感谢审阅此文的专家学者,感谢您在百忙之中对本文进行审阅,我将虚心接受各位的意见和建议。
基金项目
国家重点研发计划“深海关键技术与装备”专项“海洋微生物候选药物的高效发现与创新”项目的子课题“深海药源微生物优质菌株的发现与高产育种”(NO: 2018YFC0311001)。
文章引用
杜新阳,李欢欢,祁江峰,陈超逸,王 颖. 海洋曲霉属真菌天然产物及基因组挖掘技术的应用
The Natural Products of Marine-Derived Aspergillus Fungi and Application of Genome Mining[J]. 药物资讯, 2021, 10(03): 102-111. https://doi.org/10.12677/PI.2021.103014
参考文献
- 1. Subramani, R. and Sipkema, D.J. (2019) Marine Rare Actinomycetes: A Promising Source of Structurally Diverse and Unique Novel Natural Products. Marine Drugs, 17, Article No. 249. https://doi.org/10.3390/md17050249
- 2. Yun, C., Kim, H. and Lee, S.J. (2019) Therapeutic Application of Di-verse Marine-derived Natural Products in Cancer Therapy. Anticancer Research, 39, 5261-5284. https://doi.org/10.21873/anticanres.13721
- 3. Jannasch, H. and Taylor, C.J. (1984) Deep-Sea Microbiology. An-nual Review of Microbiology, 38, 487. https://doi.org/10.1146/annurev.mi.38.100184.002415
- 4. Haefner, B.J. (2003) Drugs from the Deep: Marine Natural Products as Drug Candidates. Drug Discovery Today, 8, 536-544. https://doi.org/10.1016/S1359-6446(03)02713-2
- 5. Simmons, T., Coates, R., Clark, B., Engene, N., Gonzalez, D., Esquenazi, E., et al. (2008) Biosynthetic Origin of Natural Products Isolated from Marine Microorgan-ism-Invertebrate Assemblages. Proceedings of the National Academy of Sciences of the United States of America, 105, 4587-4594. https://doi.org/10.1073/pnas.0709851105
- 6. Barbosa, A. and Roque, A. (2019) Free Marine Natural Products Databases for Biotechnology and Bioengineering. Biotechnology Journal, 14, Article ID: 1800607. https://doi.org/10.1002/biot.201800607
- 7. Orsi, W., Edgcomb, V., Christman, G. and Biddle, J.F. (2013) Gene Expression in the Deep Biosphere. Nature, 499, 205-208. https://doi.org/10.1038/nature12230
- 8. Yayanos, A.J. (1995) Microbiology to 10,500 Meters in the Deep Sea. Annual Review of Microbiology, 49, 777-805. https://doi.org/10.1146/annurev.mi.49.100195.004021
- 9. El-Sayed, A., Ibrahim, H. and Sitohy, M. (2014) Co-Immobilization of PEGylated Aspergillus flavipes L-Methioninase with Glutamate Dehydrogenase: A Novel Catalyt-ically Stable Anticancer Consortium. Enzyme and Microbial Technology, 54, 59-69. https://doi.org/10.1016/j.enzmictec.2013.10.004
- 10. Choudhary, A., Naughton, L., Montánchez, I., Dobson, A.D.W. and Rai, D.K. (2017) Current Status and Future Prospects of Marine Natural Products (MNPs) as Antimicrobi-als. Marine Drugs, 15, Article No. 272. https://doi.org/10.3390/md15090272
- 11. Dou, X.J. and Dong, B. (2019) Origins and Bioactivities of Natural Compounds Derived from Marine Ascidians and Their Symbionts. Marine Drugs, 17, 670. https://doi.org/10.3390/md17120670
- 12. Shin, H.J. (2020) Natural Products from Marine Fungi. Marine Drugs, 18, Article No. 230. https://doi.org/10.3390/md18050230
- 13. Bennett, J.J. (2009) Aspergillus: A Primer for the Novice. Medical My-cology, 47, S5-S12. https://doi.org/10.1080/13693780802712515
- 14. Latgé, J., Beauvais, A. and Chamilos, G. (2017) The Cell Wall of the Human Fungal Pathogen Aspergillus fumigatus: Biosynthesis, Organization, Immune Response, and Virulence. Annual Review of Microbiology, 71, 99-116. https://doi.org/10.1146/annurev-micro-030117-020406
- 15. Schubert, M., Spiegel, H., Schillberg, S. and Nölke, G. (2018) Aspergillus-Specific Antibodies—Targets and Applications. Biotechnology Advances, 36, 1167-1184. https://doi.org/10.1016/j.biotechadv.2018.03.016
- 16. Zaehle, C., Gressler, M., Shelest, E., Geib, E., Hertweck, C. and Brock, M. (2014) Terrein Biosynthesis in Aspergillus terreus and Its Impact on Phytotoxicity. Bioorganic & Medic-inal, 21, 719-731. https://doi.org/10.1016/j.chembiol.2014.03.010
- 17. Youssef, F., Ashour, M., Singab, A. and Wink, M. (2019) A Comprehensive Review of Bioactive Peptides from Marine Fungi and Their Biological Significance. Marine Drugs, 17, Article No. 559. https://doi.org/10.3390/md17100559
- 18. Kawachi, T., Tanaka, S., Fukuda, A., Sumii, Y., Se-tiawan, A., Kotoku, N., et al. (2019) Target Identification of the Marine Natural Products Dictyoceratin-A and -C as Se-lective Growth Inhibitors in Cancer Cells Adapted to Hypoxic Environments. Marine Drugs, 17, Article No. 163. https://doi.org/10.3390/md17030163
- 19. Liu, M., Sun, W., Wang, J., He, Y., Zhang, J., Li, F., et al. (2018) Bio-active Secondary Metabolites from the Marine-Associated Fungus Aspergillus terreus. Bioorganic Chemistry, 80, 525-530. https://doi.org/10.1016/j.bioorg.2018.06.029
- 20. Sun, L., Shao, C., Chen, J., Guo, Z.-Y., Fu, X.-M., Chen, M., et al. (2012) New Bisabolane Sesquiterpenoids from a Marine-Derived Fungus Aspergillus sp. Isolated from the Sponge Xestospongia testudinaria. Bioorganic & Medicinal Chemistry Letters, 22, 1326-1329. https://doi.org/10.1016/j.bmcl.2011.12.083
- 21. Cai, R., Jiang, H., Mo, Y., Guo, H., Li, C., Long, Y., et al. (2019) Ophiobolin-Type Sesterterpenoids from the Mangrove Endophytic Fungus Aspergillus sp. ZJ-68. Journal of Natural Products, 82, 2268-2278. https://doi.org/10.1021/acs.jnatprod.9b00462
- 22. Buttachon, S., Ramos, A., Inácio, Â., Dethoup, T., Gales, L., Lee, M., et al. (2018) Bis-Indolyl Benzenoids, Hydroxypyrrolidine Derivatives and Other Constituents from Cultures of the Marine Sponge-Associated Fungus Aspergillus candidus KUFA0062. Marine Drugs, 16, Article No. 119. https://doi.org/10.3390/md16040119
- 23. Zhou, R., Liao, X., Li, H., Li, J., Feng, P., Zhao, B., et al. (2018) Isola-tion and Synthesis of Misszrtine A: A Novel Indole Alkaloid from Marine Sponge-Associated Aspergillus sp. SCSIO XWS03F03. Frontiers in Chemistry, 6, Article No. 212. https://doi.org/10.3389/fchem.2018.00212
- 24. Kubota, T., Watase, S., Sakai, K., Fromont, J., Gonoi, T. and Kobayashi, J. (2015) Tyrokeradines G and H, New Bromotyrosine Alkaloids from an Okinawan Verongid Sponge. Bioorganic & Medicinal Chemistry Letters, 25, 5221-5223. https://doi.org/10.1016/j.bmcl.2015.09.061
- 25. Huang, L., Ding, L., Li, X., Wang, N., Yan, Y., Yang, M., et al. (2019) A New Lateral Root Growth Inhibitor from the Sponge-Derived Fungus Aspergillus sp. LS45. Bioorganic & Medicinal Chemistry Letters, 29, 1593-1596. https://doi.org/10.1016/j.bmcl.2019.04.051
- 26. Yang, L., Ou-Yang, H., Yan, X., Tang, B.-W., Fang, M.-J., Wu, Z., et al. (2018) Open-Ring Butenolides from a Marine-Derived Anti-Neuroinflammatory Fungus Aspergillus terreus Y10. Marine Drugs, 16, Article No. 428. https://doi.org/10.3390/md16110428
- 27. Mokhlesi, A., Stuhldreier, F., Wex, K., Berscheid, A., Hartmann, R., Rehberg, N., et al. (2017) Cyclic Cystine-Bridged Peptides from the Marine Sponge Clathria basilana Induce Apoptosis in Tumor Cells and Depolarize the Bacterial Cytoplasmic Membrane. Journal of Natural Products, 80, 2941-2952. https://doi.org/10.1021/acs.jnatprod.7b00477
- 28. Kotoku, N., Higashimoto, K., Kurioka, M., Arai, M, Fukuda, A., Sumii, Y., et al. (2014) Xylarianaphthol-1, a Novel Dinaphthofuran Derivative, Activates p21 Promoter in a p53-Independent Manner. Bioorganic & Medicinal Chemistry Letters, 24, 3389-3391. https://doi.org/10.1016/j.bmcl.2014.05.083
- 29. Xu, T., Feng, Q., Jacob, M., Avula, B., Mask, M.M., Baerson, S.R., et al. (2011) The Marine Sponge-Derived Polyketide Endoperoxide Plakortide F Acid Mediates Its Antifungal Ac-tivity by Interfering with Calcium Homeostasis. Antimicrobial Agents and Chemotherapy, 55, 1611-1621. https://doi.org/10.1128/AAC.01022-10
- 30. Gu, B., Wu, W., Jiao, F., Jiao, W.-H., Li, L., Sun, F., et al. (2018) Aspersecosteroids A and B, Two 11(9 → 10)-abeo-5,10-Secosteroids with a Dioxatetraheterocyclic Ring System from Aspergillus flocculosus 16D-1. Organic Letters, 20, 7957-7960. https://doi.org/10.1021/acs.orglett.8b03530
- 31. Abdelmohsen, U., Balasubramanian, S., Oelschlaeger, T., Grkovic, T., Pham, N.B., Quinn, R.J., et al. (2017) Potential of Marine Natural Products against Drug-Resistant Fungal, Viral, and Parasitic Infections. The Lancet Infectious Diseases, 17, e30-e41. https://doi.org/10.1016/S1473-3099(16)30323-1
- 32. Ha, M., Song, B., Chung, H. and Paek, S.-M. (2019) De-sign and Synthesis of Anti-Cancer Chimera Molecules Based on Marine Natural Products. Marine Drugs, 17, Article No. 500. https://doi.org/10.3390/md17090500
- 33. Nett, M.J. (2014) Genome Mining: Concept and Strategies for Natural Product Discovery. In: Kinghorn, A., Falk, H. and Kobayashi, J., Eds., Progress in the Chemistry of Organic Natural Products 99, Vol. 99, Springer, Cham, 199-245. https://doi.org/10.1007/978-3-319-04900-7_4
- 34. Chu, L., Huang, J., Muhammad, M., Deng, Z. and Gao, J. (2020) Genome Mining as a Biotechnological Tool for the Discovery of Novel Marine Natural Products. Critical Reviews in Biotechnology, 40, 571-589. https://doi.org/10.1080/07388551.2020.1751056
- 35. Yang, Z., He, J., Wei, X., Ju, J. and Ma, J. (2020) Explora-tion and Genome Mining of Natural Products from Marine Streptomyces. Applied Microbiology and Biotechnology, 104, 67-76. https://doi.org/10.1007/s00253-019-10227-0
- 36. Baltz, R.J. (2019) Natural Product Drug Discovery in the Genomic Era: Realities, Conjectures, Misconceptions, and Opportunities. Journal of Industrial Microbiology and Bio-technology, 46, 281-299. https://doi.org/10.1007/s10295-018-2115-4
- 37. Machado, H., Tuttle, R. and Jensen, P. (2017) Omics-Based Nat-ural Product Discovery and the Lexicon of Genome Mining. Current Opinion in Microbiology, 39, 136-142. https://doi.org/10.1016/j.mib.2017.10.025
- 38. Challis, G. (2008) Genome Mining for Novel Natural Product Discovery. Journal of Medicinal Chemistry, 51, 2618-2628. https://doi.org/10.1021/jm700948z
- 39. Costantini, M. (2020) Genome Mining and Synthetic Biology in Marine Natural Product Discovery. Marine Drugs, 18, Article No. 615. https://doi.org/10.3390/md18120615
- 40. Kenshole, E., Herisse, M., Michael, M. and Pidot, S.J. (2021) Natural Product Discovery through Microbial Genome Mining. Current Opinion in Chemical Biology, 60, 47-54. https://doi.org/10.1016/j.cbpa.2020.07.010
- 41. Jensen, P. (2016) Natural Products and the Gene Cluster Revolu-tion. Trends in Microbiology, 24, 968-977. https://doi.org/10.1016/j.tim.2016.07.006
- 42. Li, L., Jiang, W. and Lu, Y. (2017) New Strategies and Approaches for Engineering Biosynthetic Gene Clusters of Microbial Natural Products. Biotechnology Advances, 35, 936-949. https://doi.org/10.1016/j.biotechadv.2017.03.007
- 43. Albarano, L., Esposito, R., Ruocco, N. and Costantini, M. (2020) Genome Mining as New Challenge in Natural Products Discovery. Marine Drugs, 18, Article No. 199. https://doi.org/10.3390/md18040199
- 44. De La Calle, F. (2017) Marine Microbiome as Source of Natural Prod-ucts. Microbial Biotechnology, 10, 1293-1296. https://doi.org/10.1111/1751-7915.12882
- 45. O’neill, E., Saalbach, G. and Field, R. (2016) Gene Discovery for Synthetic Biology: Exploring the Novel Natural Product Biosynthetic Capacity of Eukaryotic Microalgae. Methods in Enzymology, 576, 99-120. https://doi.org/10.1016/bs.mie.2016.03.005
- 46. Van Der Lee, T. and Medema, M. (2016) Computational Strate-gies for Genome-Based Natural Product Discovery and Engineering in Fungi. Fungal Genetics and Biology, 89, 29-36. https://doi.org/10.1016/j.fgb.2016.01.006
- 47. Ju, K.S., Gao, J.T., Doroghazi, J.R, Wang, K.-K.A., Thibodeaux, C.J., Li, S., et al. (2015) Discovery of Phosphonic Acid Natural Products by Mining the Genomes of 10,000 Actinomy-cetes. Proceedings of the National Academy of Sciences of the United States of America, 112, 12175-12180. https://doi.org/10.1073/pnas.1500873112
- 48. Li, Z., Zhu, D. and Shen, Y.J. (2018) Discovery of Novel Bioactive Natural Products Driven by Genome Mining. Drug Discoveries & Therapeutics, 12, 318-328. https://doi.org/10.5582/ddt.2018.01066
- 49. Mo, T., Liu, W., Ji, W., Zhao, J., Chen, T., Ding, W., et al. (2017) Biosynthetic Insights into Linaridin Natural Products from Genome Mining and Precursor Peptide Mutagenesis. ACS Chemical Biology, 12, 1484-1488. https://doi.org/10.1021/acschembio.7b00262
NOTES
*通讯作者。