Botanical Research
Vol.3 No.03(2014), Article
ID:13611,13
pages
DOI:10.12677/BR.2014.33014
病毒诱导基因沉默(VIGS)在禾本科植物中的 研究进展
Huanhuan Tian1, Rui Qin1, Hong Liu1, Qingyun Liu2, Gang Li1*1Engineering Research Centre for the Protection and Utilization of Bioresource in Ethnic Area of Southern China, College of Life Science, South-Central University for Nationalities, Wuhan
2Hubei Xishui Agricultural Bureau, Huanggang
Email: *lglg863@163.com
Copyright © 2014 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



Received: Mar. 5th, 2014; revised: Apr. 16th, 2014; accepted: May 2nd, 2014
ABSTRACT
Virus-induced gene silencing (VIGS) is a new kind of reverse genetic technique which has developed in recent years. By transferring the recombinant virus vector containing the target gene into host plants to inhibit the expression of endogenous genes of plants, VIGS makes host plants show different phenotypes, such as loss of function or decline in the gene expression of the target gene. VIGS has the obvious advantages over the traditional technology: easy operation, without constructing the transgenic plants, quickly obtaining the phenotype of gene silencing and so on. It has been used in studying functional genomics of plant widely nowadays. This article makes a description of the mechanisms of VIGS, the process of operation, the factors that influence efficiency of silencing, and the application field of VIGS; then, this article will focus on available vectors, progresses and prospects of Gramineous.
Keywords:Virus-Induced Gene Silencing, VIGS Vector, Silence Efficiency, Gramineae

病毒诱导基因沉默(VIGS)在禾本科植物中的 研究进展
田焕焕1,覃 瑞1,刘 虹1,刘清云2,李 刚1*
1中南民族大学生命科学学院,南方少数民族地区生物资源保护与综合利用工程中心,武汉
2湖北浠水县农业局,黄冈
Email: *lglg863@163.com
收稿日期:2014年3月5日;修回日期:2014年4月16日;录用日期:2014年5月2日

摘 要
病毒诱导基因沉默(virus-induced gene silencing, VIGS)是近几年新发展起来的一种反向遗传学技术。它是通过将含有目的基因的重组病毒载体导入到宿主植物中,抑制植物内源基因表达,使其表现出目标基因功能丧失或表达水平下降的表型,属于转录后基因沉默(post-transcriptional gene silencing, PTGS)。与传统的技术相比,VIGS技术具有操作简便、无需构建转基因植株、能快速获得沉默表型等明显优点,现已被广泛应用于植物功能基因组学的研究。本文对VIGS的作用机制、操作过程、影响沉默效率的因素、应用领域各个方面进行阐述,其中会对禾本科植物中的可用载体、研究进展、前景展望进行重点介绍。
关键词
病毒诱导基因沉默(VIGS),VIGS载体,沉默效率,禾本科

1. 引言
目前,对植物功能基因的研究主要是基于基因的超量表达、基因沉默或敲除的原理建立的,其中后者采用的方法主要是辐射、化学诱变、插入突变、RNA干扰(RNA interference, RNAi)等,而且RNAi技术已在禾本科植物中被证实是基因沉默最有效的技术之一,广泛应用于水稻、玉米等作物研究中。常规的RNAi技术在功能基因组研究中,往往耗时耗力、较难得到稳定转化的植株,而瞬时表达的VIGS (virus-induced gene silencing, VIGS)技术可以克服传统技术的某些瓶颈,具有其独特的优势:快速、有效、高通量,无需构建转基因植株,因此VIGS技术在植物功能基因组研究中得到了广泛应用。
VIGS反应原本是植物体内抗病毒侵染的自然机制,可以阻止入侵病毒在植物细胞内的增殖和细胞间扩散。该反应在抑制入侵病毒的同时,也会抑制与插入片段同源的植物内源基因的表达,属于一种PTGS的类RNAi现象,普遍存在于植物体中[1] 。最早是在1990年,Napol和Stuitje分别在矮牵牛植株发现超量表达与粉色色素合成有关的查耳酮合成酶基因(CHS)[2] ;随后在1992年,Romano和Maciano在粗糙脉孢菌中发现了转基因超量表达引起的目的基因的沉默现象[3] ;在1995年,Kumagai等利用烟草花叶病毒(Tobacco mosaic virus, TMV)作载体成功沉默烟草中的八氢番茄红素脱氢酶(Phytoene desaturase, PDS)基因标志了VIGS技术的产生[4] 。从此以后,VIGS技术就逐渐发展为快速高效研究基因功能的技术,尤其是在双子叶植物研究中。目前,该技术在单子叶植物研究中的应用是较为欠缺的,主要原因是单子叶中可以使用的病毒载体较少。而水稻、小麦等主要的粮食作物又多属单子叶中的禾本科,所以将VIGS技术更好的应用于禾本科就显得尤为重要。下面将以禾本科植物为研究对象,从VIGS技术的作用机理、载体的选择、影响效率的因素等方面简要陈述,最后对VIGS技术的在禾本科中的研究进展及前景进行总结。
2. VIGS的作用机制
2.1. 基本诱导机制
VIGS反应的诱导因子同样是双链RNA (double-strand, dsRNA),另外还需要两种关键酶:依赖于RNA的RNA聚合酶(RNA-dependent RNA polymerase, RDRP)和类Dicer酶(Dicer-like, DCL),其中RDRP负责引导重组病毒的RNA形成dsRNA,DCL是一种RNaseIII酶,作用与动物体内RNAi中Dicer酶相同,都是对dsRNA进行切割。
当含有植物目标基因的重组病毒载体导入植物体后,先合成转基因的RNA,然后在RDRP酶作用下复制成dsRNA,继而引发PTGS反应。DCL酶识别dsRNA并将其切割成21~23 nt的小干扰RNA (short-interfering RNA, siRNA),基因沉默复合体(RNA-induced silencing comple, RISC)与siRNA结合,其中的RNase将双链siRNA降解为单链siRNA,然后RISC通过单链的siRNA对植物的mRNA进行扫描,找到互补序列并特异性降解,从而使植物体内的该基因在mRNA水平发生沉默[5] -[7] 。
以上所述是VIGS反应的基本机制(图1),但是这种反应往往具有局限性,仅在病毒侵染的特定部位起作用。若以特异性的单链siRNA为模板,通过一定的途径合成双链siRNA,这样双链siRNA又能产生更多的活性单链siRNA,可以进一步增强目标基因的沉默效果;若siRNA在植物体内扩增后转移,就会引起远距离的目标特定基因沉默,甚至系统性地引起植物体全身各部分的基因沉默[8] 。
2.2. 不同沉默载体间的差异
目前,绝大多数的VIGS体系都是建立于本氏烟(Nicotianan benthamiana),进而推广应用于其他物种。而开发出来的沉默载体中主要是RNA病毒,少数为DNA病毒,以及基于两者的卫星载体,这三类载体的诱导机理又略有不同。
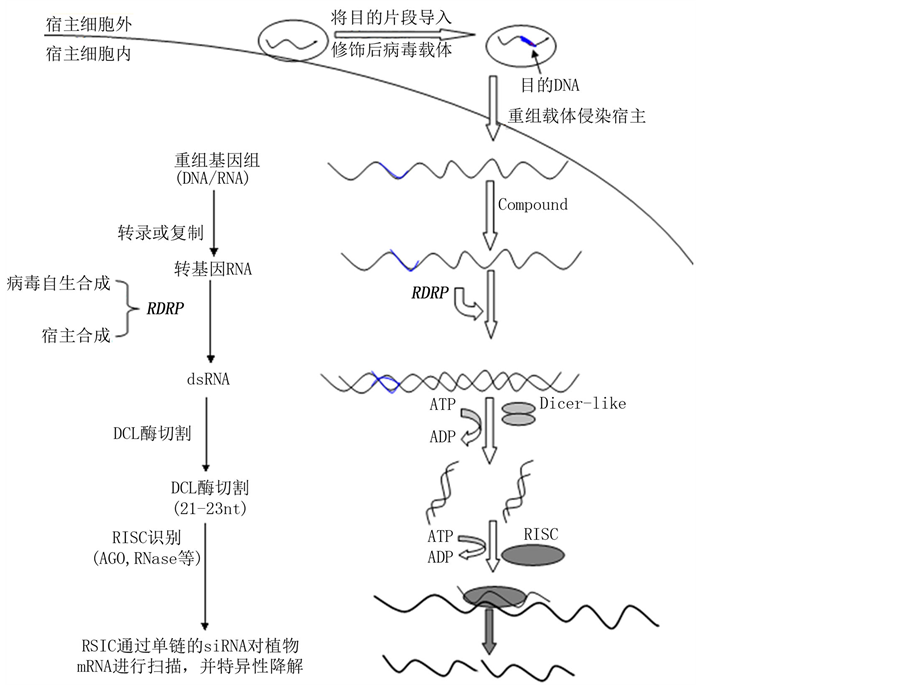
Figure 1. The basic induction mechanism of VIGS
图1. VIGS的基本诱导机制
2.2.1. RNA沉默载体
VIGS体系中,应用的RNA病毒载体绝大多是(+)ssRNA,像马铃薯X病毒(Potato virus X, PVX),豌豆早枯病毒(Peaearly browning virus, PEBV),黄瓜花叶病毒(Cucumber mosaic virus, CMV),烟草花叶病毒(Tobacco mosaic virus, TMV),烟草脆裂病毒(Tobacco rattle virus X, TRV),雀麦草花叶病毒(Brome mosaic virus, BMV),大麦条纹花叶病毒(Barley stripe mosaic virus, BSMV)等。其中,最早被开发利用的是TMV 和PVX 载体,但这两种病毒对宿主的致病症状较重,对基因沉默的时间短、效率低,且对顶端分生组织无效[9] -[14] 。在禾本科VIGS的研究中,BMSV是最早应用的病毒载体[12] ,此外可用的RNA病毒载体还有BMV。
当重组的(+)ssRNA 载体进入植物体后,会在类囊泡结构中利用病毒自身编码的RDRP酶先形成(−)ssRNA,然后在宿主体内的内质网同源蛋白(Reticulon homology proteins, RHPs)作用下复制成为dsRNA,进而进行PTGS过程。其中的RHPs是病毒在植物细胞中复制的起始因子,当病毒进入宿主后与RHPs相互作用,在宿主膜结构上形成病毒复制复合体(Viral replication complexes, VRCs),来保证重组(+)ssRNA的成功复制与作用[15] [16] 。
2.2.2. DNA沉默载体
应用于VIGS体系中的DNA病毒载体有番茄金色花叶病毒(Tomato golden mosaic virus, TGMV)、非洲木薯花叶病毒(African cassava mosaic virus, ACMV)、大白菜曲叶病毒(Cabbage leaf curl virus, CbLCV)、棉花皱叶病毒(Cotton leaf crumple virus, CLCrV)、水稻东格鲁杆状病毒载体(Rice tungro bacilliform virus, RTBV)、中国番茄黄化曲叶病毒(Tomato yellow leaf curl China virus, TYLCCV)等[17] -[22] ,其中双链的RTBV已成功应用于水稻,其他均为单链环状的(+)ssDNA。
DNA病毒自身是没有RDRP基因,当(+)ssDNA进入植物体后,先利用宿主体内的DNA聚合酶形成dsDNA,在病毒产生的复制起始蛋白Rep作用下,进行滚环复制(Rolling-circle Replication, RCR),形成大量的ssDNA的拷贝[23] 。当这些重组的ssDNA转录后只能利用植物宿主的RDRP酶合成dsRNA,进而诱导PTGS过程。目前在水稻中发现的可用于VIGS的载体RTBV是dsDNA,它的具体作用机制未见报道,猜测可能是重组的dsDNA直接进行RCR形成ssDNA,在进行以后的过程。
2.2.3. 卫星载体
DNA和RNA两类病毒载体作用过程中可能会需要卫星载体和协助载体来诱导远距离的表型症状[20] 。在基于RNA病毒的VIGS体系中,TMV的卫星RNA和协助RNA发现于本氏烟中,并证实了两者共同作用可能比卫星RNA单独作用的沉默表型更明显,常被称为卫星病毒诱导的沉默系统(satellite virus-induced silencing system, SVISS)[24] 。此外,TYLCCNV的伴随卫星分子DNAβ构建成的DNAmβ载体,已用于烟草属多个种和番茄等多种茄科植物,且被证实TYLCCNV通过根吸收法有利于对功能基因进行高通量分析[21] [25] [26] 。目前在禾本科植物研究中还未发现可用的卫星载体。
SVISS体系和DNAmβ也常被称作卫星载体,两者诱导基因沉默的具体作用机制不清,猜测可能是病毒由于基因组缺失先形成异常的(−)ssRNA或mRNA,再通过协助RNA的作用合成dsRNA,进而诱导植物发生基因沉默;此外还有可能是在周围辅助病毒和宿主特定的作用下来完成诱导过程。
2.3. VIGS的抑制
自然界中很多植物病毒自身含有编码抑制蛋白的基因,其合成的蛋白可抑制某些宿主RNA的沉默。在VIGS体系中,当大量抑制蛋白存在时,病毒的积累量减少,目的基因的沉默效果就会降低;而抑制蛋白只有在病毒大量复制时才会合成,所以在VIGS体系中只会影响病毒积累而不会完全抑制沉默效率。如水稻黑条矮缩病毒(Rice black streaked dwarf virus, RBSDV)作用于本氏烟的研究中,发现其编码的S6蛋白就能强烈抑制本氏烟中绿色荧光蛋白(green fluorescent protein, GFP)基因沉默[27] ,但是这种结果在水稻等禾本科植物中还未证实。
来源于不同病毒的沉默抑制蛋白,它们的抑制机理又存在差别。其中,来源于CMV的抑制蛋白CmvZb和TavZb,可以抑制沉默信号的系统性转移,使新长出的叶片沉默效果逐渐减弱,另外来源于番茄丛矮病毒(Tomato bushy stunt virus, RBSV)的P19蛋白与CmvZb作用机制相同[28] ;来源于马铃薯的HC-pro可抑制转GFP基因植株中的目的基因沉默,实验中发现HC-pro是通过抑制siRNA (21~25 nt)的积聚来影响沉默表型,但并不影响沉默信号的产生和系统性转移[29] ;来源于PVX的抑制蛋白P25可能是通过与产生系统性信号的上游作用而抑制沉默[30] 。
3. VIGS体系的整体流程
在对功能基因进行研究时,VIGS技术的基本操作过程是一致的。首先需要准确找到目标基因,选择最佳长度的基因片段;再选择合适的病毒,并构建重组病毒沉默载体;然后将重组载体导入到宿主;沉默处理后特定条件下培养宿主植物;最后对沉默表型和基因功能进行鉴定。下面将以禾本科植物中为例阐述该流程,并侧重介绍禾本科中载体的选择、已使用的载体的构建、以及不同重组载体侵染宿主的方法。
3.1. 目的片段的选择
插入载体中的序列与目的序列至少应该有23 bp完全一致,且同源性越高沉默效果就会越好[31] 。插入片段的长度也是有限制的,一般不能超过1.5 kb,片段太长会降低其稳定性,且会阻碍载体在宿主中移动,影响沉默效果,以300~500 bp间最合适[32] 。如果对某个基因家族进行研究时,应该选择该基因家族的保守序列;而当研究家族中某特定基因时,就应该选择该基因的非保守区域[32] [33] 。
3.2. 病毒载体的确定
用于VIGS技术的病毒载体应该满足的条件:既易感于宿主又易接受外源基因,能在植物体内复制增殖,可以系统性浸染宿主植物,且病毒子代最好可以均匀分布在器官内的各个组织;决定病毒载体浸染、复制、转移等必不可少的基因序列最好可以集中在较小的范围内。这些条件限制了载体的选择,致使VIGS技术在禾本科植物中推广受阻。与双子叶植物VIGS体系中可用病毒载体相比,单子叶植物中可用的病毒载体很少,主要有BSMV、BMV、RTBV和兰花花叶病毒(Cymbidium mosaic virus, CymMV)[20] ,而目前只有前三种载体在禾本科作物中得到应用。
BSMV是含α、β、γ三部分基因组的ssRNA病毒,其重组的沉默载体已经应用于大麦、小麦、水稻、玉米等多数单子叶植物中VIGS体系,还包括最近已经测序完成的谷类模式植物二穗短柄草,以及沉默效果不太好的燕麦属(一般实际中不用)。BSMV的宿主除了禾本科外,还包括热带作物姜(Zingiber officinale)以及很有研究前景的香蕉(Musa acuminata)和车前草(M. xparadisiaca)[20] 。该载体以往主要是用于沉默植物地面上的器官中的功能基因,现已在大麦中被证实了也可以作用于其根部组织,但是并不能说明可以作用于所有的根部组织中,因为沉默效果会受插入病毒基因组中的目的基因稳定性影响[34] 。另一种可用的RNA病毒载体BMV也是有RNA1、RNA2、RNA3三部分,现已用于大麦、水稻、栽培玉米Va35等植物中,成功完成了对目的基因的沉默;另外该载体在玉米中也可以用来研究活体营养寄生物的相互作用,例如在宿主与病原体互作中,通过玉米本身携带的真菌玉蜀黍黑粉菌(Ustilago maydis)来抑制宿主基因的表达[20] 。在水稻中发现的dsDNA病毒载体RTBV,目前仅应用于水稻。通过农杆菌转化法将重组载体导入水稻中,可以抑制处理过的水稻叶片中的PDS基因,效率高达90%[22] 。最近研究者又在蝴蝶兰(Phalaenopsis spec.)中运用对其无毒性的CymMV成功完成了对目标基因的沉默[20] 。
3.3. 构建沉默载体
在构建沉默载体时,应该尽量降低病毒本身对宿主的病毒害作用,除去非必须的基因片段,加入必需的反式作用序列、宿主和病毒识别的序列、合适的多克隆位点、tRNA结合位点等,必要时加入泛素化的启动子来保证组成型表达。下面以禾本科植物的三种载体为例简单介绍其构建方法。
BSMV构建的载体多是利用修饰过的RNAβ、RNAγ来进行重组,Holzberg 等通过对BSMV的ND18品系进行修饰,包括除去RNAβ中外壳蛋白基因,在γb基因的下游加入多克隆位点(Pac I、Not I)[12] ;Bruun-Rasmussen等则是在γb基因的下游插入含不同限制酶酶切的位点(Sma I、Pac I、Bam HI)的序列,但保留RNAβ中的外壳蛋白基因,结果在感染的大麦叶片上叶片病症较轻[35] ;后来Meng等用Holzberg构建的BSMV中的α、β、γ三部分的cDNA分别插入到单独的双亲载体中(35s启动和核酸酶终止子之间),其中β部分是利用的是含有活性外壳蛋白基因的cDNA[36] 。Campbell等将β、γ上分别携带两种不同的基因,用此重组载体浸染大麦,结果这两个基因同时沉默[37] 。另外BSMV载体沉默时间短,且稳定沉默仅发生在前2~3个感染的叶片中,实际试验中往往将PDS基因与目的基因一起导入到载体中来,以PDS作报告基因[38] 。
BMV病毒是研究(+)ssRNA的模式病毒,主要有RNA1,RNA2,RNA3三部分组成,其中RNA1编码一个单顺反子的ORF,转录的蛋白质与RNA的甲基化、N-端加帽活性、C-端RNA解旋酶区域有关;RNA2编码一个单顺反子的ORF,翻译出的蛋白与RDRP同源;RNA3却编码双顺反子的ORF,编码运动蛋白(MP)和外壳蛋白(CP)作用于系统性病毒感染。有两品系F-BMV(Fescue-BMV)和R-BMV (RussianBMV),Ding等在构建时使用F-BMV的RNA1、RNA2和R-BMV的RNA3的组合体,前者是其易于感染宿主,后者中含有唯一的HindIII酶切位点使其易于接受外源基因。操作中F-RNA为基础,RNA1、RNA2不作处理,而RNA3用R-BMV中RNA3内的外壳蛋白和运动蛋白之间的序列替代,并将外源片段插入到R-RNA3外壳蛋白终止子的下游这样可以增加嵌合体RNA3的积累[39] 。这种含有嵌合RNA3的BMV载体(C-BMV RNA3)作用效果更好,比F-BMV增殖快而比R-BNV致病性低[40] 。随后又被改进,将RNA1、RNA2和嵌合的RNA3导入到处理后的双亲载体pCAMBIA的35s启动子和核酸酶终止子间[41] 。之前较早时还有一种构建方法,也是Ding构建的载体的起源。将R-BMV中RNA3的外壳蛋白ORF的3端的更换两个限制酶,这两个酶切位点间有337-nt的空间来方便外源基因的插入[42] 。
RTBV病毒是一种具有dsDNA的逆转录病毒,其DNA全长约8 kb编码4个开放阅读框(ORFs I-IV),其中III负责编码有外壳蛋白、天冬氨酸蛋白酶和转录核糖核酸酶H组成的多聚蛋白,其他ORF功能现在还未鉴定出来[43] 。Purkayastha等构架载体时,直接把ORFs I、II全部去除,将组织特异性的启动子换成了玉米泛素化的启动子,并引进MCP、tRNA结合位点。通过农杆菌法将重组载体导入水稻时,结果发现叶片中出现白色条纹[22] 。
3.4. 沉默载体导入宿主的方法
浸染过程中,不同的浸染方式会影响沉默反应的初始底物浓度,进而影响沉默的效率。所以应该根据不同的载体、不同的要求选择最适方法。当用TRV载体浸染烟草和番茄时多采用真空渗透和高压喷射法,但若只想在其局部浸染且取得较好的效果应采用注射浸润法[44] 。目前根据已经报道的文章,可以总结出三大类:摩擦接种法、农杆菌介导转化法、微粒轰击法。
摩擦接种法主要用于RNA病毒载体,它的浸染液多是采用体外转录或从已感染的叶片中提取的病毒载体RNA,再通过牙签或者是石英砂处理待浸染的叶片。这种方法的沉默效果还是很好的,尤其是对不能用其他方法的植物(拟南芥等)[45] 。农杆菌转化法可以介导DNA和RNA病毒载体对宿主的浸染,先将含有宿主目的片段的VIGS载体导入到农杆菌中,再通过农杆菌液对植物进行接种。在该方法中使用的是根癌农杆菌(Agrobacterium tumefaciens),具体操作又可以分为真空渗透法、高压喷射法、注射器浸润法、灌根法,牙签穿刺法。其中,灌根法是最简单易行的方法, 但沉默效率较低、适用范围较小(本氏烟、番茄个别品种)[46] ;注射器浸润法适用于只能通过韧皮部浸染的DNA病毒(如RTBV作用于水稻)[20] ;注射器浸润法和牙签穿刺法工作量大,沉默效率低,不适于大规模操作;而真空浸润法却可以使TRV在番茄上获得100%沉默效率,可高通量、大规模操作[47] 。微粒轰击法,即使用基因枪通过金粉等金属粒子的轰击作用将重组载体导入宿主中,多用于DNA病毒载体,已经成功应用于TGMV[48] ,CbLCV[18] 及ACMV[49] ,后来又证实了对BSMV也适用[36] 。
就上述用于单子叶植物的三种载体来说,浸染方法也不尽相同。BSMV、BMV载体浸染宿主时只需对种子进行摩擦就可以感染,而RTBV则采用农杆菌介导的注射法。BSMV载体的浸染液是将含有目的基因序列的重组质粒线性化后,在体外转录,然后加入FES缓冲液[12] [38] ,通过牙签、石英砂对种子摩擦进行接种[12] [50] ,这种方法效果较好,但是操作复杂,且体外转录酶较昂贵。最近,Meng等通过微粒轰击的方法将含BMSV重组基因的载体成功导入了宿主中[36] ,这种基于基因枪的转化方法无需体外转录酶,但是需要购买基因枪和金粉颗粒。BMV载体也可运用BSMV中的方法对种子进行摩擦接种,先在体外对线性重组质粒转录,再加入EFS缓冲液直接接种[12] ;若重组BMV载体以DNA的形式浸染宿主时,则可以用农杆菌介导的真空渗透法将重组载体导入水稻,目前这种方法已成功用于水稻、高粱、谷子,但并非是其他植物的最佳方法[40] 。另外,摩擦接种的方法得到了改进,可以先用BMV载体对本氏烟进行免疫,从中介植物感染的组织中提取汁液作为浸染液,再对宿主进行接种,这样浸染宿主的载体沉默效果更高效[42] 。
3.5. 处理后植物的培育
由于处理后植物的培育条件会直接影响病毒的增殖、转移和植物的生长,而不同的沉默载体、不同的植物间的最适条件又不同,所以需要对分别对宿主、沉默载体的最适条件进行实验调查,其中主要是温度调查。然后进行VIGS研究时可以用植物培养箱来严格控制温度、湿度、光照等条件。
在单子叶植物的研究中已经证实,低温可以增强沉默表型。以BMV作用小麦的实验中发现18-22℃时沉默效果最好,对目标基因mRNA的降解力比26℃时强[50] ;在大麦中20℃~24℃比16℃、28℃的沉默效果好[35] ;另外,在研究兰花环斑病毒(cymbidium ringspot virus, CymRSV)时,发现24℃以下温度可以抑制植物对CymRSV基因的沉默,其原因是由于该条件抑制了siRNA的产生和积累[51] 。
3.6. 沉默效果的鉴定
当培育到一定时间后,我们需要对植物中目的片段的沉默效果进行鉴定,最常用也是最简单的方法就是通过观察表型的变化。如PDS沉默会出现光漂白现象[22] 、绿色荧光蛋白GFP沉默时绿色荧光会消失[27] ,这两者也常作为报告基因与目的片段偶联导入沉默载体中,根据宿主中表型的改变来推断目的基因是否被成功沉默;若目的基因是抗病相关基因也可以通过抗病性变化来判断。此外,也可以通过对目的基因进行定量分析来检测沉默效果,反转录PCR (reverse transcription PCR, RT-PC)可进行半定量检测[22] ,实时定量PCR (Real time-PCR)更精确些[35] [52] ,但是两者都是可以显示出PDS、GFP等基因mRNA的降解程度。
4. VIGS沉默效率的影响因子
VIGS体系中的几乎每个过程都可以影响沉默效率,但是主要集中在以下几个方面:合适的重组载体、外源基因片段的插入方向、重组载体中插入片段的稳定性、重组病毒载体的接种方法、处理后植物的培育温度。
4.1. 构建合适的载体
虽然基于BSMV、BMV沉默载体已经在部分禾本植物中得到了重大进展,但还未达到最佳的沉默效果,而且在其他的禾本植物中的有效性还未鉴定出来。原因可能是病毒对宿主的感染性不好、复制受阻或者是在宿主内的转移受阻。小麦中,栽培种“Bobwhite”表现出更强的光漂白现象,且22℃时对目标基因的沉默水平比“Clark”强[50] 。目前所有实验中,并没有可以表现完全沉默的结果,因此应该是存在比目前更好的沉默效果。而在后来的研究中发现由于BSMV载体表达了GFP基因的部分片段,在小麦栽培种Renan中引发了与植物防御系统相关的基因表达,研究者推断出这些基因的表达与稻瘟病菌的抗性相关,而与小麦的白粉病的发生无关[53] 。水稻中,BMV可以在栽培稻IR64、Drew和Cypress中会引起中度花叶症状,在栽培稻Moroberekan和IAC165中引起严重症状、甚至会引起也变得局部坏死,但却没有BMV可以感染日本粳稻(Nipponbare) [40] ;另外,BMV在玉米的多个栽培品种中会引起严重的系统性坏死,所以不适合做其宿主植物[20] 。也就是说病毒可以浸染目标植株并不代表适用于该植物中的VIGS体系。
4.2. 插入片段的方向
多数目的基因片段反向插入到重组载体中的带来的沉默效果优于正向插入的效果[35] [45] [54] 。早期的研究中发现,在BSMV载体中插入反向重复的目标序列可以增强沉默的表型或者是下调目的基因的表达量[54] 。然而,近期实验者在BSMV、BMV载体介导的VIGS体系中发现:这些短的反向重复序列非常不稳定性、沉默的有效性不如正向插入的片段[34] ,所以需要进一步对禾本科植物沉默体系进行研究,来确定反向重复序列的普遍有效性。
4.3. 重组载体中插入片段的稳定性
维持重组载体中插入片段的稳定性也是获得良好沉默效果的重要因素,其稳定性与siRNA的产生、目标mRNA的沉默和沉默表型的维持均有密切关系[35] [50] 。研究者利用TRV在本氏烟中沉默的基因,很少可以在子代该组织以及相关组织的所有细胞中出现沉默表型,这些表型的出现多与插入片段的稳定性密切相关[55] 。然而在禾本科植物中,VIGS在系统性感染的组织中多为瞬时表型,仅有2~3个叶片或者茎组织间出现沉默表型;而且叶片中出现的沉默表型多为不完全的斑点状,在纵向的维管束中表型为或大或小的条纹。这些瞬时、不完全、可观察的沉默表型一般与外源基因的部分丢失相关联[35] 。而外源基因片段的丢失机制还未完全破解,可能的原因有RNA聚合酶活性引起的插入片段的缺失、RNAs间的重组,这也就引起了宿主植物表现出复杂多样的表型[56] 。
而插入片段的稳定性受片段大小、核酸组成的影响[35] [54] ,这也可以解释两个基因同时插入载体中沉默效果不好的原因[50] 。另外,插入片段的稳定性受宿主和病毒因素共同影响,因此在宿主间沉默的结果是多样的。目前,研究者多致力于研发可大量增殖的病毒载体、有更好沉默表型的宿主,而插入序列的稳定性研究还处于早期阶段。
4.4. 重组病毒载体的接种方法
农杆菌介导的VIGS最早用于本氏烟实验中,研究者通过对其优化,成功用于茄科、莴苣、拟南芥、番茄中并得到了稳定的沉默结果[46] [57] 。目前在禾本科中,一些DNA病毒通过这种方法已成功应用于水稻、玉米、小麦中。在2009年,第一次报道了农杆菌介导的真空浸润法成功用于水稻中[41] ,对栽培稻IR64进行真空浸润产生的沉默效果比机械接种的得到的效果更强、持续时间更长[40] 。
机械接种过程中,需要烟草等作为中介宿主植物,操作较复杂。先将重组BMV载体导入到烟草中,一定时间后从中提取病毒RNA的转录产物,然后通过摩擦接种到目标植物中。而农杆菌介导的转化中,需要体外转录酶,费用较高。其中含有重组载体的接种液是线性化的重组质粒在体外转录得到的,再经真空浸润法导入到水稻中。另外,微粒轰击法也是可以选择的方法,但是需提前购买基因枪和金粉。
4.5. 处理后植物的培育温度
在双子叶植物中已经证实了:低温可以增强沉默的表型,高温易引起病毒基因组的沉默[32] ,原因可能是低温利于不断积累病毒载体而高温会破坏病毒。对BMV实验研究中发现,低温条件(24℃/20℃,白天/晚上)时,病毒的浸染和积累主要发生在大麦维管束细胞内及附近细胞,这一结果提示了控制沉默条件是很重要的。因此,温度影响多用于分析新的植物–病原体组合[20] 。
另外,选择的宿主植物所处的生长期也可以影响沉默效率。所有的宿主植物多为较小的苗期,此时重组载体较易侵入,Purkayastha等实验中所用的水稻为生长了15天的幼苗[22] 。
5. VIGS技术的应用进展
近几年,病毒诱导基因沉默(VIGS)的研究在各个方面都有了很大的突破,人们逐渐发现VIGS技术独特的研究优势:快速、有效、高通量,不需要耗时耗力得到转基因植株,往往在1~2周就可以表现出沉默表型;可以通过将某基因的最保守序列整合进重组载体中,来对基因家族中的单个基因进行研究,反之也可以共沉默同源基因;可以在植物的苗期进行突变致死基因的功能研究;可以通过含有同源基因片段的重组载体感染不同物种,来对不同物种间进行比较基因组的研究[58] ;还可以在未知全基因序列的条件下对该基因进行VIGS分析研究,另外还可以通过将cDNA文库中的片段随机的插入到载体中进行高通量的分析[59] 。也正是这些优势使其受到越来越多研究者的青睐,目前应用最多的领域是在植物的功能基因研究中,包括植物抗病机制的研究、抗旱信号途径[60] 、光合作用途径[61] 、无义介导的mRNA的降解(Nonsense-mediated mRNA decay, NMD)[62] 等方面,最近在动植物(寄宿主间)互作方面也展开了研究[20] 。
由于VIGS技术可以特定地诱导植物体内各个部位基因的沉默,因此常作为高效的反向遗传技术应用于植物体的功能基因研究中,尤其是在某些抗病基因功能、抗病信号路径的研究中发挥着越来越重要的作用。以BSMV为载体成功诱导大麦叶片中PDS基因的沉默,开辟了VIGS技术用于禾本科植物功能基因研究的先河。BMSV载体在使用中往往表现出沉默时间短、有效沉默仅发生在前2~3个感染的叶片中,所以可以将宿主的PDS基因也一起导进载体中,这样就可以通过光漂白现象来作为沉默的报告基因,而PDS的mRMA的降解情况可用RT-PCR进行检测。目前BSMV已经发展为大麦、小麦中VIGS的稳定载体,已广泛用于抗病基因相关研究,包括小麦中的叶锈病抗性基因(Lr21/Lr10)[63] [64] 、茎锈病抗性基因(TaRLK1/2/3)[65] 、丝氨酸/苏氨酸蛋白激酶基因(Stpk-V)[66] 、条纹病真菌抗性基因(TaHsp90.2、TaHsp90.3)[67] ;大麦中的白粉病抗病基因(Hsp90)[68] 、Blufensin1 (Bln1)[36] 、WRKYs1/2[69] 、编码阳离子交换蛋白HvCAX1的NecS1基因[70] ,及大麦中其他非宿主抗病基因。另外,BMSV载体也成功用于小麦中抗虫基因的研究(蚜虫抗性基因WRKY53和Pal)[71] ,种子萌发相关基因(TaHsp90.1)[68] ;大麦中有关细胞壁代谢基因(P23k、CesA)[72] [73] 、有关磷酸盐吸收的根尖基因(IPS1、PHR1、PHO2)[34] 。后来发展的可用于禾本科BMV载体也成功用于以下研究:玉米中与玉蜀黍黑霉菌相互作用的基因Tps6/11、Ecb、Bti,其中沉默Tps6/11基因可以增强组织对玉米真菌病原体的易感性[74] 。最近报道,BMV载体可以沉默玉米中的m-型的硫氧还蛋白[75] 。而且,这些利用BMV载体使基因沉默表达的组织比甘蔗花叶病毒(Sugarcane mosaic virus, ScMV)感染系统更容易得到[20] 。
6. VIGS技术在禾本科中的前景展望
水稻、小麦、玉米等重要的粮食作物都是禾本科,怎样将VIGS技术更好的应用于农业生产中也就成了重要的课题。但由于VIGS技术在禾本科中还不成熟,不能特异性沉默基因的表达;就算在已经成功应用的植物品种中,也需要改进操作方案来获得更高沉默效率。另外,开发新载体、非转基因性状的可遗传性也将会是未来的研究热点。
VIGS技术在禾本科植物功能基因组研究方面虽然取得了重大的进展,但是目前禾本科中应用的病毒载体,多数是仅能在宿主叶片中瞬时表达,并不能表现出最佳沉默效果。所以,我们可以将现有的BSMV和BMV载体进行修饰,使其能够在35s启动子后更易连接目的片段、更易表达,这样就可以使操作简便、节省时间[13] [36] 。除了对现有载体进行修饰、在已知的ESTs数据库中选择最适目标片段,我们还可以提高目的片段在重组载体中的稳定性。这样就需要对现有载体病毒的DNA/RNA结构、顺式/反式作用因子进行分析,希望能得到更好地维持插入片段稳定性的载体。
在现有载体的基础上开发新载体也是今后重要的研究领域。目前新发现的绝大多数载体都会诱发宿主植物严重症状,我们还需要致力于研发通过昆虫和种子不能传递的新型病毒载体,这样就可以在较宽松的条件下进行研究。例如,黍花叶病(Panicum mosaic virus, PMV)是一种ssRNA,可以引起多数单子叶植物轻微的花叶症状,通过机械接种可以在植物间转移,而通过昆虫载体和种子不会进行传递[76] ;X.S. Ding等基于该病毒的特点,成功构建出的PMV载体可以允许外源基因表达,但作为沉默载体的有效性还在研究中[20] 。此外,Tatineni等构建出小麦条纹花叶病毒(Wheat streak mosaic virus, WSMV)的一系列载体,这些载体可以在大麦、小麦、玉米等多种禾本科植物中表达GFP基因;但是由于该病毒会编码一种RNA沉默的抑制蛋白,WSMV载体还需要进行仔细的评估[77] 。所以目前为止,对现有载体进行优化是提高禾本科植物VIGS效果的最有效方法。
除了功能基因的研究,VIGS技术也经常用于植物–病原体的相互作用和植物发育领域的研究。尽管昆虫学家和植物学家不常用VIGS技术来研究植物–昆虫间的相互作用,但是也都意识到了VIGS具有的巨大潜力,所以这方面必将会成为VIGS技术研究的另一热点。用VIGS技术来研究植物–昆虫相互作用最早是有Stratman研究组提出并应用于番茄与M. sexta的研究中[78] ,得到与Wu等[79] 相同的结论:番茄对M. sexta幼虫的抗性是通过调节JA的生物合成来影响MAPKs的结果。另外,在黑芥(Brassica nigra)和拟南芥研究中,通过VIGS技术来研究菜粉蝶(Pieris rapae)在其宿主植物叶片上的产卵行为[80] 。这是VIGS技术首次用于生态领域,来研究昆虫和植物的相互作用。这一研究思想也可应用于水稻、小麦等禾本科植物的中的研究中,进而研究寄宿主间的相互作用。VIGS在生态领域的应用也将会成为未来研究的重要领域。
项目基金
湖北省自然科学基金重点项目BZZ11001。
参考文献 (References)
- Baulcombe, D. (1999) Viruses and gene silencing in plants. Archives of Virology, 15, 189-201.
- Napoli, C., Lemieux, C. and Jorgensen, R. (1990) Introduction of a chemeric chalcone synthase gene into petunia results in reversible cosuppression of homologous genes in trans. The Plant Cell, 2, 279-289.
- Romano, N. and Macino, G. (1992) Quelling: Transient inactivation of gene expression in Neurospora crassa by transformation with homologous sequences. Molecular microbiology, 6, 3343-3353.
- Kumagai, M.H., Donson, J., Della-Cioppa, G., et al. (1995) Cytoplasmic inhibition of carotenoid biosynthesis with virus-derived RNA. Proceedings of the National Academy of Sciences of the USA, 92, 1679-1683.
- Ding, S.W. and Voinnet, O. (2007) Antiviral immunity directed by small RNAs. Cell, 130, 413-426.
- Waterhouse, P.M. and Fusaro, A.F. (2006) Viruses face a double defense by plant small RNAs. Plant Science, 313, 54-55.
- Hammond, S.M., Bernstein, E., Beach, D., et al. (2000) An RNA-directed nuclease mediates post transcriptional gene silencing in Drosophila cells. Nature, 404, 293-296.
- Kalantidis, K.S.H., Alexiadis, T. and Helm, J.M. (2008) RNA silencing movement in plants. Biological Cell, 100, 13- 26.
- Purkayastha, A. and Dasgupta, I. (2009) Virus induced gene silencing: A versatile tool for discovery of gene functions in plants. Plant Physiology Biochemistry, 47, 967-976.
- Robertson, D. (2004) VIGS vector for gene silencing: Many targets many tools. Annual Review of Plant Biology, 55, 495-519.
- Cakir, C., Gillespie, M.E. and Scofield, S.R. (2010) Rapid determination of gene function by virus-induced gene silencing in wheat and barley. Crop Science, 50, 77-84.
- Holzberg, S., Brosio, P., Gross, C., et al. (2002) Barley stripe mosaic virus-induced gene silencing in a monocot plant. The Plant Journal, 30, 315-327.
- Pacak, A., Strozycki, P.M., Barciszewska-Pacak, M., et al. (2010) The Brome mosaic virus-based recombination vector triggers a limited gene silencing response depending on the orientation of the inserted sequence. Archives of Virology, 155, 169-179.
- Ruiz, M.T., Voinnet, O. and Baulcombe, D.C. (1998) Initiation and maintenance of virus induced gene silencing. The Plant Cell, 10, 937-946.
- Noueiry, A.O. and Ahlquist, P. (2003) Brome mosaic virus RNA replication: revealing the role of the host in RNA virus replication. Annual Review of Phytopathology, 41, 77-98.
- Rao, A.L.N. (2006) Genome packaging by spherical plant RNA viruses. Annual Review of Phytopathology, 44, 61-87.
- Peele, C., Jordml, C.V., Muangsan, N., Turnage, M., Egelkrout, E., et al. (2001) Silencing of a meristematic gene using geminivirus-derived vectors. The Plant Journal, 27, 357-366.
- Turnage, M.A., Muangsan, N., Peele, C.G. and Robertson, D. (2002) Geminivirus-based vectors for gene silencing in Arabidopsis. The Plant Journal, 30, 107-114.
- Kjemtrup, S., Sampson, K.S., Peele, C.G., Nguyen, L.V., Conkling, M.A., et al. (2009) Gene silencing from plant DNA carried by a Geminivirus. The Plant Journal, 14, 91-100.
- Becker, A. (2013) Methods and protocols: Virus-induced gene silencing. Plant Evodevo Group, Justus-Liebig-Universität Gießen, Germany.
- Tao, X.R. and Zhou, X.P. (2004) A modified viral satellite DNA that suppresses gene expression in plants. The Plant Journal, 38, 850-860.
- Purkayastha, A., Mathur, S., Verma,V., Sharma, S. and Dasgupta, I. (2010) Virus-induced gene silencing in rice using a vector derived from a DNA virus. Planta, 232, 1531-1540.
- Gutierrez, C. (2002) Strategies for geminivirus DNA replication and cell cycle interference. Physiological and Molecular Plant Pathology, 60, 219-230.
- Gosselé, V., Faché, I., Meulewaeter, F., Cornelissen, M. and Metzlaff, M. (2002) SVISS—A novel transient gene silencing system for gene function discovery and validation in tobacco plants. The Plant Journal, 32, 859-866.
- Xu, Y.P., Zheng, L.P., Xu, Q.F., Wang, C.C., Zhou, X.P., et al. (2007) Efficiency for gene silencing induction in Nicotiana species by a viral satellite DNA vector. Journal of Integrative Plant Biology, 49, 1726-1733.
- 张召军, 王晓彬, 王慧, 等 (2014) 中国番茄黄化曲叶病毒利用根吸收法诱导基因沉默(VIGS)的初步研究. 生物技术通报, 1, 143-146.
- 张凌娣, 王朝辉, 王献兵, 等 (2005) 两种植物病毒编码蛋白的基因沉默抑制子功能鉴定. 科学通报, 3, 219-224.
- Brigneti, G., Voinnet, O., Li, W.X., Ji, L.H., Ding, S.W., et al. (1998) Viral pathogenicity determinants are suppressors of transgene silencing in Nicotiana benthamiana. The EMBO Journal, 17, 6739-6746.
- Mallory, A.C., Ely, L., Smith, T.H., Marathea, R., Anandalakshmi, R., et al. (2001) HC-pro suppression of transgene silencing eliminates the small RNAs but not transgene methylation or the mobile signal. The Plant Cell, 13, 571-583.
- Voinnet, O., Lederer, C. and Baulcombe, D.C. (2000) A viral movement protein preventes spread of the gene silencing signal in Nicotiana benthamiana. Cell, 103, 157-167.
- Thomas, C.L., Jones, L., Baulcombe, D.C. and Maule, A.J. (2001) Size constraints for targeting post-transcriptional gene silencing and for RNA-directed methylation in Nicotiara benathamiana using a potato virus X vector. The Plant Journal, 25, 417-425.
- Burch-Smith, T.M., Anderson, J.C., Dinesh-Kumar, S.P. and Martin, G.B. (2004) Applicntions and advantages of virus-induced silencing for gene function studies in plants. The Plant Journal, 39, 734-746.
- Lu, R., Martin-Hernandez, A.M., Peart, J.R., Malcuit, I. and Baulcombe, D.C. (2003) Virus-induced gene silencing in plants. Methods, 30, 296-303.
- Pacak, A., Geisler, K., Jørgensen, B., Barciszewska-Pacak, M., Nilsson, L., et al. (2010) Investigations of barley stripe mosaic virus as a gene silencing vector in barley roots and in Brachypodium distachyon and oat. Plant Methods, 6, 26.
- Bruun-Rasmussen, M., Madsen, C.T., Jessing, S. and Albrechtsen, M. (2007) Stability of Barley stripe mosaic virusinduced gene silencing in barley. Molecular Plant-Microbe Interactions, 20, 1323-1331.
- Meng, Y., Moscou, M.J. and Wise, R.P. (2009) Blufensin1 negatively impacts basal defense in response to barley powdery mildew. Plant Physiology, 149, 271-285.
- Campbell, J. and Li, H. (2010) Silencing of multiple genes in wheat using barley stripe mosaic virus. Journal of Biotech Research, 2, 12-20.
- Tai, Y.S., Bragg, J. and Edwards, M.C. (2005) Virus vector for gene silencing in wheat. BioTechniques, 39, 310-312.
- Ding, X.S., Schneider, W.L., Chaluvadi, S.R., Mian, M.A. and Nelson, R.S. (2006) Characterization of a Brome mosaic virus strain and its use as a vector for gene silencing in monocotyledonous hosts. Molecular Plant-Microbe Interactions, 19, 1229-1239.
- Ding, X.S., Ballard, K., Nelson, R.S. (2009) Improving virus-induced gene silencing (VIGS) in rice through Agrobacterium in filtration. Proceedings of the 14th International Congress on Molecular Plant-Microbe Interactions, Quebec City, 19-23 July 2009, 7.
- Alejska, M., Figlerowicz, M., Malinowska, N., Urbanowicz, A. and Figlerowicz, M. (2005) A universal BMV-based RNA recombination system—How to search for general rules in RNA recombination. Nucleic Acids Research, 33, e105-e105.
- Ding, X.S., Rao, C.S. and Nelson, R.S. (2007) Analysis of gene function in rice through virus-induced gene silencing. Methods in Molecular Biology, 354, 145-160.
- Hay, J.M., Jones, M.C., Blakebrough, M.L., Dasgupta, I., Davies, J.W., et al. (1991) An analysis of an infectious clone of rice tungro bacilliform virus, a plant pararetrovirus. Nucleic Acids Research, 19, 2615-2621.
- Fu, D.Q., Zhu, B.Z., Zhu, H.L., Jiang, W.B. and Luo, Y.B. (2005) Virus-induced gene silencing in tomato fruit. The Plant Journal, 43, 299-308.
- Ratcliff, F., Martin-Hernandez, A.M., Baulcombe, D.C. (2001) Tobacco rattle virus as a vector for analysis of gene function by silencing. The Plant Journal, 25, 237-245.
- Ryu, C.M., Anand, A., Kang, L. and Mysore, K.S. (2004) Agrodrench: A novel and effective agroinoculation method for virus-induced gene silencing in roots and diverse Solanaceous species. The Plant Journal, 40, 322-331.
- Ekengren, S.K., Liu, Y.L., Schiff, M., Dinesh-Kumar, S.P. and Martin, G.B. (2003) Two MAPK cascades, NPR1, and TGA transcription factors play a role in Pto-mediated disease resisitance in tomato. The Plant Journal, 36, 905-917.
- Watson, J.M., Fusaro, A.F., Wang, M. and Waterhouse, P.M. (2005) RNA silencing platforms in plants. FEBS Letters, 579, 5982-5987.
- Fofana, I.B.F, Sangaré, A., Collier, R., Taylor, C. and Fauquet, C.M. (2004) A geminivirus-induced gene silencing system for gene function validation in cassava. Plant Molecular Biology, 56, 613-624.
- Cakir, C. and Tör, M. (2010) Factors influencing Barley stripe mosaic virus-mediated gene silencing in wheat. Physiological and Molecular Plant Pathology, 74, 246-253.
- Szittya, G., Silhavy, D., Molnár, A., Havelda, Z., Lovas, Á., et al. (2003) Low temperature inhibits RNA silencing-mediated defence by the control of siRNA generation. The EMBO Journal, 22, 633-640.
- Rotenberg, D., Thompson, T.S., German, T.L. and Willis, D.K. (2006) Methods for effective real-time RT-PCR analysis of virus-induced gene silencing. Journal of Virological Methods, 138, 49-59.
- Tufan, H.A., Stefanato, F.L., McGrann, G.R., MacCormack, R. and Boyd, L.A. (2011) The Barley stripe mosaic virus system used for virus-induced gene silencing in cereals differentially affects susceptibility to fungal pathogens in wheat. Plant Physiology, 168, 990-994.
- Lacomme, C., Hrubikova, K. and Hein, I. (2003) Enhancement of virus-induced gene silencing through viral-based production of inverted-repeats. The Plant Journal, 34, 543-553.
- Senthil-Kumar, M. and Mysore, K.S. (2011) Virus-induced gene silencing can persist for more than 2 years and also be transmitted to progeny seedlings in Nicotiana benthamiana and tomato. Plant Biotechnol Journal, 9, 797-806.
- Sztuba-Solińska, J., Dzianott, A. and Bujarski, J.J. (2011) Recombination of 5’ subgenomic RNA3a with genomic RNA3 of Brome mosaic bromovirus in Vitro and in Vivo. Virology, 410, 129-141.
- Wroblewski, T., Tomczak, A. and Michelmore, R. (2005) Optimization of Agrobacterium-mediated transient assays of gene expression in lettuce, tomato and Arabidopsis. Plant Biotechnol Journal, 3, 259-273.
- Liu, Y., Burch-Smith, T., Schiff, M., Feng S. and Dinesh-Kumar, S.P. (2004) Molecular chaperone Hsp90 associates with resistance protein N and its signaling proteins SGT1 and Rar1 to modulate an innate immune response in plants. Journal of Biological Chemistry, 79, 2101-2108.
- Dong, Y., Burch-Smith, T.M., Liu, Y., Mamillapalli, P. and Dinesh-Kumar, S.P. (2007) A ligation-independent cloning tobacco rattle virus vector for high-throughput virus-induced gene silencing identifies roles for NbMADS4-1 and -2 in floral development. Plant Physiology, 145, 1161-1170.
- Senthil-Kumar, M., Govind, G., Kang, L., Mysore, K.S. and Udayakumar, M. (2007) Functional characterization of Nicotiana benthamiana homologs of peanut water deficit-induced genes by virus-induced gene silencing. Planta, 225, 523-539.
- 崔艳红, 贾芝琪, 李颖, 等. (2009) 利用 VIGS 研究马铃薯晚疫病抗性基R3a和RB的信号传导. 园艺学报, 7, 997-1004.
- Benkovics, A.H., Nyikó, T., Mérai, Z., Silhavy, D. and Bisztray, G.D. (2011) Functional analysis of the grapevine paralogs of the SMG7 NMD factor using a heterolog VIGS-based gene depletion-complementation system. Plant Molecular Biology, 75, 277-290.
- Scofield, S.R., Amanda, L.H., Brandt, S. and Gill, B.S. (2005) Development of a virus-induced gene silencing system for hexaploid wheat and its use in functional analysis of the Lr21-mediated leaf rust resistance pathway. Plant Physiology, 138, 2165-2173.
- Loutre, C., Wicker, T., Travella, S., Galli, P., Scofield, S., et al. (2009) Two different CC-NBS-LRR genes are required for Lr10-mediated leaf rust resistance in tetraploid and hexaploid wheat. Plant Journal, 60, 1043-1054.
- Zhou, H.B., Li, S.F., Deng, Z.Y., Wang, X., Chen, T., et al. (2007) Molecular analysis of three new receptor-like kinase genes from hexaploid wheat and evidence for their participation in the wheat hypersensitive response to stripe rust fungus infection. The Plant Journal, 52, 420-434.
- Cao, A.H., Xing, L.P., Wang, X.Y., Yang, X., Wang, W., et al. (2011) Serine/threonine kinase gene Stpk-V, a key member of powdery mildew resistance gene Pm21, confers powdery mildew resistance in wheat, confers powdery mildew resistance in wheat. Proceedings of the National Academy of Sciences of the United States of America, 108, 7727-7732.
- Wang, G.F., Wei, X., Fan, R., Zhou, H., Wang, X., et al. (2011) Molecular analysis of common wheat genes encoding three types of cytosolic heat shock protein 90 (Hsp90): functional involvement of cytosolic Hsp90s in the control of wheat seedling growth and disease resistance. New Phytologist, 191, 418-431.
- Hein, I., Pacak, M.B., Hrubikova, K., Williamson, S., Dinesen, M., et al. (2005) Virus-induced gene silencing-based functional characterization of genes associated with powdery mildew resistance in barley. Plant Physiology, 138, 2155- 2164.
- Shen, Q.H., Saijo, Y., Mauch, S., Biskup, C., Bieri, S., et al. (2007) Nuclear activity of MLA immune receptors links isolate-specific and basal disease-resistance responses. Science, 315, 1098-1103.
- Zhang, L., Lavery, L., Gill, U., Gill, K., Steffenson, B., et al. (2009) A cation/proton-exchanging protein is a candidate for the barley NecS1 gene controlling necrosis and enhanced defense response to stem rust. Theoretical and Applied Genetics, 118, 385-397.
- Van Eck, L., Schultz, T., Leach, J.E., Scofield, S.R., Peairs, F.B., et al. (2010) Virus-induced gene silencing of WRKY53 and an inducible phenylalanine ammonia-lyase in wheat reduces aphid resistance. Plant Biotechnology Journal, 8, 1023-1032.
- Oikawa, A., Rahman, A., Yamashita, T., Taira, H. and Kidou, S. (2007) Virus-induced gene silencing of P23k in barley leaf reveals morphological changes involved in secondary wall formation. Journal of Experimental Botany, 58, 2617-2625.
- Held, M.A., Penning, B., Brandt, A.S., Kessansa, S.A., Yong, W., et al. (2008) Small-interfering RNAs from natural antisense transcripts derived from a cellulose synthase gene modulate cell wall biosynthesis in barley. Proceedings of the National Academy of Sciences of the United States of America, 105, 20534-20539.
- Van der Linde, K., Kastner, C., Kumlehn, J., Kahmann, R. and Doehlemann, G. (2011) Systemic virus-induced gene silencing allows functional characterization of maize genes during biotrophic interaction with Ustilago maydis. New Phytologist, 189, 471-483.
- Shi,Y., Qin, Y., Cao, Y., Sun, H., Zhou, T., et al. (2011) Influence of an m-type thioredoxin in maize on potyviral infection. European Journal of Plant Pathology, 131, 317-326.
- Niblett, C.L. and Paulsen, A.Q. (1975) Purification and further characterization of Panicum Mosaic Virus. Phytopathology, 65, 1157-1160.
- Tatineni, S., McMechan, A.J., Hein, G.L. and French, R. (2011) Efficient and stable expression of GFP through Wheat streak mosaic virus-based vectors in cereal hosts using a range of cleavage sites: formation of dense fluorescent aggregates for sensitive virus tracking. Virology, 410, 268-281.
- Stratmann, J.W. and Hind, S.R. (2011) Gene silencing goes viral and uncovers the private life of plants. Entomologia Experimentalis et Applicata, 140, 91-102.
- Wu, J., Hettenhausen, C., Meldau, S. and Baldwin, I.T. (2007) Herbivory rapidly activates MAPK signaling in attacked and unattacked leaf regions but not between leaves of Nicotiana attenuata. Plant Cell, 19, 1096-1122.

NOTES
*通讯作者。