Computer Science and Application
Vol.
10
No.
02
(
2020
), Article ID:
34213
,
9
pages
10.12677/CSA.2020.102025
Calculation of Cell Growth Confluence Based on Region growth
Yibo Shao, Zhilei Xu, Tuozhong Yao
School of Electronics and Information Engineering, Ningbo University of Technology, Ningbo Zhejiang
Received: Jan. 26th, 2020; accepted: Feb. 11th, 2020; published: Feb. 18th, 2020

ABSTRACT
Cell growth confluence is the main parameter in the in vitro culture of cells, which has an important impact on subsequent subculture. The main method for calculating the cell growth convergence degree by manual observation is judged according to the empirical value, and the result has a large error, which is not suitable for large-scale mass production. The development of digital image processing is largely based on the calculation of cell growth convergence to improve efficiency and objective accuracy. To this end, this paper proposes a method for calculating the cell growth convergence based on region growth. First, the improved Gauss-Laplacian operator is used to enhance the contrast of the image preprocessing; secondly, the mean shift algorithm is used to separate the cell region from the non-cell region; finally, the mathematical region is used to filter the blank region with smaller area. In turn, the effective separation of the cell region from the background is achieved, and the growth confluence of the cells is obtained accurately. In this paper, the algorithm is applied to the image of cells collected in the laboratory and the network, and the effectiveness of the algorithm is verified by experiments, which effectively replaces the traditional artificial method for cell growth convergence calculation.
Keywords:Cell Growth Convergence, Mean Shift, Gauss-Laplacian Operator, Region Growth

基于区域生长的细胞生长汇合度 计算
邵一波,许志磊,姚拓中
宁波工程学院电子与信息工程学院,浙江 宁波

收稿日期:2020年1月26日;录用日期:2020年2月11日;发布日期:2020年2月18日

摘 要
细胞生长汇合度一般指细胞贴壁并且完全舒展之后,细胞所占的面积的占培养表面面积的百分比,它作为细胞体外培养环节中的主要参数,对后续的传代培养有着重要影响。传统计算细胞生长汇合度主要的方法通过人工观察,根据经验值判断,对结果有较大的误差,不适用于大规模批量生产。为此本文采用数字图像处理技术代替人工,提出了一种基于区域生长的细胞生长汇合度计算方法。首先,采用改进的高斯-拉普拉斯算子对图像预处理增强对比度;其次,应用均值漂移算法将细胞区域和非细胞区域分离;最后,使用数学形态学操作过滤面积较小的空白区域,进而实现细胞区域与背景的有效分离,得到准确细胞的生长汇合度。本文通过实验室采集的Hela细胞数据集与人工方式相比较,保证了准确性的同时提高了计算速度,在批量生产过程中有效替代传统的人工方法进行细胞生长汇合度计算。
关键词 :细胞生长汇合度,均值漂移,高斯-拉普拉斯算子,区域生长

Copyright © 2020 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
近年来,随着细胞培养技术的不断发展,为了满足细胞在研究和医学生产等的巨大需求,需要将细胞进行体外扩增培养。细胞在培养瓶中的不断扩增,由于培养瓶空间有限,当细胞生长铺满一层时因为接触抑制,细胞会停止分裂进入老化凋亡的状态,因此需要及时进行传代培养。在实际操作中,通过传代操作将贴壁细胞从一个培养瓶中取下来在稀释分装到X个培养瓶,相当于将原来的面积扩大了X倍。其中的X我们通过“生长汇合度”来确定。目前主要的方法是专业人员手工观察,采用高精密仪器,成本较大,且个人的主观性较强,没有一个客观评价的标准,不利于批量生产。
细胞显微图像的处理识别是生物学和医学领域研究的重要手段,图像处理技术有可能为实现细胞汇合度的客观准确评价提供新的手段。近年来随着图像处理技术在细胞研究领域的深入研究,针对不同细胞类别图像的特征,涌现了许多推进细胞领域发展的文献。然而,目前的相关研究工作主要针对切片细胞图像,对于形态多样化的体外培养贴壁细胞图像研究鲜见报道。相较于切片细胞图像,贴壁细胞由于未经染色、图像的前景和背景不易区分,更加具有挑战性。区别于图像处理细胞生长汇合度计算,传统的人工方法存在以下问题:1) 需要专业人员观察;2) 观察结果受到操作人的主观性;3) 不利于应用于大规模生产。因此,采用图像处理技术对细胞图像进行分析已经成为细胞研究领域的热门发展方向。
2. 相关研究
目前已有许多比较成熟的切片细胞分割处理算法,针对细胞分割问题,已经催生出多种方法 [1] - [8]。目前,传统的方法大致可分为如下几类:第一类基于图像阈值的方法,例如直方图阈值的双峰法、迭代法、自适应阈值法等;第二类基于边缘检测的方法,例如Canny算子、Sobel算子、Marr算子等;第三类基于聚类的方法,例如模糊C均值聚类算法、K-Means算法等。
基于图像阈值的分割方法是一种广泛使用的图像分割技术,它利用图像前景和背景在灰度空间上的差异,将图像视为不同灰度级的组合。目前主要方法针对前后景有较大灰度差的情况下有较好的结果,而观察贴壁细胞对象和背景不易区分的特点发现不适用该类方法。
基于边缘检测基本思想是利用边缘增强算子,突出图像中的局部边缘,再通过某一阈值确定边缘强度从而来提取边缘点集。但是由于图像噪声和图像模糊的干扰,检测到的边界可能出现间断的情况。对比了常用的Sobel算子、Canny算子、Log算子等发现每一种算子都针对某一图像特性有较好的效果,但是对较为复杂的细胞图像分割效果不理想。
近年来随着数学各分支在理论和应用上的不断突破,产生不少新的分割算法,例如杨小青等提出的基于形态学的显微细胞图像处理与应用 [9],对于离散的背景单一情况有较好的效果,不适用复杂背景;伏雪等提出的一种基于Mean-Shift改进的图像分割算法,既考虑了图像的边缘信息又减少了训练和分类的复杂度,但还是会产生过分割的情况;基于神经网络的图像分割算法 [5] 是目前研究较多的分割算法,机器学习依赖人工设计特征表达,如提取图像形状、大小等得到最有特征集。但是这种人工的选取以来大量专业知识,且无法将要预先得到所有特征,在实际使用中有局限性。例如U-Net也只是应用在实验研究和视觉比赛中。
基于区域的分割方法是近年来计算机视觉研究中十分关注的图像分割算法,其基本思想是将具有相似性质的像素集合起来构成区域。在实际应用中发现需要解决三个问题:(1) 找到一组代表所有去要区分区域的种子点;(2) 在种子正常过程中准确找到相同区域的像素点;(3) 判定终止生长的条件。常见的有区域增长分割、区域分离合并分割算法,如蒋秋霖等 [10] 提出的基于区域生长算法的脑肿瘤图像分割,通过使用迭代法确定自适应阈值,精度达到97.41%,误检率和漏检率均大幅度减少;潘家辉等 [11] 提出的基于区域生长算法的CT序列图像分割,与人工分割相比,平均相对误差为1.06%;刘应乾提出的基于Gabor滤波与区域生长的细胞分割 [12] 对于噪声不敏感,有较好结果。
考虑上述方法的优缺点本文提出一种改进的区域生长的方法,能够基本解决显微细胞图像的前后景分离问题,在不破坏细胞生长环境的条件下,应用图像处理技术对细胞生长汇合度客观准确的自动检测,为细胞显微图像自动分析技术的建立提供了理论和实验依据。
3. 算法概述
本文提出的基于区域生长的细胞生长汇合度计算方法,其基本流程如图1所示。
1) 通过滤波先消除噪声影响,再锐化边缘,开始默认进行两次拉普拉斯锐化处理;
2) 采用均值漂移算法对预处理的图像进行粗分,将颜色相似的区域合并(对图像多维度数据颜色值(RGB)与空间位置 ,有颜色半径cr和空间半径sr。对于图像内任一点为中心,sr为半径区域内的所有点,先找到RGB值在cr范围内的点作为样本点,通过计算样本点与中心点的向量均值,作为新的中心,移动直至收敛。此时之前所有的样本点都会连通并且像素值等于该收敛点的值),并用漫水填充算法填充各个类区域,分离色彩相近的不同区域;
3) 通过并查集的数据结构存取各个类区域,通过颜色、大小等属性提取出背景区域;
4) 形态学运算过滤较小面积区域;
5) 计算得到细胞区域面积占图像大小比例如果大于75%,输出结果,否则重新从步骤一开始计算,此时只需进行一次拉普拉斯变化,最后直接输出结果。
3.1. 图像增强预处理
观察显微镜细胞图像,我们可以清楚看到细胞边缘区域与背景区域有较大差异,而内部区域与背景对比度低,且存在干扰的纹理、杂质物质等导致错误分割的因素,因此需要对图像进行预处理操作。本文预处理流程为:对原图进行几何变换和高斯模糊,用来降低计算量,再通过边缘检测算法,可有效的将前景细胞图像增强并进一步降低背景干扰因素。

Figure 1. The main flowchart of our proposed algorithm
图1. 细胞生长汇合度计算框架
根据Marr和Hildreth [1] 提出的观点,本文采用 滤波器进行边缘提取。
其中 为拉普拉斯算子,G是标准差为 的二维高斯函数,
预处理步骤如下:
(1) 用一个 取样的3 × 3的高斯低通滤波器对输入图像 进行滤波平滑;
(2) 用拉普拉斯滤波对步骤(1)得到的图像进行图像增强,本文针对细胞图像多次实验发现对于细胞区域占整幅图像面积75%及以上的图像需要进行两次拉普拉斯锐化计算,对于低于75%图像占比的只需进行一次锐化计算。
得到结果如图2所示。
通过左右图像对比,可以观察到细胞区域与背景有明显差异,但是由于背景特征存在噪点,干扰纹理等信息,对实验结果的准确性有极大的干扰,因此对图像进行后处理是十分必要的。
3.2. 分离非细胞区域
预处理之后,图像中细胞和背景极大地增强了对比度.我们的目标是对细胞图像进行前后景分割,传统的图像分割方法是采用迭代图割的交互式提取方法(Graph Cuts)。但是当遇到背景复杂或者背景和目标相似度很大的细胞图像,无法有效分离背景区域。本文提出基于均值漂移的背景提取算法。通过均值漂移算法可以对图像中的每一个像素初步分类,将该像素与它邻域中所有像素的RGB值的均值位置的元素标记为同一类,不断重复直到收敛。调整漂移物理空间半径和色彩空间半径大小,得到最佳效果。均值漂移算法对彩色图像实现了平滑操作,为了达到分割的目的需要使用漫水填充算法进一步处理。
漫水填充算法是给定一个连通域内的一个点,以此为起点找到这个连通域的其余所有点并将其填充为指定的颜色。
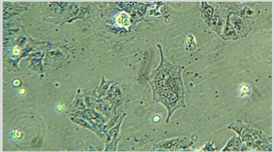
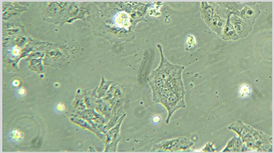

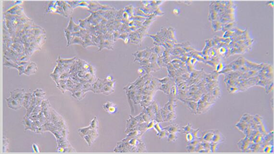
Figure 2. Comparison of cell images before and after image enhancement
图2. 图像增强前后细胞图对比
具体算法流程如下:
1) 选定种子点;
2) 检查种子点颜色,如果该点颜色与边界色和填充色均不同,则用填充色填充,否则不填充;
3) 检查种子八邻域位置,重复步骤2,直到遍历所有像素点。

Figure 3. Preliminary segmentation of the cell background
图3. 初步分割的细胞背景图
此时已经基本对细胞区域和背景区域实现分割,由于漫水填充的随机性无法确定背景区域的颜色信息,因此采用并查集(Union-Find)的数据结构,将具有相同特征值的像素点合并到同一类中,可以有效地避免每次计算颜色随机性的影响。
具体算法流程如下:
1) 在RGB颜色空间下,设k为当前像素为图像的第几个像素,将预处理好的图像的每一个像素记录在一个二维维数组id中,赋value值,value表示当前像素的父节点像素值,此时value等于k,表示父节点是本身;
2) 对图像顺序扫描,对像素k,遍历其四邻域,若RGB值相同,则合并,在id中记录这两个像素点的值为k;
3) 遍历id,将结果记录在一维数组index,其中index[k]表示第k个位置的像素点作为父节点包含子节点的个数,也就是连通区域的面积;
4) 最后将连通区域面积大于阈值的区域设置为背景。
根据上一步骤结果,我们得到了背景二值图 和生长汇合度rate。实际情况中出现了边缘连接处断裂情况,参考白华 [13] 等对其他贴壁细胞生长汇合度的研究,实际对于不同细胞的 ,进行膨胀操作的核参数par选取需要大量测试。根据本文使用数据集得到如下式的近似关系:
(1)
最终得到的结果图4如下。

Figure 4. The resulting cell background image
图4. 最终得到的部分细胞背景图
4. 实验结果与分析
将图像经过一系列处理算法,实现了细胞和背景的有效分离。通过计算最终图像亮色区域面积最终可以得到细胞生长汇合度:
本文在实验室采集不同生长汇合度细胞图像共四类,每一类二十四张作为实验样本如图5所示。
图6显示的是人工标注的不同瓶细胞平均生长汇合度和算法估计的细胞平均生长汇合度。其中(a)、(b)、(c)、(d)四类代表了上图不同汇合度级别的细胞,细胞1、2表示在该类别下随机抽取的两瓶细胞。实验结果表明,本算法自适应的选择计算方法与实际人工标注的平均误差小于5%,准确性较高。本文提出的图像处理方法可有效代替传统人工方法对细胞生长汇合度进行自动计算。
深度学习作为图像处理新的研究方向,在医学领域有着越来越广泛地应用。本文采用Mask-RCNN对现有的数据集做训练,与本文算法对比,得到的效果如图7所示。
上图左侧图片彩色区域显示为Mask-RCNN计算的细胞区域,中间图片为原图,右边为本文算法结果图,其中白色区域为细胞区域。图7(a)、图7(c)左边彩色区域基本将细胞区域检测出来,但是其他采用深度学习的方法对于细胞区域和背景都是大面积时有较好的结果,但是当细胞区域较密集时,效果十分不理想。观察图7(b),红色方框中显示了细胞与背景边界模糊,但是本文算法有效的将边缘模糊区域检测出来。如图7(d)、图7(e)所示,当细胞呈现聚集而将背景分割成较小区域,此时左图深度学习的方法由于没有较好的训练集数据导致欠分割和过分割,而本文算法依然有较好的稳定性和准确性。
对比本文算法有较好的适应性,在不同细胞生长汇合度下都可以准确计算出实际结果。通过比较,深度学习的方法受前期人工标注和训练模型的影响,不能够保证结果的准确性和稳定性,在相同电脑配置下,Mask-RCNN平均处理1张图的时间为3~5 s,而本文算法平均时间为2~3 s,具有较高的准确性和速度。
 (a) 汇合度0%~25%
(a) 汇合度0%~25%
 (b) 汇合度25%~50%
(b) 汇合度25%~50%
 (c) 汇合度50%~75%
(c) 汇合度50%~75%
 (d) 汇合度75%~100%
(d) 汇合度75%~100%
Figure 5. Experimental data set of different confluence
图5. 不同汇合度实验样本数据集
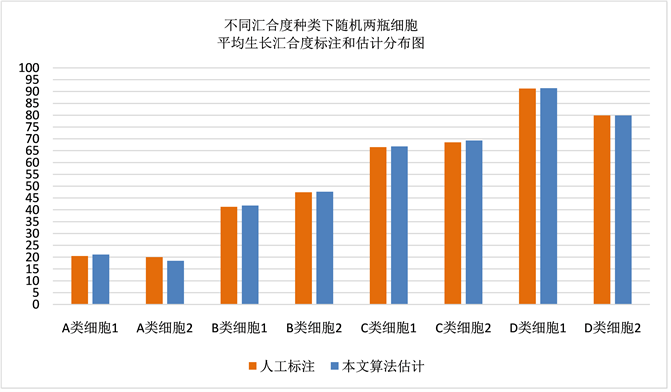
Figure 6. Partial cell growth confluence annotation and estimated distribution map
图6. 部分细胞生长汇合度标注和估计分布图
 (a)
(a)
 (b)
(b)
 (c)
(c)
 (d)
(d)
 (e)
(e)
Figure 7. Comparison results of Mask-RCNN and this algorithm
图7. Mask-RCNN与本文算法对比结果
5. 结论
本文提出一种基于区域生长的细胞生长汇合度计算方法,采用了自定义高斯–拉普拉斯算子对初始图像进行增强,然后通过色彩平滑滤波和漫水填充的方式对预处理的图像进行初步分割。接着采用并查集的数据结构记录分割的结果,计算每一块区域面积若是大于阈值,则认为是背景区域标记为黑色,否则标记为白色。由于在实际细胞图像中观察到干扰的污染点,所以我们最后采用形态学处理,将较小的分割区域也认为是背景区域,进一步提高算法的准确率。
由于不同生物细胞特性不同,本文的方法只在Hela细胞图中应用,对于其他贴壁细胞的生长汇合度计算有不确定性,未来期望将深度学习技术结合进来,应用于不同种类的贴壁细胞观察中,提高算法的自适应性和准确性。
文章引用
邵一波,许志磊,姚拓中. 基于区域生长的细胞生长汇合度计算
Calculation of Cell Growth Confluence Based on Region growth[J]. 计算机科学与应用, 2020, 10(02): 236-244. https://doi.org/10.12677/CSA.2020.102025
参考文献
- 1. Otsu, N. (2007) A Threshold Selection Method from Gray-Level Histograms. IEEE Transactions on Systems Man & Cybernetics, 9, 62-66. https://doi.org/10.1109/TSMC.1979.4310076
- 2. Ali, S. and Madabhushi, A. (2012) An Integrated Region-, Boundary-, Shape-Based Active Contour for Multiple Object Overlap Resolution in Histological Imagery. IEEE Transactions on Medical Imaging, 31, 1448-1460. https://doi.org/10.1109/TMI.2012.2190089
- 3. Liu, B., Jia, X., Jiang, Q., et al. (2015) A Segmentation System Based on Clustering Method for Pediatric DTI Images. International Journal of Imaging Systems and Technology, 25, 102-113. https://doi.org/10.1002/ima.22126 .
- 4. Wu, H.S., Barba, J. and Gil, J. (1998) A Parametric Fitting Algorithm for Segmentation of Cell Images. IEEE Transactions on Biomedical Engineering, 45, 400-407. https://doi.org/10.1109/10.661165
- 5. Hunter, I.A. and Soraghan, J.J. (1995) Neural Network Edge Detection-Successes and Failures. IEE Colloquium on Applications of Neural Networks to Signal Processing (Digest No. 1994/248), London, 15-15 December 1994, 2/1-2/3.
- 6. Zhang, Y. and Cheng, X. (2010) Medical Image Segmentation Based on Watershed and Graph Theory. 2010 3rd International Congress on Image and Signal Processing, Yantai, 16-18 October 2010, 1419-1422. https://doi.org/10.1109/CISP.2010.5646332
- 7. 沙盛中, 翁桂荣, 秦树伟. 基于数学形态学的白细胞分割及提取[C]//中国自动化学会, 中南大学, 湖南省自动 化学会. 第26届中国控制会议, 2007: 497-499.
- 8. 刘锡民, 白丽华, 张庆刚, 许炳章, 邢岳林. 彩色切片细胞的识别与计数研究[J]. 山东师范大学学报(自然科学版), 18(2): 32-34.
- 9. 杨小青, 杨秋翔, 杨剑. 基于形态学的显微细胞图像处理与应用[J]. 计算机系统应用, 2016, 25(3): 222-226.
- 10. 蒋秋霖, 王昕. 提出了基于区域生长算法的脑肿瘤图像分割[J]. 长春工业大学学报, 2018(5): 490-493.
- 11. 潘家辉, 朱玲利, 鲍苏苏. 提出的基于区域生长算法的CT序列图像分割[J]. 洛阳师范学院学报, 2015(5): 58-61.
- 12. 刘应乾, 曹茂永. 基于Gabor滤波与区域生长的细胞分割[J]. 山东科技大学学报(自然科学版), 2012, 31(2): 99-103.
- 13. 白华, 张凤凤, 张成, 赵军发. 间充质干细胞生长汇合度自动分析算法研究[J]. 中国细胞生物学学报, 2017, 39(11): 72-79.