Bioprocess
Vol.
13
No.
02
(
2023
), Article ID:
67477
,
9
pages
10.12677/BP.2023.132016
蛋白质S-棕榈酰化在T细胞信号传导中的研究进展
戴心忱1,2*,张为国2,陆美玲1#
1中国药科大学生命科学与技术学院,江苏 南京
2中国医学科学院北京协和医学院苏州系统医学研究所,江苏 苏州
收稿日期:2023年4月21日;录用日期:2023年6月9日;发布日期:2023年6月21日

摘要
蛋白质S-棕榈酰化是一种高度动态的翻译后脂质化修饰,由DHHC蛋白家族催化,在调节蛋白质定位、稳定性和细胞内转运等方面发挥重要作用。近年来,随着T细胞免疫疗法的兴起,对于T细胞信号通路的研究也愈发深入。大量证据表明,T细胞活化过程中,有很多关键信号分子是S-棕榈酰化的,它们的稳定表达和质膜定位对于免疫信号的传递是必需的。因此,本文总结了DHHC蛋白家族和蛋白质S-棕榈酰化的机制,并基于S-棕榈酰化在T细胞信号传导中的不同作用,分析了靶向S-棕榈酰化治疗免疫疾病的潜力和挑战。
关键词
S-棕榈酰化,DHHC蛋白家族,T细胞信号传导,进展

Research Progress on Protein S-Palmitoylation in T-Cell Signaling Transduction
Xinchen Dai1,2*, Weiguo Zhang2, Meiling Lu1#
1College of Life Science and Technology, China Pharmaceutical University, Nanjing Jiangsu
2Suzhou Institute of Systematic Medicine, Chinese Academy of Medical Sciences and Peking Union Medical College, Suzhou Jiangsu
Received: Apr. 21st, 2023; accepted: Jun. 9th, 2023; published: Jun. 21st, 2023

ABSTRACT
Protein S-palmitoylation is a highly dynamic post-translational lipid modification catalyzed by the DHHC protein family which plays an important role in regulating protein localization, stability and intracellular translocation. In recent years, there has been a surge of interest in T-cell-based immunotherapy, leading to a deeper investigation of T-cell signaling pathways. A large amount of evidence indicates that many pivotal signaling molecules involved in T-cell activation are modified by S-palmitoylation, which is essential for their stable expression and plasma membrane localization, and thus for proper immune signaling. Therefore, in this review, we summarize the DHHC protein family and the mechanism of protein S-palmitoylation, and analyze the potential and challenges of targeting S-palmitoylation for immune diseases based on the different roles of S-palmitoylation in T-cell signaling transduction.
Keywords:S-Palmitoylation, DHHC Protein Family, T-Cell Signaling Transduction, Progress

Copyright © 2023 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
迄今为止,已经发现了200多种不同的翻译后修饰,其中包括磷酸化、乙酰化、甲基化、泛素化及脂质化等 [1] ,这些修饰为蛋白质的结构和功能增加了极其复杂的化学特性。作为最重要的翻译后修饰之一,脂质化修饰能够增加蛋白质的疏水性,从而导致其构象、稳定性、细胞内转运和定位等方面发生变化 [2] [3] [4] 。
棕榈酰化修饰是常见的脂质化修饰,已知大约有4000多种理论上棕榈酰化的蛋白质 [5] 。因为其显著的生理作用和广泛性,使其成为当下研究的热点之一。棕榈酰化是在膜的细胞质一侧发生的脂质化修饰,根据其添加的棕榈酸酯和所涉及的氨基酸分为三个不同的类别:O-棕榈酰化通过氧化酯键将棕榈酸或棕榈油添加到丝氨酸或苏氨酸上;N-棕榈酰化通过酰胺键将棕榈酸添加到N-末端甘氨酸、赖氨酸或半胱氨酸上;S-棕榈酰化通过硫酯键将棕榈酸添加到半胱氨酸上 [6] 。其中,可逆且动态的S-棕榈酰化是蛋白质棕榈酰化的主要形式,不稳定的硫酯键允许蛋白质在几秒到几小时的时间范围内在棕榈酰化和去棕榈酰化之间循环,从而赋予蛋白质多样的生理功能 [7] 。因此,本文讨论的棕榈酰化均为S-棕榈酰化。
为了深入研究棕榈酰化,研究者们开发了多种技术,如代谢标记法、酰基–生物素置换法和质谱分析法等。另外,点击化学反应也为棕榈酰化的研究提供了新的思路 [8] 。这些技术的应用导致新发现的棕榈酰化蛋白质数量迅速增加,目前已有约350种蛋白质被明确证实会被棕榈酰化 [1] 。其中,有多种蛋白质在T细胞活化和分化中起着关键作用,包括Lck、ZAP-70、LAT、IP3R和PD-1/PD-L1等。棕榈酰化修饰可以调节它们的膜定位和稳定性,为蛋白质重组和信号传导的形成提供分子基础,这对T细胞的激活具有重大意义 [9] [10] [11] 。因此,本文将聚焦于棕榈酰化,总结并讨论它在T细胞激活过程中的作用,为免疫疾病提供新的解决思路。
2. DHHC蛋白家族
尽管蛋白质棕榈酰化早在40多年前就被发现,但是什么酶参与棕榈酰化却让研究者困惑了很长时间,直到十多年前才有人发现哺乳动物蛋白的棕榈酰化反应是由一类具有蛋白棕榈酰基转移酶(protein acyltransferase, PAT)活性的蛋白来催化的 [12] 。这些蛋白都含有典型的DHHC (Asp-His-His-Cys)基序,因此被称为DHHC蛋白家族 [7] 。至今已在哺乳动物中鉴定出23种DHHC蛋白,分别命名为DHHC1~DHHC24,但排除了DHHC10 [13] 。大量的实例表明,一个给定的DHHC蛋白可以棕榈酰化多种不同的底物,而一个给定的蛋白质可以被几个DHHC蛋白棕榈酰化 [14] 。这表明DHHC蛋白的功能可能会有一定程度的冗余。
2.1. DHHC蛋白的结构
大多数DHHC蛋白是完整的膜蛋白,至少含有四个跨膜结构域(trans-membrane domain, TMD),N端和C端均暴露于细胞质(图1(a)),介导蛋白质–蛋白质相互作用 [2] 。最近,有研究人员纯化并解析了人类DHHC20蛋白的晶体结构,发现DHHC20的四个TMD在膜中采用类似圆锥形帐篷的形式排列,在该空腔内的氨基酸残基决定了该酶对酰基辅酶A链长度的选择性。此外,在C端区域发现了一个疏水环和一个两亲性螺旋,形成了TMD的支撑结构 [15] 。特征性的DHHC-CRD (cysteine-rich domain),位于TMD2和TMD3之间,具有酶的催化活性。棕榈酰基转移反应发生在DHHC基序的半胱氨酸残基上,该半胱氨酸还参与协调两个锌原子,它们虽然不发挥催化作用,但对DHHC蛋白的稳定性至关重要 [16] 。除了DHHC-CRD之外,还有三个短的保守序列(图1(a)):DPG (Asp-Pro-Gly)基序、TTxE (Thr-Thr-xxx-Glu)基序和棕榈酰基转移酶保守C末端(PaCCT)基序 [17] [18] 。系统发育分析将人类DHHC蛋白聚类成几个亚家族(图1(b))。不同DHHC蛋白之间的序列存在显著差异,有些具有独特的结构域,比如PDZ结合基序(图1(c))、SH3结构域以及锚蛋白重复序列等。研究表明,DHHC蛋白与其底物的相互作用还通过DHHC-CRD以外的四个结构域进行。DHHC5、DHHC8和DHHC14的PDZ结合基序可与含有PDZ结构域的相应蛋白结合,在DHHC招募底物的过程中发挥重要作用 [19] 。PDZ结合基序在其他DHHC蛋白中可能具有类似的功能。
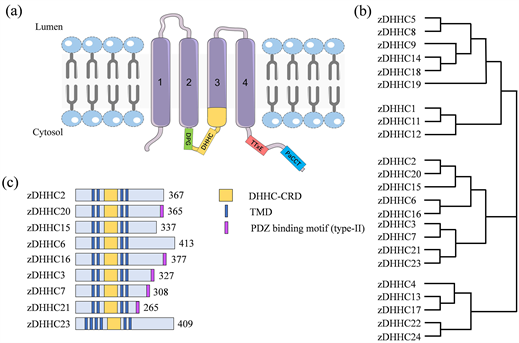
Figure 1. Topology and conserved motifs of DHHC family protein membranes [17] . (a) Most DHHC proteins contain four transmembrane domains (TMDs), DHHC-CRD, DPG, TTxE and PaCCT motifs; (b) Phylogenetic analysis of DHHC proteins; (c) Some DHHC proteins have PDZ-binding motifs
图1. DHHC家族蛋白膜拓扑结构和保守基序 [17] 。(a) 大多数DHHC蛋白含有四个跨膜区(TMD)、DHHC-CRD、DPG、TTxE和PaCCT基序;(b) DHHC蛋白的系统发育分析;(c) 部分DHHC蛋白质有PDZ结合基序
2.2. DHHC蛋白的定位
在过表达DHHC蛋白(GFP标记)的HEK293细胞中,不同DHHC蛋白定位在细胞的不同区域,包括质膜(如DHHC5, 20和21)、高尔基体(如DHHC3, 4和7)和内质网(如DHHC1, 6和10),甚至出现在多个位置(如DHHC2, 9和12) [20] 。因此,有学者提出,DHHC蛋白的不同亚细胞位置可能调节不同但互补的功能,并且这种协同作用可以介导细胞内的蛋白质转运 [21] [22] 。但是,这些定位结果是通过在HEK293细胞中过表达获得的,由于过表达可能导致蛋白定位异常,所以我们尚不清楚这些DHHC蛋白在正常细胞以及T细胞中是否具有相同的定位。
2.3. DHHC蛋白与疾病
DHHC蛋白的自然突变、实验性缺失或过表达会导致严重的表型变化,其中大部分与人类疾病有关 [23] 。例如,DHHC8和DHHC9的突变可以导致神经系统疾病 [24] 。乳腺、肺和前列腺癌细胞存在DHHC2基因的缺失和突变 [25] 。与此同时,过表达DHHC18和DHHC23可以促进原癌基因BMI1的棕榈酰化,进而提高神经胶质瘤细胞在应激性肿瘤微环境下的存活 [26] 。DHHC13可以棕榈酰化EGFR,使其能够定位在质膜上,降低DHHC13的表达水平可以破坏其定位,有望成为EGFR过表达肿瘤治疗的新方法 [27] 。
3. 蛋白质棕榈酰化修饰机制
目前,蛋白质棕榈酰化的机制仍不太明确,但是对酵母Erf2/Erf4蛋白和哺乳动物DHHC2和DHHC3蛋白的研究表明,该修饰过程可能采用两步反应机制(图2) [28] [29] 。第一步是DHHC基序中半胱氨酸的自酰化。研究表明,DHHC基序的天冬氨酸和第一位组氨酸可以形成氢键对,从而接受来自半胱氨酸的质子,最终对棕榈酰基–辅酶A的羰基硫酯进行亲核攻击,以产生棕榈酰基中间体。第二步是棕榈酰基中间体发挥转移酶活性,将棕榈酰基转移到蛋白底物中的半胱氨酸巯基上,完成目的蛋白的棕榈酰化。若没有底物蛋白,棕榈酰基中间体会被缓慢水解并进入新的棕榈酰基循环 [29] 。在整个过程中,DHHC基序中半胱氨酸或第一个组氨酸的突变都会阻断目的蛋白的棕榈酰化 [30] 。然而,有研究发现,至少有两种酵母DHHC蛋白没有发生两步反应机制中的自酰化,并且依然具有部分棕榈酰基转移酶功能,这表明可能存在一步棕榈酰化的机制 [31] 。
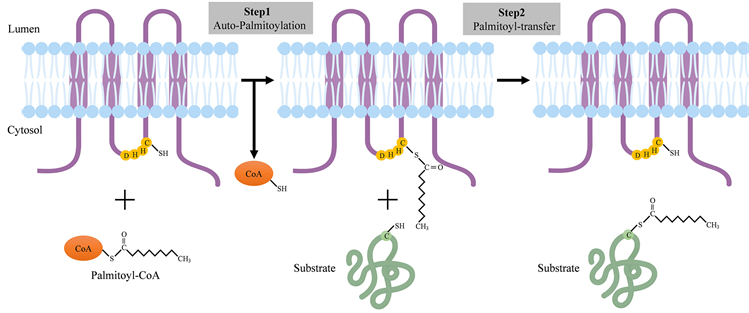
Figure 2. Protein palmitoylation modification process (Created by https://www.biorender.com/)
图2. 蛋白质棕榈酰化修饰过程(Created by https://www.biorender.com/)
4. 蛋白质棕榈酰化在T细胞信号通路中的作用
T细胞在特异性免疫中扮演着重要角色,其增殖和激活需要接受T细胞受体(T-cellreceptor, TCR)信号。当TCR和其辅助受体(CD4/CD8)识别抗原呈递细胞(APC)上表达的主要组织相容性复合物(MHC)及其呈递的抗原肽时,T细胞被激活 [32] 。TCR受到刺激后,淋巴细胞特异性蛋白酪氨酸激酶(Lck)被活化并招募至CD3ζ链上的免疫受体酪氨酸活化基序(ITAM)附近,随后将ITAM磷酸化。磷酸化的ITAM可以进一步募集ZAP-70与之结合,此时附近的Lck便会磷酸化ZAP-70使之活化。活化的ZAP-70可以磷酸化并激活T细胞活化接头(LAT),在此处的下游,PLCγ1、Grb2、MAPK和PKC等蛋白被活化。这将引起NFAT、NFκB和Ras通路的激活,最终导致基因表达和T细胞活化 [33] [34] 。已知TCR信号通路中有多个蛋白受到棕榈酰化的调节。
4.1. Lck
Lck是非受体酪氨酸激酶Src家族的成员,在T细胞活化的早期阶段扮演着关键角色。研究表明,Lck在Cys3和Cys5被DHHC2和DHHC21棕榈酰化,同时还是N-豆蔻酰化修饰的 [35] 。这两个脂质化修饰发挥着不同的作用,豆蔻酰化最有可能将Lck靶向质膜,而棕榈酰化则赋予了Lck与脂筏结合的能力,最终Lck能够在质膜上稳定定位 [36] 。据报道,Lck C3A和C5A双突变体无法定位于质膜,不能磷酸化ITAM和ZAP-70,引起T细胞下游信号缺失。但是,Lck C3A或Lck C5A单突变体却不会完全消除棕榈酰化和质膜定位,表明这两个位点被棕榈酰化后均可以发挥作用。然而,在大多数研究中,Lck C5A的突变体仅有少量出现质膜定位缺陷,而Lck C3A突变体中大约一半无法与质膜结合 [37] [38] [39] [40] 。因此,Lck Cys3上的棕榈酰化修饰占主导作用,同时,单棕榈酰化和双棕榈酰化Lck很可能共存 [35] 。鉴于棕榈酰化的Lck在TCR信号通路中的重要作用,有学者设计了一种新型肽,可以特异性干扰DHHC21-Lck相互作用,抑制Lck棕榈酰化的同时,特异性阻断T细胞功能 [41] ,在预防免疫紊乱方面具有重要价值。
4.2. Fyn
同样作为Src家族成员的Fyn也参与了T细胞的活化,并且功能与Lck部分重叠。与Lck相似的是,Fyn也需要N-豆蔻酰化和S-棕榈酰化联合作用才能正确定位在质膜上发挥作用。然而,与Lck不同的是,在T细胞未激活前,Fyn便位于脂筏,并且未棕榈酰化的Fyn也有一小部分能与脂筏结合 [35] 。Fyn可以在Cys3和Cys6被棕榈酰化,其中Cys3棕榈酰化的Fyn占大多数,只有一小部分是Cys6棕榈酰化的,但是这些Fyn可以绕过高尔基途径,直接靶向质膜与脂筏结合 [42] [43] ,其中的机制有待进一步探究。
4.3. ZAP-70
ZAP-70是T细胞免疫反应中必不可少的酪氨酸激酶。在TCR的参与下,ZAP-70可被募集到特定的质膜结构域,并被Lck激活。活化的ZAP-70通过磷酸化LAT和SLP-76这两个重要的连接分子来传递TCR信号,这对于促进T细胞的发育和激活非常重要 [33] 。最近,有学者提出,ZAP-70激酶结构域的Cys564能够被棕榈酰化,并进一步报道其棕榈酰基转移酶为DHHC21。他们在一名免疫缺陷的患者体内发现了棕榈酰化缺陷的ZAP-70 C564R突变体,它可以与Lck相互作用并被磷酸化,但是无法磷酸化LAT和激活T细胞 [11] 。因此,ZAP-70棕榈酰化缺陷被认为是该疾病的原因。然而,新近研究发现,ZAP-70 C564R突变体的功能缺陷似乎不是由于棕榈酰化损失造成的。研究人员设计了ZAP-70的C564A突变体,它与ZAP-70 C564R突变体相似,也是不可棕榈酰化的。然而,在ZAP-70缺陷的P116 T细胞中,该突变体能被活化并磷酸化LAT,从而传递信号。此外,ZAP-70 C564A增强了Lck的活性并提高了其与TCR的亲和力。因此,表达ZAP-70 C564A的ZAP-70缺陷型的P116 T细胞表现出ITAM和ZAP-70的过度磷酸化 [44] 。总的来说,ZAP-70Cys564位点对于Lck活性和近端TCR信号的调节非常重要,但其能否被棕榈酰化对于ZAP-70发挥功能似乎并没有影响,或者说还存在着未知的调控机制。
4.4. LAT
LAT被ZAP-70磷酸化后,可以与Grb2、PLCγ1、Gads和SLP-76等下游分子结合,组成TCR信号复合物,从而启动Ras-MAPK活化通路、PKC-NF-κB活化通路和钙调蛋白NF-AT活化通路等,最终导致多个转录因子活化入核激活T细胞 [45] [46] 。但是,构成这一过程的前提是LAT被棕榈酰化。目前,已知Cys26和Cys29是LAT的棕榈酰化位点,其中Cys26的棕榈酰化对LAT的功能和定位至关重要 [47] 。有体外实验证明,DHHC18能够棕榈酰化LAT [48] 。与wt LAT相比,棕榈酰化缺陷的LAT主要定位在高尔基体上,无法定位在质膜被ZAP-70磷酸化 [49] ,进而影响其与Grb2和PLCγ1等形成TCR信号复合物,阻碍了T细胞的激活。同时,棕榈酰化缺陷的LAT稳定性也大大降低,相比于wt LAT,它对泛素依赖的降解途径更为敏感 [47] 。在一些无反应T细胞中,LAT棕榈酰化程度严重下降,而其他关键蛋白的棕榈酰化却不受影响 [49] ,表明可能是LAT相关的DHHC蛋白受到某种未知的调节。
4.5. 钙离子通道相关蛋白
钙离子信号在调节T细胞信号传导中具有重要作用。磷脂酰肌醇酰转移酶γ1 (PLCγ1)被激活会产生肌醇三磷酸(IP3),它与内质网膜上的IP3受体(IP3R)结合,引发内质网中钙离子储存耗竭,进而促进钙释放激活钙通道(CRAC)的激活,并允许钙离子进入细胞,从而协调T细胞中的各种下游信号事件,最终导致NFAT通路的激活 [50] 。PLCγ1能够被DHHC21棕榈酰化,并受TCR信号动态调节,敲低DHHC21表达水平会导致钙流通急剧下降 [10] ,但其中的机制还知之甚少。同样,IP3R也是棕榈酰化的,有人利用质谱法发现IP3R可以在Cys56和Cys849被棕榈酰化,并由硒蛋白K (SelK)依赖性的DHHC6催化。与Lck这类质膜定位蛋白不同的是,棕榈酰化缺陷不会影响IP3R的内质网膜定位,但依然会减少钙流通,可能是蛋白质稳定性和表达量下降 [51] 。综合这些数据表明,棕榈酰化可以调节钙离子流量来影响T细胞活化。
4.6. PD-1/PD-L1
免疫检查点抑制剂的开发一直是肿瘤免疫治疗中最热门的方向,其中以程序性细胞死亡蛋白1 (PD-1)/程序性死亡配体1 (PD-L1)为靶点的药物更是多达上百种。T细胞激活也受免疫检查点信号控制。T细胞上的PD-1在与其配体PD-L1和PD-L2结合后,可以向T细胞传递抑制信号。这有助于自我耐受和肿瘤逃逸 [52] 。据报道,DHHC3可以棕榈酰化结直肠癌细胞中的PD-L1 Cys272位点,阻断其泛素化,从而抑制溶酶体对PD-L1的降解。通过2-溴棕榈酸酯(2-BP)抑制PD-L1棕榈酰化,可在体外和小鼠体内激活抗肿瘤免疫杀死肿瘤细胞 [53] 。然而,DHHC3并不是PD-L1唯一的棕榈酰基转移酶,在乳腺癌细胞中,DHHC9可以棕榈酰化PD-L1以维持它的稳定性和膜表面分布 [54] 。最近的一项研究中发现,PD-1也可以被DHHC9棕榈酰化,棕榈酰化对其在癌细胞中激活mTOR信号传导的能力很重要,相关研究人员还设计了一种肽用于竞争性抑制PD-1棕榈酰化 [55] 。因此,抑制PD-1/PD-L1棕榈酰化会增加T细胞对肿瘤细胞的杀伤力,为克服PD-1/PD-L1介导的肿瘤逃逸提供了的新策略。
5. 总结与展望
随着蛋白质组学、化学生物学和CRISPR技术的不断发展,大量的研究表明,棕榈酰化对T细胞介导的细胞免疫发挥着至关重要的作用。棕榈酰化可以调节蛋白之间相互作用,促进信号传递并加强免疫反应,而棕榈酰化缺陷会导致T细胞功能降低甚至无能。因此,增强棕榈酰化可能会进一步提高机体免疫力,而抑制棕榈酰化则可能有助于治疗自身免疫性疾病。但需要注意的是,棕榈酰化修饰也会维持PD-1/PD-L1稳定性及膜定位,对于T细胞活化具有抑制作用。总之,以棕榈酰化修饰为靶标是治疗免疫相关疾病最有前途的方向之一。
然而,要充分利用棕榈酰化来治疗人类疾病,还面临着诸多挑战。首先,从抑制棕榈酰化的角度来看,2-BP是最常用和最有效的抑制剂,但其广谱抑制性限制了临床应用。目前,特异性DHHC蛋白抑制剂的开发主要集中在能棕榈酰化PD-1/PD-L1的DHHC蛋白上 [44] ,对于LAT等传统意义上的T细胞信号分子,仍缺乏相关的研究。从增强棕榈酰化来看,目前没有任何DHHC蛋白激动剂的报道。但是考虑到去棕榈酰基转移酶的存在,可以根据它的最新晶体结构设计相关的竞争性抑制剂,反向增强棕榈酰化。但遗憾的是,当前对于去棕榈酰基转移酶在T细胞信号传导过程中的研究还处于一个起步的阶段,大量关键蛋白质的去棕榈酰基转移酶尚未被发现。因此,棕榈酰化修饰这一已有四十年历史的翻译后修饰,在免疫系统中仍有大量探索的空间。
文章引用
戴心忱,张为国,陆美玲. 蛋白质S-棕榈酰化在T细胞信号传导中的研究进展
Research Progress on Protein S-Palmitoylation in T-Cell Signaling Transduction[J]. 生物过程, 2023, 13(02): 115-123. https://doi.org/10.12677/BP.2023.132016
参考文献
- 1. Bijlmakers, M.J. (2009) Protein Acylation and Localization in T Cell Signaling. Molecular Membrane Biology, 26, 93-103. https://doi.org/10.1080/09687680802650481
- 2. Kabouridis, P.S., Magee, A.I. and Ley, S.C. (1997) S-acylation of LCK Protein Tyrosine Kinase Is Essential for Its Signalling Function in T Lymphocytes. The EMBO Journal, 16, 4983-4998. https://doi.org/10.1093/emboj/16.16.4983
- 3. Kosugi, A., Hayashi, F., Liddicoat, D.R., et al. (2001) A Pivotal Role of Cysteine 3 of Lck Tyrosine Kinase for Localization to Glycolipid-Enriched Microdomains and T Cell Activation. Immunology Letters, 76, 133-138. https://doi.org/10.1016/S0165-2478(01)00174-2
- 4. Kwong, J. and Lublin, D.M. (1995) Amino-Terminal Palmi-tate or Polybasic Domain Can Provide Required Second Signal to Myristate for Membrane Binding of P56lck. Biochemi-cal and Biophysical Research Communications, 207, 868-876. https://doi.org/10.1006/bbrc.1995.1266
- 5. Yurchak, L.K. and Sefton, B.M. (1995) Palmitoylation of either Cys-3 or Cys-5 Is Required for the Biological Activity of the Lck Tyrosine Protein Kinase. Molecular and Cellular Biology, 15, 6914-6922. https://doi.org/10.1128/MCB.15.12.6914
- 6. Gauthaman, A., Jacob, R., Pasupati, S., et al. (2022) Novel Pep-tide-Based Inhibitor for Targeted Inhibition of T Cell Function. Journal of Cell Communication and Signaling, 16, 349-359. https://doi.org/10.1007/s12079-021-00660-0
- 7. Sato, I., Obata, Y., Kasahara, K., et al. (2009) Differ-ential Trafficking of Src, Lyn, Yes and Fyn Is Specified by the State of Palmitoylation in the SH4 Domain. Journal of Cell Science, 122, 965-975. https://doi.org/10.1242/jcs.034843
- 8. West, S.J., Boehning, D. and Akimzhanov, A.M. (2022) Regulation of T Cell Function by Protein S-acylation. Frontiers in Physiology, 13, Article 2404. https://doi.org/10.3389/fphys.2022.1040968
- 9. Schultz, A., Schnurra, M., El Bizri, A., et al. (2022) A Cysteine Residue within the Kinase Domain of Zap70 Regulates Lck Activity and Proximal TCR Signaling. Cells, 11, Article 2723. https://doi.org/10.3390/cells11172723
- 10. Martinez-Florensa, M., Garcia-Blesa, A., Yélamos, J., et al. (2011) Serine Residues in the LAT Adaptor Are Essential for TCR-Dependent Signal Transduction. Journal of Leukocyte Biol-ogy, 89, 63-73. https://doi.org/10.1189/jlb.0509342
- 11. Shim, E.K., Jung, S.H. and Lee, J.R. (2011) Role of Two Adaptor Molecules SLP-76 and LAT in the PI3K Signaling Pathway in Activated T Cells. The Journal of Immunology, 186, 2926-2935. https://doi.org/10.4049/jimmunol.1001785
- 12. Zhang, W.G., Trible, R.P. and Samelson, L.E. (1998) LAT Palmitoylation: Its Essential Role in Membrane Microdomain Targeting and Tyrosine Phosphorylation dur-ing T Cell Activation. Immunity, 9, 239-246. https://doi.org/10.1016/S1074-7613(00)80606-8
- 13. Morrison, E., Wegner, T., Zucchetti, A.E., et al. (2020) Dy-namic Palmitoylation Events Following T-Cell Receptor Signaling. Communications Biology, 3, Article No. 368. https://doi.org/10.1038/s42003-020-1063-5
- 14. Hundt, M., Tabata, H., Jeon, M.S., et al. (2006) Impaired Activa-tion and Localization of LAT in Anergic T Cells as a Consequence of a Selective Palmitoylation Defect. Immunity, 24, 513-522. https://doi.org/10.1016/j.immuni.2006.03.011
- 15. Trebak, M. and Kinet, J.P. (2019) Calcium Signalling in T Cells. Nature Reviews Immunology, 19, 154-169. https://doi.org/10.1038/s41577-018-0110-7
- 16. Fredericks, G.J., Hoffmann, F.W., Rose, A.H., et al. (2014) Sta-ble Expression and Function of the Inositol 1,4,5-Triphosphate Receptor Requires Palmitoylation by a DHHC6/Selenoprotein K Complex. Proceedings of the National Academy of Sciences, 111, 16478-16483. https://doi.org/10.1073/pnas.1417176111
- 17. Lin, H.N. (2021) Protein Cysteine Palmitoylation in Immunity and Inflammation. The FEBS Journal, 288, 7043-7059. https://doi.org/10.1111/febs.15728
- 18. Yao, H., Lan, J., Li, C.S., et al. (2019) Inhibiting PD-L1 Palmitoylation Enhances T-Cell Immune Responses against Tumours. Nature Biomedical Engineering, 3, 306-317. https://doi.org/10.1038/s41551-019-0375-6
- 19. Zmuda, F. and Chamberlain, L.H. (2020) Regulatory Effects of Post-Translational Modifications on zDHHC S-Acyltransferases. Journal of Biological Chemistry, 295, 14640-14652. https://doi.org/10.1074/jbc.REV120.014717
- 20. Yang, Y., Hsu, J.M., Sun, L., et al. (2019) Palmitoylation Stabi-lizes PD-L1 to Promote Breast Tumor Growth. Cell Research, 29, 83-86. https://doi.org/10.1038/s41422-018-0124-5
- 21. Jiang, H., Zhang, X.Y., Chen, X., et al. (2018) Protein Lipidation: Occurrence, Mechanisms, Biological Functions, and Enabling Technologies. Chemical Reviews, 118, 919-988. https://doi.org/10.1021/acs.chemrev.6b00750
- 22. Linder, M.E. and Deschenes, R.J. (2007) Palmitoylation: Polic-ing Protein Stability and Traffic. Nature Reviews Molecular Cell Biology, 8, 74-84. https://doi.org/10.1038/nrm2084
- 23. Morris, G., Walder, K., Puri, B.K., et al. (2016) The Deleterious Effects of Oxidative and Nitrosative Stress on Palmitoylation, Membrane Lipid Rafts and Lipid-Based Cellular Signalling: New Drug Targets in Neuroimmune Disorders. Molecular Neurobiology, 53, 4638-4658. https://doi.org/10.1007/s12035-015-9392-y
- 24. Blanc, M., David, F.P. and Van Der Goot, F.G. (2019) Swis-sPalm 2: Protein S-palmitoylation Database. In: Linder, M., Eds., Protein Lipidation, Vol. 2009, Humana, New York, NY, 203-214. https://doi.org/10.1007/978-1-4939-9532-5_16
- 25. Cassinelli, S., Viñola Renart, C., Benavente Garcia, A., et al. (2022) Palmitoylation of Voltage-Gated Ion Channels. International Journal of Molecular Sciences, 23, Article 9357. https://doi.org/10.3390/ijms23169357
- 26. Ko, P.J. and Dixon, S.J. (2018) Protein Palmitoylation and Cancer. EMBO Reports, 19, e46666. https://doi.org/10.15252/embr.201846666
- 27. Zhang, M.M., Zhou, L.X., Xu, Y.J., et al. (2020) A STAT3 Pal-mitoylation Cycle Promotes TH17 Differentiation and Colitis. Nature, 586, 434-439. https://doi.org/10.1038/s41586-020-2799-2
- 28. Fan, Y., Shayahati, B., Tewari, R., et al. (2020) Regulation of T Cell Receptor Signaling by Protein Acyltransferase DHHC21. Molecular Biology Reports, 47, 6471-6478. https://doi.org/10.1007/s11033-020-05691-1
- 29. Shayahati, B., Fan, Y., West, S., et al. (2020) Calci-um-Dependent Protein Acyltransferase DHHC21 Controls Activation of CD4+ T Cells. bioRxiv, 135, jcs258186. https://doi.org/10.1101/2020.09.01.277947
- 30. Tewari, R., Shayahati, B., Fan, Y., et al. (2021) T Cell Recep-tor-Dependent S-acylation of ZAP-70 Controls Activation of T Cells. Journal of Biological Chemistry, 296, Article ID: 100311. https://doi.org/10.1016/j.jbc.2021.100311
- 31. Fukata, M., Fukata, Y., Adesnik, H., et al. (2004) Identifi-cation of PSD-95 Palmitoylating Enzymes. Neuron, 44, 987-996. https://doi.org/10.1016/j.neuron.2004.12.005
- 32. Jin, J.Y., Zhi, X.L., Wang, X.H., et al. (2021) Protein Pal-mitoylation and Its Pathophysiological Relevance. Journal of Cellular Physiology, 236, 3220-3233. https://doi.org/10.1002/jcp.30122
- 33. Ladygina, N., Martin, B.R. and Altman, A. (2011) Dynamic Palmitoylation and the Role of DHHC Proteins in T Cell Activation and Anergy. Advances in immunology, 109, 1-44. https://doi.org/10.1016/B978-0-12-387664-5.00001-7
- 34. Rana, M.S., Kumar, P., Lee, C.J., et al. (2018) Fatty Acyl Recognition and Transfer by an Integral Membrane S-Acyltransferase. Science, 359, eaao6326. https://doi.org/10.1126/science.aao6326
- 35. González Montoro, A., Quiroga, R. and Valdez Taubas, J. (2013) Zinc Co-Ordination by the DHHC Cysteine-Rich Domain of the Palmitoyltransferase Swf1. Biochemical Journal, 454, 427-435. https://doi.org/10.1042/BJ20121693
- 36. Malgapo, M.I.P. and Linder, M.E. (2021) Substrate Recruit-ment by zDHHC Protein Acyltransferases. Open Biology, 11, Article ID: 210026. https://doi.org/10.1098/rsob.210026
- 37. Stix, R., Lee, C.J., Faraldo Gómez, J.D., et al. (2020) Structure and Mechanism of DHHC Protein Acyltransferases. Journal of Molecular Biology, 432, 4983-4998. https://doi.org/10.1016/j.jmb.2020.05.023
- 38. Thomas, G.M., Hayashi, T., Chiu, S.L., et al. (2012) Palmitoylation by DHHC5/8 Targets GRIP1 to Dendritic Endosomes to Regulate AMPA-R Trafficking. Neuron, 73, 482-496. https://doi.org/10.1016/j.neuron.2011.11.021
- 39. Yao, H., Li, C.S., He, F., et al. (2021) A Peptidic Inhibitor for PD-1 Palmitoylation Targets Its Expression and Functions. RSC Chemical Biology, 2, 192-205. https://doi.org/10.1039/D0CB00157K
- 40. Ohno, Y., Kihara, A., Sano, T., et al. (2006) Intracellular Local-ization and Tissue-Specific Distribution of Human and Yeast DHHC Cysteine-Rich Domain-Containing Proteins. Bio-chimica et Biophysica Acta (BBA)—Molecular and Cell Biology of Lipids, 1761, 474-483. https://doi.org/10.1016/j.bbalip.2006.03.010
- 41. De, I. and Sadhukhan, S. (2018) Emerging Roles of DHHC-Mediated Protein S-Palmitoylation in Physiological and Pathophysiological Context. European Journal of Cell Biology, 97, 319-338. https://doi.org/10.1016/j.ejcb.2018.03.005
- 42. Wang, J., Hao, J.W., Wang, X., et al. (2019) DHHC4 and DHHC5 Facilitate Fatty Acid Uptake by Palmitoylating and Targeting CD36 to the Plasma Membrane. Cell Reports, 26, 209-221. https://doi.org/10.1016/j.celrep.2018.12.022
- 43. Cho, E. and Park, M. (2016) Palmitoylation in Alzheimer’s Dis-ease and Other Neurodegenerative Diseases. Pharmacological Research, 111, 133-151. https://doi.org/10.1016/j.phrs.2016.06.008
- 44. Raymond, F.L., Tarpey, P.S., Edkins, S., et al. (2007) Mutations in ZDHHC9, Which Encodes a Palmitoyltransferase of NRAS and HRAS, Cause X-Linked Mental Retardation Associated with a Marfanoid habitus. The American Journal of Human Genetics, 80, 982-987. https://doi.org/10.1086/513609
- 45. Runkle, K.B., Kharbanda, A., Stypulkowski, E., et al. (2016) Inhibition of DHHC20-Mediated EGFR Palmitoylation Creates a Dependence on EGFR Signaling. Molecular Cell, 62, 385-396. https://doi.org/10.1016/j.molcel.2016.04.003
- 46. Chen, X.R., Hu, L., Yang, H.R., et al. (2019) DHHC Protein Family Targets Different Subsets of Glioma Stem Cells in Specific Niches. Journal of Experimental & Clinical Cancer Research, 38, Article No. 25. https://doi.org/10.1186/s13046-019-1033-2
- 47. Guo, H.L., Wang, J., Ren, S., et al. (2022) Targeting EGFR-Dependent Tumors by Disrupting an ARF6-Mediated Sorting System. Nature Communications, 13, Article No. 6004. https://doi.org/10.1038/s41467-022-33788-7
- 48. Jennings, B.C. and Linder, M.E. (2012) DHHC Protein S-Acyltransferases Use Similar Ping-Pong Kinetic Mechanisms but Display Different Acyl-CoA Specificities. Journal of Biological Chemistry, 287, 7236-7245. https://doi.org/10.1074/jbc.M111.337246
- 49. Mitchell, D.A., Mitchell, G., Ling, Y.P., et al. (2010) Mutational Analysis of Saccharomyces cerevisiae Erf2 Reveals a Two-Step Reaction Mechanism for Protein Palmitoylation by DHHC Enzymes. Journal of Biological Chemistry, 285, 38104-38114. https://doi.org/10.1074/jbc.M110.169102
- 50. Gottlieb, C.D. and Linder, M.E. (2017) Structure and Function of DHHC Protein S-acyltransferases. Biochemical Society Transactions, 45, 923-928. https://doi.org/10.1042/BST20160304
- 51. Montoro, A.G., Ramirez, S.C. and Taubas, J.V. (2015) The Canonical DHHC Motif Is Not Absolutely Required for the Activity of the Yeast S-acyltransferases Swf1 and Pfa4. Journal of Bi-ological Chemistry, 290, 22448-22459. https://doi.org/10.1074/jbc.M115.651356
- 52. Brownlie, R.J. and Zamoyska, R. (2013) T Cell Receptor Signalling Networks: Branched, Diversified and Bounded. Nature Reviews Immunology, 13, 257-269. https://doi.org/10.1038/nri3403
- 53. Gaud, G., Lesourne, R. and Love, P.E. (2018) Regulatory Mechanisms in T Cell Receptor Signalling. Nature Reviews Immunology, 18, 485-497. https://doi.org/10.1038/s41577-018-0020-8
- 54. Hwang, J.R., Byeon, Y., Kim, D., et al. (2020) Recent Insights of T Cell Receptor-Mediated Signaling Pathways for T Cell Activation and Development. Experimental & Molecular Medi-cine, 52, 750-761. https://doi.org/10.1038/s12276-020-0435-8
- 55. Zhang, Y.Q., Qin, Z.R., Sun, W.H., et al. (2021) Function of Protein S-palmitoylation in Immunity and Immune-Related Diseases. Frontiers in Immunology, 12, Article 661202. https://doi.org/10.3389/fimmu.2021.661202
NOTES
*第一作者。
#通讯作者。