Pure Mathematics
Vol.
09
No.
10
(
2019
), Article ID:
33283
,
8
pages
10.12677/PM.2019.910137
Clustering of Cancer Subtypes Based on Similarity Network Fusion of Normalized Euclidean Distance and Spectral Neighbor
Kuan Yang1, Yaping Zhao2
1School of Mathematical Sciences, Ocean University of China, Qingdao Shandong
2Qingdao Foreign Affairs Service Vocational School, Qingdao Shandong

Received: Nov. 14th, 2019; accepted: Nov. 27th, 2019; published: Dec. 4th, 2019

ABSTRACT
Similarity network fusion (SNF) is an effective clustering method to identify cancer subtypes. By using SNF, patient similarity networks for each of their data types are integrated into a single similarity network, which contains all the information of patients. In this paper, a similarity network fusion based on normalized Euclidean distance and spectral clustering (NSSNF) is proposed by using normalized Euclidean distance and redefined neighbor of the patient to reduce the noise of data analysis in similarity networks and increase the complementarity between the data from different similarity networks. Finally, for the five cancer data types from the TCGA database, the data analysis was performed by the NSSNF method, and the results of the evaluation indexes DB and CH showed that the NSSNF method is superior to the SNF method, NSNF method and CSNF method.
Keywords:Subtype, Network Fusion, Normalization, Euclidean Distance, Neighbor, Spectral Clustering, Similarity Network

基于归一化欧氏距离和谱分邻的融合网络对癌症亚型聚类
杨宽1,赵亚萍2
1中国海洋大学数学科学学院,山东 青岛
2青岛外事服务职业学校,山东 青岛
收稿日期:2019年11月14日;录用日期:2019年11月27日;发布日期:2019年12月4日

摘 要
相似网络融合(SNF)是一种确定癌症亚型的有效聚类方法,可以将病人不同数据类型的相似网络融合成一个相似网络,融合后的相似网络包含病人的所有信息。本文使用归一化的欧氏距离和重新定义的病人的邻居,提出了基于归一化欧氏距离和谱聚类分邻的相似网络融合(NSSNF),减少了相似网络中数据分析时产生的噪声,并增加了不同相似网络的数据间的互补性,最后,针对数据库TCGA中的五种癌症数据,利用NSSNF方法进行数据分析,评价指标DB和CH的结果表明NSSNF方法优于传统SNF方法、NSNF方法和CSNF方法。
关键词 :亚型,网络融合,归一化,欧氏距离,邻居,谱聚类,相似网络

Copyright © 2019 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
2018年4月5日,《Cell》公布了泛癌症图谱(Pan-Cancer Altas),其分子特征是20,000多种原发性癌症和匹配的33种癌症类型的正常样本,其中,居于癌症发病率首位的肺癌是一种高度错综复杂的异质性疾病,目前肺癌的精确治疗已发展到新的阶段,已确定EGFR突变、ALK融合等七种不同的肺癌亚型,这七种不同的肺癌亚型需要不同的靶向药物进行精确治疗,然而,肺癌以及其它癌症的治疗也存在着许多问题,如何寻找未知的靶基因,确定未知的癌症亚型还需要更多地努力 [1] [2]。相似网络融合是一种有效确定癌症亚型的聚类方法,可以将病人不同的数据类型整合到一个网络中,通过相似网络融合,病人之间的弱相似性减少,降低了数据分析时的噪声,强相似性增加,增加了不同相似网络的数据间的互补性。2014年由Bo Wang [3] 等人首次提出相似网络融合方法((Similarity Network Fusion) SNF),利用TCGA计划中病人的三种特征DNA甲基化、mRNA表达以及microRNA表达三种数据构建病人的相似融合网络,最后对融合后的网络进行聚类,对比于其它利用单一种数据进行聚类的方法提高了聚类的性能,但也存在着不足,少量的数据样本对应着高维数据,降低数据噪声,扩大数据间的互补性还需要提高。2014年,Jiang Xingpeng [4] 改进了邻居的定义,提出了一致相似网络融合(CSNF),用一致k近邻代替原来的k近邻方法,以此来推断微生物之间的关系,提高了聚类性能。2017年,Zhang Yong [5] 等人提出一种新的基于相似网络融合的多视图聚类算法(RSNF),这种方法结合了随机森林的强度优势以及SNF聚类方法的鲁棒性优势,并将其应用于分析人体微生物数据,改善了SNF的聚类性能。2017年,Zhao Yaping [6] 提出了包含邻居信息的相似网络融合(NSNF),用包含邻居信息的多重紧密k近邻方法代替原来的k近邻方法来构建邻居间的相似网络,将其运用于癌症亚型聚类,与原有方法相比,聚类效果提高很多。2018年,张月 [7] 等人,提出了一种基于一致相似度网络融合的极化SAR图像非监督地物分类方法,修正了误分像素的类别标签,提高了地物分类的精度,和传统极化SAR图像地物分类方法相比,聚类性能有了很大的提高。2018年,Ning Chen [8] 提出了基于利用上下文信息改进SNF的信息检索(CI-SNF),对SNF融合相似性采用Jaccard距离,增强了样本的局部一致性,引入倒排索引技术,提高了聚类的效率,并且将其应用于歌曲识别、图像分类、癌症亚型识别以及药物识别中。
相似网络融合经过这几年有了很大的发展,但是,如何更精确地区分亚型,提高聚类的有效性一直是一个发展中的难题。本文提出了基于归一化欧氏距离和谱分邻相似网络融合方法(NSSNF),用两种方式改进了相似网络融合方法(SNF),一方面,用归一化欧氏距离对病人相似性中的距离重新定义,这样求得的更具有全局性,减少了病人相似数据的噪声;另一方面,用谱聚类分邻的方法重新定义病人邻居间相似网络中的邻居,分邻方法更加科学,同一组邻居强相似性增加,不同组邻居弱相似性减少,增加了数据间的互补性,融合网络更具有全局性,聚类效果优于SNF方法、NSNF方法和CSNF方法。
2. 方法
2.1. 数据来源
数据来源于TCGA网站,本文下载了五种癌症数据:肺癌(LSCC)、肾癌(KRCCC)、肠癌(COAD)、乳腺癌(BIC)和胶质瘤(GBM)。每种癌症数据都包含3种数据类型:DNA甲基化、mRNA表达、miRNA表达。每种癌症详细信息见表1。
Table 1. Detailed data for each cancer
表1. 每种癌症的详细数据
2.2. 相似网络融合
本文提出的方法归一化欧氏距离和谱分邻的相似网络融合(NSSNF)一共有五个步骤:构建病人相似网络、病人相似网络标准化、构建邻居间的相似网络、相似网络融合和对融合网络进行谱聚类。
2.2.1. 构建病人相似网络
假设有 个病人和 种数据类型(比如:DNA甲基化,mRNA表达miRNA表达)。病人的相似网络可以表示成 ,其中顶点集V表示病人 ,边集E表示病人之间相似性程度,病人之间的相似网络可以用 的相似矩阵来表示,其中 表示病人与病人间的相似性,受Danfeng Qin [9] 等人的启发,病人之间的相似性定义如下
, (1)
其中, 的值越大,代表着病人 和病人 相似性越高, 表示超参数并且可以经验性的给出, [3] 是消除缩放的尺度参数,定义如下
, (2)
其中, 表示病人 病人 病人之间归一化欧氏距离,定义为
, (3)
表示病人 和 之间的欧氏距离, 表示距病人 最近的k个邻居归一化欧氏距离的平均值,Ni是指包含病人 在内距它归一化欧氏距离最近的k个病人组成的邻居。
本文用归一化欧氏距离为基础描述这种相似关系,构建病人之间的相似网络更具有全局性,和SNF方法相比,减少了数据噪声。
2.2.2. 病人相似网络标准化
因为要把不同数据类型的相似矩阵融合成一个矩阵,所以要对不同数据类型的相似网络进行标准化 [3]。病人相似网络被重新定义之后,病人相似性具有全局性,病人 和病人 之间的相似性 ,标准化过程定义如下
(4)
从而,可以求出标准化后的病人相似网络 。
2.2.3. 构建邻居间的相似网络
下面通过谱分邻的方法构建邻居间的相似网络,谱分邻就是用谱聚类 [10] 算法定义病人邻居间相似网络中的邻居,步骤如下
1) 矩阵W作为相似性矩阵。
2) 求度矩阵D,其中 。
3) 求拉普拉斯矩阵 。
4) 求L最小的k个特征值对应的特征向量V。
5) 对特征向量V进行K-means聚类。
通过谱分邻方法,n个病人被聚成C组,一般情况下,病人邻居间的聚类数目为 。病人邻居间的相似网络定义如下
(5)
其中, 表示每一组邻居,每一组内的病人之间的相似性很高,不同组病人间的相似性为零,从而,可以求出病人邻居间的 。
本文通过谱分邻方法构建病人邻居间的相似网络,和SNF方法相比,同一组邻居间的相似性更高,不同组邻居间的相似性更低,提高了聚类效果。
2.2.4. 相似网络融合
相似网络融合采用了一种基于消息传递理论的非线性方法 [11],矩阵P包含所有病人的相似信息,而矩阵S只包含病人邻居间的相似信息,将矩阵P作为初始矩阵,矩阵S作为核矩阵进行迭代融合,进行一定次数的迭代,每种数据类型的网络逐渐收敛。
对于一种癌症,有n个病人和3种数据类型(DNA甲基化,mRNA表达和miRNA表达),通过公式(1)、(4)、(5),可以分别计算出病人DNA甲基化的相似网络 、标准化矩阵 和邻居间相似矩阵 ,同理,计算出mRNA表达矩阵 、 和,miRNA表达矩阵 、 和 。令 ,,,,,。迭代步骤如下
, (6)
, (7)
, (8)
其中,t是迭代的次数。经过t次迭代之后,网络融合矩阵可用下列矩阵表示
。 (9)
从而,可以求出包含病人全局信息的融合网络 。
2.2.5. 对融合网络进行谱聚类
包含全局信息的融合矩阵P作为相似性矩阵进行谱聚类 [10],谱聚类的步骤如下:
1) 融合矩阵P作为相似性矩阵。
2) 求度矩阵D,矩阵D是相似矩阵P的度矩阵。
3) 求拉普拉斯矩阵L,其中 。
4) 求出L的最小的k个特征值和对应的特征向量V。
5) 将特征向量V进行K-means聚类。
从而,n个病人被聚成了k个不同的类别。
3. 结果
评价聚类的有效性指标主要有两种。第一种是Davies Bouldin (DB) [12] 指标,DB指标主要描述病人的类内散度与各聚类中心的间距,定义为
, (10)
其中, 和 分别是第i类和第j类的中心点, 和 分别是第i类和第j类的类内数量。从DB指标可以看出DB越小表示类与类之间的相似性越低,从而对应最佳的聚类结果。
第二种是Calinski Harabasz (CH) [13] 指标,CH中类内离差矩阵描述紧密度,类间离差矩阵描述分离度,定义为
, (11)
其中, 是整个数据集的中心。从CH指标可以看出CH越大代表着类自身越紧密,类与类之间越分散,即聚类结果更好。
通过SNF、CSNF、NSNF和NSSNF方法,分别对5种癌症进行了相似网络融合,并计算了4种方法的聚类性能。详细结果见表2,表3。
Table 2. Detailed data for CH Value
表2. 每种癌症CH指标的详细数据
Table 3. Detailed data for DB Value
表3. 每种癌症DB指标的详细数据
对比四种SNF、NSSNF、NSNF和CSNF方法,数据表明NSSNF方法的DB值小于其它三种方法的DB值,NSSNF方法的CH值大于其它三种方法的CH值,因此本文改进的NSSNF方法聚类性能优于其它三种方法。具体对比结果见图1,图2。
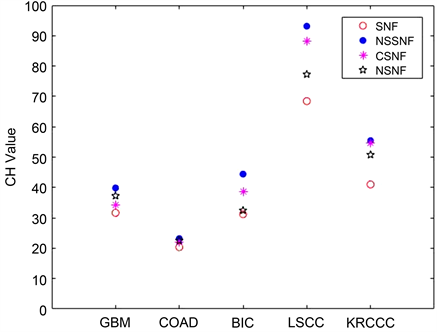
Figure 1. Comparison of CH values of five cancers data
图1. 五种癌症数据CH值的比较

Figure 2. Comparison of DB values of five cancers data
图2. 五种癌症数据DB值的比较
4. 结论
本文采用基于归一化欧氏距离的NSSNF方法,通过谱聚类改进邻居定义,构建患者相似度网络,将不同数据类型的相似性网络融合到一个相似性网络中,然后进行谱聚类。最后,通过DB值和CH值来评价谱聚类的有效性,数据分析结果均表明,NSSNF聚类方法明显优于SNF方法、CSNF方法和NSNF方法。
基金项目
国家自然科学基金项目(11271341)资助。
文章引用
杨 宽,赵亚萍. 基于归一化欧氏距离和谱分邻的融合网络对癌症亚型聚类
Clustering of Cancer Subtypes Based on Similarity Network Fusion of Normalized Euclidean Distance and Spectral Neighbor[J]. 理论数学, 2019, 09(10): 1115-1122. https://doi.org/10.12677/PM.2019.910137
参考文献
- 1. Wu, D., Wang, D.C., Cheng, Y., et al. (2016) Roles of Tumor Heterogeneity in the Development of Drug Resistance: A Call for Precision Therapy. Seminars in Cancer Biology, 42, 13-19. https://doi.org/10.1016/j.semcancer.2016.11.006
- 2. 周彩存, 刘桑田. 肺癌的靶向治疗与精准医学[J]. 医学研究生学报, 2017, 30(11): 1132-1139.
- 3. Wang, B., Mezlini, A., Demir, F., et al. (2014) Similarity Network Fusion for Aggregating Data Types on a Genomic Scale. Nature Methods, 11, 333-337. https://doi.org/10.1038/nmeth.2810
- 4. Jiang, X.P. and Hu, X.H. (2014) Inferring Microbial Interaction Net-works Based on Consensus Similarity Network Fusion. College of Computing and Informatics, 57, 1115-1120. https://doi.org/10.1007/s11427-014-4735-x
- 5. Zhang, Y., Hua, X. and Jiang, X.P. (2017) Multi-View Cluster-ing of Microbiome Samples by Robust Similarity Network Fusion and Spectral Clustering. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 14, 264-271. https://doi.org/10.1109/TCBB.2015.2474387
- 6. Zhao, Y.P., Zhao, X.Q., Wang, K.D., et al. (2017) Clustering Cancer Subtypes Based on Neighborhood-Information Embedded Similarity Network Fusion. Periodical of Ocean University of China, A1, 155-160.
- 7. 张月, 邹焕新, 邵宁远, 等. 基于相似度网络融合的极化SAR图像地物分类[J]. 系统工程与电子技术, 2018, 40(2): 295-302.
- 8. Chen, N. (2019) CI-SNF: Exploiting Contextual Information to Improve SNF Based Information Retrieval. Information Fusion, 52, 175-186. https://doi.org/10.1016/j.inffus.2018.08.004
- 9. Qin, D.F., Wengert, C., van Gool, L., et al. (2013) Query Adaptive Similarity for Scale Object Retrieval. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Portland, 23-28 June 2013, 1610-1617.
- 10. Von Luxburg, U. (2007) A Tutorial on Spectral Clustering. Statistics and Computing, 17, 395-416. https://doi.org/10.1007/s11222-007-9033-z
- 11. Nigro, J.M., et al. (2005) Integrated Array-Comparative Genomic Hybridization and Expression Array Profiles Identify Clinically Relevant Molecular Subtypes of Glioblastoma. Cancer Research, 65, 1678-1686. https://doi.org/10.1158/0008-5472.CAN-04-2921
- 12. Davies, D.L. and Bouldin, D.W. (1979) A Cluster Separation Measure. IEEE Transactions on Pattern Analysis Machine Intelligence, 1, 224. https://doi.org/10.1109/TPAMI.1979.4766909
- 13. Caliski, T. and Harabasz, J. (1974) A Dendrite Method for Cluster Analysis. Communications in Statistics, 3, 1-27. https://doi.org/10.1080/03610917408548446
