Computer Science and Application
Vol.
08
No.
11
(
2018
), Article ID:
27393
,
7
pages
10.12677/CSA.2018.811180
A Nuclei Segmentation Research Based on Convolutional Neural Network
Weidong Mai, Guan Chen, Weijie Ye*
School of Statistics and Mathematics, Guangdong University of Finance and Economics, Guangzhou Guangdong

Received: Oct. 15th, 2018; accepted: Oct. 26th, 2018; published: Nov. 2nd, 2018

ABSTRACT
In the pathological diagnosis of many diseases, the change of the shape and characteristics of the nucleus is an important symptom for the occurrence of the disease. Applying computer intelligence to segment the nuclei in the pathological tissue section can provide more advices for disease diagnosis. In this study, convolutional neural network was applied to the nuclei segmentation of breast cancer histopathological section image. After optical preprocessing the images, each of them was divided into multiple small images and used to train the improved AlexNet model. Then, the trained model is used in the nucleus segmentation of the test set. We divided the whole image into multiple small images, the small images were processed parallelly by the trained model, and finally integrated all the output to a whole nucleus segmentation image. The results show that the nucleus recognition rate in the training set reach to 92%. The trained model can accurately recognize all nuclei which are not labeled in the artificially labeled image, indicating that the model has successfully learned the main features of the nucleus. Finally, the result of image segmentation in test set shows that the trained model successfully segmented the nucleus of pathological tissue slice image accurately and quickly, which proves that our method of cutting image to parallelly process and then integrating all outputs ensures both accuracy and calculation efficiency.
Keywords:Nucleus, Pathological Section, AlexNet, Features Segmentation
基于卷积神经网络的细胞核智能分割研究
麦伟东,陈冠,叶伟杰*
广东财经大学统计与数学学院,广东 广州

收稿日期:2018年10月15日;录用日期:2018年10月26日;发布日期:2018年11月2日

摘 要
在许多疾病的病理学诊断中,细胞核的形状、特征的变化是病变发生与否的重要依据,利用计算机智能分割出病理组织切片中的细胞核能为疾病诊断提供更多的参考。本研究将卷积神经网络应用在乳腺癌病理组织切片图像中的细胞核分割上。在对图像进行光学预处理后,将其分割成多个小图像输入到改进的Alexnet模型中对模型进行训练,使其能自动识别细胞核特征。随后,将训练后的模型用于测试集图像的细胞核分割中,把图像分割成多个小图像让模型并行处理,并最终整合所有的输出结果生成一张完整的细胞核分割图,达到细胞核分割的目的。结果表明,模型对训练集中的细胞核识别率达到92%,训练后的模型对人工标记图像中并没有标记出来的细胞核都能准确地识别出来,表明模型已成功的学习到细胞核的主要特征。最后,对测试集图片进行分割的结果显示,训练后的模型成功地把病理组织切片图像中的细胞核准确且快速地分割出来,证明这种切分图像进行细胞核分割最后再整合的方法在保证准确性的同时也能提高计算效率。
关键词 :细胞核,病理组织切片,AlexNet,特征检测

Copyright © 2018 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
细胞核的病变是许多疾病发生的重要标志之一,尤其是癌症,癌细胞的细胞核较正常细胞有着明显的区别,因此细胞核的性状常常作为病理学诊断的一个重要依据 [1] 。近年来,深度学习算法的发展使得计算机辅助诊断技术的应用能力有了较大的提高,而如何将细胞核这一重要特征既精确又高效地从一些病理学组织切片中分割出来已成为该领域极具挑战的一个重要课题 [2] 。
针对细胞核分割技术,国内外的学者都提出了不少模型。项磊与徐军 [4] 使用方向梯度直方图(Histogram of Oriented Gradient, HOG)以及滑动窗口技术,搭建softmax分类器、HOG以及支持向量机SVM(Support Vector Machine, SVM)等多个模型,以达到对乳腺癌病理切片图像进行细胞检测,其中HOG+SVM模型的准确率达到了71.5%。此外,张敏淑 [5] 针对白细胞图像的特征提取问题,提出了一个基于阈值分割算法的白细胞定位并提取细胞核特征的模型,并利用大律法进一步排除图片其余成分对细胞核检测的干扰,取得了较好的成果,但存在着将细胞核与淋巴细胞混淆的情况。虽然这些研究都对细胞核分割进行了较深入的探讨,但仍存在着正确率较低或精度较差等问题,因此相关研究开始将目光转向深度学习算法。2016年,Xu等 [6] 提出了基于堆叠稀疏自编码器(Stacked Sparse Autoencoder, SSAE)的乳腺癌细胞细胞核检测模型,结果发现模型对细胞核的识别率达88.84%。随后,Win等人 [7] 利用全连接卷积神经网络对细胞学胸腔积液图像进行细胞核分割,并进一步运用形态学运算以及分水岭算法,分割出整个细胞,正确率达到了92%。尽管这些模型的识别率较高,但由于其计算量庞大而使得处理一张图像要耗费较长时间,极不适合实际应用。
本研究针对上述问题,采用切分图像的方法将较大的图像分割成多个小图像,随后根据这些小图像的特点改进经典的卷积神经网络结构Alexnet并进行训练,使得训练后的模型能识别出图像中的细胞核,并最终将所有小图像的结果整合成一张完整的细胞核分割图。结果表明,模型成功地把病理组织切片图像中的细胞核分割出来。把图像分割成多个小图像使得模型能减少计算量,且能并行处理,降低整体的处理时长,而同时又能保持较高的识别率,便于实际应用。
2. 方法
2.1. AlexNet深度学习网络
本研究将由Alex Krizhevsky [8] 提出的卷积神经网络结构Alexnet为基础进行改进来完成特征分类任务。经典的AlexNet由3层卷积+池化层和3层全连接层组成,其中的卷积层都采用5 * 5的卷积核以及池化采用最值池化,最后的分类器采用4096 * 4096 * 1000的全连接层。本次任务因为识别对象为细胞核,故采用的是3 * 3的卷积以步长为1进行卷积,用更小的卷积核是为了捕获到细小的细胞核特征。加上2 * 2步长为3的最大值池化层,分类器选用了两层1024个节点的全连接层,采用更少的全连接节点会更好的避免模型过拟合,最后通过softmax层计算分类结果(图1)。
模型采用线性整流函数(Rectified Linear Unit, ReLU)作为激活函数:

Figure 1. Improved Alexnet Neural Network structure Diagram
图1. 改进的Alexnet神经网络结构图
2.2. 数据集
本文将使用医学研究机构Andrew Janowczyk [9] 提供了一个开源数据集供学者研究使用,里面包含140张2000 * 2000的细胞组织切片及其由专业人士进行科学标记的特征分割图。根据科学标记的数据集产生了大批量用于训练识别特定特征的训练集以及验证集。
2.3. 图片预处理
鉴于不同条件下获取的HE染色病理切片会存在色彩上的出入,对实验结果造成影响,因此在实验开展前我们会对图片进行标准化,从而把色差的影响降低到最小。图片标准化的方法采用Marc Macenko等 [10] 提出的光学变换方法。
第一步,把图片中所有的颜色值转化为光密度(optical density, OD)。第二步,将OD敏感度小于β的删除,此处β值默认设置为0.15。第三步,将第二步得到的结果进行奇异值分解。最后将得到的数据转化为颜色值,得到标准化后的病理切片。
2.4. 细胞核分割
要分割病理图像中的细胞核,一整张的图片输入学习会造成大量计算资源的浪费,也会使计算效率大幅降低。通过观察这些乳腺癌病理学组织切片,可以发现图片中一个细胞核普遍占据了一个直径为3到11个像素的空间,若把图片分割成32 * 32的大小输入学习,能够把切片的各种病理学特征涵盖进去,同时也有利于AlexNet的训练。因此,我们这里给出一个先切分图像并行输入到模型进行识别最后进行整合的方法。在训练阶段,训练集图片已经是32 * 32大小,直接将这些图片输入到模型中进行训练。在应用阶段,使用训练后的模型进行细胞核切割就需要用到先切分再识别最后整合的完整步骤。首先,先对原图的长和宽进行31个像素的全零填充使得最终得到的分割图和原图大小一致,然后以1 * 1为步长将填充完的图片切分成多张32 * 32的局部图片。由于不需要改变模型的参数,所以这些图片可以并行地输入到模型里进行识别,模型输出结果就是局部图片中点坐标对应最终细胞核分割图坐标的RGB值。将所有局部图片处理完后所得到的结构按照坐标排列即可得到最终细胞核分割图的像素矩阵,这个像素矩阵乘255并输出为图片即得到一张细胞核为白色,非细胞核为黑色的细胞核分割图。
3. 结果
3.1. 图片预处理结果
在图片处理阶段,通过光学变换将原本多种特征糅合在一起的细胞组织切片原图变成病理特征突出的图片。由图2的原图部分可以看出在光学显微镜下细胞组织切片颜色过于鲜艳,而且特征模糊并且多种干扰因素掺杂在其中。通过光学变换处理可以明显看出,处理过后的图片中病理学特征十分鲜明可见,例如其中的细胞核轮廓清晰可见,明显区别于细胞质以及小泡;其中的导管部分也更加突出,不会与肌肉组织糅合在一起,显现出明显的导管形状。通过光学变换处理,效果是肉眼可见的,这样的处理对于神经网络的识别更加有利,不仅把我们所需的特征独立呈现,而且把众多的干扰因素排除在外,使得在神经网络的训练中得到比较高的识别率。
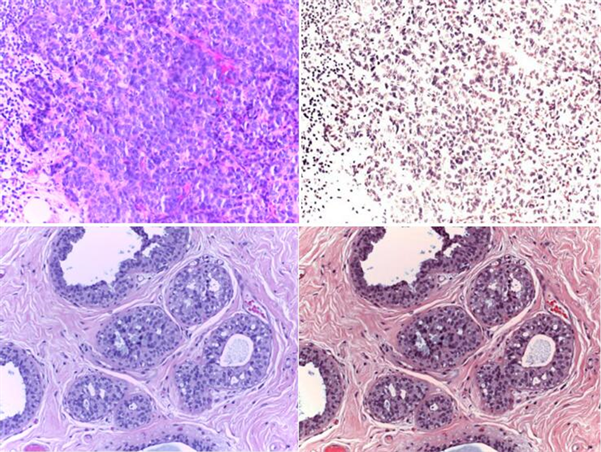
Figure 2. The results of pretreatment. The figures on the left are the original tissue sections, and the figures on the right are tissue sections after optically transforming
图2. 两组图片,左边一组为显微镜下的细胞组织切片图,右边一组经过光学变换的图
3.2. 模型的训练
在通过搭建Alexnet识别细胞核区域和非细胞核区域中,随着训练的进行,在200,000轮训练中,神经网络在每隔50轮训练就对采样集进行识别率统计得到的识别率变化如图3所示,基本已经稳定在85%以上,以及每个批次训练数据的损失值变化图如图4所示,损失值已经下降到足够小,满足进行细胞核的特征分割,最终对于测试集的识别统计中可达到92%的识别率。

Figure 3. Recognition rate sampling diagram of neural network in 200,000 steps training process
图3. 在200,000轮训练中神经网络的识别率采样图

Figure 4. The variation of the loss function per batch in 200,000 trials of training
图4. 在200,000轮训练中每个批次的损失值下降过程
3.3. 用训练后的模型进行细胞核分割
经过训练后的模型可以直接用于组织切片的细胞核分割,将原始图片输入到模型里,会输出一张细胞核特征图,图中只有白色和黑色两种像素分别代表着原图中不同的两个区域:细胞核区域以及非细胞核区域。
首先,我们将训练集中的图片输入测试模型的效果。由图5可以看出,模型有效地把组织切片中的细胞核进行定位识别,并将分割出来的细胞核特征并在输出的双通道特征图片。将经过神经网络分割的细胞核特征图与人工科学标记的细胞核图对比,人工科学标记图片仅标示出其中一小部分的细胞核,而模型的输出结果能把绝大部分的细胞核标示出来,模型已经把细胞核特征学会。由此可知人工科学标记图片不仅耗费大量的时间和人工,而且存在严重的漏标现象,但是使用通过少量的人工科学标记图训练得到的神经网络来分割,不仅极大的提高了效率,而且分割的更为精细。
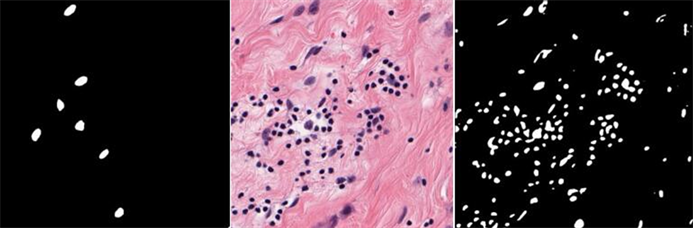
Figure 5. The left chart is the artificially scientific tag map for the tissue section, the middle chart is the original tissue section, and the right chart is the corresponding output of the neural network
图5.左边图为科学标记图,中间为原图,右边为神经网络分割图
最后,我们用训练后的模型去分割测试集中的组织切片图的细胞核。由于测试集中的图片并没有输入到模型中进行训练,所以其输出结果是对模型性能的直接验证。在图6的对比图中可见,模型成功地将切片图中的细胞核分割出来,图中的白色部分对应原图中的细胞核部分,可见效果图与科学标记图有着同样的分割效果,都将细胞核从一张复杂的细胞组织切片图中分割出来了。通过分割图可以轻松的定位到细胞核并且了解到细胞核的形状大小等等信息,都是对于疾病诊断提供十分重要的依据。
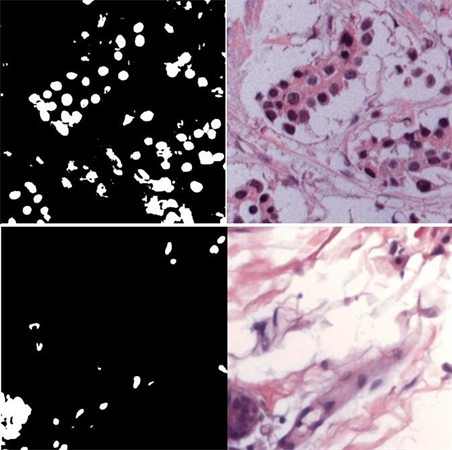
Figure 6. The test set segmentation results of the trained neural network, the left charts are the segmentation results, and the right charts are the original tissue sections
图6. 训练后的网络对测试集的分割结果,左边一组为切割完成的特征图,右边一组为原图
4. 结论
本文训练了一个改进的AlexNet卷积神经网络模型对乳腺组织切片图像中的细胞核进行了识别和分割。得益于卷积神经网络的特点,该模型能够准确地识别细胞核的特征,成功地将训练集和测试集的切片图中的细胞核分割出来。由于细胞核病变是乳腺癌的一个重要特征,本研究所得的细胞核分割图将会有助于对乳腺癌的诊断,这也是本研究的下一步工作:将各种乳腺癌组织切片特征提取出来并用于构建乳腺癌分类模型。此外,本研究中的细胞核分割模型不仅可以用于乳腺癌组织切片中,也可以被用于其他疾病的组织切片,提取对应的细胞核特征。
基金项目
本研究由国家自然科学基金(NO.11702064)和国家创新训练项目(NO.201810592004)支持。
文章引用
麦伟东,陈 冠,叶伟杰. 基于卷积神经网络的细胞核智能分割研究
A Nuclei Segmentation Research Based on Convolutional Neural Network[J]. 计算机科学与应用, 2018, 08(11): 1643-1649. https://doi.org/10.12677/CSA.2018.811180
参考文献
- 1. 杨槐. 乳腺癌病理学诊断研究[J]. 局解手术学, 2011, 20(2): 177-179.
- 2. 邓杨, 包骥. 数字病理中计算机辅助诊断研究展望[J]. 实用医院临床杂志, 2017, 14(5): 10-12.
- 3. 蔡海洋. 胃腺癌病理切片CAD系统的研究与实现[D]: [硕士学位论文]. 西安: 西安电子科技大学, 2008.
- 4. 项磊, 徐军. 基于HOG特征和滑动窗口的乳腺病理图像细胞检测[J]. 山东大学学报(工学版), 2015, 45(1): 37-44.
- 5. 张敏淑. 白细胞图像的特征提取与分类算法研究[D]: [硕士学位论文]. 杭州: 中国计量大学, 2016.
- 6. Xu, J., Xiang, L., Liu, Q., Gilmore, H., Wu, J., Tang, J. and Madabhushi, A. (2016) Stacked Sparse Autoencoder (SSAE) for Nuclei Detection on Breast Cancer Histopathology Images. IEEE Transactions on Medical Imaging, 35, 119-130. https://doi.org/10.1109/TMI.2015.2458702
- 7. Win, K.Y., Choomchuay, S., Hamamoto, K. and Raveesunthornkiat, M. (2018) Artificial Neural Network Based Nuclei Segmentation on Cytology Pleural Effusion Images. International Conference on Intelligent Informatics and Biomedical Sciences, Okinawa, 24-26 November 2017, 245-249.
- 8. Krizhevsky, A., Sutskever, I. and Hinton, G.E. (2012) ImageNet Classification with Deep Convolutional Neural Networks. International Conference on Neural Information Pro-cessing Systems, Curran Associates Inc., 1097-1105.
- 9. Janowczyk, A. and Madabhushi, A. (2016) Deep Learning for Digital Pa-thology Image Analysis: A Comprehensive Tutorial with Selected Use Cases. Journal of Pathology Informatics, 7, 29-47. https://doi.org/10.4103/2153-3539.186902
- 10. Macenko, M., Niethammer, M., Marron, J.S., Borland, D., Woosley, J.T., Guan, X., Schmitt, C. and Thomas, N.E. (2009) A Method for Normalizing Histology Slides for Quantitative Analysis. IEEE International Symposium on Biomedical Imaging: From Nano to Macro, Boston, 28 June-1 July 2009, 1107-1110.
NOTES
*通讯作者。