Hans Journal of Computational Biology
Vol.4 No.02(2014), Article
ID:13644,6
pages
DOI:10.12677/HJCB.2014.42002
Based on BioPerl Realize Accurately Download LEA Gene Sequences from the NCBI
School of Life Sciences, Xinjiang Normal University, Urumchi
Email: 313741033@qq.com, #379483304@qq.com
Copyright © 2014 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



Received: Apr. 11th, 2014; revised: Apr. 18th, 2014; accepted: Apr. 22nd, 2014
ABSTRACT
Recently, researchers have paid more and more attention to the resistance gene research; in Xinjiang, especially the research of drought resistance gene has attached great importance. Based on these factors, according to the conservative domain structure of LEA gene (late embriogenesis abundant gene, LEA) and the corresponding keywords, this paper designed a program that LEA gene sequences was downloaded accurately from NCBI based on the BioPerl. This procedure not only solves the precise acquisition of LEA gene, but also provides a better solution to download different types of sequence exactly.
Keywords:BioPerl, Conservative Structure Domain, Feature List, Key Words, LEA Gene

基于BioPerl实现从NCBI中精确下载
LEA基因序列
张晓婧*,曹兴芹,潘伟民#
新疆师范大学生命科学学院,乌鲁木齐
Email: 313741033@qq.com, #379483304@qq.com
收稿日期:2014年4月11日;修回日期:2014年4月18日;录用日期:2014年4月22日

摘 要
近年来关于抗逆基因的研究,越来越受研究者们的关注,在新疆尤其重视抗干旱基因的研究。基于这些因素,本文根据LEA基因(late embriogenesis abundant gene, LEA)的保守结构域所在片段(下文简称保守结构片段)和相应的关键词,基于BioPerl设计了从NCBI中精确下载LEA基因序列的程序。此程序不仅解决了LEA基因的精确获取,同时也为不同类型序列的精确下载,提供了一种较好的解决办法。
关键词
BioPerl,保守结构域,特征表,关键词,LEA基因

1. 引言
随着人类基因组计划的完成,生物数据急剧增长,海量的数据已经不是传统生物实验所能分析的,需要借助生物信息学对生物数据进行搜集、分析,为了更好的研究生物数据,构建二次生物数据库是必不可少的,但构建二次生物数据库的前提就是,需要获取准确、完整的生物数据,以往人们是从NCBI中手动获取数据,或者是实验积累过程中发现的数据,这些工作都比较费时费力,是不可行的,但是利用生物信息学实现大规模数据的获取是非常便利的。BioPerl是Perl为生物学提供的专业处理生物数据的软件包[1] ,不仅可以从本地或远程数据库获取序列,还可以对序列进行各种处理功能,对于现今大量的生物数据来说,这是一个非常专业、便利的工具。
在新疆地区,绿洲面积仅约占全区面积的5%,适宜植物生长的土地资源非常稀少, 植物不仅面临干旱、盐碱胁迫,还面临着高温、低温等非生物胁迫,这些因素或多或少会制约农业经济的发展,因此抗逆基因的研究,对于新疆地区或是全球来说都具有重要的意义,而要研究抗逆基因,构建抗逆基因二次数据库是或不可缺的。晚期胚胎发生丰富蛋白(LEA)存在于大部分生物体中,它是一类与渗透调节有关的家族蛋白[2] ,当生物体受到干旱、低温、盐胁迫等环境胁迫时,LEA基因会在生物体中大量积累[3] -[5] ,保护生物对抗非生物胁迫,简单的说LEA基因就是一种抗逆基因。精确、完整地下载LEA基因序列数据是构建抗逆基因二次数据库的一部分关键工作,也能为需要精确序列的工作研究奠定良好的基础,并且这种搜索下载序列的方法模式,能为以后这方面的研究做一个参考。
本程序基于bioperl,用Perl语言结合保守结构片段及它所对应的关键词,来精确检索LEA基因,并将其下载下来,打破了以往模糊下载的模式[6] -[8] ,提供了一种更精确、更可靠的方法来远程获取序列数据,不仅可用于数据库的构建,也可用于平时搜索数据使用,为生物学家带来便利,对于生命科学的研究有重要意义。
2. 程序方法设计
2.1. 匹配条件选择
一般要达到精确检索的这个目的,匹配条件对于目标序列来说一定是唯一的,即根据这个匹配条件只能确定目标序列,而不能确定别的序列,这样才能做到精确下载。从大量文献中可以发现,LEA基因被分为七个族,每个族都有各自的保守结构域[9] [10] (详见参考文献[9] [10] ),而保守结构域就是指在生物进化或者一个蛋白质家族中具有不变或相同的结构域,他们不能被改变,由保守结构域的定义可以知道每个LEA基因族所具有的保守结构域是不变的,这样刚好构成了本文所需要的匹配条件,只要在程序中用保守结构片段筛选LEA基因序列,就能达到精确下载的目的。因此本文将LEA基因的保守结构片段作为本程序的匹配条件。
2.2. 生物特征与程序之间的中间媒介
由于此程序的匹配条件用到的是LEA基因的保守结构片段,这个属于生物特征,要想与计算机程序联系起来,就必须需要一个中间媒介,来将两者联系起来。GenBank格式中有一个非常重要的部分,就是特征表(FEATURES)部分,它用大量的词汇来描述核酸序列的结构、功能等大量重要的信息,并巧妙的处理它们,它具体对以下信息进行描述:执行一个生物学功能;影响或是一个生物学功能表达的产物;与其他分子之间的相互作用;影响一个序列的复制;影响或是不同序列重组的结果;是一个可识别的重复单元;有第二级或第三级结构;显示变异,或有被修改。
可以看出特征表(FEATURES)中基本上包含了基因的所有信息,当然也包含了本文需要的中间媒介,这就是“translation”这个标签,在此标签中能将基因序列翻译成蛋白质序列,因为保守结构片段是蛋白质序列,所以刚好能与此标签联系起来,作为此程序中联系生物特征的中间媒介。程序只需要在这个标签中来匹配保守结构片段,就能精确的在NCBI中查找LEA基因。以下是从BioPerl网站的HowTo中引用的标签介绍表,及特征表(FEATURES)格式[11] :(图1,图2)。
2.3. 缩小检索范围
本程序设计初期时,是希望将特征表中含有“CDS”主标签的序列先检索出来(因为“CDS”主标签包含“translation”标签),但是由于NCBI中的数据量太过于庞大,检索时导致内存不足,无法实现,所以后期笔者进行了LEA基因族关键词筛选,将筛选出的关键词与保守结构片段结合起来检索,这样便可起到缩小检索范围的作用,不至于导致内存不足,程序无法执行。
LEA基因族被分类以来,每一个家族都有自己特定的名称,并不是都称为LEA,本文为了得到可靠准确的关键词,对LEA基因族保守结构片段进行blastp,在得出的众多同源序列中,手动筛选,发现每一个族的同源序列中都有一个固定的名称,即每个LEA基因家族特有的名称,例如,LEA2家族,研究者一般都不把它称为LEA或LEA2,而是称为dehydrin(脱水素),在很多文献中也有介绍过(详见参考文

Figure 1. Tag examples of the feature table (belong to the BioPerl’s HowTo)
图1. 特征表中的标签例子(引用于BioPerl网站中的HowTo文档)
献[9] [10] ),准确性可以保证。由此能将每个族的固定名称作为检索数据库时的关键词条件,这里的检索是模糊检索,并不能精确的检索到LEA基因(因为用关键词检索NCBI时是全文检索,即只要L-E-A三个字母挨在一起就会被检索出,准确率比较低),所以要将关键词与保守结构片段结合起来查询下载,才可完成本程序的目的。
表1为保守结构片段与关键词结合列表。
本文以LEA2中的“SSSSSEDD”这个保守结构片段为例,来介绍程序(其他族的程序与此类似,只需将对应的关键词及保守结构片段换掉即可)。
2.4. 程序流程设计
程序流程设计如图3。程序首先根据相对应的关键词进行模糊检索,并将这些序列下载下来,程序开始读入序列文件,每次读取1个序列(next_seq),取得序列成功后,程序指向序列的FEATURES部分(get_SeqFeatures),首先判断此特征表中是否有“CDS”主标签,如果有就继续判断是否有“translation”标签,有就获取其值,并将此值与保守结构片段($val)进行匹配,匹配成功便将此序列下载下来,并打印其display_id,如果不匹配则读取下一条序列,如此反复循环,最终将所有符合条件的LEA基因下载下来。图3中虚线框部分可以替换,以适应不同特点的序列的获取。
3. 程序运行环境及核心代码
3.1. 程序的运行环境
程序环境:Windows XP + ActivePerl 5.16.1 Build + BioPerl 1.6.1,以上的安装配置均参照BioPerl网站中Installing BioPerl on Windows文件[12] 。
3.2. 获取LEA2程序的核心代码
如图4所示,是本程序的核心代码。程序第一步是利用LEA2族关键词“dehydrin”来缩小检索范围,

Figure 2. Feature example
图2. 特征表的例子
表1. 保守结构片段与关键词结合列表
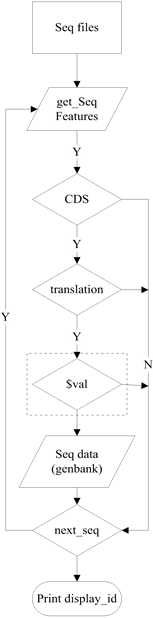
Figure 3. Flow sheet of program
图3. 程序流程图
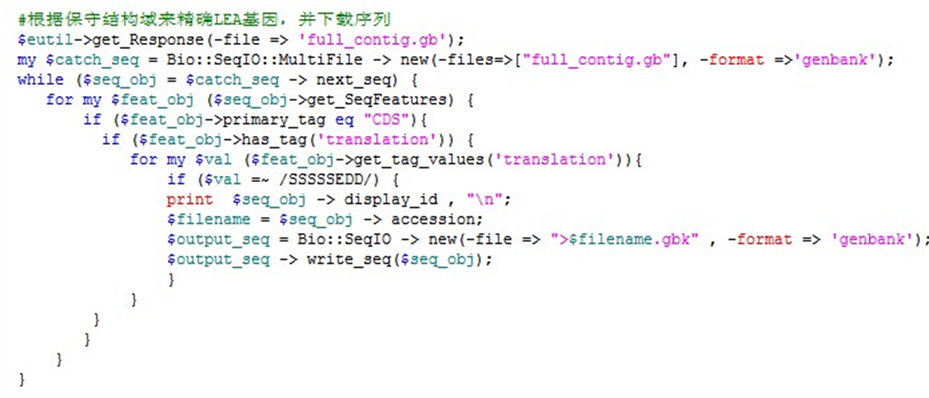
Figure 4. The core code of program
图4. 程序核心代码
并将此检索到的序列下载下来,存为“full_contig.gb”这个文件;
第二步,利用LEA2中“SSSSSEDD”这个保守结构片段来匹配上一步检索到的序列是否符合条件[13] ,如果匹配成功就将其下载下来,并以其“accession号”来命名序列文件,匹配不成功,便再次进入循环,直到匹配成功,最后便完成了精确下载LEA2基因的任务。
4. 程序的实现及结果
将此程序在命令符中运行,执行Perl脚本,等待几分钟后,LEA2基因成功下载到本地文件中,并以其“Accession”编号为文件名存储。以“SSSSSEDD”为例的程序共下载到294条LEA2基因序列,一一查看过,都是准确的。为此本程序成功实现了根据LEA基因保守结构域,精确远程下载LEA基因序列。
5. 讨论
从运行的结果可以证实,程序能从NCBI中有效精确地下载到LEA基因序列数据,并且在精确度方面有显著的优势。同时,在程序设计时,用变量$val来表示保守结构片段,这样做一方面可以进行保守结构片段的替换,且在不输入值的情况下,程序也可以很好的执行;另一方面,能大大提高此程序的适用性,根据别的基因蛋白质序列特征来替换$val变量,便可以精确下载到指定基因序列(这里需要将关键词也换成对应关键词)。还有一点需要强调的是,本文在查找LEA基因关键词时,用到blast程序,很多读者可能会有这样的疑惑,直接用保守结构域+blast查找不是也可以吗?这里笔者也做了一个对比,此程序相比这个方法有以下几点优势:首先一点,此方法是自动获取的,运行程序便可获取到LEA基因,而blast的方法只是检索不能下载;其次,笔者试了一下用blast的方法查找,将结果与程序得到的结果相对比,程序的结果包含blast结果,blast只检索出50条序列,也就是说本文提供的程序可以下载到更全面的序列。
现在交叉学科很多,在做这方面的研究时,应该充分利用两门学科中的优势,并剖析其中的深层联系,就像此程序中,将生物描述的基因特征与计算机形式描述的基因特征结合起来,做到了以往所不能达到的基因精确下载,说明利用两门学科中紧密联系的部分,是在做交叉学科研究时,必须具备的能力和思维方式。且从文章中能看出,genbank格式中的特征表(FEATURES)对于处理序列来说非常重要,它其中包含的都是序列最重要的信息,不管是在生物特征分析,还是更深层次的研究上,都可以用到这块内容,并且特征表是生物与计算机学科最好的连接媒介,在将来研究这块领域时,应该好好地利用特征表中的信息,挖掘出其中更加有用的信息及特征[14] [15] ,相信在不久的将来一定能编写出更加方便,具有意义的生物程序,推进生命科学的研究。
项目基金
自治区自然科学基金(批准号:2010211022)资助项目,新疆师范大学研究生科技创新基金资助项目,(项目编号:20131203)。
参考文献 (References)
- [1] BioPerl操作指南. http://www.bbioo.com
- [2] 白永琴, 杨青川 (2009) LEA蛋白研究进展. 生物技术通报, 9, 1-5.
- [3] 李剑, 赵常玉, 张富生等 (2010) LEA蛋白与植物抗逆性. 植物生理学通讯, 11, 1101-1108.
- [4] 杨天旭, 汪耀富, 宋世旭等 (2006) 逆境胁迫下植物LEA蛋白的研究进展. 干旱地区农业研究, 11, 120-124.
- [5] 孙海丹, 兰英, 刘昀等 (2004) LEA蛋白质11-氨基酸基序与植物抗旱性. 东北师大学报自然科学版, 9, 85-90.
- [6] 向福, 余龙江, 栗茂腾等 (2005) 用bioperl实现种子植物18srRNA基因序列的大规模获取. 华中农业大学学报, 24, 330-333.
- [7] 向福, 余龙江, 陈悟等 (2004) 基于Bioperl的基因序列获取的程序设计与实现. 生物技术, 14, 64-66.
- [8] 白琳 (2012) 植物抗逆基因资源平台的构建与分析. 浙江大学生命科学学院, 7-9.
- [9] 刘洋, 刑鑫, 李德全等 (2011) LEA蛋白的分类与功能研究进展. 生物技术通报, 8, 36-43.
- [10] 李乐, 许红亮, 杨兴露等 (2011) 大豆LEA基因家族全基因组鉴定、分类和表达. 中国农业科学, 5, 3945-3954.
- [11] BioPerl. HowTo. http://www.bioperl.org/wiki/HOWTOs
- [12] BioPerl. Installation. http://www.bioperl.org/wiki/Installing_BioPerl
- [13] Phoenix, T., 等著 (2012) 盛春译. Perl语言入门(第六版). 东南大学出版社, 130-179.
- [14] 许丹, 朱斐等 (2013) 从PubMed数据库中挖掘生物医学中的十大热点话题. 计算机与现代化, 1, 192-199.

NOTES
*第一作者。
#通讯作者。
