Hans Journal of Computational Biology
Vol.05 No.02(2015), Article ID:15600,11
pages
10.12677/HJCB.2015.52003
Analysis of Circular RNA in Caenorhabditis elegans
*通讯作者。
Junwu Liu, Lingling Chen*
College of Informatics, Huazhong Agricultural University, Wuhan Hubei
Email: *llchen@mail.hzau.edu.cn
Received: Jun. 11th, 2015; accepted: Jun. 25th, 2015; published: Jun. 30th, 2015
Copyright © 2015 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



ABSTRACT
Exonic circular RNA (ecircRNA) is a class of special RNA molecule which is processed from pre- mRNA through back-to-splice. Recently, a lot of researches identified thousands of circular RNA in eukaryotes and found that circRNA could function as gene regulator. In our research, we predicted 1112 canonical circular RNAs in RNase R treated RNA-seq from different development stages of Caenorhabditis elegans. We discovered that these circular RNAs had developmental specificity, and had no obvious correlation with linear RNA. Through analyzing the sequence information of flanking introns, we inferred that circular RNAs in C. elegans were produced by intron- pairing-driven circularization model.
Keywords:Exonic Circular RNA, C. elegans, Developmental Specificity, Intron-Pairing-Driven Circularization

线虫环状RNA分析
刘骏武,陈玲玲*
华中农业大学信息学院,湖北 武汉
Email: *llchen@mail.hzau.edu.cn
收稿日期:2015年6月11日;录用日期:2015年6月25日;发布日期:2015年6月30日

摘 要
外显子环状RNA (ecircRNA)是前体信使RNA中外显子经过反向剪接首尾相连而成环的一类特殊的RNA分子。近期多项研究发现,这类环状RNA广泛存在于真核生物细胞中,并具有调控基因表达的重要功能。本文根据线虫不同生长发育阶段的RNA测序数据,共预测出1112个典型的外显子环状RNA。对这些环状RNA进行分析,发现它们具有生长发育阶段特异性,并同线性的mRNA分子不具有直接的相关性。通过分析环状RNA侧翼内含子序列信息,证明在线虫中环状RNA的形成支持内含子配对驱动环化模型。
关键词 :外显子环状RNA,线虫,生长发育特异性,内含子配对驱动环化

1. 引言
外显子环状RNA指以pre-mRNA的外显子为主体,通过反向剪接的方式而头尾相连的一类特殊RNA分子(图1)。外显子环状RNA最先发现可由人类的Ets-1[1] [2] 和小鼠的SRY[3] 基因产生,但是由于这一类环状RNA分子的表达量较低并且对它们的生物学功能缺乏足够的认识,因此被认为是由于剪切错误而形成的产物[4] 。随着高通量测序技术和生物信息学的发展,在多种人类细胞组织[5] -[8] 、小鼠[8] 、果蝇[9] 、线虫[8] 等真核生物中以及古细菌[10] 中预测出大量这类不带poly(A)尾的环状RNA分子。近年研究表示,一些环状RNA可以作为miRNA分子海绵而大量结合miRNA分子[8] [11] ,从而阻碍miRNA对靶基因的调控作用,为一类新型的竞争性内源RNA (competing endogenous RNA, ceRNA)。
环状RNA的3'端和5'端共价闭合,缺乏poly(A)尾巴,一般认为不具有编码蛋白质功能。在细胞内比较稳定,并对核酸外切酶不敏感,可结合miRNA分子也可被miRNA切割降解[12] 。外显子环状RNA通常定位于细胞质中,并认为环状RNA在形成过程中同时会将内含子剪切掉。而最近的研究表明,一些环状RNA的内含子会保留下来,并在细胞核中行使增强转录起始的功能[13] 。
对于环状RNA的产生机制,Jeck等[5] 提出了环状RNA形成的两种模型:套索驱动的环化(lariat-driven circularization)和内含子配对驱动环化(intron-pairing-driven circularization)。这两种模型的区别在于:套索驱动的环化先由环状RNA两侧外显子的剪接供体SD(splice donor)和剪接受体SA(splice acceptor)共价结合形成套索结构,然后剪接体切除剩余内含子形成circRNA;而内含子配对驱动的环化则先由两侧内含子互补配对结合,然后剪接体切除内含子形成环状RNA。近期的相关研究中[14] -[16] ,都支持了内含子配对驱动环化这一模型。

Figure 1. Formation of circular RNA
图1. 环状RNA的形成
本文以模式生物线虫为例,通过经核酸外切酶RNase R富集后的RNA-seq数据,重新预测了线虫中存在的环状RNA,并具体分析了环状RNA的一些基本特征,环状RNA同线性转录本的关系,环状RNA侧翼内含子序列间反向互补配对情况,环状RNA作为miRNA分子海绵的可能性,并对产生环状RNA的基因进行GO功能注释。以期为其他物种环状RNA及非编码RNA的研究提供一些参考信息。
2. 材料和方法
2.1. 数据来源
线虫RNA-seq数据来自NCBI GEO datasets数据库中的GSE63823 [16] ,为一组线虫不同生长发育阶段:胚胎期(embryo)、幼虫期(L1~L4)、成虫期(adult)的total RNA(rRNA-deplete)及对应时期经核酸外切酶RNase R处理后的RNA-seq数据,测序数据信息统计如表1所示。
线虫参考基因组(Ce6)及基因组注释信息从数据库UCSC[17] 中下载得到。
线虫miRNA成熟体序列从miRNA收录数据库miRBase (v21) [18] 中下载得到。
2.2. 数据分析方法
2.2.1. 数据预处理
使用FastQC (v0.10.1)(http://www.bioinformatics.bbsrc.ac.uk/projects/fastqc/)对测序数据进行质量检测,确保质量可以进行后续的分析;如果测序质量不好,可用Trimmomatic (v0.30)[19] 进行质量控制。
2.2.2. 环状RNA的预测
本文使用find_circ流程[8] 对线虫环状RNA进行预测。find_circ流程首先将不能和基因组比对上的读段两端各取20 bp作为锚点,再将锚点作为独立的读段往基因组上比对并寻找唯一匹配位点,如果两个锚点的比对位置在线性上方向呈反向,就延长锚点的读段,直至找到环状RNA的接合位置,若此时两侧的序列分别为GT/AG剪接信号,则判断为环状RNA。相关程序可在circBase[20] 数据库中下载得到。预测步骤如下:
Table 1. The RNA-seq datasets of C. elegans
表1. 线虫RNA-seq数据集
1) 基因组建索引:bowtie2-build genome.fa genome.fa(bowtie2v2.2.1)。
2) 安装python包:numpy (http://www.numpy.org/)和pysam (https://pypi.python.org/pypi/pysam)。
3) 建立一个文件夹genome,里面存放基因组中各个染色体的fasta文件,文件命名需同基因组fasta文件里的id相同。
4) 将处理后的读段比对到参考基因组上并将结果排序和转换成BAM格式:bowtie2 –p 20 - -very-sensitive --score-min=C,-15,0 –q –x genome.fa -1sample_1.fastq -2 sample_2.fastq 2>log/sam- ple.bowtie2.log| samtools view -hbuS - |samtools sort – sample。
5) 取出未匹配上的读段:samtools view -hf 4 sample.bam|samtools view -Sb -> BAM/unmapped_sample. bam。
6) 将未匹配的读段两端各取20 bp作为锚点序列:python unmapped2anchors.py BAM/unmapped_ sample.bam|gzip>gzip/sample_anchors.qfa.gz。
7) 将锚点同基因组比对,并得到可能的环状RNA位点:bowtie2 –p 23 --reorder --score-min=C,-15,0 -q -x genome.fa -U gzip/sample_anchors.qfa.gz |python find_circ.py -G genome -p sample -s log/find_ circ.log >bed/ sample.sites.bed 2>bed/sample.sites.reads。
8) 筛选候选环状RNA:grep circ bed/sample.sites.bed | grep -v chrM | python sum.py -2,3 |python scorethresh.py -16 1|python scorethresh.py -15 2|python scorethresh.py -14 2 |python scorethresh.py 7 2 |python scorethresh.py 8,9 35 |python scorethresh.py -17 100000 > bed/sample.circ_candidates.bed。
2.2.3. 表达量统计
Cufflinks (v2.2.0) [21] 是一款常用的转录本组装工具,它利用Tophat (v2.0.11) [22] 比对的结果来组装转录本,估计这些转录本的丰度并且可以检测样本间的表达差异及可变剪接。
利用FPKM[23] (Reads per kilobase of exon model per Million mapped reads)归一化各个转录本的表达量。
FPKM计算公式:

流程如下:
1) 基因组建索引:bowtie2-build genome.fa genome.fa;
2) Tophat比对:tophat -a 8 -m 0 -I 300000 -p 20 --library-type fr-unstranded --no-novel-indels --segment- mismatches 2 -G genome.gtf -o tophat/sample genome.fa sample.fastq
3) Cufflinks组装转录本并估计表达量:cufflinks -p 20 -G genome.gtf --library-type fr-unstranded -o cufflinks/sample tophat/sample/accepted_hits.bam
我们选取Y
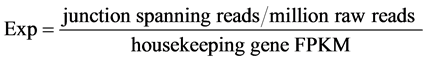
2.2.4. miRNA结合位点统计
我们使用滑动窗口的方法确定线虫各个miRNA在环状RNA上的结合位点数,窗口大小为7 bp,窗口的滑动步长为1 bp,先用perl脚本提取线虫成熟体miRNA的种子序列(第2~8 nt),然后每滑动一次窗口,判断该窗口是否同种子序列反向互补,若反向互补,则判定为该miRNA的一个结合位点。
2.2.5. 内含子反向互补配对
根据内含子互补配对驱动环化模型,我们使用blast将环状RNA的上下游内含子序列进行比对,为了尽可能使blast 2.2.29+结果倾向于更长的匹配,我们设定参数为:-ungapped -word_size 6 -penalty -1,编写perl脚本统计内含子内部和内含子间的反向互补配对数目。
3. 结果与分析
3.1. 线虫环状RNA的基本特征
我们共鉴定出1531个环状RNA,舍去79个位于线虫基因组rRNA簇chrI:15,060,128~15,071,020上的环状RNA,共1452个作为候选的circRNA。根据circRNA的供体和受体所处的位置(图2),统计得出:
1) canonical:circRNA的起始和终止点都在相同基因的外显子边界上,共1112个;
2) alt_donor:circRNA的起始位点在外显子边界上而终止位点不在,共68个;
3) alt_acceptor:circRNA的终止位点在外显子边界上而起始位点不在,共65个;
4) antisense:circRNA与基因区重叠且处于反义链上,共20个;
5) intergenic:circRNA与基因间区重叠,共162个;
6) other:其他情况,共25个。
在这1452个环状RNA中,仅119个在线虫的各个时期中均有表达。在某一特定时期表达的环状RNA数目相对于其他时期而言,具有相同环状RNA的比例都不高(表2),说明线虫环状RNA在不同生长发育阶段会表达不同的环状RNA,具有生长发育阶段特异性。
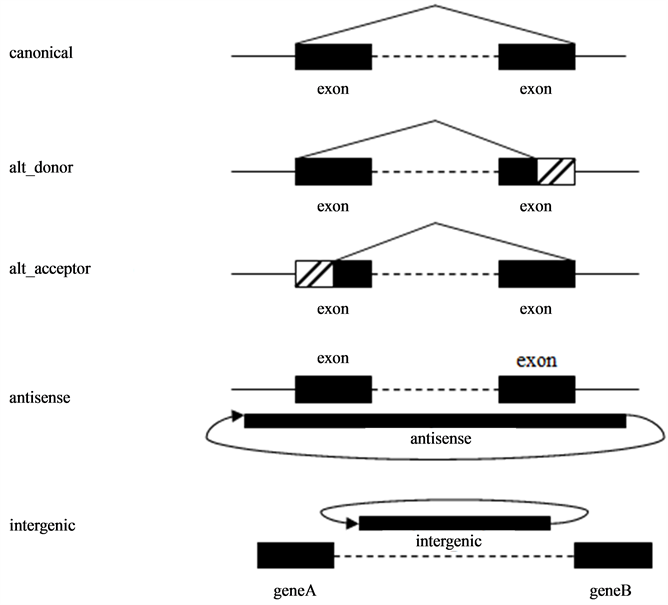
Figure 2. Type of circular RNA
图2. 环状RNA的类型
Table 2. The number of the same circular RNA among different stages
表2. 各个生长时期间相同的环状RNA数目
此外,我们将预测出的1112个canonical环状RNA映射回基因上,由于一个基因的选择性剪切会形成多个不同组成的转录本,我们无法判断环状RNA真实来自于哪一个转录本,所以我们假设只要含有该环状RNA边界外显子的转录本都会形成,那么我们一共可以对应到2056个转录本。我们对这些环状RNA使用的外显子数目和长度分布进行统计,可以看到环状RNA由中间外显子组成的比例最大(90.08%),并且值得注意的是由第一个外显子的数目组成的环状RNA有203个(图3(a)),这个数目和比例远远超过了人类环状RNA的情况,而环状RNA形成的两个模型中也并没有描述由第一个外显子形成环状RNA的情况;由两个外显子组成的环状RNA数目最多,其次为由三个外显子和一个外显子组成的情况(图3(c)),包含外显子越多的环状RNA数目越少,最多的环状RNA可由12个外显子构成(cel_circ_F08B
我们将环状RNA对应的转录本和环状RNA之间的表达量进行相关性分析,发现在97个在所有时期都表达的canonical环状RNA中,仅有11个环状RNA同其线性转录本相关系数大于0.8,1个小于负0.8。表明多数环状RNA同线性RNA表达量间并没有明显的相关性,环状RNA并不是简单地由线性RNA中的一部分形成,它还受到其他多种因素的调控,呈现出复杂的调控机制。
3.2. RNase R对环状RNA丰度的影响
RNase R是一种3' -> 5'核酸外切酶,可以切割线性的RNA分子,因此认为它在mRNA降解过程中扮演着重要的角色[25] 。在环状RNA的研究中,认为环状RNA是对RNase R不敏感的,因此RNase R的处理会对环状RNA起富集作用,并且为判定RNA是否成环的一个重要条件。在本文的实验中,我们通过对比前后经过RNase R处理的RNA-seq数据,虽然RNase R处理后的RNA-seq数据深度提高了,但经过归一化比较后发现大部分环状RNA在经过RNase R处理后的确是富集了,但是仍有一部分没有富集,还有一小部分环状RNA对RNase R敏感(图4)。
在对RNase R敏感的环状RNA中,大部分是intergenic类型,图4所展示的chrX: 925,367~926,302是唯一表现出敏感的canonical类型的环状RNA。而目前尚没有关于intergenic类型环状RNA的研究,故不能确定这一类环状RNA是否真的存在并对RNase R敏感还是软件预测的假阳性。
3.3. 环状RNA的多样性和可选择性
环状RNA可以由一个外显子或多个外显子构成,一个基因中也会有形成多个环状RNA的情况:环

Figure 3. Basic feature analysis of circular RNAs in C.elegans
图3. 线虫环状RNA的基本特征分析

Figure 4. Read counts variation before and after RNase R treatment: enrichment, sensitive and medium
图4. RNase R处理前后环状RNA读数变化:共富集、敏感、中等三种情况
状RNA可能由基因的不同外显子组成,也可能供体或受体为相同的外显子,也可能多种情况在一个基因中同时发生,不同的可变剪接体间形成的环状RNA还会竞争外显子等情况(图5)。环状RNA对外显子具有选择性,称为可变环化[14] 。
3.4. 线虫环状RNA侧翼内含子特征
线虫环状RNA上游和下游内含子的中位数长度分别达到了907 bp和962 bp,它们之间没有显著差异性(p = 0.157),但同线虫所有内含子的中位数长度(69 bp)有极其显著差异(p < 0.0001) (图3(d)),说明具有较长的侧翼内含子序列是环状RNA的一个基本特征。

Figure 5. Examples that illustrate the diversity of circularizing exons
图5. 线虫环状RNA多样的环化方式
Alu元件是人类基因组上大量存在的短散在重复序列,也是人类环状RNA形成的主要原因,同时环状RNA两侧的内含子序列内(IRwithin)和序列间(IRacross)的反向互补配对的竞争(图6(a))也是造成环状RNA形成的重要原因[14] 。由于在线虫基因组中83%都是非重复序列,因此线虫环状RNA形成的主要原因不会是因为基因组上的重复序列。我们根据环状RNA的位点信息,提取出环状RNA侧翼内含子序列,并用blast来统计IRwithin和IRacross的情况。我们发现96.33%的环状RNA侧翼内含子都存在序列反向互补情况(图6(b)),只有约0.3%的内含子间不含有IRacross,在1852个序列对中,IRacross − IRwithin >= 1的有1374对,占74.19%。说明具有反向互补配对的侧翼内含子序列也是线虫环状RNA的一个重要的基本特征。
同时我们也统计了203个由第一个外显子构成的环状RNA的上游基因间区序列和其下游第一个内含子之间的序列比对情况,发现具有IRacross的有180对,占88.67%,IRacross − IRwithin >= 1的有107对,占52.71%(图6(b))。真核基因转录产物一般是单顺反子,但线虫中约25%的基因会产生多顺反子,因此我们推测这些顺反子之间的基因间区可能会和intron区的序列反向互补配对,从而造成第一个外显子也参与成环的现象。
3.5. miRNA在环状RNA上的结合位点
在canonical和antisense类型的环状RNA中,每个环状RNA都有一个或多个miRNA的靶位点,最多的可以有95个不同miRNA的靶位点,但可结合相同miRNA的数目却大多只有1~3个。其中环状RNA位点(chrIII:6758887-6763523)可由K07E
我们的结果中可以看出,在线虫中环状RNA可作为分子海绵的数量和能力十分有限,而在目前已报导的circRNA中,也只有CDR1as和SRY两个环状RNA被证明具有分子海绵作用。
3.6. GO功能富集
我们将可以产生环状RNA的转录本进行GO功能富集分析[26] (图7),GO富集结果表明多数基因在细胞成分上为细胞和细胞器成分,具有结合蛋白,催化,传输等分子功能,参与到生物调节,细胞过程,生长过程,免疫,代谢等多种生物过程中。
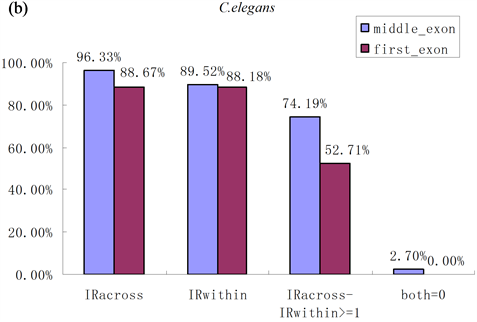
Figure 6. Reverse complementary matches of circular RNA flanking intron in C.elegans
图6. 线虫环状RNA侧翼内含子序列反向互补情况
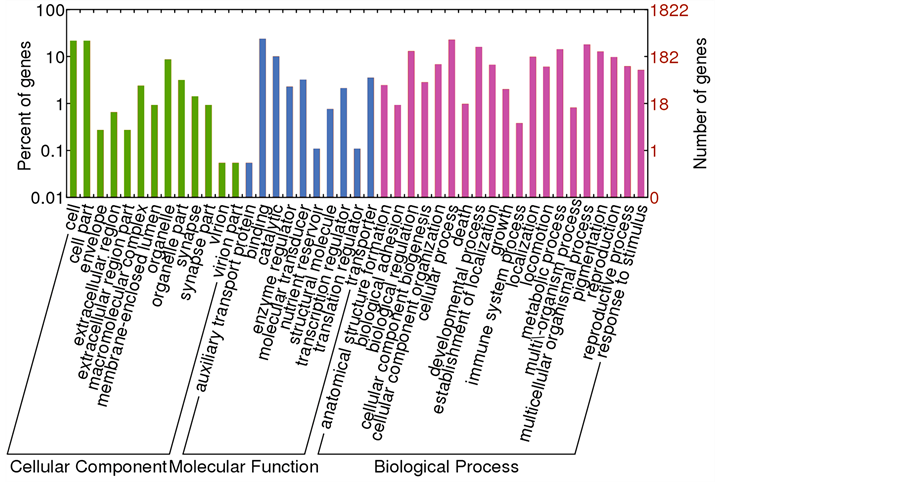
Figure 7. GO annotation for genes which can generate circular RNA
图7. 可产生环状RNA的基因GO功能注释
4. 讨论
本文根据核酸外切酶RNase R处理后的RNA-seq测序数据,重新预测线虫中存在的环状RNA,数目由原先的725个提升到1452个。在得到的环状RNA中,有一些经过RNase R处理后读段降低或消失的其他类型环状RNA。通过对环状RNA的构成和基本特征进行分析,发现线虫中环状RNA主要由中间两个外显子构成,但是由第一个外显子形成环状RNA的比例较高,我们认为原因是由于线虫可以产生多顺反子。通过分析环状RNA侧翼内含子序列,发现线虫环状RNA内含子长度较长且上下游区间多含有反向互补配对序列,同样支持了内含子配对驱动环化模型。
在环状RNA作为miRNA分子海绵的可能性上,线虫中虽然每个环状RNA上都有miRNA的结合位点,但我们只发现一个环状RNA可以结合16个相同的miRNA分子,可能为潜在的分子海绵,而其他的环状RNA都没有10个以上的miRNA结合位点。此外,我们还检测了人类CDR1as序列上其他miRNA分子的结合位点,发现有119个miR-5011-5p位点、116个miR
目前关于环状RNA功能的研究较少,有研究表明在果蝇和小鼠的脑组织中环状RNA表达的丰度会高于其他组织,环状RNA可能和神经发育相关[9] [27] ,并且CDR1as的表达也会阻碍中脑的发育。尽管因此,环状RNA究竟为什么而产生,还可以行使怎样的功能尚值得我们进一步研究。而准确的建库方式以及专门寻找环状RNA的生物信息学工具[28] 的开发会使我们更好地研究这类新型的RNA分子。
文章引用
刘骏武,陈玲玲, (2015) 线虫环状RNA分析
Analysis of Circular RNA in Caenorhabditis elegans. 计算生物学,02,17-28. doi: 10.12677/HJCB.2015.52003
参考文献 (References)
- 1. Nigro, J.M., Cho, K.R., Fearon, E.R., Kern, S.E., Ruppert, J.M., Oliner, J.D., Kinzler, K.W. and Vogelstein, B. (1991) Scrambled exons. Cell, 64, 607-613.
- 2. Cocquerelle, C., Daubersies, P., Majerus, M.-A., Kerckaert, J.-P. and Bailleul, B. (1992) Splicing with inverted order of exons occurs proximal to large introns. The EMBO Journal, 11, 1095-1098.
- 3. Capel, B., Swain, A., Nicolis, S., Hacker, A., Walter, M., Koopman, P., Goodfellow, P. and Lo-vell-Badge, R. (1993) Circular transcripts of the testis-determining gene Sry in adult mouse testis. Cell, 73, 1019-1030.
- 4. Cocquerelle, C., Mascrez, B., Hetuin, D. and Bailleul, B. (1993) Mis-splicing yields circular RNA molecules. The FASEB Journal, 7, 155-160.
- 5. Jeck, W.R., Sorrentino, J.A., Wang, K., Slevin, M.K., Burd, C.E., Liu, J., Marzluff, W.F. and Sharpless, N.E. (2013) Circular RNAs are abundant, conserved, and associated with ALU repeats. RNA, 19, 141-157.
- 6. Salzman, J., Chen, R.E., Olsen, M.N., Wang, P.L. and Brown, P.O. (2013) Cell-type specific features of circular RNA expression. PLoS Genet, 9, e1003777.
- 7. Guo, J.U., Agarwal, V., Guo, H. and Bartel, D.P. (2014) Expanded identification and characterization of mammalian circular RNAs. Genome Biology, 15, 304-313.
- 8. Memczak, S., Jens, M., Elefsinioti, A., Torti, F., Krueger, J., Rybak, A., Maier, L., Mackowiak, S.D., Gregersen, L.H., Munschauer, M., Loewer, A., Ziebold, U., Landthaler, M., Kocks, C., le Noble, F. and Rajewsky, N. (2013) Circular RNAs are a large class of animal RNAs with regulatory potency. Nature, 495, 333-338.
- 9. Westholm, J.O., Miura, P., Olson, S., Shenker, S., Joseph, B., Sanfilippo, P., Celniker, S.E., Graveley, B.R. and Lai, E.C. (2014) Genome-wide analysis of drosophila circular RNAs reveals their structural and sequence properties and age-dependent neural accumulation. Cell Reports, 9, 1966-1980.
- 10. Danan, M., Schwartz, S., Edelheit, S. and Sorek, R. (2012) Transcriptome-wide discovery of circular RNAs in Archaea. Nucleic Acids Reserach, 40, 3131-3142.
- 11. Hansen, T.B., Jensen, T.I., Clausen, B.H., Bramsen, J.B., Finsen, B., Damgaard, C.K. and Kjems, J. (2013) Natural RNA circles function as efficient microRNA sponges. Nature, 495, 384-388.
- 12. Hansen, T.B., Wiklund, E.D., Bramsen, J.B., Villadsen, S.B., Statham, A.L., Clark, S.J. and Kjems, J. (2011) miRNA- dependent gene silencing involving Ago2-mediated cleavage of a circular antisense RNA. EMBO Journal, 30, 4414-4422.
- 13. Li, Z., Huang, C., Bao, C., Chen, L., Lin, M., Wang, X., Zhong, G., Yu, B., Hu, W., Dai, L., Zhu, P., Chang, Z., Wu, Q., Zhao, Y., Jia, Y., Xu, P., Liu, H. and Shan, G. (2015) Exon-intron circular RNAs regulate transcription in the nucleus. Nature Structural Molecular Biology, 22, 256-264.
- 14. Zhang, X.O. and Wang, H.B., Zhang, Y., Lu, X., Chen, L.L., Yang, L. (2014) Complementary sequence-mediated exon circularization. Cell, 159, 134-147.
- 15. Liang, D. and Wilusz, J.E. (2014) Short intronic repeat sequences facilitate circular RNA production. Genes and Development, 28, 2233-2247.
- 16. Ivanov, A., Memczak, S., Wyler, E., Torti, F., Porath, H.T., Orejuela, M.R., Piechotta, M., Levanon, E.Y., Landthaler, M., Dieterich, C. and Rajewsky, N. (2015) Analysis of intron sequences reveals hallmarks of circular RNA biogenesis in animals. Cell Reports, 10, 170-177.
- 17. Kent, W.J., Sugnet, C.W., Furey, T.S., Roskin, K.M., Pringle, T.H., Zahler, A.M. and Haussler, D. (2002) The human genome browser at UCSC. Genome Research, 12, 996-1006.
- 18. Kozomara, A. and Griffiths-Jones, S. (2014) miRBase: Annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research, 42, D68-D73.
- 19. Bolger, A.M., Lohse, M. and Usadel, B. (2014) Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics, 30, 2114-2120.
- 20. Glažar, P., Papavasileiou, P. and Rajewsky, N. (2014) circBase: A database for circular RNAs. RNA, 20, 1666-1670.
- 21. Trapnell, C., Roberts, A., Goff, L., Pertea, G., Kim, D., Kelley, D.R., Pimentel, H., Salzberg, S.L., Rinn, J.L. and Pachter, L. (2012) Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nature Protocols, 7, 562-578.
- 22. Kim, D., Pertea, G., Trapnell, C., Pimentel, H., Kelley, R. and Salzberg, S.L. (2013) TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology, 14, R36.
- 23. Mortazavi, A., Williams, B.A., McCue, K., Schaeffer, L. and Wold, B. (2008) Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods, 5, 621-628.
- 24. Zhang, Y., Chen, D., Smith, M.A., Zhang, B. and Pan, X. (2012) Selection of reliable reference genes in Caenorhabditis elegans for analysis of nanotoxicity. PloS ONE, 7, e31849.
- 25. Cheng, Z.-F. and Deutscher, M.P. (2005) An important role for RNase R in mRNA decay. Molecular Cell, 17, 313-318.
- 26. Ye, J., Fang, L., Zheng, H., Zhang, Y., Chen, J., Zhang, Z., Wang, J., Li, S., Li, R. and Bolund, L. (2006) WEGO: A web tool for plotting GO annotations. Nucleic Acids Research, 34, W293-W297.
- 27. You, X., Vlatkovic, I., Babic, A., Will, T., Epstein, I., Tushev, G., Akbalik, G., Wang, M., Glock, C., Quedenau, C., Wang, X., Hou, J., Liu, H., Sun, W., Sambandan, S., Chen, T., Schuman, E.M. and Chen, W. (2015) Neural circular RNAs are derived from synaptic genes and regulated by development and plasticity. Nat Neuroscience, 18, 603-610.
- 28. Gao, Y., Wang, J. and Zhao, F. (2015) CIRI: An efficient and unbiased algorithm for de novo circular RNA identification. Genome Biology, 16, 4.
