Hans Journal of Computational Biology
Vol.
12
No.
04
(
2022
), Article ID:
57619
,
7
pages
10.12677/HJCB.2022.124008
新冠病毒Omicron变异株S蛋白变异位点 及进化分析
吴秀兰1,李绮晴2,郑珊丽2,黄万信1,钟华斌1,唐文武2*
1肇庆学院食品与制药工程学院,广东 肇庆
2肇庆学院生命科学学院,广东 肇庆
收稿日期:2022年9月26日;录用日期:2022年11月2日;发布日期:2022年11月9日

摘要
新冠病毒Omicron变异株是2021年11月首次在南非发现,并已成为当前传染性最高的主流变异株,其传染性和S蛋白位点变异存在相关性。本文通过生物信息学方法对Omicron变异株的S蛋白变异位点进行分析,并与多种冠状病毒S蛋白进行多序列比对及构建系统进化树。结果显示Omicron变异株S蛋白序列保守性较高,共发现26个变异位点,其中K417N、Q493R、N501Y、Y505H变异位点可能增强病毒感染性。序列比对显示该变异株与Zeta变异株一致性最高,达到98.11%,其次与猫、猴的冠状病毒S蛋白序列一致性皆为98.04%,与Lambda变异株相似性最低,其一致性为97.01%。本研究有助于了解Omicron变异株S蛋白的功能,并为后续筛选靶向药物提供理论依据。
关键词
新冠病毒,Omicron,S蛋白,变异位点,进化分析

Mutation Sites and Evolutionary Analysis of SARS-CoV-2 Omicron Spike Protein
Xiulan Wu1, Qiqing Li2, Shanli Zheng2, Wanxin Huang1, Huabin Zhong1, Wenwu Tang2*
1College of Food and Pharmaceutical Engineering, Zhaoqing University, Zhaoqing Guangdong
2College of Life Science, Zhaoqing University, Zhaoqing Guangdong
Received: Sep. 26th, 2022; accepted: Nov. 2nd, 2022; published: Nov. 9th, 2022

ABSTRACT
The omicron variant was first detected in South Africa in November 2021 and has become the most prevalent variant, with a correlation with variation in its Spike protein locus. Using bioinformatics methods and software, we analyzed the mutation sites of Spike protein with the Omicron variant and compared it with other coronaviruses to construct the phylogenetic tree. The results showed that the sequence of Spike protein with the Omicron variant is highly conserved. A total of 26 mutation sites have been found in the Spike protein, among which K417N, Q493R, N501Y, and Y505H may enhance virus infectivity. Sequence alignment showed that the omicron variant had the highest identity with the Zeta variant, reaching 98.11%, followed by coronaviruses such as Felinae and Primates, with 98.04% identity. The lowest identity is with the Lambda variant, with an identity of 97.01%. This study will help to understand the function of the Spike protein of the Omicron variant and provide a theoretical basis for further research and screening of new antiviral drug targeting Spike protein.
Keywords:SARS-CoV-2, Omicron, Spike Protein, Mutation Sites, Evolution Analysis

Copyright © 2022 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
2019年12月出现的新型冠状病毒(Severe Acute Respiratory Syndrome Coronavirus 2,SARS-CoV-2,简称新冠病毒)感染引发的新型冠状病毒肺炎(Coronavirus Disease 2019,COVID-19,简称新冠肺炎),已在全球范围内肆虐传播两年多,对全球公共卫生及人类生命健康造成巨大威胁 [1]。尽管全球各地已采取各种防疫措施,但每日新增确诊病例数及死亡病例数仍在快速增加。据世界卫生组织(World Health Organization,简称WHO)实时统计数据,截至北京时间2022年5月30日,全球累计新冠肺炎确诊病例252,467,084例,累计死亡病例6,285,171例 [2]。新冠病毒在大规模传播的同时不断发生变异进化,产生基本传播系数(Basic reproductive number,简称R0)或致病性更强的变异株,如Alpha变异株、Beta变异株、Gamma变异株、Delta变异株,对新冠防控和疫苗开发造成巨大影响 [3]。2021年11月9日于南非首次检测到的Omicron (B.1.1.529)变异株,在短短一周时间即成为南非豪登省主流新冠变异株,并于同月26日被WHO定性为最高级别的“关切变异株” [4]。目前全球有150多个国家及地区检测出Omicon变异株 [5],该变异株已成为全球优势变异株 [6]。
作为Omicron变异株发现前传染性最高的变异株,Delta变异株的R0值约为5~8 [7],而Omicron变异株的R0数值在全球范围内可能高达10 [8]。流行病学追踪显示,Omicron变异株比Delta变异株更易于传播,其传播速度亦高于Delta变异株 [9]。病毒基因测序显示,2022年2月底开始爆发,导致感染人数超过52万的上海市新冠疫情,主要是Omicron变异株传播造成 [10]。新冠病毒主要有四种结构蛋白,分别为表面刺突蛋白(S蛋白)、膜蛋白(M蛋白)、核衣壳蛋白(N蛋白)及包膜蛋白(E蛋白) [11]。其中S蛋白具有识别人体细胞表面血管紧张素转化酶II (简称ACE2蛋白)受体蛋白,并与之特异性结合,从而介导细胞膜融合的功能,能够影响冠状病毒感染宿主细胞、特异性识别特定宿主的能力 [12]。S蛋白与ACE2受体蛋白的亲和力决定了新冠病毒感染能力,因此S蛋白的氨基酸位点变异与新冠病毒传染性密切相关 [13]。Omicron变异株是当前R0数值最高的新冠病毒变异株,其S蛋白的变异位点及其特征研究具有重要意义。本文对新冠病毒Omicron变异株的S蛋白变异位点进行分析,与其他冠状病毒的S蛋白进行多序列比对及构建系统进化树,以便加深人们对Omicron变异株S蛋白功能的理解,并对后续研究、筛选靶向S蛋白的新型抗病毒药物提供理论依据。
2. 材料与方法
2.1. Omicron变异株的S蛋白序列获取
从NCBI (https://www.ncbi.nlm.nih.gov)下载原始新冠病毒Wuhan-Hu-1 (NC_045512.2) S蛋白氨基酸序列及核苷酸序列。以来源于印度的Omicron变异单株(EPI_ISL_10204983)为例,通过GISAID (https://www.gisaid.org/)数据库下载该测序株的基因组序列。通过线上网站EMBOSS Needle (https://www.ebi.ac.uk/Tools/psa/emboss_needle/)获取其S蛋白核苷酸序列,通过线上网站Expasy (https://web.expasy.org/translate/)将其转换为氨基酸序列。
2.2. Omicron变异株S蛋白变异位点预测
通过线上网站Nextclade (https://clades.nextstrain.org)对Omicron变异株S蛋白进行变异位点分析,并利用在线工具Phyre2对S蛋白三级结构进行模拟构建,使用PyMOL对结果进行可视化操作,标记主要突变位点的所在空间位置。
2.3. 不同新冠病毒变异株的多序列比对
从NGDC (https://ngdc.cncb.ac.cn/ncov/)及GISAID数据库中,下载WHO认可的5个关注VOC变种(Alpha, Beta, Gamma, Delta, Omicron)和7个需留意的VOI变种(Epsilon, Zeta, Eta, Theta, Lota, Kappa, Lambda)的全基因组序列,按照上述2.1方法获取新冠病毒变异株的S蛋白序列。将12种新冠病毒变异株和Wuhan-Hu-1、猫、狗、猴、马来菊头蝠等冠状病毒的S蛋白序列,利用MEGA11软件进行多序列比对。
2.4. 不同冠状病毒的系统进化树构建
从NCBI网站中获取水貂(Neovison vison)、孟加拉虎(Panthera tigris)、亚洲狮(Panthera leo persica)、白鼬(Mustela erminea)、马来亚虎(Panthera tigris jacksoni)、白尾鹿(Odocoileus virginianus)、骆驼(Camelus)、蝙蝠(Chiroptera)、猪(Sus)、牛(Bovini)、麻雀(Passer)、兔(Rabbit)等13种动物的新冠病毒S蛋白序列,并与前文的Alpha、Beta、Gamma、Delta、Omicron 5种新冠病毒变异株,以及猫(Felinae)、狗(Canis lupus familiaris)、猴(Primates)、马来菊头蝠(Rhinolophus malayanus)等冠状病毒和Wuhan-Hu-1共24种冠状病毒的S蛋白序列,利用MEGA11软件的邻接法构建系统进化树。
3. 结果与分析
3.1. Omicron变异株S蛋白变异位点分析
由线上网站Nextclade比对分析显示,所选Omicron变异株S蛋白与最早发现的Wuhan-Hu-1标准毒
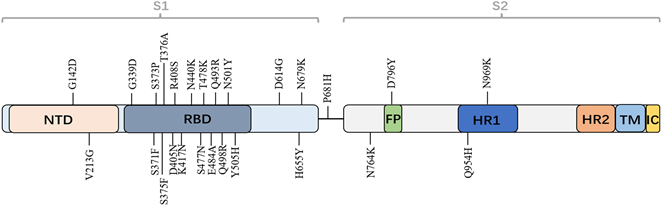
Figure 1. The S protein mutation site of the SARS-COV-2 Omicron variant
图1. 新冠病毒Omicron变异株的S蛋白变异位点
株S蛋白进行比对,共有26个位点发生突变,分别为G142D、V213G、G339D、S371F、S373P、S375F、T376A、D405N、R408、K417N、N440K、S477N、T478K、E484A、Q493R、Q498R、N501Y、Y505H、D614G、H655Y、N679K、P681H、N764K、D796Y、Q954H、N969K (见图1)。其中与人ACE2受体结合特异性和亲和力相关的RBD区域,共发现16个氨基酸突变位点,分别为G339D、S371F、S373P、S375F、T376A、D405N、R408S、K417N、N440K、S477N、T478K、E484A、Q493R、Q498R、N501Y、Y505H。
3.2. Omicron变异株S蛋白三级结构分析
利用在线工具Phyre2对Omicron变异株S蛋白的三级结构进行模型构建,在获得的结果中下载pbd文件,使用PyMOL进行可视化分析并标记出RBD区域的主要突变位点,相关结果见图2。图2(a)显示出S1和S2蛋白的三维结构,以及S1蛋白的RBD区域(灰色)和NTD区域(麦色)。图2(b)显示,在Omicron变异株的S蛋白RBD区域的16个氨基酸变异位点中,K417N、N440K、T478K、E484A、Q493R、Q498R、N501Y、Y505H共8个氨基酸位点与人受体蛋白ACE2结合域相互作用。因此上述位点变异可能增强新冠病毒与人ACE2的结合能力,进而提高病毒感染力。
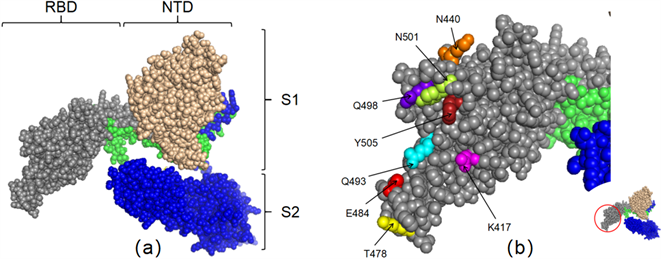
Figure 2. The tertiary structure of S protein and RBD mutation position with SARS-COV-2 Omicron variant; (a) The tertiary structure model of Omicron variant’s S protein; (b) RBD mutation position of SARS-COV-2 Omicron variant
图2. Omicron 变异株的S蛋白三级结构和RBD区域重要突变位点示意图;(a) Omicron变异株的S蛋白三级结构;(b) S蛋白RBD区域重要突变位点标记示意图
3.3. 不同新冠病毒变异株的多序列比对
对5个关注VOC变种,7个需留意VOI变种,以及4种动物冠状病毒的S蛋白进行多序列比对,部分比对集合图见图3。结果显示,17种冠状病毒S蛋白中,完全一致的氨基酸残基有1187个,占93.17%,表明S蛋白的氨基酸序列保守性较高。其中与Omicron变异株S蛋白序列一致性最高的是Zeta变异株,两者序列一致性高达98.11%;其次是猫、猴的新冠病毒S蛋白序列,序列一致性皆为98.04%;与新冠病毒Lambda变异株相似性最低,其序列一致性仅为97.01%。
3.4. 不同冠状病毒的S蛋白系统进化分析
对17种不同动物冠状病毒、5种新冠病毒变异株以及SARS病毒共24种冠状病毒的S蛋白进行多序列比对,并采用邻接法构建系统进化树,分析结果见图4。结果显示,骆驼、麻雀、猪、蝙蝠、牛、兔以及人类SARS等冠状病毒聚为一类,自展值为100,说明它们在进化上相距较近,可能来源于同一祖先。人类Omicron变异株(EPI_ISL_10204983)、Gamma变异株(OU066503.1)以及Beta变异株(MZ314998.1)

图3 不同冠状病毒的S蛋白多序列比对集合图
与上述冠状病毒遗传距离较近,进而可推测这些冠状病毒在进化上方向一致。
聚类分析显示,白鼬、亚洲狮、狮子、白尾鹿、马来菊头蝠的冠状病毒以及人类新冠病毒Delta变异株(Z706155.1)聚为一支,马来亚虎、虎、猫、猴的冠状病毒以及最早发现的新冠病毒Wuhan-Hu-1 (NC_045512.2)聚为一支,上述结果说明冠状病毒进化可能与宿主物种无关,冠状病毒S基因的RNA序列变异具有随机性和多样化,导致其进化与宿主物种及发生时间等没有相关性。
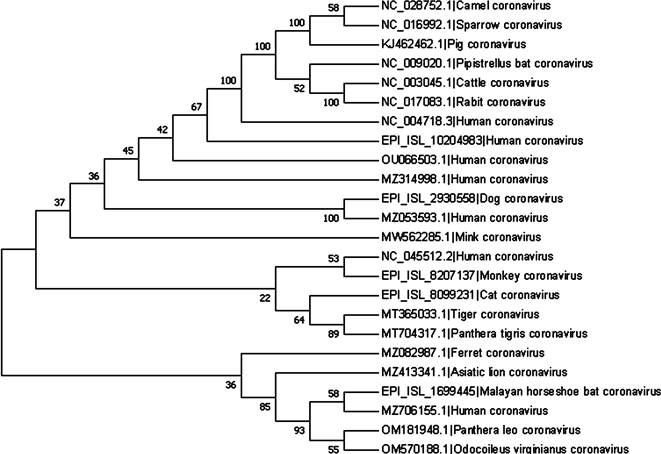
注:SARS冠状病毒(NC_004718.3);水貂Neovison vison (MW562285.1):马来亚虎Panthera tigris jacksoni (MT365033.1);亚洲狮Panthera leo persica (MZ413341.1);狮子Panthera leo (OM181948.1);白鼬Mustela ermine (MZ082987.1);虎Panthera tigris (MT704317.1);白尾鹿Odocoileus virginianus (OM570188.1);牛Bovini (NC 003045.1);骆驼Camelus (NC_028752.1);蝙蝠Pipistrellus (NC_009020.1);猪Sus (KJ462462.1);麻雀Passer (NC_016992.1);兔Rabbit (NC_017083.1);Alpha变异株(MZ053593.1);Beta变异株(MZ314998.1);Gamma变异株(OU066503.1);Delta变异株(Z706155.1);Omicron变异株(EPI_ISL_10204983);猫Felinae (EPI_ISL_8099231);狗Canis lupus familiaris (EPI_ISL_2930558);猴Primates (EPI_ISL_8207137);马来菊头蝠Rhinolophus malayanus (EPI_ISL_1699445);Wuhan-Hu-1 (NC_045512.2)
图4. 各类冠状病毒S蛋白的系统进化树
4. 讨论
新冠肺炎是当前国际社会关最受关注的公共卫生事件,SARS-CoV-2的快速传播危及全世界人民的生命健康及全球经济的发展 [14] [15]。2021年11月首次发现的Omicron变异株,不仅传染速度高于其它新冠病毒变异株,而且容易让康复后的感染者再次感染 [16]。S蛋白对于冠状病毒入侵宿主细胞至关重要 [17],其S1亚基的RBD结合域能与人ACE2结合,协助病毒穿过细胞膜进入宿主细胞,进而感染人类,因此RBD区域变异位点与新冠病毒传染性强弱密切相关 [18] [19] [20]。本研究显示,Omicron变异株与原始新冠病毒Wuhan-Hu-1相比,S蛋白中发现26个氨基酸突变位点,其中16个变异位点位于RBD区域。Shang等报道新冠病毒RBD区域内的417、449、487、489、493、500、501、502、505共9个变异位点与ACE2结合力直接相关 [21],本研究发现K417N、Q493R、N501Y、Y505H 4个相同的变异位点,这些变异位点可能增强新冠病毒与人ACE2的结合能力,进而提高病毒感染性 [22]。
Omicron变异株的4个关键变异位点中,N501Y变异位点广泛存在于Alpha、Beta、Gamma等高传染性变异株中 [23]。Frampton等人研究证明,N501位氨基酸是冠状病毒受体结合域的关键接触残基之一,直接参与RBD和ACE2的结合作用,而N501Y突变增强了RBD和ACE2的结合亲和力 [24]。刘洋等研究表明,N501Y变异位点能增加新冠病毒S蛋白对上呼吸道细胞受体的亲和力,在巴西、南非等国的趋同进化表明,N501Y是最主要的关注适应性突变位点,是上呼吸道感染和冠状病毒传播性增强的关键因素 [25]。Q493能与S蛋白中和抗体LY-CoV555相结合从而形成三个稳定力,当S蛋白发生Q493R突变时,会导致该位点与抗体无法形成相互作用力,表明Q493R突变体可能是LY-CoV555 逃逸突变体。K417N突变不直接与ACE2相互作用,但其有助于病毒更有效地与hACE2结合 [3]。
5. 结论
综上所述,本研究对Omicron变异株S蛋白变异位点进行分析,与其他新型冠状病毒变异株的S蛋白进行多序列比对及构建系统进化树,有助于深入了解Omicron变异株S蛋白的功能,为后续研究、筛选靶向S蛋白的新型抗病毒药物提供理论依据。
基金项目
广东省大学生创新训练计划项目(S202010580047)。
文章引用
吴秀兰,李绮晴,郑珊丽,黄万信,钟华斌,唐文武. 新冠病毒Omicron变异株S蛋白变异位点及进化分析
Mutation Sites and Evolutionary Analysis of SARS-CoV-2 Omicron Spike Protein[J]. 计算生物学, 2022, 12(04): 59-65. https://doi.org/10.12677/HJCB.2022.124008
参考文献
- 1. 任彪, 廖彬佑, 王建楠, 等. 奥密克戎(Omicron)变异株及其对口腔诊疗的潜在影响[J]. 四川大学学报(医学版), 2022, 53(2): 175-180.
- 2. WHO (2022) Coronavirus (COVID-19) Dashboard. https://covid19.who.int
- 3. 吴俣, 刘珏, 刘民, 梁万年. 新型冠状病毒Omicron变异株的流行病学特征及防控研究[J]. 中国全科医学, 2022, 25(1): 14-19.
- 4. WHO (2021) Classification of Omicron (B.1.1.529): SARS-CoV-2 Variant of Concern. https://www.who.int/news/item/26-11-2021-classification-of-omicron-(b.1.1.529)-sars-cov-2-variant-of-concern
- 5. 今冬疫情又起, 但黎明的曙光已不再遥远[EB/OL]. http://www.CRNTT.com, 2022-01-21.
- 6. 美国专家反驳“疫情终结论”: 奥秘克戎传越广, 下一个变种越早来[EB/OL]. https://new.qq.com/omn/20220118/20220118A0BLND00.html, 2022-01-19.
- 7. 14秒就能人传人, 钟南山口中“传染性极强”的德尔塔毒株是怎么回事?[EB/OL]. http://finance.sina.com.cn/tech/csj/2021-07-29/doc-ikqciyzk8293182.shtml#~:text, 2021-12-09.
- 8. Burki, T.K. (2022) Omicron Variant and Booster COVID-19 Vaccines. The Lancet Respiratory Medicine, 10, E17. https://www.thelancet.com/journals/lanres/article/PIIS2213-2600(21)00559-2
- 9. 新冠病毒又双叒变异! 加息进程受阻?[EB/OL]. https://finance.sina.com.cn/money/forex/forexinfo/20211129/docikyakumx0681628.shtm, 2021-11-28.
- 10. 上海疫情已超52万人感染, 多为奥秘克戎BA.2和BA.2.2 [EB/OL]. https://cj.sina.com.cn/articles/view/1643971635/61fd043300100xa47?finpagefr=p_104, 2022-4-29.
- 11. 徐本锦, 宣焱, 杜淼, 等. 新冠病毒刺突糖蛋白结构与功能的生物信息学分析及原核表达[J]. 病毒学报, 2022, 38(1): 41-56.
- 12. 何金娇, 刘洋洋, 毛雪飞, 等. SARS-CoV-2 S蛋白结构与功能的生物信息学分析[J]. 基因组学与应用生物学, 2021, 40(Z1): 2346-2355.
- 13. 王嘉君. 浅谈Omicron新型冠状病毒变异株的特点和防控措施[J]. 福建轻纺, 2022(2): 33-37.
- 14. Li, Q., Guan, X., Wu, P., et al. (2020) Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia. New England Journal of Medicine, 382, 1199-1207.
- 15. Huang, C., Wang, Y., Li, X., et al. (2020) Clinical Features of Patients Infected with 2019 Novel Coronavirus in Wuhan, China. The Lancet, 395, 497-506. https://doi.org/10.1016/S0140-6736(20)30183-5
- 16. 南非卫生部: 奥密克戎毒株或致新冠二次感染风险增加[EB/OL]. http://www.news.cn/world/2021-12/11/c_1128153618.htm, 2021-12-12.
- 17. Wrapp, D., Wang, N., Corbett, K.S., et al. (2020) Cryo-EM Structure of the 2019-nCoV Spike in the Prefusion Conformation. Science, 367, 1260-1263. https://doi.org/10.1126/science.abb2507
- 18. Astuti, I. (2020) Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2): An Overview of Viral Structure and Host Response. Diabetes & Metabolic Syndrome: Clinical Re-search & Reviews, 14, 407-412. https://doi.org/10.1126/science.abb2507
- 19. Kumar, S. and Saxena, S.K. (2021) Structural and Molecular Perspec-tives of SARS-CoV-2. Methods, 195, 23-28. https://doi.org/10.1016/j.ymeth.2021.03.007
- 20. Rodríguez-Maldonado, A.P., Vázquez-Pérez, J.A., Cedro-Tanda, A., et al. (2021) Emergence and Spread of the Potential Variant of Interest (VOI) B.1.1.519 of SARS-CoV-2 Predomi-nantly Present in Mexico. Archives of Virology, 166, 3173-3177. https://doi.org/10.1007/s00705-021-05208-6
- 21. Shang, J., Ye, G., Shi, K., et al. (2020) Structural Basis of Re-ceptor Recognition by SARS-CoV-2. Nature, 581, 221-224. https://doi.org/10.1038/s41586-020-2179-y
- 22. Qin, S., Cui, M., Sun, S., et al. (2021) Genome Characterization and Potential Risk Assessment of the Novel SARS-CoV-2 Variant Omicron (B.1.1.529). Zoonoses, 1, 1-5. https://doi.org/10.15212/ZOONOSES-2021-0024
- 23. 穆雪纯, 李丽, 王凌航. 新型冠状病毒流行变异株的分型及研究进展[J]. 国际病毒学杂志, 2021, 28(4): 336-340.
- 24. Frampton, D., Rampling, T., Cross, A., et al. (2021) Genomic Characteristics and Clinical Effect of the Emergent SARS-CoV-2 B.1.1.7 Lineage in London, UK: A Whole-Genome Sequencing and Hospital-Based Cohort Study. The Lancet Infectious Diseases, 21, 1246-1256. https://doi.org/10.1016/S1473-3099(21)00170-5
- 25. Liu, Y., Liu, J., Plante, K.S., et al. (2022) The N501Y Spike Substitution Enhances SARS-CoV-2 Transmission. Nature, 602, 294-299. https://doi.org/10.1038/s41586-021-04245-0