Asian Case Reports in Oncology
Vol.
10
No.
01
(
2021
), Article ID:
40046
,
23
pages
10.12677/ACRPO.2021.101001
影响人脑胶质瘤细胞发生、增殖关键lncRNA、miRNA的筛选与分析
李明扬,王仕超,凌宇,刘春霞,曹俊伟,张焱如*
内蒙古农业大学内蒙古自治区生物制造重点实验室,内蒙古 呼和浩特

收稿日期:2020年12月20日;录用日期:2021年1月20日;发布日期:2021年1月27日

摘要
脑胶质瘤是一种极度常见颅腔内的原发性恶性肿瘤,患病人群占据所有颅内肿瘤患者的60%。IV级胶质瘤为最高恶性程度,也称为胶质母细胞瘤(胶质母细胞瘤,GBM),约占全部胶质瘤的55%,且诊断为GBM的患者5年生存率低于6%。许多研究表明,脑胶质瘤中异常表达的lncRNA不仅调控着与癌细胞增殖、凋亡有关的信号转导通路,还与病人的预后有关。利用转录组高通量测序方法,筛选影响脑胶质瘤发生、发展及复发过程中关键通路中的重要基因,对脑胶质瘤的分子诊断、靶向药物的研发及新治疗方案的选择具有重要的价值。本研究通过RNA-seq和生物信息学分析,筛选出在脑胶质瘤组织中差异表达的lncRNA 106种,其中上调表达52种,下调表达54种。microRNA共计15个,其中上调表达8种;下调表达7种。这些差异表达的ncRNA主要参与有机循环化合物代谢过程、胞内运输、细胞蛋白质定位、蛋白质运输、有丝分裂细胞周期、细胞周期调节、DNA代谢过程的正调控、水解酶活性的调节等生物过程、生物调节、应激反应、多细胞有机体过程、细胞成分的组织或生物发生、细胞定位、信号途径、发育过程、生物过程的正调控与负调控。经lncRNA-miRNA-mRNA关联分析,建立了ceRNAs调控网络,明确了lncRNA、miRNA与靶标mRNA之间的对应关系。这些关键lncRNA的筛选,为脑胶质瘤的诊断及治疗提供了新的思路与方向。
关键词
胶质瘤,lncRNA,miRNA,ceRNA网络,联合分析
Screening and Analysis of Key lncRNA and miRNA Affecting the Occurrence and Proliferation of Human Glioma Cells
Mingyang Li, Shichao Wang, Yu Ling, Chunxia Liu, Junwei Cao, Yanru Zhang*
Key Laboratory of Biomanufacturing of Inner Mongolia Autonomous Region, Inner Mongolia Agricultural University, Hohhot Inner Mongolia

Received: Dec. 20th, 2020; accepted: Jan. 20th, 2021; published: Jan. 27th, 2021

ABSTRACT
Glioma is an extremely common primary malignant tumor in the cranial cavity, which accounts for 60% of all patients with intracranial tumors. Grade IV glioma is the highest degree of malignancy, also called glioblastoma (glioblastoma, GBM), accounting for about 55% of all gliomas, and the 5-year survival rate of patients diagnosed with GBM is lower than 6%. Many studies have shown that the abnormally expressed lncRNA in brain gliomas not only regulates the signal transduction pathways related to cancer cell proliferation and apoptosis, but is also related to the patient’s prognosis. Using transcriptome high-throughput sequencing to screen important genes in key pathways that affect the development, development and recurrence of glioma is of great value for molecular diagnosis of glioma, research and development of targeted drugs and selection of new treatment options. In this study, through RNA-seq and bioinformatics analysis, 106 types of lncRNAs that are differentially expressed in glioma tissues were screened, of which 52 were up-regulated and 54 were down-regulated. There are a total of 15 microRNAs, of which 8 are up-regulated and 7 are down-regulated. These differentially expressed ncRNAs are mainly involved in the metabolic process of organic circulating compounds, intracellular transport, cell protein localization, protein transport, mitotic cell cycle, cell cycle regulation, positive regulation of DNA metabolism, regulation of hydrolase activity and other biological processes and biological regulation, Stress response, multicellular organism process, tissue or biogenesis of cell components, cell location, signal pathway, development process, positive and negative regulation of biological processes. After lncRNA-miRNA-mRNA association analysis, a ceRNAs regulatory network was established, and the correspondence between lncRNA, miRNA and target mRNA was clarified. The screening of these key lncRNAs provides new ideas and directions for the diagnosis and treatment of glioma.
Keywords:Glioma, lncRNA, miRNA, ceRNA Network, Conjoint Analysis

Copyright © 2021 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
脑胶质瘤是一种极度常见颅腔内的原发性恶性肿瘤,患病人群占据所有颅内肿瘤患者的60%。常因神经突触中的实质细胞或间质细胞发生畸变所致 [1]。浸润性生长、极高的原位复发率和死亡率是其最明显的三大特征。神经胶质瘤可以导致颅内压增高以及部分神经功能丧失,引起患者语言困难,行动能力异常,癫痫抽搐并伴随持续强烈疼痛感,重症患者将伴随脑疝甚至死亡。依据世界人民卫生服务组织(World Health Organization, WHO)公布的分类管理标准 [2],将胶质瘤按组织学类型分为I~IV级,其中I、II级为低级别系统胶质瘤,III、IV级为高级别神经胶质瘤。IV级胶质瘤为最高恶性程度,也称为胶质母细胞瘤(胶质母细胞瘤,GBM),约占全部胶质瘤的55% [3] [4]。目前,GBM尚无治疗方案,尽管有深入的治疗研究,但诊断为GBM的患者生存率仍然极低,5年生存率低于6% [5]。近几年的研究表明,一些相关信号通路网络中关键基因突变是肿瘤的发生原因。异常的信号通路影响到身体组织器官的功能,进而产生异常的细胞群,导致肿瘤形成 [6] [7]。随着脑胶质瘤分子机制研究的不断深入,在脑胶质瘤的发生、恶化和复发过程中靶向阻断主要信号通路的关键因子已成为一种具有巨大潜力的新方法 [5] [6] [7]。
lncRNA是一类长度大于200 bp的RNA,缺少有意义的开放阅读框,但能以RNA的形式在表观遗传修饰、转录水平、转录后水平调控肿瘤的增殖、诱导凋亡、血管生成、侵袭和转移。lncRNA可通过以下两种形式调控信号转导通路:Cis-lncRNA一般通过转录干扰或募集染色质修饰复合物进行染色质修饰来调控与其临近的上下游的蛋白编码基因;trans-lncRNA可以独立于序列互补,靶向基因位点发挥功能,除了染色质修饰复合物外,还可以与转录延伸因子RNA聚合酶结合,从而影响转录。一些具有结合miRNA位点的lncRNA,可作为竞争性内源性RNA (competing endogenous RNA, ceRNA)与miRNA靶基因竞争性结合miRNA,进而调控miRNA及miRNA靶基因的表达,从而影响肿瘤增殖、凋亡过程 [8]。但lncRNA的表达并不稳定,其表达量会随着肿瘤发展阶段的变化而出现上调或下调表,而这些异常表达的分子往往与肿瘤的发生与恶化有着密切的联系。大量研究表明,一些lncRNA够调节胶质瘤干细胞特性,进而导致肿瘤复发和不良预后。
二代测序(Next-generation sequencing, NGS),也称为大规模平行测序 [9],它可以同时测序数百万个DNA片段。与传统的测序方法 [10] 相比,具有短时、高效、成本节约的优点。基于二代测序技术的全转录组测序是监测细胞所能转录出来的所有类型RNA的总和,包括mRNA和非编码RNA (non-coding RNA, ncRNA)。目前针对非编码RNA的研究主要集中在微小RNA (microRNAs, miRNA)、长链非编码RNA (Long non-coding RNA, lncRNA)和环形RNA (circularRNA, circRNA) [11]。CeRNA (competing endogenous RNAs,内源竞争RNA)是一种调控机制,是对mRNA表达调控机制的一种补充。在细胞内,lncRNA,mRNA,circRNA都能够和miRNA进行结合。当细胞内lncRNA的表达量下降时,更多的miRNA与mRNA结合,由于miRNA对mRNA起负控作用,导致mRNA的表达量下降 [12]。从ceRNA的调控原理可以看出,在这个调控关系中有两个核心:1) 两个RNA都能够和miRNA结合;2) 两个RNA的表达量呈现正相关 [13]。因此,筛选差异表达的lncRNA和差异表达的mRNA,首先分别分析lncRNA-mRNA和mRNA-miRNA之间的靶标关系,再以miRNA为桥梁构建lncRNA-miRNA-mRNA关系对,并筛选lncRNA和mRNA表达量呈现正相关性的关系对,获得lncRNA-miRNA-mRNA的ceRNA网络。
综上,本研究在脑胶质瘤病例中,取脑胶质瘤组织为样本,通过RNA-Seq [14] (Chu et al., 2012, Wang et al., 2009)筛选脑胶质瘤中差异表达的lncRNA、miRNA及其靶基因,以期获得具体的lncRNA- micRNA-mRNA调控网络 [15],并对其靶基因进行GO和KEGG富集,对其所在通路及所调控的生物过程进行分析,探究lncRNA、miRNA对脑胶质瘤发生发展调控的影响,为靶向ncRNA的脑胶质瘤治疗及预后提供分子理论依据。
2. 材料与方法
2.1. 样本采集
2017年11月至2018年2月共采集四例符合《中国中枢神经系统胶质瘤诊断与治疗指南(2015)》 [16] 诊断标准的患者的肿瘤组织及癌旁组织,病理学诊断分期为IV级神经胶质瘤。所有组织标本的取材均需事先向患者及/或家属详细解释,征得患者及/或家属的同意后签署《知情同意书》。标本采集在手术室进行,并得到相关管理人员的许可,同时争取得到相关人员的积极配合以保证标本的及时获得。选取的标本包括肿瘤组织、正常组织和(或)癌旁组织。标本切取的方法与时机:首先进行大体标本的观察或拍照,确认肿瘤的部位、范围,注意与周围组织及坏死组织的鉴别,在标本离体后应在最短的时间内切取标本(<30 min),将癌组织及癌旁组织切取成多块直径为0.5 cm左右的组织块,分别装入已编号的1.5 ml灭菌冻存管中,迅速放入液氮转移罐并做好长期保存准备。记录样本编号、姓名、住院号、肿瘤类型、取材数量(分别用T = 肿瘤,C = 正常,T及C后面的数字表示取材份数)、离体时间(分钟);四位患者的肿瘤样本分别编号为AT、BT、CT、DT;相应的对照组织为AC、BC、CC、DC。四组混测的肿瘤组织为Q1,癌旁组织为Q2 (表1)。
Table 1. Sample grouping record form
表1. 样本分组情况记录表
2.2. RNA的分离、纯化和质量控制
RNA的分离、纯化和质量控制使用Qiagen RNeasy Mini Kit (Qiagen,德国)从脑胶质瘤样本组织中提取总RNA。再对提取得到的RNA使用Nano Photometer分光光度计(IMPLEN,美国)、含Qubit RNA Assay Kit (Life Technologies,美国)的Qubit 2.0 Flurometer和含RNA Nano 6000 Assay Kit (Agilent Technologies,美国)的Bioanalyzer 2100系统来检测RNA的纯度,浓度和完整性。
2.3. 文库构建及转录组测序
测序工作由天津诺禾致源基因测序公司完成,miRNA、lncRNA独立建库,即去除样本中的核糖体RNA,然后对其进行测序。
样品检测合格后,使用Small RNA Sample Pre Kit构建文库,利用Small RNA的3’及5’端特殊结构(5’端有完整的磷酸基团,3’端有羟基),以total RNA为起始样品,直接将Small RNA两端加上接头,然后反转录合成cDNA。随后经过PCR扩增,PAGE胶电泳分离目标DNA片段,切胶回收得到的即为cDNA文库。
文库构建完成后,先使用Qubit2.0进行初步定量,稀释文库至1 ng/ul,随后使用Agilent 2100对文库的insert size进行检测,insert size符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量(文库有效浓度 > 2 nM),以保证文库质量。
库检合格后,把不同文库按照有效浓度及目标下机数据量的需求pooling后进行HiSeq/MiSeq测序。
2.4. 测序数据的处理与分析
原始测序数据(raw data)经去除接头和低质量reads等处理后得到有效数据(clean data)。使用Tophat2 [17] 进行参考基因组比对,再通过cufflinks [18] 完成转录本的拼接。以是否具有编码潜能作为判断标准,利用CNCI v2分析 [19]、CPC v9.4分析 [20]、Pfam26.0蛋白结构域分析 [21] 和PhyloCSF分析 [22] 筛选出lncRNA、miRNA和mRNA,再使用cuffdiff (http://cole-trapnell-lab.github.io/cufflinks/cuffdiff/index.html)软件进行RNA表达水平分析。
根据顺式cis作用原理:lncRNA临近位置(上下游100 kb)的蛋白编码基因作为其靶基因;反式trans作用原理:样本间lncRNA与蛋白编码基因的表达量采用Pearson相关系数法分析(相关系数 > 0.95)来预测lncRNA的靶基因。对差异表达的lncRNA、miRNA和mRNA使用GOseq [23]、UniProt (https://www.uniprot.org/)、KOBAS (2.0) [24] 和Enrichr (http://amp.pharm.mssm.edu/Enrichr/)进行基因本体论(Gene Ontology, GO)富集和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)通路富集分析。
3. 结果与分析
在生物体内,不同基因相互协调行使其生物学功能,通过Pathway显著性富集能确定候选靶基因参与的最主要生化代谢途径和信号转导途径。KEGG (Kyoto Encyclopedia of Genes and Genomes)是有关Pathway的主要公共数据库 [25]。Pathway显著性富集分析以KEGG Pathway为单位,应用超几何检验,找出与整个基因组背景相比,在候选靶基因中显著性富集的Pathway [25]。该分析的计算公式:
其中,N为所有基因中具有Pathway注释的基因数目;n为N中候选靶基因的数目;M为所有基因中注释为某特定Pathway的基因数目;m为注释为某特定Pathway的候选靶基因数目。用BH的方法对p-value进行校正,得到的校正后的P-value值越小代表越显著。这里将值小于0.05的Pathway定义为在候选靶基因中显著富集的Pathway。
3.1. 差异lncRNA的筛选及富集分析
测序结果利用韦恩图显示四组间共有的差异lncRNA数目为零,如图1所示。因此以混测组(GLQ1_vs_GLQ2)所得差异表达的lncRNA为样本,对其共表达的基因进行GO富集,在混测组(GLQ1_vs_GLQ2)中差异lncRNA共计106个,其中上调52个包括lnc_000266、lnc_000276、lnc_000417、
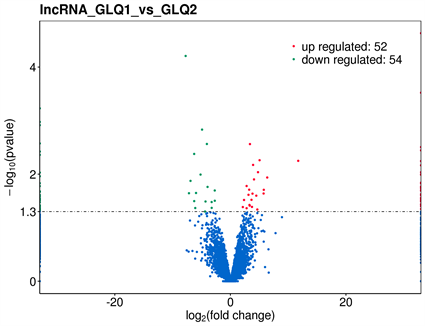
Figure 1. Differentially expressed lncRNA volcano map
图1. 差异表达lncRNA火山图
lnc_000510、lnc_000565、lnc_000662、lnc_000853、lnc_001155、lnc_001315、lnc_001370、lnc_001788、lnc_001827等;下调54个,包括ENST00000572151、lnc_000009、lnc_000021、lnc_000036、lnc_000115、lnc_000410、lnc_000467等(见表2);如图2所示。在GO富集结果分类统计图中,从左往右依次为生物学过程、细胞组分、分子功能包括,如图3所示。结果显示差异lncRNA主要参与有机循环化合物代谢过程、胞内运输、细胞蛋白质定位、蛋白质的运输、有丝分裂细胞周期、细胞周期调节、DNA代谢过程的正调控、水解酶活性的调节等生物过程。如图4所示。差异lncRNA共表达mRNA的KEGG富集通路散点图如图4,主要富集的细胞通路有血小板激活、基础转录因子及补体和凝血级联等相关途径。q-value的取值范围为[0, 1],越接近于零,表示富集结果的可信度越高。差异表达转录本(lncRNA)所对应的靶基因进行了GO富集分析,主要富集在有机循环化合物代谢过程、胞内运输、细胞蛋白质定位、蛋白质的运输、有丝分裂细胞周期、细胞周期调节、DNA代谢过程的正调控、水解酶活性的调节等生物过程。
Table 2. Mixed test group (GLQ1_vs_GLQ2) differential lncRNA screening results
表2. 混测组(GLQ1_vs_GLQ2)差异lncRNA筛选结果

Figure 2. Venn diagram of lncRNA differential expression between all groups
图2. 组间差异表达lncRNA韦恩图

Figure 3. Mixed test group (GLQ1_vs_GLQ2) difference lncRNA co-expression gene GO enrichment classification statistics
图3. 混测组(GLQ1_vs_GLQ2)差异lncRNA共表达基因GO富集分类统计图

Figure 4. Mixed test group (GLQ1_vs_GLQ2) differential lncRNA co-expression gene KEGG pathway enrichment scatter diagram
图4. 混测组(GLQ1_vs_GLQ2)差异lncRNA共表达基因KEGG途径富集散点图
3.2. 差异microRNA的筛选及富集分析
将多组比较的差异miRNA个数进行统计,绘制维恩图,直观展现出各个比较组共有及特有的差异miRNA数目(见图5)。如图6所示,四组建交集miRNA数目为零,因此以混测组(GLQ1_vs_GLQ2)所得结果进行后期分析。混测组(GLQ1_vs_GLQ2)差异microRNA共计15个(见表3),如图6所示。其中上调8个包hsa-miR-3157-3p、hsa-miR-6761-5p、hsa-miR-3124-5p、hsa-miR-5009-5p、hsa-miR-216b-3p、hsa-miR-296-3p、novel_912、novel_328;下调7个,hsa-miR-135b-5p、hsa-miR-377-3p、hsa-miR-136-5p、hsa-miR-381-3p、hsa-miR-153-5p、hsa-miR-299-3p、hsa-miR-4473。富集的GO相关的所有差异miRNA靶基因(见表4,无确定靶基因则未列出)的分类统计图,从左往右依次为生物学过程、细胞组分、分子功能包括,如图7所示。结果显示差异miRNA主要参与细胞过程、生物调节、代谢过程、应激反应、多细胞有机体过程、细胞成分的组织或生物发生、细胞定位、信号途径、发育过程、生物过程的正调控、生物过程的负调控。如图7所示。差异miRNA靶基因的KEGG富集通路散点图如图8,主要富集的细胞通路维生素的消化吸收、肿瘤坏死因子信号通路、突触囊泡循环、唾液分泌、癌症中的蛋白多糖、蛋白质输出、癌症相关通路、胰腺癌等相关途径。(q-value的取值范围为[0, 1],越接近于零,表示富集结果的可信度越高)。
Table 3. Mixed test group (GLQ1_vs_GLQ2) differential miRNA screening results
表3. 混测组(GLQ1_vs_GLQ2)差异miRNA筛选结果
Table 4. Mixed test group (GLQ1_vs_GLQ2) differential miRNA and its target gene comparison table
表4. 混测组(GLQ1_vs_GLQ2)差异miRNA及其靶基因对照
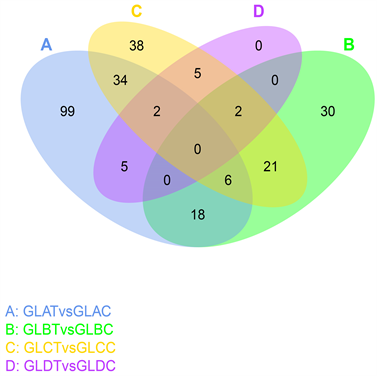
Figure 5. Venn diagram of microRNA differential expression between all groups
图5. 组间差异表达microRNA韦恩图
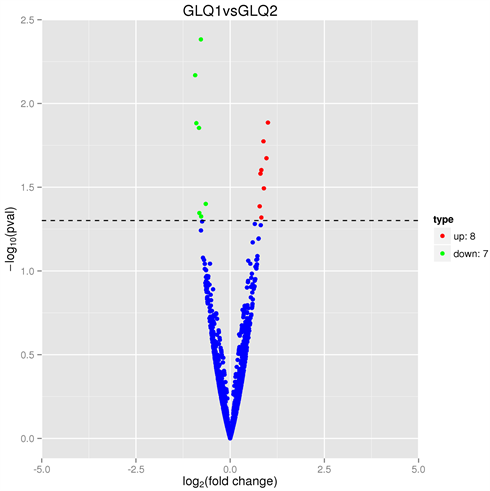
Figure 6. The volcano map of mixed test group (GLQ1_vs_GLQ2) differential microRNA
图6. 混测组(GLQ1_vs_GLQ2)差异microRNA火山图
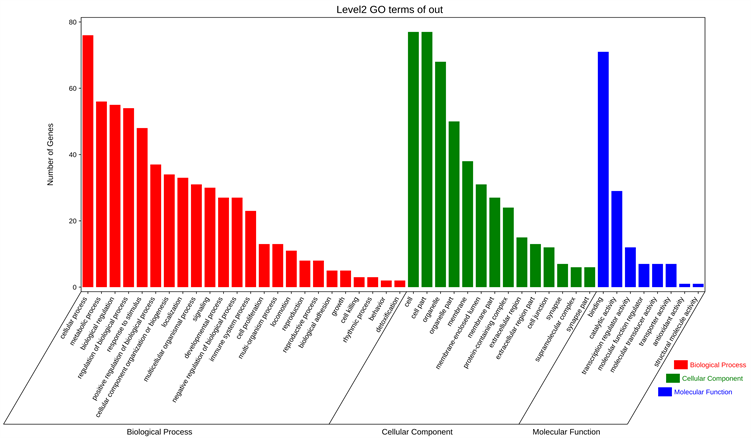
Figure 7. Mixed test group (GLQ1_vs_GLQ2) difference microRNA target gene GO enrichment classification statistics
图7. 混测组(GLQ1_vs_GLQ2)差异microRNA靶基因GO富集分类统计图

Figure 8. Mixed test group (GLQ1_vs_GLQ2) differential microRNA KEGG pathway enrichment scatter diagram
图8. 混测组(GLQ1_vs_GLQ2)差异microRNA KEGG途径富集散点图
3.3. lncRNA-miRNA-mRNA的关联分析
lncRNA具有广泛的调控作用,不仅能够直接调控DNA的结构、RNA的转录和翻译,而且具有miRNA结合位点,可以竞争性地结合miRNA,抑制miRNA对靶基因的调节作用,从而间接地调控基因表达。根据ceRNAs理论,筛选具有相同miRNA结合位点的lncRNA-gene 关系对,构建以lncRNA为起始、miRNA为核心、mRNA为靶标的lncRNA-miRNA-gene组合来构建ceRNA调控网络(图8~10)。在全转录组层面,通过ceRNA调控网络,揭示非编码RNA调控基因表达的机制。
统计筛选得到的lncRNA-miRNA-gene组合中上下调RNA的数目,可以直观展现不同组合中相关基因数目的变化。不同的形状代表不同的RNA种类,不同颜色代表上下调关系,不同的节点(node)的大小与此节点在通路中所处位置的重要程度(degree)成正比,即与此节点相连的边越多,它的重要程度越大,节点也就越大,这些节点在网络中可能处于较为核心的位置,与所研究的生物学问题更相关。
根据筛选所得数据,分别建立lncRNA (down)-miRNA (up)-mRNA (down)和lncRNA (up)-miRNA (down)-mRNA (up)三级互作关系网络(图9~11)。在lncRNA (down)-miRNA (up)-mRNA (down)网络中,以hsa-miR-296-3p为中间节点,上游lncRNA:LNC_002514、LNC_001173下调表达,使得hsa-miR-296-3p表达量增加,进而下游基因:MRAS (ENSG00000158186)、GABBR1 (ENSG00000204681)、AIFM3 (ENSG00000183773)、TRIM26 (ENSG00000234127)、ZNF875 (ENSG00000181666)、HNRNPUL1 (ENSG00000105323)、FOSL1 (ENSG00000175592)、INO80E (ENSG00000169592)、PDGFRB (ENSG00000113721)的表达量也随之下调(见表5),对以上9种下调表达的基因进行KEGG富集,共到6条通路,分别为HTLV-I感染、磷脂酶D信号通路、肌动蛋白细胞骨架的调节、Rap1信号通路、Ras信

Figure 9. Interaction map of all differential lncRNA-miRNA-mRNA
图9. 全部差异lncRNA-miRNA-mRNA互作图
号通路、MAPK信号通路。其中下调表达的lncRNA:LNC_001173与LNC_002436、LNC_001254分别调控两种miRNA:hsa-miR-6761-5p和hsa-miR-3157-3p的表达量上调,而相应的hsa-miR-6761-5p的靶基因KIAA0586 (ENSG00000100578)、hsa-miR-3157-3p的靶基因PNMA3 (ENSG00000183837)的表达量也随之下调。在lncRNA (up)-miRNA (down)-mRNA (up)互作网络中,8种上调表达的lncRNA:LNC_003613、LNC_003495、LNC_000853、LNC_000276、LNC_003672、LNC_002545、ENST00000509088、ENST00000505149可调控miRNA:hsa-miR-299-3p使之表达减少,并使下游基因SLC19A1 (ENSG00000173638)的表达量同步增加。
在以上数据筛选基础上,将上述结果与Targetscan数据库中已有的miRNA与靶基因的互作结合位点进行比对,在上述lncRNA (down)-miRNA (up)-mRNA (down)网络中只有FOSL1 (ENSG00000175592)与hsa-miR-296-3p有两个确定的互作位点,上游下调的两个:LNC_002514、LNC_001173均无文献数据支持,但LNC_001173在本次筛选结果中涉及内容过多,验证难度较大,因此拟建立由LNC_002514-hsa-miR-296-3p-FOSL1组成的轴向调控通路(图12),以备后期研究验证。
Table 5. Gene ID and its Chinese and English gene name comparison table
表5. 基因ID及其中英文基因名对照表

Figure 10. Differential lncRNA (down)-miRNA (up)-mRNA (down) interaction network diagram
图10. 差异lncRNA (down)-miRNA (up)-mRNA (down)互作网络图
Figure 11. Differential lncRNA (up)-miRNA (down)-mRNA (up) interaction network diagram
图11. 差异lncRNA (up)-miRNA (down)-mRNA (up)互作网络图

Figure 12. LNC_002514-hsa-miR-296-3p-FOSL1 axial regulation pathway diagram
图12. LNC_002514-hsa-miR-296-3p-FOSL1轴向调控通路图
4. 讨论
通过肿瘤RNA测序(RNA-Seq)监控基因和转录组的表达变化可以协助了解肿瘤的分类和发展,全转录组测序不仅可以得到每种RNA的全套分析结果,还可通过二重、三重关联分析,对目标基因的范围进行有效的缩小,进而得到准确的关键基因。本研究基于二代测序技术的全转录组测序研究,同时分析样本中差异表达的mRNA,lncRNA,miRNA,并且通过两两关联分析、三元关联分析,建立可能的ceRNA调控网络,使研究内容更加系统化,致力于深入挖掘生命现象背后的转录调控问题。
4.1. 差异lncRNA的分析
在本次测序结果中,已知有注释的差异lncRNA四种:HOXD-AS2 [26]、LINC01088 [27]、DNMBP-AS1 [28]、Metazoa_SRP [29]。其中HOXD-AS2与多种癌症密切相关,在其与胶质瘤相关研究中,研究人员对胶质瘤细胞系和正常星形胶质细胞系进行了针对lncRNA表达谱高通量的芯片检测,结果表明:HOXD-AS2在神经胶质瘤细胞中升高,与神经胶质瘤的分级和预后不良有关;HOXD-AS2的丢失可通过诱导体外细胞周期G1阻滞来抑制神经胶质瘤细胞的生长,进而胶质瘤的增殖受到抑制。除胶质瘤外还与肺癌细胞的耐药性、胃癌、卵巢癌、乳腺癌和皮肤鳞状细胞癌的发生及恶化密切相关;DNMBP-AS1已有明确研究证实其可作为肝癌患者预后的重要生物标记。Metazoa_SRP已有研究表明其与肺纤维化 [30]、骨肉瘤 [31] 的发生有关,同时在乳腺癌、结直肠癌表型的易感性标记有极高的关联性 [32],并且在人脑的发育及相关精神疾病 [29] 的研究中具有深远影响;在非小细胞肺癌(NSCLC)的研究中,linc01088和EZH2在NSCLC组织和细胞系中均高表达:抑制linc01088就抑制了NSCLC细胞的增殖,并延长了G1期,同时缩短了S和G2-M期;RIP分析揭示了linc01088与EZH2之间的直接结合关系;抑制linc01088或EZH2诱导p21表达的上调,随后抑制了肿瘤的生长;证明了linc01088可以通过与EZH2结合抑制p21来促进细胞增殖,从而加剧NSCLC的肿瘤发生。因此,linc01088可能是潜在的癌基因和新型抗肿瘤治疗的靶标 [33]。在卵巢癌中,linc01088可通过靶向miR-24-1-5p抑制卵巢上皮细胞的肿瘤发生 [34],并且与胃癌、肝癌 [35] 和子宫内膜癌 [34] 发生发展的多个重要通路相关。而CTC-458G6.2和CTB-1I21.1在目前的研究中尚未有明确机制表明其与疾病发生或预后的影响及意义,但现有数据及分析可知,这两个lncRNA在大脑中特异性表达,并且与多个已知与胶质瘤相关基因具有互作关系,可作为日后研究的重要方向。这些lncRNA与相关文献报道表达水平相一致,目前在胶质瘤发病机制研究领域也有多篇报道
本次测序也发现多个未注释的差异lncRNA,但其对胶质瘤的调控机制还有待进一步实验验证。而lncRNA:LNC_002514、LNC_001173、LNC_002436和LNC_001254目前在人类疾病方面尚未研究,因此作为潜在生物标记,为今后的研究验证工作提供方向。
4.2. 差异miRNA的分析
在本次测序结果中,差异表达的miRNA:hsa-miR-296-3p在人脐带静脉内皮细胞(HUVEC)中的miRNA和mRNA表达谱相关研究中差异调节涉及细胞粘附,血管生成,细胞周期,JAK-STAT信号传导,MAPK信号传导,一氧化氮信号传导,VEGF信号传导和伤口愈合途径的基因。此外,基于q-PCR的miRNA和mRNA上调和下调实验研究表明,hsa-miR-296-3p等miRNA有效地影响斜管恒温器刺激微重力(MG)条件下HUVEC的细胞增殖和血管功能。对相互作用组结果的共识表明,暴露于短期MG的HUVEC转录组的波动受限,这可能导致较高水平的内皮功能,如血管生成和血管形成 [36]。hsa-miR-6761-5p、hsa-miR-3157-3p在最新研究中被列为颅内动脉瘤发展的潜在靶标 [37],hsa-miR-3157-3p在大肠癌相关的ceRNA调控网络中重要节点 [38]。与hsa-miR-299-3p相关的研究较为广泛,与黑色素瘤 [39]、多形性胶质母细胞瘤 [40]、透明肾细胞癌 [41] 等多种常见癌症相关,并且被列为喉癌 [42]、结肠癌 [43]、颅内动脉瘤 [37] 等疾病的独立预后因素。
4.3. 下游靶基因分析
受差异miRNA调控的下游靶基因共12种:PDGFRB [44]、MRAS [45]、INO80E [46]、hnRNPUL1 [47]、FOSL1 [48]、ZNF875 [49]、AIFM3 [50]、GABBR1 [51]、TRIM26 [52]、PNMA3 [53]、KIAA0586 [54]、SLC19A1 [55]。
血小板衍生的生长因子(PDGF)信号通路驱动高度神经胶质瘤(HGG)进程,并且PDGF受体(PDGFRs)的表达增强是胶质母细胞瘤(GBMs)子集中的一种普遍的突变。PDGFRA在神经胶质瘤细胞中表达,而PDGFRB主要限于与神经胶质瘤相关的基质。相关研究还显示,小胶质细胞(TAMMs)的空间位置与已经获得PDGFRB在小鼠和人类低级神经胶质瘤和HCGs中表达的肿瘤细胞亚群的扩展相关。此外,M2极化的小胶质细胞诱导神经胶质瘤细胞中PDGFRB的表达并刺激其迁移能力 [56]。
MRAS是相对于经典RAS癌蛋白而言最接近的,并且共享大多数调节和效应子相互作用。但是它也具有独特的功能,包括与SHOC2和蛋白磷酸酶1 (PP1)结合时,具有磷酸酶调节亚基的功能。这种磷酸酶复合物调节RAF激酶激活周期中的关键步骤,并提供有效的ERK途径激活和RAS转化所需的关键坐标输入。MRAS突变很少在癌症中发生,但在某些情况下表达失调可能在肿瘤发生中起作用。在RAS病Noonan综合征中确实发生了MRAS的激活突变(以及SHOC2和PP1),这强调了RAS-ERK途径中MRAS的关键作用。MRAS在细胞迁移和分化中也具有独特作用,其性质与细胞极性调节中的关键作用相一致 [45]。
INO80E在评估异基因造血干细胞移植(HSCT)在胰腺导管腺癌治疗中的免疫学作用中,是及其重要的免疫应答指标。并能够对临床和生物学相关细胞免疫组库的建立产生影响 [46]。
2020年4月FOSL1在低级神经胶质瘤(LGG)的研究中,通过比较不同类型肿瘤之间的FOSL1表达,探索TCGA-LGG和亚组中FOSL1表达与总体生存(OS)之间的关联。结果表明:LGG中的FOSL1表达在21种不同的癌症中排名第一。FOSL1表达较低的LGG的OS较长(P < 0.001)。星形细胞瘤组的FOSL1表达最高,OS最短,其次是少星形细胞瘤和少突胶质细胞瘤(P < 0.05)。1p19q共缺失或IDH突变亚组具有较低的FOSL1表达和较长的OS (P < 0.001)。与相应的组相比,FOSL1表达较低的LGG的OS比以下组更长,因此0FOSL1是LGG和亚组的预后标志物 [47]。
4.4. LNC_002514-hsa-miR-296-3p-FOSL1轴向调控的分析
在lncRNA (down)-miRNA (up)-mRNA (down)网络中,经过生物信息学分析后发现LNC_002514表达量下降,释放hsa-miR-296-3p作用于下游基因:FOSL1。
在2019年12月发表的关于冠状动脉疾病(CAD)的生物标记文章中,研究人员建立一个用于诊断CAD的circRNA-miRNA-mRNA三重网络。使用接收器工作特性(ROC)曲线评估了hsa-miR-21-3p和hsa-miR-296-3p、circYOD1的预后价值。circ-YOD1的ROC曲线下面积(AUC)为0.824,hsa-miR-21-3p的AUC为0.731,hsa-miR-296-3p为0.776。因而在对cad中circRNA-miRNA-mRNA的三重调控网络的研究表明,hsa-miR-296-3p在潜在生物标记的研究过程中有着及其重要作用 [57]。
FOSL1 (Fos样抗原1,也称为Fra-1)是FOS家族的成员,它由四个基因成员c-FOS,FOSB,FOSL1和FOSL2组成,所有这些基因编码包含亮氨酸拉链的蛋白质。与Jun (c-jun, junB, junD),激活转录因子(ATF)和肌腱膜纤维肉瘤(MAF)结合的FOS是激活蛋白1 (AP-1)复合物的成员。FOSL1是调节正常细胞生长,分化和凋亡的重要核转录因子。在宫颈癌发病机制的最新研究中,研究人员发现Fra-1抑制宫颈癌细胞的增殖,同时还促进细胞凋亡并影响细胞周期分布。此外,Fra-1上调子宫颈癌细胞中的STAT1表达并调节p53信号通路活性。Fra-1的过表达通过改变HeLa细胞中的sirtuin 1 (SIRT1)表达来抑制细胞衰老,而Fra-1的过表达恢复了线粒体疾病并抑制了HeLa细胞中的代谢重编程。STAT1沉默会削弱Fra-1对宫颈癌细胞生长的抑制作用,而STAT1的敲低则逆转了由Fra-1引起的HeLa细胞对细胞衰老和线粒体功能障碍的影响。STAT1沉默也恢复了子宫颈癌细胞的代谢重编程。其研究结果表明,Fra-1通过STAT1介导的p53信号通路调节来抑制子宫颈癌细胞的生长和Warburg效应 [48]。此外,已发现它在包括肺癌在内的许多癌症中具有重要作用。在胶质瘤的相关研究中,当FOSL1沉默时,改变了恶性神经胶质瘤细胞的形态,锚定非依赖性生长和体内肿瘤形成。它的过表达赋予非致瘤性神经胶质瘤细胞系致瘤性 [56]。
LNC_002514在目前的研究中尚未有明确的研究结果,通过生物信息学分析结果可知LNC_002514与hsa-miR-296-3p及其下游基因FOSL1存在可能的调控关系,因而可作为潜在的研究方向。
在本次检测结果中,FOSL1的表达量与预期结果相反,因而有如下可能:1. 在组织切取过程中因癌组织与癌旁组织边界不明显、难以准确区分,所以所取得的样品可能不完全是胶质瘤组织,故而检测结果与理论依据不符。2. 本次测序样本容量较小,因此可能无法准确展现FOSL1在恶性胶质瘤中的表达特点。
5. 结论
本研究通过RNA-seq和生物信息学分析,筛选出了在四例脑胶质瘤样本中差异表达的lncRNA106种,上调表达52种,下调表达54种;差异表达miRNA15种,上调表达8种,下调表达7种;这些非编码RNA主要参与有机循环化合物代谢过程、胞内运输、细胞蛋白质定位、蛋白质的运输、有丝分裂细胞周期、细胞周期调节、DNA代谢过程的正调控、水解酶活性的调节等生物过程。并且根据lncRNA、miRNA、mRNA的靶向关系建立了ceRNAs调控网络,在此基础上提出了LNC_002514-hsa-miR-296-3p-FOSL1轴向调控的可能性,并对其作出简要分析。上述nc-RNA的筛选及调控轴向的提出将为以后脑胶质瘤相关研究提供新的方向。
致谢
感谢王仕超师姐提供的数据支持,感谢导师张焱如教授在各个方面无微不至的照顾与帮助,感谢舍友为我提供了安静的写作环境,感谢父母多年来的养育与经济和精神支持,感谢男友的支持与安慰,谢谢大家!
基金项目
内蒙古自治区自然科学基金项目(2017MS0307)。
文章引用
李明扬,王仕超,凌 宇,刘春霞,曹俊伟,张焱如. 影响人脑胶质瘤细胞发生、增殖关键lncRNA、miRNA的筛选与分析
Screening and Analysis of Key lncRNA and miRNA Affecting the Occurrence and Proliferation of Human Glioma Cells[J]. 亚洲肿瘤科病例研究, 2021, 10(01): 1-23. https://doi.org/10.12677/ACRPO.2021.101001
参考文献
- 1. Rees, J.H. (2011) Diagnosis and Treatment in Neuro-Oncology: An Oncological Perspective. The British Journal of Ra-diology, 84, 82-89. https://doi.org/10.1259/bjr/18061999
- 2. 中华医学会病理学分会脑神经病理学组. 2016 WHO中枢神经系统肿瘤分类第4版修订版概述及胶质瘤部分介绍[J]. 中华病理学杂志, 2016, 45(11): 745-747.
- 3. Muir, C.S., Storm, H.H. and Polednak, A. (1994) Brain and Other Nervous System Tumours. Cancer Survival, 19-20, 369-392.
- 4. 中国中枢神经系统胶质瘤诊断与治疗指南编写组. 中华医学杂志[J]. 中华医学会, 2016, 96(7): 485-509.
- 5. Strom, Q.T., Gittleman, H., Liao, P., et al. (2016) CBTRUS Statistical Report: Primary Brain and Other Central Nervous System Tumors Diagnosed in the United States in 2009-2013. Neuro-Oncology, 18, v1-v75. https://doi.org/10.1093/neuonc/now207
- 6. Ng, H.K. and Lam, P.Y. (1998) The Molecular Genetics of Central Nervous System Tumors. Pathology, 30, 196-202. https://doi.org/10.1080/00313029800169236
- 7. Schlessinger, J. (2000) Cell Signaling by Receptor Tyrosine Ki-nases. Cell, 103, 211-225. https://doi.org/10.1016/S0092-8674(00)00114-8
- 8. 王曦, 陈定. LncRNAs作为miRNA的靶模拟物调节miRNA [J]. 科技创新导报, 2016, 13(28): 176-177.
- 9. Marziali, A. and Akeson, M. (2001) New DNA Sequencing Methods. Annual Review of Biomedical Engineering, 3, 195-223. https://doi.org/10.1146/annurev.bioeng.3.1.195
- 10. Goodwin, S., McPherson, J.D. and McCombie, W.R. (2016) Coming of Age: Ten Years of Next-Generation Sequencing Technologies. Nature Reviews Genetics, 17, 333-351. https://doi.org/10.1038/nrg.2016.49
- 11. Niedzicka, M., Fijarczyk, A., Dudek, K., et al. (2016) Molecular Inver-sion Probes for Targeted Resequencing in Non-Model Organisms. Scientific Reports, 6, Article No. 24051. https://doi.org/10.1038/srep24051
- 12. Xu, J., Gao, C., Zhang, F., et al. (2016) Differentially Expressed lncRNAs and mRNAs Identified by Microarray Analysis in GBS Patients vs Healthy Controls. Scientific Reports, 6, Article No. 21819. https://doi.org/10.1038/srep21819
- 13. Xu, J., Zhang, F., Gao, C., et al. (2017) Microarray Analysis of lncRNA and mRNA Expression Profiles in Patients with Neuromyelitis Optica. Molecular Neurobiology, 54, 2201-2208. https://doi.org/10.1007/s12035-016-9754-0
- 14. Wang, Z., Gerstein, M. and Snyder, M. (2009) RNA-Seq: A Revolutionary Tool for Transcriptomics. Nature Reviews Genetics, 10, 57-63. https://doi.org/10.1038/nrg2484
- 15. Yang, Y., et al. (2018) Analyzing the Interactions of mRNAs, miRNAs, lncRNAs and circRNAs to Predict Competing Endogenous RNA Networks in Glioblastoma. Journal of Neu-ro-Oncology, 137, 493-502. https://doi.org/10.1007/s11060-018-2757-0
- 16. 《中国中枢神经系统胶质瘤诊断与治疗指南》编写组. 中华医学杂志[J]. 中华医学会, 2016, 96(7): 485-509.
- 17. Kim, D., Pertea, G., Trapnell, C., et al. (2013) TopHat2: Accurate Alignment of Transcriptomes in the Presence of Insertions, Deletions and Gene Fusions. Genome Biology, 14, R36. https://doi.org/10.1186/gb-2013-14-4-r36
- 18. Trapnell, C., Williams, B.A., Pertea, G., et al. (2010) Transcript Assembly and Quantification by RNA-Seq Reveals Unannotated Transcripts and Isoform Switching during Cell Differ-entiation. Nature Biotechnology, 28, 511-515. https://doi.org/10.1038/nbt.1621
- 19. Liang, S., Luo, H., Bu, D., et al. (2013) Utilizing Sequence Intrinsic Compo-sition to Classify Protein-Coding and Long Noncoding Transcripts. Nucleic Acids Research, 41, e166. https://doi.org/10.1093/nar/gkt646
- 20. Kong, L., Zhang, Y., Ye, Z.Q., et al. (2007) CPC: Assess the Pro-tein-Coding Potential of Transcripts Using Sequence Features and Support Vector Machine. Nucleic Acids Research, 35, W345-W349. https://doi.org/10.1093/nar/gkm391
- 21. Punta, M., Coggill, P.C., Eberhardt, R.Y., et al. (2011) The Pfam Protein Families Database. Nucleic Acids Research, 40, D290-D301. https://doi.org/10.1093/nar/gkr1065
- 22. Lin, M.F., Jungreis, I. and Kellis, M. (2011) PhyloCSF: A Comparative Genomics Method to Distinguish Protein Coding and Non-Coding Regions. Bioinformatics, 27, i275-i282. https://doi.org/10.1093/bioinformatics/btr209
- 23. Young, M.D., Wakefield, M.J., Smyth, G.K., et al. (2010) Gene Ontology Analysis for RNA-seq: Accounting for Selection Bias. Genome Biology, 11, R14. https://doi.org/10.1186/gb-2010-11-2-r14
- 24. Mao, X., Cai, T., Olyarchuk, J.G., et al. (2005) Automated Genome Annotation and Pathway Identification Using the KEGG Orthology (KO) as a Controlled Vocabulary. Bioinformatics, 21, 3787-3793. https://doi.org/10.1093/bioinformatics/bti430
- 25. Kanehisa, M., Araki, M., Goto, S., et al. (2008) KEGG for Linking Genomes to Life and the Environment. Nucleic Acids Research, 36, D480-D484. https://doi.org/10.1093/nar/gkm882
- 26. Yao, L., Ye, P.C., Tan, W., et al. (2020) Decreased Expression of the Long Non-Coding RNA HOXD-AS2 Promotes Gastric Cancer Progression by Targeting HOXD8 and Activating PI3K/Akt Signaling Pathway. World Journal of Gastrointestinal Oncology, 12, 1237-1254. https://doi.org/10.4251/wjgo.v12.i11.1237
- 27. Zhang, W., Fei, J., Yu, S., et al. (2018) LINC01088 Inhibits Tu-morigenesis of Ovarian Epithelial Cells by Targeting miR-24-1-5p. Scientific Reports, 8, Article No. 2876. https://doi.org/10.1038/s41598-018-21164-9
- 28. Xue, C., Zhao, Y., Jiang, J. and Li, L. (2020) Expression Levels of lncRNAs Are Prognostic for Hepatocellular Carcinoma Overall Survival. American Journal of Translational Research, 12, 1873-1883.
- 29. O’Brien, H.E., Hannon, E., Hill, M.J., et al. (2018) Expression Quantitative Trait Loci in the De-veloping Human Brain and Their Enrichment in Neuropsychiatric Disorders. Genome Biology, 19, 194. https://doi.org/10.1186/s13059-018-1567-1
- 30. Deist, M.S., Gallardo, R.A., Bunn, D.A., Dekkers, J.C.M., Zhou, H. and Lamont, S.J. (2017) Resistant and Susceptible Chicken Lines Show Distinctive Responses to Newcastle Disease Virus Infection in the Lung Transcriptome. BMC Genomics, 18, 989. https://doi.org/10.1186/s12864-017-4380-4
- 31. Rathe, S.K., Popescu, F.E., Johnson, J.E., et al. (2019) Identifica-tion of Candidate Neoantigens Produced by Fusion Transcripts in Human Osteosarcomas. Scientific Reports, 9, Article No. 358. https://doi.org/10.1038/s41598-018-36840-z
- 32. Pande, M., Joon, A., Brewster, A.M., et al. (2018) Genetic Sus-ceptibility Markers for a Breast-Colorectal Cancer Phenotype: Exploratory Results from Genome-Wide Association Studies. PLoS ONE, 13, e0196245. https://doi.org/10.1371/journal.pone.0196245
- 33. Liu, J.Q., Feng, Y.H., Zeng, S. and Zhong, M.Z. (2020) linc01088 Promotes Cell Proliferation by Scaffolding EZH2 and Repressing p21 in Human Non-Small Cell Lung Cancer. Life Sciences, 241, Article ID: 117134. https://doi.org/10.1016/j.lfs.2019.117134
- 34. Ai, H., Xie, W., Xiu, A.H., et al. (2018) The Down-Regulation of Long Non-Coding RNA LINC01088 Is Associated with the Poor Prognosis of Epithelial Ovarian Cancer Patients. Eu-ropean Review for Medical and Pharmacological Sciences, 22, 5836-5841.
- 35. Liu, J., Yao, Y., Hu, Z., Zhou, H. and Zhong, M. (2019) Transcriptional Profiling of Long-Intergenic Noncoding RNAs in Lung Squamous Cell Carcinoma and Its Value in Diagnosis and Prognosis. Molecular Genetics & Genomic Medicine, 7, e994. https://doi.org/10.1002/mgg3.994
- 36. Kasiviswanathan, D., Chinnasamy Perumal, R., Bhuvaneswari, S., et al. (2020) Interactome of miRNAs and Transcriptome of Human Umbilical Cord Endothelial Cells Exposed to Short-Term Simulated Microgravity. NPJ Microgravity, 6, 18. https://doi.org/10.1038/s41526-020-00108-6
- 37. Liao, B., Zhou, M.X., Zhou, F.K., et al. (2020) Exosome-Derived MiRNAs as Biomarkers of the Development and Progression of In-tracranial Aneurysms. Journal of Atherosclerosis and Thrombosis, 27, 545-610. https://doi.org/10.5551/jat.51102
- 38. Zhao, H., et al. (2020) Construction of ceRNA Coexpression Network and Screening of Molecular Targets in Colorectal Cancer. Disease Markers, 2020, Article ID: 2860582. https://doi.org/10.1155/2020/2860582
- 39. Motti, M.L., Minopoli, M., Di Carluccio, G., Ascierto, P.A. and Carri-ero, M.V. (2020) MicroRNAs as Key Players in Melanoma Cell Resistance to MAPK and Immune Checkpoint Inhibi-tors. International Journal of Molecular Sciences, 21, 4544. https://doi.org/10.3390/ijms21124544
- 40. Henriksen, M., Johnsen, K.B., Andersen, H.H., Pilgaard, L. and Duroux, M. (2014) MicroRNA Expression Signatures Determine Prognosis and Survival in Glioblastoma Multiforme—A Systematic Overview. Molecular Neurobiology, 50, 896-913. https://doi.org/10.1007/s12035-014-8668-y
- 41. He, H., Wang, L., Zhou, W., et al. (2015) MicroRNA Expression Profiling in Clear Cell Renal Cell Carcinoma: Identification and Functional Validation of Key miRNAs. PLoS ONE, 10, e0125672. https://doi.org/10.1371/journal.pone.0125672
- 42. Liu, H., Sun, Y., Tian, H., et al. (2019) Characterization of Long Non-Coding RNA and Messenger RNA Profiles in Laryngeal Cancer by Weighted Gene Co-Expression Network Anal-ysis. Aging (Albany NY), 11, 10074-10099. https://doi.org/10.18632/aging.102419
- 43. Roman-Canal, B., Tarragona, J., Moiola, C.P., et al. (2019) EV-Associated miRNAs from Peritoneal Lavage as Potential Diagnostic Biomarkers in Colorectal Cancer. Journal of Translational Medicine, 17, 208. https://doi.org/10.1186/s12967-019-1954-8
- 44. Gilbertson, R.J. and Clifford, S.C. (2003) PDGFRB Is Overex-pressed in Metastatic Medulloblastoma. Nature Genetics, 35, 197-198. https://doi.org/10.1038/ng1103-197
- 45. Young, L.C. and Rodriguez-Viciana, P. (2018) MRAS: A Close but Un-derstudied Member of the RAS Family. Cold Spring Harbor Perspectives in Medicine, 8, a033621. https://doi.org/10.1101/cshperspect.a033621
- 46. Omazic, B., Ayoglu, B., Löhr, M., et al. (2017) A Preliminary Report: Radical Surgery and Stem Cell Transplantation for the Treatment of Patients with Pancreatic Cancer. Journal of Immunotherapy, 40, 132-139. https://doi.org/10.1097/CJI.0000000000000164
- 47. Zhu, J., Zhao, Y.P. and Zhang, Y.Q. (2020) Low Expression of FOSL1 Is Associated with Favorable Prognosis and Sensitivity to Radiation/Pharmaceutical Therapy in Lower Grade Glioma. Neurological Research, 42, 522-527. https://doi.org/10.1080/01616412.2020.1748323
- 48. Zhang, M., Liang, L., He, J., et al. (2020) Fra-1 Inhibits Cell Growth and the Warburg Effect in Cervical Cancer Cells via STAT1 Regulation of the p53 Signaling Pathway. Frontiers in Cell and Developmental Biology, 8, Article ID: 579629. https://doi.org/10.3389/fcell.2020.579629
- 49. Oguri, T., Katoh, O., Takahashi, T., et al. (1998) The Krüppel-Type Zinc Finger Family Gene, HKR1, Is Induced in Lung Cancer by Exposure to Platinum Drugs. Gene, 222, 61-67. https://doi.org/10.1016/S0378-1119(98)00464-8
- 50. Chua-On, D., Proungvitaya, T., Tummanatsakun, D., et al. (2020) Apoptosis-Inducing Factor, Mitochondrion-Associated 3 (AIFM3) Protein Level in the Sera as a Prognostic Marker of Cholangiocarcinoma Patients. Biomolecules, 10, 1021. https://doi.org/10.3390/biom10071021
- 51. Li, J., Zhang, C., Yuan, X., Ren, Z. and Yu, Z. (2020) Correlations between Stemness Indices for Hepatocellular Carcinoma, Clinical Characteristics, and Prognosis. American Journal of Translational Research, 12, 5496-5510.
- 52. Wang, K., Chai, L., Qiu, Z., Zhang, Y., Gao, H. and Zhang, X. (2019) Overexpression of TRIM26 Suppresses the Proliferation, Metastasis, and Glycolysis in Papillary Thyroid Carcinoma Cells. Journal of Cellular Physiology, 234, 19019-19027. https://doi.org/10.1002/jcp.28541
- 53. Takaji, M., Ko-matsu, Y., Watakabe, A., Hashikawa, T. and Yamamori, T. (2009) Paraneoplastic Antigen-Like 5 Gene (PNMA5) Is Preferentially Expressed in the Association Areas in a Primate Specific Manner. Cerebral Cortex, 19, 2865-2879. https://doi.org/10.1093/cercor/bhp062
- 54. Wang, T., Xuan, Z., Dou, Y., et al. (2019) Identification of Novel Mu-tations in Preaxial Polydactyly Patients through Whole-Exome Sequencing. Molecular Genetics & Genomic Medicine, 7, e690. https://doi.org/10.1002/mgg3.690
- 55. Li, C., Yuan, B., Yu, X., et al. (2020) SLC19A1 May Serve as a Po-tential Biomarker for Diagnosis and Prognosis in Osteosarcoma. Clinical Laboratory, 66. https://doi.org/10.7754/Clin.Lab.2020.200246
- 56. Wallmann, T., Zhang, X.M., Wallerius, M., et al. (2018) Mi-croglia Induce PDGFRB Expression in Glioma Cells to Enhance Their Migratory Capacity. iScience, 9, 71-83. https://doi.org/10.1016/j.isci.2018.10.011
- 57. Miao, L., Yin, R.X., Zhang, Q.H., et al. (2019) A Novel circR-NA-miRNA-mRNA Network Identifies circ-YOD1 as a Biomarker for Coronary Artery Disease. Scientific Reports, 9, Article No. 18314. https://doi.org/10.1038/s41598-019-54603-2
NOTES
*通讯作者。
