Statistics and Application
Vol.
10
No.
03
(
2021
), Article ID:
42865
,
10
pages
10.12677/SA.2021.103035
基于可变剪接的乳腺癌患者预后特征分析
姜蔚
华北电力大学数理学院,北京

收稿日期:2021年5月6日;录用日期:2021年5月20日;发布日期:2021年6月1日

摘要
本文在对传统临床数据分析的基础上,引入可变剪接数据,运用单因素COX回归、Lasso回归等方法筛选得到与生存显著相关的可变剪接事件,研究了影响乳腺癌患者总生存率的关键因素,构造了较为理想的10-可变剪接事件预后模型,挖掘了可变剪接事件与乳腺癌预后的关联性。结果表明,可变剪接事件可以作为独立预后因子,较好地预测乳腺癌患者的生存情况,这为医学人员进一步认识与理解乳腺癌的预后特征提供了理论依据和数据支撑,也为进一步实验验证提供了潜在目标。
关键词
可变剪接,COX回归,Lasso回归,生存分析
Analysis of Prognostic Associated Alternative Splicing Signatures in Breast Cancer
Wei Jiang
School of Mathematics and Physics, North China Electric Power University, Beijing

Received: May 6th, 2021; accepted: May 20th, 2021; published: Jun. 1st, 2021

ABSTRACT
Based on the analysis of traditional clinical data, this paper introduces alternative splicing events, and survival-associated alternative splicing events were selected by using univariate COX regression analysis and Lasso regression analysis. Then, we study the key factors affecting the overall survival rate of breast cancer patients, construct the prognosis model of 10-survival-associated alternative splicing events, and excavate the correlation between alternative splicing events and prognosis of breast cancer patients. The results show that alternative splicing events can be used as an independent prognostic factor to predict the survival of breast cancer patients. It is a theoretical basis and data support for medical personnel to further understand the prognostic characteristics of breast cancer, and also a potential target for further experimental verification.
Keywords:Alternative Splicing, COX Regression, Lasso Regression, Survival Analysis

Copyright © 2021 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
乳腺癌(Breast Cancer, BRCA)是全球最常见的恶性肿瘤之一,占所有癌症病例的11%以上,在全球癌症发病率中位列第二;乳腺癌中99%发生在女性,是女性发病率最高的恶性肿瘤 [1] [2]。近十年来,我国乳腺癌发病率呈现明显上升趋势,大城市发病率高达万分之五左右。因此,深刻挖掘乳腺癌的预后分子机制,寻找新型治疗靶标,具有重大的现实意义。
可变剪接(Alternative Splicing, AS)在发育、分化和癌症等过程中发挥着非常重要的作用。可变剪接是产生蛋白质多样性的主要机制,是一个受严格调控的生物过程,通过这个过程,任何特定的基因产物数量都可以大大增加,因此,可变剪接的错误调节可能引起多种人类疾病。近年来,越来越多证据表明可变剪接的失调与癌症的发生发展有关 [3] [4] [5],此外,也有许多研究表明可变剪接在癌症治疗方面具有一定的临床潜力 [6] [7] [8] [9] [10]。
本文在对传统临床数据分析的基础上,对乳腺癌的可变剪接事件进行了综合挖掘,采用COX回归、Lasso回归等方法分析研究了影响乳腺癌患者总生存率(Overall Survival, OS)的关键因素,构造了较为理想的10-AS事件预后模型,揭示了可变剪接事件对乳腺癌预后的潜在价值。
2. 材料与方法
2.1. 材料
本文选取来自癌症基因组数据库(The Cancer Genome Atlas, TCGA)的乳腺癌患者数据,下载其基因表达矩阵和临床数据,其中乳腺癌患者的随访时间限定在90~8605天,这些患者的临床信息包含性别(Gender)、年龄(Age)、T (Tumor)分期、N (Node)分期、M (Metastasis)分期等。其可变剪接数据可在TCGASpliceSeq数据库中获得,TCGASpliceSeq是TCGA中mRNA可变剪接模式的公开数据库资源,选取样本的可变剪接表达估计值(Percent-spliced-in, PSI)百分比大于75%的部分。
2.2. 方法
2.2.1. 数据处理
利用Rstudio及相关软件包对数据进行处理:对乳腺癌患者样本的基因数据进行探针转换和剪接因子(Splicing Factor, SF)提取;对相应的临床数据进行补缺和数字化;对相应的AS数据进行补缺和过滤,删除主体内容中均值小于0.05和标准差小于0.01的AS事件。
2.2.2. 与生存相关AS事件的筛选
选用COX单因素回归分析筛选与生存显著相关的AS事件;选用LASSO回归,取一倍标准误差下的最简模型对应的λ值,去除相关性高的AS事件,防止模型过拟合,降低临床检测成本。
2.2.3. 模型的建立与评价
以P < 0.05为标准筛选出与乳腺癌生存相关的AS事件,建立单因素COX回归模型:
,
当回归系数β > 0时,协变量X的取值越大,风险函数 的值越大,病人死亡的风险越高;回归系数β < 0时,协变量X的取值越大,风险函数 的值越小,病人死亡的风险越低 [11]。
选择每个事件类型对应PSI值的中位数为患者分类的阈值,其中分数小于该数值记作低风险,反之记作高风险,再通过所得模型进行风险评分。
绘制Kaplan-Meier曲线比较高低风险两组乳腺癌患者的生存情况;绘制受试者工作特征(Receiver Operating Characteristic, ROC)曲线评价模型的预测准确性。
3. 结果
3.1. 可变剪接的upset图、火山图和气泡图
一般认为可变剪接有七种主要形式 [12],如图1,分别为外显子跳跃(Exon Skip, ES)、可变供体位点(Alternative Donor Site, AD)、可变受体位点(Alternative Acceptorsite, AA)、内含子保留(Retained Intron, RI)、外显子互斥(Mutually Exclusive Exons, ME)、可变启动子(Alternative Promoter, AP)和可变终止子(Alternative Terminator, AT)。
对乳腺癌患者样本的所有AS事件进行补缺和过滤,绘制其Upset图,如图2。
对乳腺癌患者样本的所有AS事件分别进行单因素COX回归,以P < 0.05为标准,共筛选出2042个与生存显著相关的AS事件,见表1;绘制对应的Upset图,如图3;绘制火山图,如图4。
对乳腺癌患者样本的7类AS事件分别绘制气泡图,横坐标为z值,纵坐标为与生存显著相关的前20个AS事件名称,如图5。

Figure 1. Alternative splicing events
图1. 可变剪接事件

Figure 2. Upset plot of alternative splicing
图2. AS事件upset图

Figure 3. Upset plot of survival-associated alternative splicing events
图3. 生存相关AS事件Upset图
Table 1. Survival-associated alternative splicing events
表1. 生存相关AS事件
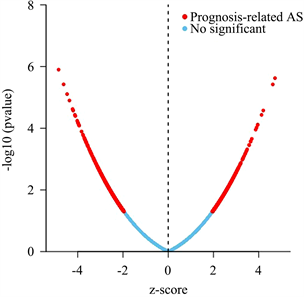
Figure 4. Volcano plot of alternative splicing
图4. 火山图

Figure 5. Bubble plot of 7 alternative splicing events
图5. 七类AS事件气泡图
3.2. 特征筛选和模型建立
利用LASSO回归对2042个与生存相关的AS事件降维,绘制相关参数选择示意图和AS事件系数分布图,如图6所示。观察左侧图象可知,随着λ的增大,相关AS事件的回归系数逐渐趋于零;再根据右侧图象选取合适的λ值:图中红色曲线最低处对应最小模型误差,穿过此处的虚线顶部为对应变量个数,此时筛选出的AS事件为16个;而其右侧虚线是在其一倍标准误内的最简模型,对应变量个数为14个。
由于这两个 对应的模型误差变化不大,这里选择更简洁的模型。

Figure 6. Parameter selection and coefficient distribution in LASSO regression
图6. LASSO回归的参数选择和系数分布
利用逐步回归对14个AS事件进一步筛选,通过向前向后算法挑选出对生存有显著影响的特征以达到最优,最终将10个AS事件纳入COX比例风险回归模型,见表2所示。并由此计算BRCA患者的风险评分,这里风险评分定义为以相应COX回归系数为权重的14个AS事件PSI值的线性组合,并按其中位值将患者分为高低风险两组,如表3所示。
Table 2. Construction of univariate COX model
表2. 构建单因素COX模型
Table 3. Risk score and classification
表3. 风险评分和分类情况
3.3. 生存分析
绘制Kaplan-Meier曲线,如图7所示。显然,低风险组的患者相较于高风险组均有着较高的中位生存时间,且患者的生存率差异显著(P < 0.01),对比患者的5年生存率,高风险组约为60%,而低风险组约为87%;绘制ROC曲线,如图8所示,此时AUC > 0.85,该模型分类效果优秀。

Figure 7. Kaplan-Meier plot of alternative splicing prognostic signatures
图7. 可变剪接预后特征的Kaplan-Meier曲线

Figure 8. ROC curve of alternative splicing prognostic signatures
图8. 可变剪接预后特征的ROC曲线
3.4. 独立预后分析
为考察模型是否能作为独立预后因子,将BRCA患者的临床数据和风险评分合并成一个矩阵,绘制相应的森林图,如图9、图10所示,若P值均小于0.05,则认为该指标可作为独立预后因子使用;中间灰色线代表HR = 1,图形出现在右边即为高风险因素,出现在左边即为低风险因素。
因此,BRCA患者AS事件风险评分可以独立于其他的临床性状作为独立的预后因子,即在临床应用上可以通过测量AS事件的PSI值,预测BRCA患者的生存期。

Figure 9. Univariate regression analysis of prognostic factors
图9. 单因素独立预后分析

Figure 10. Multi-factors regression analysis of prognostic factors
图10. 多因素独立预后分析
4. 总结与讨论
4.1. 总结
本文在对传统临床数据分析的基础上,对乳腺癌的AS事件进行了综合挖掘,采用COX回归、LASSO回归等方法分析研究了影响乳腺癌患者生存时间的关键因素,并以此提出了较为理想的10-AS事件预后模型,揭示了AS事件与乳腺癌预后的关联性,揭示了AS事件对乳腺癌预后的潜在价值。这为医学人员进一步认识与理解乳腺癌的预后特征提供了理论依据和数据支撑,也为进一步实验验证提供了具体目标。
4.2. 讨论
相对于较为完备的基因表达数据,可变剪接机制的研究还尚未成熟,全基因组选择性剪接在肺癌中的研究仍处于空白,剪接事件对癌症预后和临床诊断的敏感性还有待提升。但与此同时,这个领域的留白给研究人员带来了更具体的实验方向和更丰富的科研课题。已有研究表明与基因层次的显著差异分析相比,调控剪接事件的剪接因子在生存分析方面有更好的结果,因此,调控可变剪接的剪接因子很有可能成为癌症治疗更具潜力的靶基因。
文章引用
姜 蔚. 基于可变剪接的乳腺癌患者预后特征分析
Analysis of Prognostic Associated Alternative Splicing Signatures in Breast Cancer[J]. 统计学与应用, 2021, 10(03): 355-364. https://doi.org/10.12677/SA.2021.103035
参考文献
- 1. El-Serag, H.B. and Rudolph, K.L. (2007) Hepatocellular Carcinoma: Epidemiology and Molecular Carcinogenesis. Gastroenterology, 132, 2557-2576.
https://doi.org/10.1053/j.gastro.2007.04.061 - 2. Mikulits, W. (2018) Epithelial to Mesenchymal Transition in Hepatocellular Carcinoma. Future Oncology, 5, 1169.
https://doi.org/10.2217/fon.09.91 - 3. Fu, X.D. and Ares, M. (2014) Context-Dependent Control of Alternative Splicing by RNA-Binding Proteins. Nature Reviews Genetics, 15, 689-701.
https://doi.org/10.1038/nrg3778 - 4. Song, X., Zeng, Z., Wei, H., et al. (2017) Alternative Splicing in Cancers: From Aberrant Regulation to New Therapeutics. Seminars in Cell & Developmental Biology, 75, 13-22.
https://doi.org/10.1016/j.semcdb.2017.09.018 - 5. Li, Y., Sun, N., Lu, Z., et al. (2017) Prognostic Alternative mRNA Splicing Signature in Non-Small Cell Lung Cancer. Cancer Letters, 393, 40-51.
https://doi.org/10.1016/j.canlet.2017.02.016 - 6. 刘文斌, 王兵, 方刚, 石晓龙, 许鹏. 基于中值的JS散度可变剪接差异分析研究[J]. 电子与信息学报, 2020, 42(6): 1392-1400.
- 7. Park, E., Pan, Z., Zhang, Z., et al. (2018) The Expanding Landscape of Alternative Splicing Variation in Human Populations. American Journal of Human Genetics, 102, 11-26.
https://doi.org/10.1016/j.ajhg.2017.11.002 - 8. Lin, J.C. (2018) Therapeutic Applications of Targeted Alternative Splicing to Cancer Treatment. International Journal of Molecular Sciences, 19, 75.
https://doi.org/10.3390/ijms19010075 - 9. Martinez-Montiel, N., Rosas-Murrieta, N.H., Ruiz, M.A., et al. (2018) Alternative Splicing as a Target for Cancer Treatment. International Journal of Molecular Sciences, 19, 545.
https://doi.org/10.3390/ijms19020545 - 10. 王科俊, 吕俊杰, 冯伟兴, 等. 可变剪接与疾病的生物信息学研究概况[J]. 生命科学研究, 2011, 15(1): 86-94.
- 11. Mauger, E.A., Wolfe, R.A. and Port, F.K. (1995) Transient Effects in the Cox Proportional Hazards Regression Model. Statistics in Medicine, 14, 1553-1565.
https://doi.org/10.1002/sim.4780141406 - 12. Wang, E.T., Sandberg, R., Luo, S.J., et al. (2008) Alternative Isoform Regulation in Human Tissue Transcriptomes. Nature, 456, 470-476.
https://doi.org/10.1038/nature07509
