 Advance in Microbiology 微生物前沿, 2013, 2, 83-89 http://dx.doi.org/10.12677/amb.2013.23016 Published Online September 2013 (http://www.hanspub.org/journal/amb.html) Role of DNA Damage Response (SOS response) on the Control of Bacterial Resistance Yan Zhang1,2, Du ohong She ng1* 1State Key Laboratory of Microbial Technology, Shandong University, Jinan 2Shandong Shuili Hospital, Jinan Email: *dhsheng@sdu.edu.cn Received: Feb. 13th, 2013; revised: Mar. 26th, 2013; accepted: Apr. 5th, 2013 Copyright © 2013 Yan Zhang, Duohong Sheng. This is an open access article distributed under the Creative Commons Attribution License, which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited. Abstract: Bacterial drug resistance is one of the world’s most pressing public health threats. Especially in today, new antimicrobial drugs are no longer being developed at a rate that can keep pace with microbial evolution. The mechanism for the emergence, spread and reversion of bacterial resistance has become a research hotspot. Recently, it has been proved that bacterial DNA damage response (also named SOS response) acts an important role in the evolution of drug resistance. Here, we reviewed the relation between SOS response and bacterial drug resistance. Keywords: Bacterium; SOS Response; Drug Resistance; RecA 细菌 DNA 损伤诱导反应(SOS 反应) 在控制细菌耐药性中的作用 张 岩1,2,盛多红 1* 1山东大学微生物技术国家重点实验室,济南 2山东水利医院,济南 Email: *dhsheng@sdu.edu.cn 收稿日期:2013 年2月13 日;修回日期:2013 年3月26 日;录用日期:2013 年4月5日 摘 要:细菌耐药性是当前最紧迫的公众健康问题之一,尤其在目前新型抗菌药物研发落后于耐药菌进化速度 的情况下,细菌耐药性的发生、传播以及消除的机制,已经成为人们关注的热点。近来研究发现,细菌DNA 损 伤诱导反应(SOS 反应)在细菌进化耐药性的过程中扮演着重要角色。在这里,本文对 SOS反应与细菌耐药性的 关系、研究进展以及当前存在问题作一阐述。 关键词:细菌;SOS 反应;耐药性;RecA 1. 引言 DNA 是生命遗传信息的载体,是细胞内最重要的 组成成分,它的稳定性和细胞的生死突变直接相关, 直接或间接损伤 DNA 是临床常用的杀死细胞的手段。 在正常的细胞里,损伤的 DNA 可以被损伤诱导反应 (DNA Damage Response, DDR)迅速识别,并激活 DNA 修复蛋白,修复药物造成的DNA 损伤,因此,DNA 损伤诱导反应被认为是细胞产生耐药性的重要原因。 比如人类肿瘤细胞可以通过诱导DNA 修复,抵御放 化疗对 DNA 的损伤进而表现为耐药性[1]。细菌 DNA 损伤诱导反应又称为 SOS 反应,是指细菌应对基因组 *通讯作者。 Copyright © 2013 Hanspub 83  细菌 DNA 损伤诱导反应(SOS 反应)在控制细菌耐药性中的作用 DNA 损伤的重要保护机制。关于 SOS 反应与细菌耐 药性的关系已经引起人们的关注,控制 SOS 反应一度 被认为是解决细菌耐药性的最终手段。在这里,我们 对SOS 反应与细菌耐药性之间的关系做一简单综述。 2. 耐药基因的稳定性是细菌耐药性泛滥的 重要原因 80 年代以来,由于抗菌药物(Antimicrobial or Antibiotic)的大量使用,尤其是不合理使用,导致细菌 产生对抗菌药物的耐药性和多重耐药性的现象日趋 严峻,加重了细菌感染的致病率,致死率以及患者的 治疗费用支出。世界卫生组织在 2007 年就把细菌耐 药性列为威胁人类的重大公共安全问题之一;2011年 世界卫生日更是提出了“抵御细菌耐药性”的主题, 向全世界发出抵抗细菌耐药性的呼声。细菌耐药性在 我国广泛存在,《柳叶刀》杂志 2008 年曾刊文指出, 中国医院内感染的致病菌有40%为耐药菌,耐药菌的 增长率达26%,居世界首位。2011 年全国细菌耐药监 测结果表明,对常用的β-内酰胺类,喹诺酮类抗菌药 物的耐药性普遍达到50%以上,有些更是达到70%~ 90%。细菌耐药性和耐药菌感染已经成为我们人类面 临的最大难题之一,目前细菌不仅对天然来源的抗菌 药物和半合成抗菌药物存在耐药性,而且对人工新合 成的抗菌药物也产生了耐药性,目前几乎所有的抗菌 药物,均出现了耐药菌株。而新型抗菌药物的研发速 度远远落后于细菌耐药性的发展速度。人类在对抗细 菌的战斗中,几乎快到了无药可用的尴尬境地[2,3]。 耐药菌株产生的主要原因是不规范使用或滥用 抗菌药物,减少抗菌药物的使用可以明显降低耐药菌 株的发生,比如在匈牙利,当医生减少使用青霉素控 制肺炎感染时,抗青霉素的肺炎链球菌耐药菌株明显 减少[4]。然而事情没有想象的那么简单,控制抗菌药 物的使用,耐药菌只是减少并没有消失,当重新使用 相应抗菌药物时,耐药性迅速反弹,残余的耐药菌株 又会重新占据优势[5]。 耐药基因的稳定性是细菌耐药性泛滥的另一个 原因,细菌容易接受耐药基因,却不容易丢失耐药基 因,即使在没有抗菌药物的环境中,细菌耐药性也往 往能稳定存在。1) 耐药基因或单独或以整合子、转座 子及温和噬菌体的形式整合到细菌染色体上,随染色 体一起复制而稳定存在;2) 耐药基因往往成簇存在或 与其他抗逆基因一起存在,一种环境因子的压力均可 以保持多种耐药基因的稳定遗传;3) 细菌的耐药性在 环境中普遍存在,耐药基因存在于致病菌以及非致病 菌、大量使用抗菌药物的医疗机构以及外部正常环境 中,在土壤、水体和空气中广泛分布,形成一个耐药 基因可以相互交流的大环境[6];4) 现行的污染处理方 法不能有效抑制耐药性传播。比如现在,在医院、工 厂、家庭常用的季铵盐类消毒剂,杀死细菌,却不能 消除耐药基因,在消毒的同时还促进了抗菌药物耐药 性的发展和传播[7]。南京大学环境学院的马黎平等的 研究证明了常用的污泥法处理城市废水不能消除反 而促进了带有耐药基因整合子的扩散[8]。 因此,为了应对急剧增加的耐药菌感染,除了加 强抗菌药物的管理工作,控制耐药菌株的出现,研究 细菌耐药机制,遏制当前耐药性的迅速扩散,以及消 除或者减少已经普遍存在的耐药菌株或耐药基因,是 当前科研工作者急需解决的问题之一。 3. 抗菌药物诱导的细菌 SOS 反应 抗菌药物的作用机理主要是抑制细菌细胞内特 有的重要代谢过程,临床用抗菌药物发挥作用需要以 下几个步骤:首先是进入细菌细胞、在细胞内达到抑 制浓度、和靶目标结合,干扰细菌代谢,抑制细菌生 长甚至死亡。任何一个步骤的破坏都会影响抗菌药物 的效果。目前对抗菌药物与作用靶位的作用机制以及 对细胞造成的直接影响,已经了解的比较多。然而对 药物处理后细菌是如何反应,以及如何导致细菌死 亡,都还不清楚。最近,Kohanski 等提出了一个通用 的抗菌药物杀菌模型[9,10]。如图1所示,抗菌药物与 细菌细胞内初级靶位结合后,通过一系列代谢反馈活 性调节,激活细胞内还原力的过度氧化消耗。NADPH 通过电子传递链进行氧化,在氧化过程中产生的超氧 基化合物,和铁硫蛋白中的铁发生芬顿反应(Fenton reaction),进而生成氧化力极强的羟自由基,严重破 坏细菌细胞内的DNA、蛋白质和细胞膜等细胞结构, 最终导致细胞死亡。提高细胞内的抗氧化能力,可以 明显提高细菌对抗菌药物的耐受性[11]。在此过程中, 无论是抗菌药物作用于靶位DNA 产生的直接损伤, 还是随后产生的羟自由基等对DNA 的间接损伤,都 Copyright © 2013 Hanspub 84 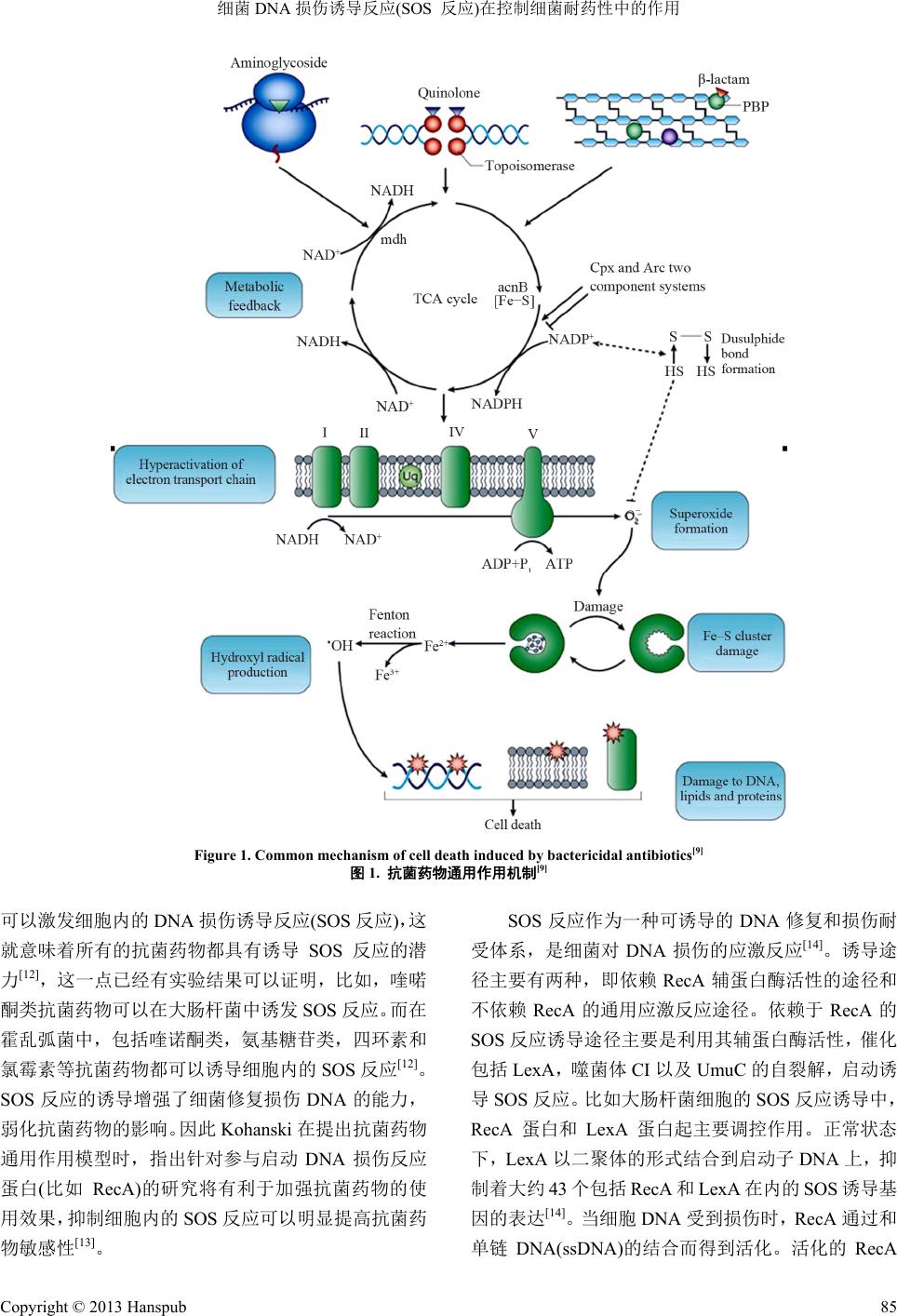 细菌 DNA 损伤诱导反应(SOS 反应)在控制细菌耐药性中的作用 Copyright © 2013 Hanspub 85 Figure 1. Common mechanism of cell death induced by bactericidal antibiotics[9] 图1. 抗菌药物通用作用机制[9] SOS反应作为一种可诱导的 DNA修复和损伤耐 受体系,是细菌对 DNA损伤的应激反应[14]。诱导途 径主要有两种,即依赖 RecA 辅蛋白酶活性的途径和 不依赖 RecA 的通用应激反应途径。依赖于 RecA的 SOS 反应诱导途径主要是利用其辅蛋白酶活性,催化 包括 LexA,噬菌体 CI以及UmuC 的自裂解,启动诱 导SOS 反应。比如大肠杆菌细胞的 SOS 反应诱导中, RecA 蛋白和 LexA 蛋白起主要调控作用。正常状态 下,LexA 以二聚体的形式结合到启动子DNA 上,抑 制着大约 43个包括RecA 和LexA在内的 SOS 诱导基 因的表达[14]。当细胞DNA 受到损伤时,RecA 通过和 单链 DNA(ssDNA)的结合而得到活化。活化的RecA 可以激发细胞内的 DNA 损伤诱导反应(SOS 反应),这 就意味着所有的抗菌药物都具有诱导 SOS反应的潜 力[12],这一点已经有实验结果可以证明,比如,喹喏 酮类抗菌药物可以在大肠杆菌中诱发SOS反应。而在 霍乱弧菌中,包括喹诺酮类,氨基糖苷类,四环素和 氯霉素等抗菌药物都可以诱导细胞内的SOS 反应[12]。 SOS反应的诱导增强了细菌修复损伤 DNA 的能力, 弱化抗菌药物的影响。因此 Kohanski 在提出抗菌药物 通用作用模型时,指出针对参与启动DNA 损伤反应 蛋白(比如RecA) 的研究将有利于加强抗菌药物的使 用效果,抑制细胞内的 SOS反应可以明显提高抗菌药 物敏感性[13]。 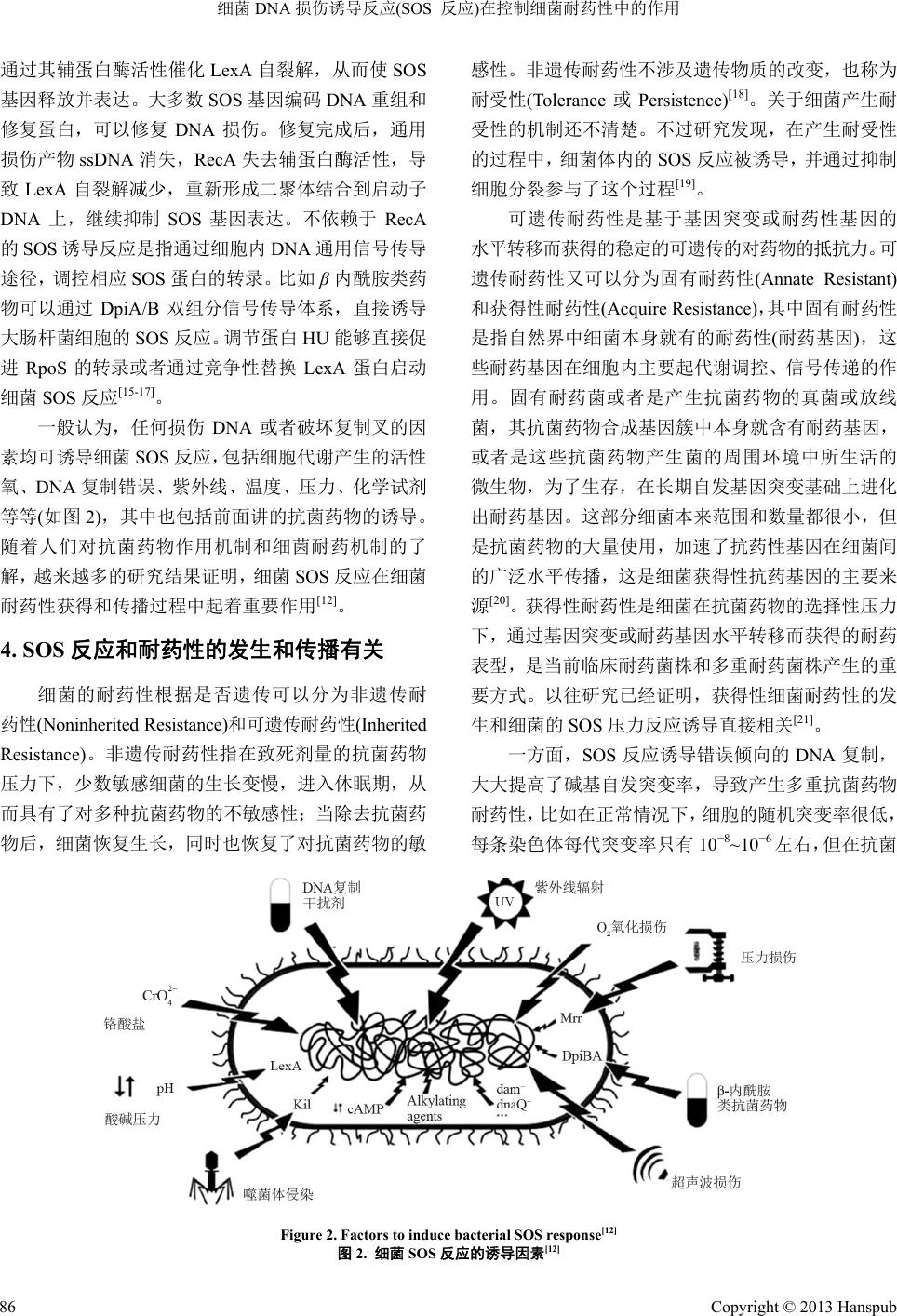 细菌 DNA 损伤诱导反应(SOS 反应)在控制细菌耐药性中的作用 通过其辅蛋白酶活性催化 LexA 自裂解,从而使 SOS 基因释放并表达。大多数SOS 基因编码 DNA 重组和 修复蛋白,可以修复 DNA损伤。修复完成后,通用 损伤产物ssDNA消失,RecA 失去辅蛋白酶活性,导 致LexA 自裂解减少,重新形成二聚体结合到启动子 DNA 上,继续抑制SOS基因表达。不依赖于 RecA 的SOS 诱导反应是指通过细胞内 DNA通用信号传导 途径,调控相应 SOS 蛋白的转录。比如 β内酰胺类药 物可以通过 DpiA/B 双组分信号传导体系,直接诱导 大肠杆菌细胞的SOS 反应。调节蛋白HU能够直接促 进RpoS 的转录或者通过竞争性替换LexA蛋白启动 细菌 SOS反应[15-17]。 一般认为,任何损伤 DNA或者破坏复制叉的因 素均可诱导细菌SOS 反应,包括细胞代谢产生的活性 氧、DNA 复制错误、紫外线、温度、压力、化学试剂 等等(如图 2),其中也包括前面讲的抗菌药物的诱导。 随着人们对抗菌药物作用机制和细菌耐药机制的了 解,越来越多的研究结果证明,细菌SOS 反应在细菌 耐药性获得和传播过程中起着重要作用[12]。 4. SOS反应和耐药性的发生和传播有关 细菌的耐药性根据是否遗传可以分为非遗传耐 药性(Noninherited Resistance)和可遗传耐药性(Inherited Resistance)。非遗传耐药性指在致死剂量的抗菌药物 压力下,少数敏感细菌的生长变慢,进入休眠期,从 而具有了对多种抗菌药物的不敏感性;当除去抗菌药 物后,细菌恢复生长,同时也恢复了对抗菌药物的敏 感性。非遗传耐药性不涉及遗传物质的改变,也称为 耐受性(Tolerance 或Persistence)[18]。关于细菌产生耐 受性的机制还不清楚。不过研究发现,在产生耐受性 的过程中,细菌体内的SOS反应被诱导,并通过抑制 细胞分裂参与了这个过程[19]。 可遗传耐药性是基于基因突变或耐药性基因的 水平转移而获得的稳定的可遗传的对药物的抵抗力。可 遗传耐药性又可以分为固有耐药性(Annate Resistant) 和获得性耐药性(Acquire Resistance),其中固有耐药性 是指自然界中细菌本身就有的耐药性(耐药基因),这 些耐药基因在细胞内主要起代谢调控、信号传递的作 用。固有耐药菌或者是产生抗菌药物的真菌或放线 菌,其抗菌药物合成基因簇中本身就含有耐药基因, 或者是这些抗菌药物产生菌的周围环境中所生活的 微生物,为了生存,在长期自发基因突变基础上进化 出耐药基因。这部分细菌本来范围和数量都很小,但 是抗菌药物的大量使用,加速了抗药性基因在细菌间 的广泛水平传播,这是细菌获得性抗药基因的主要来 源[20]。获得性耐药性是细菌在抗菌药物的选择性压力 下,通过基因突变或耐药基因水平转移而获得的耐药 表型,是当前临床耐药菌株和多重耐药菌株产生的重 要方式。以往研究已经证明,获得性细菌耐药性的发 生和细菌的SOS 压力反应诱导直接相关[21]。 一方面,SOS 反应诱导错误倾向的DNA 复制, 大大提高了碱基自发突变率,导致产生多重抗菌药物 耐药性,比如在正常情况下,细胞的随机突变率很低, 每条染色体每代突变率只有10−8~10−6左右,但在抗菌 Figure 2. Factors to induce bacterial SOS response[12] 图2. 细菌SOS反应的诱导因素[12 ] Copyright © 2013 Hanspub 86  细菌 DNA 损伤诱导反应(SOS 反应)在控制细菌耐药性中的作用 药物选择压力下,随着SOS 反应的诱导,细菌的突变 频率提高了10~10,000 倍,大大增加了抗菌药物抗性 出现的几率[22];另外,SOS 反应诱导蛋白大多数是 DNA 修复蛋白,可以修复抗菌药物带来的 DNA 损伤, 提高细菌对抗菌药物的耐药性,比如链球菌 (Strepto- coccus pneumoniae)热休克蛋白 ClpL 的诱导表达可以 增强链球菌对青霉素的耐药性;SOS 基因RecA的缺 失可以明显降低细菌对多种抗菌药物的耐药性。另一 方面,SOS 反应还可以促进耐药基因的水平转移。早 在1986 年,就有报道称亚致死剂量的β内酰胺抗菌 药物可以促进四环素耐药质粒水平转移率提高 100~ 1000 倍。另外,DNA 重组酶、整合酶、以及转座酶 都是 SOS反应诱导蛋白,它们分别通过促进 DNA 链 交换、整合子耐药基因盒的捕获、以及转座子的移动, 加速了耐药基因在细菌内以及细菌间的广泛传播。同 时,SOS 反应还可以促进大肠杆菌许多噬菌体阻遏蛋 白CI 的自裂解,噬菌体阻遏蛋白的裂解启动了温和 噬菌体的脱阻遏。噬菌体本身携带的大量基因,通过 噬菌体的转染,进一步促进了耐药基因的水平传播 [22-24]。 5. 存在的问题 抗菌药物、细菌耐药性和 SOS 反应存在着紧密关 联。抗菌药物可以诱导SOS 反应;SOS 反应可以弱化 抗菌药物的效果,发展耐药性,细菌耐药性可以抵御 抗菌药物的压力,让细菌更好的生存。理论上讲,阻 断从抗菌药物到耐药性的任何一个环节,均可以控制 耐药性的发生发展。因此,控制细菌SOS反应诱导作 为一个新的药物靶点受到热捧,被认为是解决细菌耐 药性的最终途径[25]。由于对抗菌药物诱导SOS 反应的 具体过程、诱导机制、以及 SOS 对耐药性产生和消除 的影响和具体机制还不清楚,以 SOS为靶位,研制新 药和治疗方法的进展并不顺利,还存在很多问题。 归纳起来有下面几点:1) 抗菌药物作用模式的统 一性,按照 Kohanski 提出的抗菌药物通用作用模式, 抗菌药物诱导的过氧化氢损伤包括DNA 在内的生物 大分子,损伤的DNA 可以诱导细菌SOS 反应,理论 上,所有抗菌药物均有诱导SOS 反应的潜力[9]。然而 对于有些致病菌,比如大肠杆菌,细胞内的SOS 反应 不能被青霉素、四环素或者卡那霉素等抗菌药物诱导 [12],但可以被低浓度的 H2O2(1~3 mM)诱导发生[26]。 这种现象的存在是因为大肠杆菌中没有类似的统一 诱导模式,还是因为有不同的诱导的方式或者其他未 明因素的影响,都还需要进一步的研究。2) 到目前为 止,关于抗菌药物可以诱导基因转变的观点已经得到 共识。尽管 SOS 反应可以促进突变的发生,但是耐药 性突变不一定通过SOS诱导反应诱导。比如 β内酰胺 类药物可以直接和 dinB(PolIV)相互作用,通过不依赖 于SOS 反应的方式诱导基因突变[27]。Elena Lopez的 研究也显示[28],不同浓度的亚致死浓度环丙沙星在不 诱导 SOS反应的情况下,明显促进了大肠杆菌同源性 重组率,但 SOS 诱导可以进一步增加 DNAS 同源性 重组水平。因此,简单抑制 SOS 反应不能完全抑制突 变发生。3) 尽管 RecA/LexA 在很多细菌的SOS反应 诱导中起重要作用,但是抑制 RecA 活性或者抑制 LexA 自裂解并不能有效抑制 SOS 反应。抗菌药物诱 导的细菌 SOS反应不像现在所了解的那么简单,在 SOS反应关键蛋白RecA 或LexA缺失的情况下,细 菌的同源重组反应也可以被活化,修复蛋白也可以被 诱导激活[12,29]。另外,很多致病菌,比如奈瑟氏淋球 菌、不动杆菌等,细胞内缺少 LexA和RecA为核心 的典型 SOS 反应[30]。4) SOS诱导基因既包括促进基 因突变的相关基因,比如错误倾向的DNA聚合酶等, 也包括很多维持基因组稳定的DNA 损伤修复蛋白, 对抗菌药物诱导产生的 DNA损伤进行修复,修复突 变的基因,理论上降低了耐药突变的形成。抑制细菌 SOS 反应,细菌基因组更容易突变,增加了出现耐药 性突变的发生几率。5) 细菌 SOS 反应可以通过突变 和促进基因水平转移来引入耐药性,理论上也可以通 过逆向作用方式消除耐药性。比如通过DNA 修复作 用、通过耐药基因回复突变、通过激活转座酶切除转 座子、通过激活整合酶切除耐药基因盒或改变基因盒 排列等方式,可以加速逆转细菌耐药性。因此,在目 前耐药性广泛分布现状下, 如何有效利用细菌 SOS 反应这种适应进化机制,既能增强现有抗菌药物效 果,减少耐药性发生,又能消除现有细菌耐药性才是 我们需要的结果。 6. 结论 以往对细菌耐药性的研究多是从彻底消灭致病 Copyright © 2013 Hanspub 87  细菌 DNA 损伤诱导反应(SOS 反应)在控制细菌耐药性中的作用 菌作为出发点,包括分析耐药菌株和耐药机制寻找新 药进行抑制,大规模筛选新型抗菌药物用于杀死细菌 和耐药细菌,以及现在各国普遍推广的抗菌药物的有 效使用管理等,都是为了强化我们对付细菌的武器(抗 菌药物)。然而对于我们的对手细菌的研究却相对较 少,从细菌的角度来讲,细菌发展耐药性只是为了更 好的生存,是细菌本身的一种进化适应机制,只是当 前人们对抗菌药物的大量使用加速了细菌耐药性的 进化过程[31]。细菌耐药性的发展史也是一直伴随着抗 菌药物的存在,比如β-内酰胺酶已经存在于细菌中有 10 亿多年了[32]。面对一定浓度的抗菌药物,细菌如果 能降解利用抗菌药物,则把它用作营养底物;不能利 用则不吸收或者排出,这类细菌对相应的抗菌药物具 有不敏感性;如果既不能利用、又不能排出则可能对 细胞正常代谢造成影响,代谢的受阻可以激发细胞内 的应激反应(包括SOS反应),此时的抗菌药物又是一 种诱导信号分子。诱导应激反应的结果又可分为三 种,诱导相关的酶系、消除抗菌药物影响;通过遗传 学改变(基因突变和基因水平转移)或非遗传学改变 (改变细胞结构或生活周期产生耐受性)产生耐药性或 抗药性;细胞死亡。因此,了解抗菌药物的作用机制, 了解抗菌药物存在时细菌是如何诱导 SOS反应的? 诱导的 SOS 反应对细菌耐药性的发生、转移的机制是 什么?了解这种进化机制可能更有利于我们认识和 控制细菌耐药性。 7. 致谢 感谢山东大学微生物技术国家重点实验室徐海 教授对本文的审校与指导。本文得到山东省优秀中青 年科学家科研奖励基金(BS2010SW014)的支持。 参考文献 (References) [1] K. Barakat, M. Gajewski and J. A. Tuszynski. DNA repair inhibitors: The next major step to improve cancer therapy. Current Topics in Medicinal Chemistry, 2012, 12(12): 1376- 1390. [2] M. O. Sommer, G. Dantas. Antibiotics and the resistant micro- biome. Current Opinion in Microbiology, 2011, 14(5): 556-563. [3] P. C. Appelbaum. 2012 and beyond: Potential for the start of a second pre-antibiotic era? Journal of Antimicrobial Chemothera- py, 2012, 67(9): 2062-2068. [4] R. Nowak. Hungary sees an improvement in penicillin resistance. Science, 1994, 264: 364. [5] D. N. Gerding, T. A. Larson, R. A. Hughes, M. Weiler, C. Shan- holtzer and L. R. Peterson. Aminoglycoside resistance and ami- noglycoside usage: Ten years of experience in one hospital. Antimicrobial Agents and Chemotherapy, 1991, 35: 1284-1290. [6] N. Han, D. Sheng and H. Xu. Role of Escherichia coli strain subgroups, integrons, and integron-associated gene cassettes in dissemination of antimicrobial resistance in aquatic environ- ments of Jinan, China. Water Science and Technology, 2012, 66 (11): 2385-2392. [7] K. Hegstad, S. Langsrud, B. T. Lunestad, A. Scheie, M. Sunde and S. P. Yazdankhah. Does the wide use of quaternary ammo- nium compounds enhance the selection and spread of antimic- robial resistance and thus threaten our health? Microbial Drug Resistance, 2010, 16(2): 91-104. [8] L. Ma, X. Zhang, S. Cheng, Z. Zhang, .P Shi, B. Liu, B. Wu and Y. Zhang. Occurrence, abundance and elimination of class 1 integrons in one municipal sewage treatment plant. Ecotoxico- logy, 2011, 20(5): 968-973. [9] M. A. Kohanski, D. J. Dwyer and J. J. Collins. How antibiotics kill bacteria: From targets to networks. Nature Reviews Micro- biology, 2010, 8(6): 423-435. [10] M. A. Kohanski, D. J. Dwyer and B. Hayete. A common mecha- nism of cellular death induced by bactericidal antibiotics. Cell, 2007, 130(5): 797-810. [11] P. Belenky, J. J. Collins. Antioxidant strategies to tolerate anti- biotics. Science, 2011, 334(6058): 915-916. [12] Z. Baharoglu, D. Mazel. Vibrio cholerae triggers SOS and muta- genesis in response to a wide range of antibiotics: A route to- wards multiresistance. Antimicrobial Agents and Chemotherapy, 2011, 55(5): 2438-2441. [13] V. Sharma, Y. Sakai, K. A. Smythe and Y. Yokobayashi. Knock- down of recA gene expression by artificial small RNAs in Es- cherichia coli. Biochemical and Biophysical Research Com- munications, 2013, 430(1): 256-259. [14] I. Erill, S. Campoy and J. Barbé. Aeons of distress: An evo- lutionary perspective on the bacterial SOS response. FEMS Microbiology Reviews, 2007, 31(6): 637-656. [15] C. Miller, L. E. Thomsen, C. Gaggero, R. Mosseri, H. Ingmer and S. N. Cohen. SOS response induction by beta-lactams and bacterial defense against antibiotic lethality. Science, 2004, 305: 1629-1631. [16] A. Balandina, L. Claret, R. Hengge-Aronis and J. Rouviere- Yaniv. The Escherichia coli histone-like protein HU regulates rpoS translation. Molecular Microbiology, 2011, 39: 1069-1079. [17] O. Preobrajenskaya, A. Boullard, F. Boubrik, M. Schnarr and J. Rouviere-Yaniv. The protein HU can displace the LexA repress- sor from its DNA-binding sites. Molecular Microbiology, 1994, 13: 459-467. [18] B. R. Levin. Microbiology. Noninherited resistance to antibiotics. Science, 2004, 305(5690): 1578-1579. [19] T. Dorr, K. Lewis and M. Vulić. SOS response induces persis- tence to fluoroquinolones in Escherichia coli. PLoS Genetics, 2009, 5(12): Article ID: e1000760. [20] V. M. D’Costa, C. E. King and L. Kalan. Antibiotic resistance is ancient. Nature, 2011, 477(7365): 457-461. [21] A, Giedraitiene, A.Vitkauskiene and R. Naginiene. Antibiotic resistance mechanisms of clinically important bacteria. Medicina (Kaunas), 2011, 47(3): 137-146. [22] A. Jolivet-Gougeon, B. Kovacs and S. Le Gall-David. Bacterial hypermutation: Clinical implications. Journal of Medical Micro- biology, 2011, 60(Pt 5): 563-573. [23] T. D. Tran, H. Y. Kwon and E. H. Kim. Decrease in penicillin susceptibility due to heat shock protein ClpL in Streptococcus pneumoniae. Antimicrobial Agents and Chemotherapy, 2011, 55(6): 2714-2728. [24] J. Aranda. Acinetobacter baumannii RecA protein in repair of DNA damage, antimicrobial resistance, general stress response, and virulence. Journal of Bacteriology, 2011, 193(15): 3740- 3747. [25] T. J. Wigle, S. F. Singleton. Directed molecular screening for RecA ATPase inhibitors. Bioorganic & Medicinal Chemistry Letters, 2007, 17: 3249-3253. Copyright © 2013 Hanspub 88  细菌 DNA 损伤诱导反应(SOS 反应)在控制细菌耐药性中的作用 Copyright © 2013 Hanspub 89 [26] J. A. Imlay, S. Linn. Mutagenesis and stress responses induced in Escherichia coli by hydrogen peroxide. Journal of Bacteri- ology, 1987, 169: 2967-2976. [27] T. Mori, T. Nakamura, N. Okazaki, A. Furukohri, H. Maki and M. T. Akiyama. Escherichia coli DinB inhibits replication fork progression without significantly inducing the SOS response. Genes and Genetic Systems, 2012, 87(2): 75-87. [28] E. Lopez, J. Blazquez. Effect of subinhibitory concentrations of antibiotics on intrachromosomal homologous recombination in Escherichia coli. Antimicrobial Agents and Chemotherapy, 2009, 53(8): 3411-3415. [29] R. Singh, K. R. Ledesma, K. Chang and V. H. Tam. Impact of recA on levofloxacin exposure-related resistance development. Antimicrobial Agents and Chemotherapy, 2010, 54(10): 4262- 4268. [30] J. Hare, J. Bradley and C. Lin. Diverse DNA damage responses in Acinetobacter include the capacity for DNA damage-induced mutagenesis in the opportunistic pathogens Acinetobacter bau- mannii and Acinetobacter ursingii. Microbiology, 2011, 158(Pt 3): 601-611. [31] R. Jayaraman. Antibiotic resistance: An overview of mecha- nisms and a paradigm shift. Current Science, 2009, 96(11): 1475-1484. [32] B. Hall, M. Barlow. Evolution of the serine β-lactamases: Past, present and future. Drug Resistance Updates, 2004, 7: 111-123. |