Advances in Clinical Medicine
Vol.
13
No.
01
(
2023
), Article ID:
60898
,
14
pages
10.12677/ACM.2023.131134
生物信息学和网络药理学分析槲皮素靶向BIRC5抑制胶质母细胞瘤的增殖并影响预后
胡阳春1,陈宇2,舒磊2,夏志宇2,代兴亮1*
1安徽医科大学第一附属医院神经外科,安徽 合肥
2安徽医科大学第一临床学院临床医学,安徽 合肥
收稿日期:2022年12月28日;录用日期:2023年1月21日;发布日期:2023年1月31日

摘要
目的:本文旨在通过生物信息学和网络药理学方法分析影响胶质母细胞瘤预后关键基因和潜在中药靶点。方法:从基因表达数据库(GEO)获取脑胶质母细胞瘤数据集,GEO2R在线分析得到胶质瘤和正常脑组织之间差异基因。通过Venn图取三个差异基因集交集,共筛选出共有差异基因(DEGs),并进行GO和KEGG分析富集DEGs的分子功能和信号通路。将差异基因映射入STRING数据库构建蛋白相互作用(PPI)网络,并利用Cytoscape软件筛选Hub基因。GEPIA2数据库分析Hub基因转录表达及预后。TCMSP数据库获取车前子、丹参、金银花、连翘四种中药的生物信息并通过设置药物动力学标准获得活性组分,再通过perl软件从差异基因中获取对应的靶点基因,Cytoscape软件构建“中药成分-DEGs靶点”蛋白质互作网络,分析节点枢纽筛选出关联度较高的靶点基因和活性成分,与Hub基因对比获取最终的潜在关键基因和中药成分,在AutoDock和PyMol软件中进行中药成分和关键基因的分子对接验证。结果:下载得到三个数据库GSE14805、GSE29796、GSE35493,进行GEO2R分析和交集后共得到193个共有的差异表达基因,其中上调基因148个,下调基因45个,并借助Cytoscape软件获取蛋白质互作网络并筛选到15个Hub基因。GO和KEGG分析提示Hub基因主要参与细胞周期、细胞外基质的生物学过程和细胞组分中的细胞粘附连接、粘着斑及胞浆。通过网络药理学分析,建立活性成分–靶点基因相互作用网络后,筛选出潜在的治疗靶点BIRC5和靶向治疗胶质母细胞瘤的中药活性成分槲皮素。分子对接结果显示BIRC5蛋白受体和中药成分配体结合较稳定且有氢键相互作用的形成。结论:通过生物信息学和网络药理学方法分析提示槲皮素有潜在通过靶点BIRC5影响胶质母细胞瘤的增殖、侵袭和迁移,进而影响胶质母细胞瘤患者预后的作用,为胶质瘤治疗和新化疗药的研发提供了新思路。
关键词
胶质母细胞瘤,生物信息学,网络药理学,BIRC5,中药靶点,槲皮素

Quercetin Targeting BIRC5 Inhibits the Proliferation of Glioblastoma and Affects the Prognosis Based on Bioinformatics and Network Pharmacology Analysis
Yangchun Hu1, Yu Chen2, Lei Shu2, Zhiyu Xia2, Xingliang Dai1*
1Department of Neurosurgery, The First Affiliated Hospital of Anhui Medical University, Hefei Anhui
2Department of Clinical Medicine, The First Clinical College of Anhui Medical University, Hefei Anhui
Received: Dec. 28th, 2022; accepted: Jan. 21st, 2023; published: Jan. 31st, 2023

ABSTRACT
Objective: This study aims to analyze the key genes and potential targets of traditional Chinese medicine that affect the prognosis of glioblastoma through bioinformatics and network pharmacology methods. Methods: The data set of glioblastoma was obtained from the Gene Expression Omnibus (GEO), and the differential expressing genes (DEGs) between glioma and normal brain tissue were obtained by online GEO2R analysis. Three sets of differential genes were collected by Venn map, DEGs were screened, and the molecular functions and signal pathways of DEGs were analyzed and enriched by GO and KEGG. The DEGs were mapped into STRING database to construct protein interaction (PPI) network, and the Hub genes were screened by using Cytoscape software. GEPIA2 database was used to analyze the transcriptional expression and prognosis of Hub genes. The biological information of plantain seed, salvia miltiorrhiza, honeysuckle, and forsythia suspensa, and obtains the active components were obtained by setting pharmacokinetic standards. Then, the corresponding target genes are obtained from the DEGs via perl software. The Cytoscape software constructs the protein interaction network of “traditional Chinese medicine components-DEGs targets”, and analyzes the node Hub to screen out the target genes and active components with high correlation, compare with the Hub genes to obtain the final potential key genes and Chinese medicine ingredients, and carry out molecular docking verification of Chinese medicine ingredients and key genes in AutoDock and PyMol software. Results: Three databases GSE14805, GSE29796 and GSE35493 were downloaded, and after GEO2R analysis and intersection, 193 common differential expression genes were obtained, including 148 up-regulated genes and 45 down-regulated genes. With the help of Cytoscape software, the protein interaction network was obtained and 15 Hub genes were screened. GO and KEGG analysis indicated that Hub genes were mainly involved in cell cycle, biological process of extracellular matrix and cell adhesion, adhesion spot and cytoplasm in cell components. Through network pharmacological analysis, the potential therapeutic target BIRC5 and the active ingredient quercetin of traditional Chinese medicine targeting the treatment of glioblastoma were screened after the establishment of active ingredient target gene interaction network. The results of molecular docking showed that the binding of BIRC5 protein receptor to the ligand of Chinese traditional medicine was stable and hydrogen bond interaction was formed. Conclusion: Through the analysis of bioinformatics and network pharmacology methods, it is suggested that quercetin has the potential to affect the proliferation, invasion and migration of glioblastoma through the target BIRC5, thereby affecting the prognosis of glioblastoma patients, providing a new idea for the treatment of glioma and the development of new chemotherapeutic drugs.
Keywords:Glioblastoma, Bioinformatics, Network Pharmacology, BIRC5, Traditional Chinese Medicine Target, Quercetin

Copyright © 2023 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
成人胶质母细胞瘤(Glioblastoma, GBM)是所有脑恶性实体瘤中最致命和最顽固的肿瘤之一,每年确诊人数超10,000例,其五年生存期仅为5% [1]。因胶质瘤的发生机制复杂,具体通路还未探明,所以目前的多模式治疗手段主要包括可行的最大手术切除,辅以放疗和/或化疗的同步治疗,必要时可以结合中频交流电场治疗,但病人的中位生存期依旧很短,只有大约15个月 [2]。
由于胶质母细胞瘤的高度侵袭浸润性,术后的高复发率也是胶质母细胞瘤治疗的一大难题。随着基因技术的发展,越来越多的靶向治疗手段投入临床恶性肿瘤的治疗并取得良好的疗效,这也使得这种高度精准、疗效显著的治疗方式在胶质瘤中的应用研究成为潜在的热点。当前,探索胶质瘤治疗靶点往往首先通过数据库进行生物信息学大数据分析,筛选差异表达基因,预测潜在的靶基因,在进一步通过体内外细胞和动物实验探究靶基因的功能,以及基因上下游靶点研究致病分子机制,对分子通路的干预尝试治疗效果,达到治疗胶质瘤的目的 [3]。
如何获取胶质母细胞瘤相关的基因成为了问题的关键,随着生物信息的应用,获取靶向基因的手段也由传统的费时费力筛选转向由生物信息集合筛选的新方法,可以快速地获得胶质母细胞瘤的差异基因。然而,由于胶质母细胞瘤的肿瘤微环境内存在的差异性和异质性 [4],单独的靶向基因的治疗手段往往无法取得较好的治疗效果,所以寻找相对应靶向治疗药物十分关键。研究显示中医药作为一种天然的药物,其众多的产物具有抗癌活性,包括抗增殖、促凋亡、抗转移、抗血管生成作用,以及调节自噬、逆转多药耐药、平衡免疫和增强体内外化疗的效果 [5]。
本研究旨在通过数据库获取可能与胶质瘤治疗及预后相关的差异基因,提高胶质瘤的诊断率和提供新的治疗靶点,并通过中药数据库获取与胶质瘤相关中药单体成分,最终获取与胶质瘤治疗相关的靶点和中药成分。
2. 资料与方法
2.1. 资料来源
胶质瘤数据来源于NCBI的GEO数据库中,从25426个人脑胶质瘤芯片数据库中审查筛选获取载胶质母细胞瘤的基因表达谱的数据集GSE14805、GSE29796、GSE35493。车前子、丹参、金银花、连翘等中药的成分及相关靶基因均从中药系统药理学数据库和分析平台(TCMSP:https://old.tcmsp-e.com/tcmsp.php)中获得,其中以药物成分同时满足OB (口服生物利用度值) ≥ 40%和DL (药物相似性值) ≥ 0.18被视为有意义的数据。所有数据均可在线免费获得,本研究不涉及任何作者对人类或动物进行的任何实验。
2.2. 数据的筛选及差异基因的分析
利用在线GEO分析工具GEO2R (https://www.ncbi.nlm.nih.gov/geo/geo2r/)分析胶质母细胞瘤组织与癌旁脑组织差异表达基因,并获取其调整后的P值(adj.P.Val)和|logFC|的值。并以调整后的P < 0.01并且|logFC| ≥ 2作为判断基因是否有意义的条件,满足条件的基因即为差异基因(DEGs)。对GSE14805、GSE29796、GSE35493g这三个基因数据集分别进行分析筛选获取差异基因,再利用网络工具Venn diagram webtool (Draw Venn Diagram, http://bioinformatics.psb.ugent.be/webtools/Venn/)对这三个基因集的数据取交集并进行可视化处理获得Venn图。
2.2.1. DEGs的GO和KEGG分析
Gene Ontology (GO)是关于基因功能的最全面、应用最广泛的知识库 [6],主要包括三种本体论描述基因:生物过程(biological process)、细胞组分(cellular component)和分子功能(molecular function)。其丰富而巨大的数据贮存使得GO分析在生物信息的功能富集研究中广泛使用。KEGG (京都基因与基因组百科全书)是基因组集成的系统分析基因功能并链接基因组信息和功能信息的数据库资源,包括新陈代谢、其他细胞过程、有机体功能和人类疾病等 [7]。本文利用DAVID生物信息学资源 (https://david.ncifcrf.gov/tools.jsp)在线分析对差异基因进行GO和KEGG分析,以P < 0.01和基因计数 ≥ 10被认为具有统计学意义。最后利用生物信息学平台(https://hiplot.com.cn/)对数据进行可视化处理,得到GO分析和KEGG富集分析的气泡图。
2.2.2. PPI网络构建及Hub基因确定
蛋白质相互作用网络(protein-protein interaction network, PPI)在神经元、神经胶质细胞和其他细胞类型中存在,在神经科学中广泛利用并可能解决目前分子神经科学中未解决的问题 [8]。本文利用交互基因检索搜索工具(STRING)数据库(http://string-db.org)得到PPI网络的构造数据。将EDGs映射到STRING数据库中,随后将PPI网络通过Cytoscape (3.9.0版)这一开源的可视化软件(https://www.cytoscape.org/)进行可视化处理。再采用Cytoscape的CytoNCA软件包来进行DC (度)分析,满足degree value > 35,得到Hub基因。
2.2.3. Hub基因的相关分析
GEPIA2是基于来自TCGA和GTEx数据库的肿瘤和正常样本进行基因表达分析的有价值且被高度引用的资源具有198,619种亚型和84种癌症亚型,将基因表达定量从基因水平扩展到转录本水平,并支持特定癌症亚型的分析以及亚型之间的比较 [9]。通过GEPIA2 (http://gepia2.cancer-pku.cn/#analysis)进行Hub基因的生存分析,P < 0.05的生存曲线,被认为具有统计学意义。并同时绘制Hub基因在胶质母细胞瘤和正常组织的差异表达。
2.2.4. 蛋白质–蛋白质相互作用(PPI)网络和核心靶点分析
用perl软件(版本5.2.8.1;https://www.perl.org/)将从TCMSP上获取的车前子、丹参、金银花、连翘这四种中草药的数据进行分析,获取他们的有效成分及对应的基因靶点。再利用获得中药成分数据使用Cytoscape得到活性成分和靶点的PPI网络。将获得的靶点基因与之前获得的Hub基因取交集获得相对应的基因靶点及中药活性成分。
2.2.5. 分子对接
分子对接是一种基于计算机结构方法建立的方法,广泛用于药物发现,能够识别具有治疗意义的新型化合物,在分子水平上预测配体–靶标相互作用,并且够以相当高的准确度预测适当靶结合位点内小分子配体的构象 [10] [11]。最后将获得基因靶点和中药的活性成分进行分子对接判断该药物对应靶点作用的可能性。
3. 结果
3.1. 差异表达基因筛选
本研究利用GEO数据库中的三个基因表达谱GSE14805、GSE29196、GSE35493,以P < 0.05和|logFC| ≥ 2作为有效数据的筛选标准,经过分析GSE14805含出1284个DEG,其中上调基因446个,下调基因838个;GSE29196含出2854个DEG,其中上调基因2639个,下调基因215个;GSE35493含出3818个DEG,其中上调基因1606个,下调基因2212个。利用韦恩交集得到三个基因表达谱共同上调基因148,下调基因45 (图1(A)-(C))。
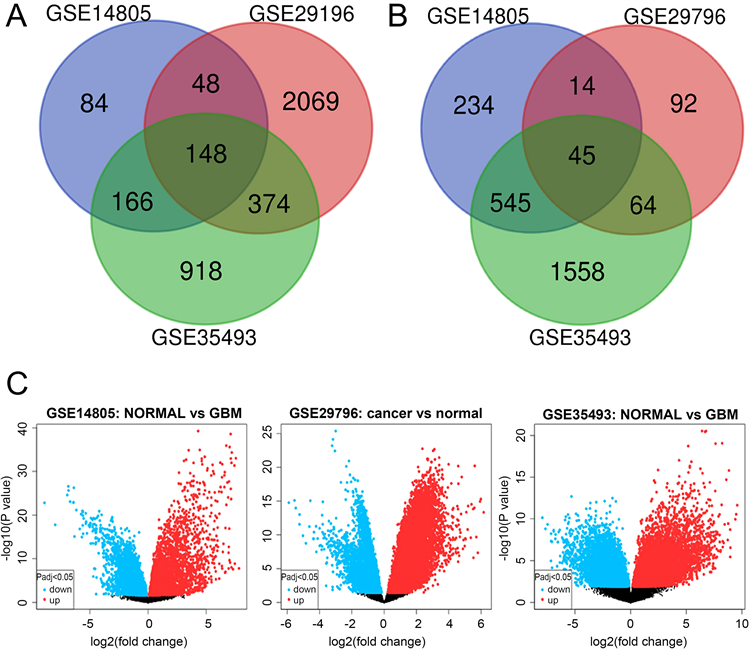
Figure 1. Screening of differentially expressed genes: Venn diagram of differentially expressed genes up-regulated by (A); Venn map of (B) down regulated differential genes; (C) Volcanic map of differential genes of GSE14805, GSE29196 and GSE35493
图1. 差异表达基因筛选:(A) 上调差异基因Venn图;(B) 下调差异基因Venn图;(C) GSE14805、GSE29196、GSE35493分别的差异基因的火山图
3.2. GO和KEGG分析
采用DAVID在线分析方法进行DEGs的GO和KEGG分析,在HIPLOT网站使用GO-KEGG-Bubble插件进行可视化处理(图2)。GO分析结果表明,DEGs主要富集在CC中,主要包括细胞外基质、细胞外间隙、细胞粘附连接、粘着斑及胞浆、核质、细胞质有关。MF分析显示DEGs主要富集在细胞黏附和细胞外基质有关,而其生物过程也主要与细胞的分裂黏附和细胞基质组织相关(图2(A),表1)。此外KEGG分析也表明EDGs主要富集在细胞周期和黏附的相关通路上(图2(B),表2)。

Figure 2. GO and KEGG: (A) GO analysis bubble chart; (B) KEGG analysis bubble chart
图2. GO和KEGG:(A) GO分析气泡图;(B) KEGG分析气泡图
Table 1. GO analysis
表1. GO分析
Table 2. KEGG analysis
表2. KEGG分析
3.3. PPI网络构建及Hub基因确定和分析
将DEGs的数据结果映射到STRING数据库中,得到PPI网络的构造及数据,并在此基础上以minimum required interaction score (截止标准)设置为大于0.4得到节点数192,边数1054,平均节点度11的蛋白质互作网络。此外将PPI网络得到数据通过Cytoscape (3.9.0版)进行可视化处理,得到蛋白质相互作用网络,并以红色标记上调基因,绿色标记下调基因(图3)。因为具有较高连接度的节点对于维护整个网络的稳定性往往更为重要,本研究以每个蛋白质节点的连接程度为筛选关键基因的条件。CytoHubba是Cytoscape中的一个插件,用于计算每个蛋白质节点的程度,在我们的研究中,以连接度大于35为条件,获取十四个基因CDK1,CCNB1,TOP2A,AURKA,CCNB2,KIF11,BUB1B,VEGFA,MCM7,MCM4,RRM2,CHEK1,CDKN3,BIRC5 (图4(A)),以及相互作用相关性(图4(B))。在GEPIA2生物信息分析平台分析获取关键基因转录水平表达差异及预后生存曲线(总生存期和无病生存期分析)。发现14个关键基因转录水平表达差异显著,差异具有统计学意义(图5),生存分析显示Logrank P值均小于0.001,表明这些生存曲线均具有意义(图6)。
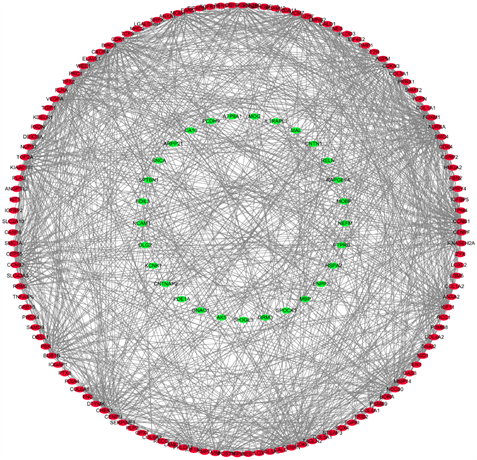
Figure 3. PPI network of DEGs (Red is over-expressed gene, green is low-expressed gene)
图3. 差异基因PPI网络图(红色为过表达基因,绿色为低表达基因)
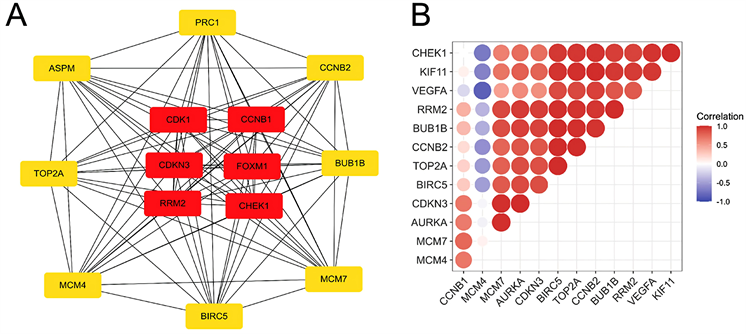
Figure 4. Hub genes: (A) PPI network of Hub genes; (B) Correlation heatmap plot of DEGs
图4. Hub基因:(A) Hub基因PPI网络图;(B) DEGs相关性热图

Figure 5. Expression of Hub genes in normal and glioma tissues (Box plot)
图5. Hub基因在正常组织和胶质瘤组织中的表达(箱式图)
3.4. 中药成分相互作用的蛋白质–蛋白质相互作用(PPI)网络构建
通过perl软件获得车前子、丹参、金银花、连翘四种中药数据导入药Cytoscape得到活性成分和靶点的PPI网络(图7),黄色代表中药成分的潜在靶基因。图中结果显示MOL000098 (槲皮素)的关联度最高,它作用的潜在靶点最多。同时将潜在的中药靶点与前面获取的关键基因取交集获取到6个共有基因CDK1、CCNB1、TOP2A、VEGFA、CHEK1、BIRC5。
3.5. 分子拼接
Hub基因与中药靶点交集基因中5个基因与槲皮素有关联,其中与中药成分的关系最为密切的是BIRC5,因此推测BIRC5是槲皮素的潜在靶点。为了验证中药成分槲皮素和对应的基因靶点是否有相互作用的可能性,本研究利用AutoDockTools-1.5.6和PyMol软件,将靶点蛋白BIRC5和中药活性成分槲皮素进行分子拼接(图8)。结果提示槲皮素与BIRC5的氨基酸残基GLU-100 (长度2.0 Å、2.4 Å),GLY-99 (3.4 Å),ARG-66 (2.2 Å)具有形成氢键,形成较稳定化合物的潜能。分子拼接的结果表明,槲皮素可以对BIRC5直接或间接的起到作用,而BIRC5与胶质瘤的增殖和侵袭迁移的相关,所以分子拼接可以作为槲皮素对胶质瘤起到抑制作用的关键证据,即槲皮素潜在靶向BIRC5发挥抑制胶质瘤增殖和侵袭迁移的作用。
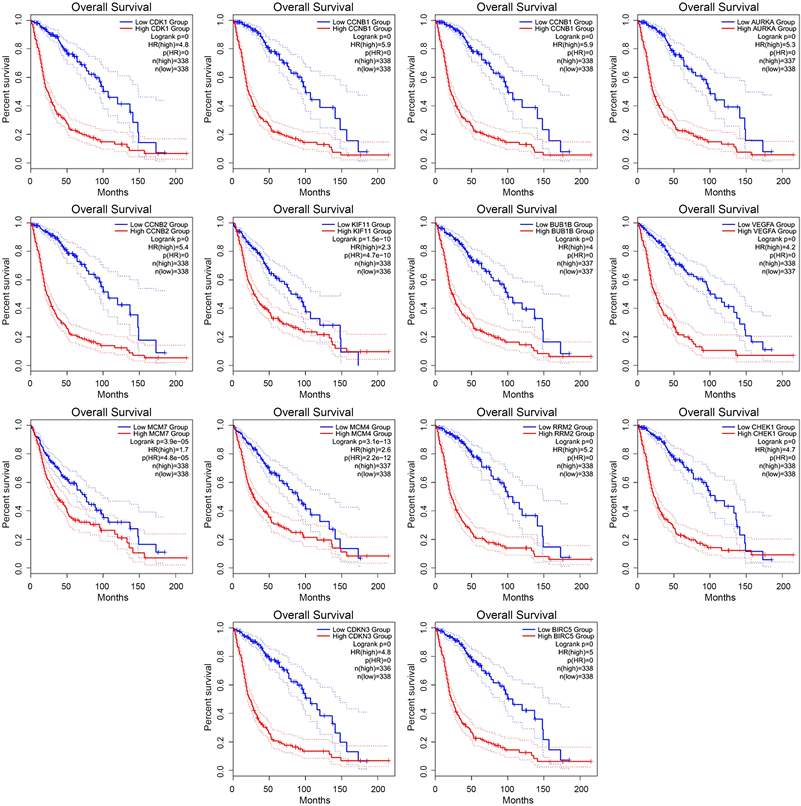
Figure 6. Survival analysis of Hub genes—Overall survival
图6. Hub基因的生存分析——总生存率
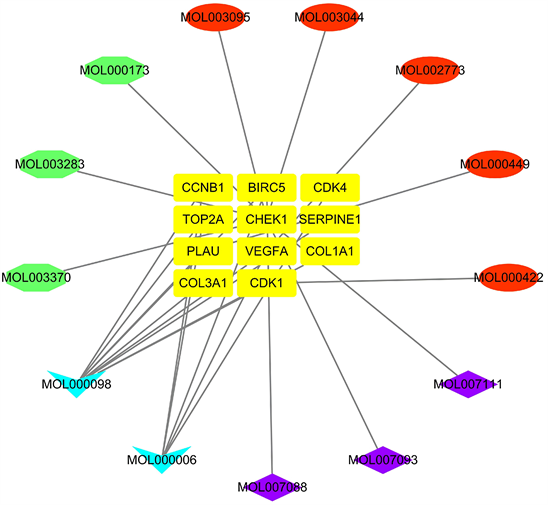
Figure 7. Interaction network between active ingredients of traditional Chinese medicine and differentially expressed genes of glioblastoma
图7. 中药活性成分与胶质母细胞瘤差异表达基因的互作网络
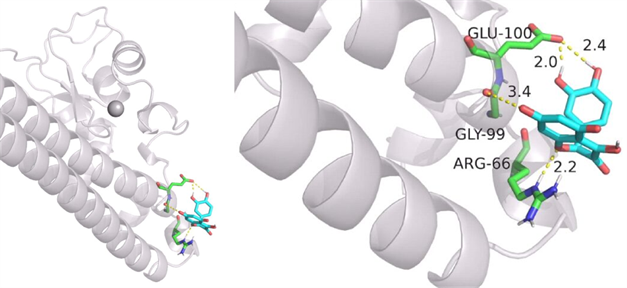
Figure 8. Molecular docking: BIRC5-quercetin
图8. 分子对接:BIRC5-槲皮素
4. 讨论
胶质母细胞瘤是最常见的原发性成人中枢神经系统恶性肿瘤,年发病率为每100,000人5.26例或每年17,000例新诊断 [12],因其高发病率和不良的生存预后成为困扰人类的疾病。其传统的治疗手段主要以GBM的切除技术和放化疗放疗联合替莫唑胺为主 [13],虽然目前有研究显示胶质母细胞瘤免疫疗法和免疫调节对与胶质母细胞瘤的治疗有着较好的效果 [14] [15],但是因为免疫细胞亚型的多样和构建治疗环境的复杂性以及较高的成本导致这种治疗方法进展缓慢。同时基因靶点药物在其他恶性肿瘤逐渐被利用,靶向药极高的特异性和灵敏性使其在治疗中取得了较好的疗效,如靶向药来那度胺在多发想骨髓瘤中应用极大的提高了患者的无病生存期减少了化疗的副作用 [16] [17]。而获取胶质瘤相关的灵敏度和特异度比较高的分子标记物是研究新型的靶向化疗药必不可少的过程,同时特异性的分子标记物还可以辅助诊断胶质瘤及分型,此外有研究显示传统中药通过基因途径对恶性肿瘤如胶质瘤、肺癌、乳腺癌的治疗具有作用 [18] [19] [20] [21]。基于生物分子途径和相互作用网络,揭示疾病的机制并将它们与药物的化学空间和网络足迹联系起来,利用这种整体方法在开发的早期阶段确定最有希望的目标和化合物 [22]。本研究致力于通过生物分子突进和互作网络获取传统中药中的有效成分及其作用的胶质瘤靶点为更有效的药物研发提供思路。
在本次研究我们基于公开的基因和中药数据库筛选出符合要求的数据,并对其进行了分子通路和蛋白质网络等方面的深层分析。通过胶质瘤相关基因的分析获取得到了14个与胶质瘤预后及组织表达量相关的关键基因,GO和KEGG分析显示这些基因主要富集在与胶质瘤细胞粘附和细胞周期相关的信号通路上。同时对获取的车前子、丹参、金银花、连翘四种的中药数据进行蛋白质网络分析获取到了与胶质瘤关联度最高的中药有效成分MOL000098 (槲皮素),并通过中药靶点对应的相关基因和14个关键进行比较获得了与中药槲皮素相关性最高的基因BIRC5。
槲皮素作为黄酮醇类黄酮亚类的主要代表,它具有多重生物活性,如抗氧化、抗病毒、抗炎作用,作为膳食补充剂和抗糖尿病的潜力,具有重要药用和科研价值 [23] [24] [25]。在抗癌作用方面,有文章报道槲皮素通过调节PI3K/Akt/mTOR促进细胞活力丧失、细胞凋亡和自噬的能力,Wnt/β-catenin和MAPK/ERK1/2通路特别是靶向参与葡萄糖代谢和线粒体功能 [26],并且槲皮素还可诱导EB介导的溶酶体活化,增加铁蛋白降解,导致铁死亡和Bid参与的细胞凋亡,诱导p53非依赖性癌细胞死亡 [27]。还有研究发现槲皮素可以与特定的纳米药物载体粒子缀和形成槲皮素共轭纳米粒子 [28],发挥载体靶向递送系统用于肝癌的治疗 [29]。
杆状病毒抑制剂5 (BIRC5)也称生存素(survivin)是1997年发现的人类细胞凋亡抑制剂(IAP)蛋白的最小家族成员 [30],BIRC5在癌症中的过表达可能会抑制凋亡检查点,并有利于转化细胞的异常有丝分裂,可抑制细胞凋亡并促进细胞增殖 [31]。它在胚胎组织(例如神经元前体细胞和胚胎成纤维细胞)和肿瘤(包括癌细胞和癌症干细胞)中高度表达,其表达与肿瘤细胞分化,增殖,侵袭和迁移有关,并且作为“桥接分子”,其调节胚胎和癌细胞中有丝分裂,细胞凋亡和自噬之间的相互作用 [32],还与多种肿瘤中免疫细胞浸润显著相关 [33]。此外有研究报道BIRC5是线粒体代谢的关键参与者,BIRC5在神经母细胞瘤中的线粒体高表达,起到募集Drp1使线粒体片段化减少了线粒体呼吸和ROS的产生的作用,介导了晚期神经母细胞瘤的化学抗性并起到重要的抗凋亡功能 [34]。Survivin位于许多癌细胞信号网络的十字路口,许多上游细胞信号分子控制和调节生存素及其功能 [35],p53具有调节BIRC5表达的作用,p53在正常组织中转录抑制BIRC5表达,影响p53的突变会导致BIRC5上调 [36] [37]。而槲皮素具有诱导p53非依赖性癌细胞死亡和靶向参与葡萄糖代谢和线粒体功能,而BIRC5是槲皮素在胶质瘤中的靶向基因,故推测槲皮素可以通过上述的信号通路影响BIRC5进而通过BIRC5对胶质瘤细胞线粒体功能和能量代谢产生作用进而影响胶质瘤的增殖和迁移。
5. 结论
本研究基于生物信息学方法分析提示CDK1,CCNB1,TOP2A,AURKA,CCNB2,KIF11,BUB1B,VEGFA,MCM7,MCM4,RRM2,CHEK1,CDKN3,BIRC5在胶质瘤中异常高表达,通过网络药理学方法确定胶质瘤差异基因BIRC5潜在中药靶点成分槲皮素。因此,槲皮素潜在通过靶点基因BIRC5作用于胶质母细胞瘤,抑制胶质瘤的增殖、侵袭和转移,为胶质瘤治疗和新化疗药的研发提供了新思路。
基金项目
该研究受到安徽医科大学临床医学国家一流本科专业建设项目(胡阳春,2021年),安徽医科大学科学研究基金项目(2021xkj131, 2021xkj135);安徽省高校自然科学研究项目(KJ2021A0293)支持。
文章引用
胡阳春,陈 宇,舒 磊,夏志宇,代兴亮. 生物信息学和网络药理学分析槲皮素靶向BIRC5抑制胶质母细胞瘤的增殖并影响预后
Quercetin Targeting BIRC5 Inhibits the Proliferation of Glioblastoma and Affects the Prognosis Based on Bioinformatics and Network Pharmacology Analysis[J]. 临床医学进展, 2023, 13(01): 928-941. https://doi.org/10.12677/ACM.2023.131134
参考文献
- 1. Alexander, B.M. and Cloughesy, T.F. (2017) Adult Glioblastoma. Journal of Clinical Oncology, 35, 2402-2409. https://doi.org/10.1200/JCO.2017.73.0119
- 2. Alifieris, C. and Trafalis, D.T. (2015) Glioblastoma Multiforme: Pathogenesis and Treatment. Pharmacology & Therapeutics, 152, 63-82. https://doi.org/10.1016/j.pharmthera.2015.05.005
- 3. Touat, M., Idbaih, A., Sanson, M. and Ligon, K.L. (2017) Glioblastoma Targeted Therapy: Updated Approaches from Recent Biological Insights. Annals of Oncology, 28, 1457-1472. https://doi.org/10.1093/annonc/mdx106
- 4. Le Rhun, E., Preusser, M., Roth, P., et al. (2019) Molec-ular Targeted Therapy of Glioblastoma. Cancer Treatment Reviews, 80, Article ID: 101896. https://doi.org/10.1016/j.ctrv.2019.101896
- 5. Luo, H., Vong, CT., Chen, H., et al. (2019) Naturally Occurring Anti-Cancer Compounds: Shining from Chinese Herbal Medicine. Chinese Medicine, 14, 48. https://doi.org/10.1186/s13020-019-0270-9
- 6. The Gene Ontology Consortium (2019) The Gene Ontology Re-source: 20 Years and Still Going Strong. Nucleic Acids Research, 47, D330-D338. https://doi.org/10.1093/nar/gky1055
- 7. Kanehisa, M., Furumichi, M., Tanabe, M., Sato, Y. and Morishima, K. (2017) KEGG: New Perspectives on Genomes, Pathways, Diseases and Drugs. Nucleic Acids Research, 45, D353-D361. https://doi.org/10.1093/nar/gkw1092
- 8. Basu, A., Ash, P.E., Wolozin, B. and Emili, A. (2021) Protein Interac-tion Network Biology in Neuroscience. Proteomics, 21, e1900311. https://doi.org/10.1002/pmic.201900311
- 9. Tang, Z., Kang, B., Li, C., Chen, T. and Zhang, Z. (2019) GEPIA2: An Enhanced Web Server for Large-Scale Expression Profiling and Interactive Analysis. Nucleic Acids Research, 47, W556-W560. https://doi.org/10.1093/nar/gkz430
- 10. Pinzi, L. and Rastelli, G. (2019) Molecular Docking: Shift-ing Paradigms in Drug Discovery. International Journal of Molecular Sciences, 20, 4331. https://doi.org/10.3390/ijms20184331
- 11. Ferreira, L.G., Dos Santos, R.N., Oliva, G. and Andricopulo, A.D. (2015) Molecular Docking and Structure-Based Drug Design Strategies. Molecules, 20, 13384-13421. https://doi.org/10.3390/molecules200713384
- 12. Omuro, A. and DeAngelis, L.M. (2013) Glioblastoma and Other Malignant Gliomas: A Clinical Review. JAMA, 310, 1842-1850. https://doi.org/10.1001/jama.2013.280319
- 13. Carlsson, S.K., Brothers, S.P. and Wahlestedt, C. (2014) Emerging Treatment Strategies for Glioblastoma Multiforme. EMBO Molecular Medicine, 6, 1359-1370. https://doi.org/10.15252/emmm.201302627
- 14. Li, Y., Sharma, A., Maciaczyk, J. and Schmidt-Wolf, I.G.H. (2022) Recent Development in NKT-Based Immunotherapy of Glioblastoma: From Bench to Bedside. International Journal of Molecular Sciences, 23, 1311. https://doi.org/10.3390/ijms23031311
- 15. Peng, J., Liang, Q., Xu, Z., et al. (2022) Current Understanding of Ex-osomal MicroRNAs in Glioma Immune Regulation and Therapeutic Responses. Frontiers in Immunology, 12, Article ID: 813747. https://doi.org/10.3389/fimmu.2021.813747
- 16. Facon, T., Kumar, S., Plesner, T., et al. (2019) Daratumumab plus Lenalidomide and Dexamethasone for Untreated Myeloma. The New England Journal of Medicine, 380, 2104-2115. https://doi.org/10.1056/NEJMoa1817249
- 17. Siegel, D.S., Schiller, G.J., Samaras, C., et al. (2020) Pomalidomide, Dexamethasone, and Daratumumab in Relapsed Refractory Multiple Myeloma after Lenalidomide Treatment. Leukemia, 34, 3286-3297. https://doi.org/10.1038/s41375-020-0813-1
- 18. Wang, J., Qi, F., Wang, Z., et al. (2020) A Review of Traditional Chinese Medicine for Treatment of Glioblastoma. BioScience Trends, 13, 476-487. https://doi.org/10.5582/bst.2019.01323
- 19. Xiang, Y., Guo, Z., Zhu, P., Chen, J. and Huang, Y. (2019) Tradition-al Chinese Medicine as a Cancer Treatment: Modern Perspectives of Ancient but Advanced Science. Cancer Medicine, 8, 1958-1975. https://doi.org/10.1002/cam4.2108
- 20. Su, X.L., Wang, J.W., Che, H., et al. (2020) Clinical Application and Mechanism of Traditional Chinese Medicine in Treatment of Lung Cancer. Chinese Medical Journal (England), 133, 2987-2997. https://doi.org/10.1097/CM9.0000000000001141
- 21. Yang, Z., Zhang, Q., Yu, L., et al. (2021) The Signaling Pathways and Targets of Traditional Chinese Medicine and Natural Medicine in Triple-Negative Breast Cancer. Journal of Ethnopharmacology, 264, Article ID: 113249. https://doi.org/10.1016/j.jep.2020.113249
- 22. Fotis, C., Antoranz, A., Hatziavramidis, D., Sakellaropoulos, T. and Alexopoulos, L.G. (2018) Network-Based Technologies for Early Drug Discovery. Drug Discovery Today, 23, 626-635. https://doi.org/10.1016/j.drudis.2017.12.001
- 23. Tang, S.M., Deng, X.T., Zhou, J., Li, Q.P., Ge, X.X. and Miao, L. (2020) Pharmacological Basis and New Insights of Quercetin Action in Respect to Its Anti-Cancer Effects. Biomedi-cine & Pharmacotherapy, 121, Article ID: 109604. https://doi.org/10.1016/j.biopha.2019.109604
- 24. Andres, S., Pevny, S., Ziegenhagen, R., Bakhiya, N., et al. (2018) Safety Aspects of the Use of Quercetin as a Dietary Supplement. Molecular Nutrition & Food Research, 62, Arti-cle ID: 1700447. https://doi.org/10.1002/mnfr.201700447
- 25. Eid, H.M. and Haddad, P.S. (2017) The Antidia-betic Potential of Quercetin: Underlying Mechanisms. Current Medicinal Chemistry, 24, 355-364. https://doi.org/10.2174/0929867323666160909153707
- 26. Reyes-Farias, M. and Carrasco-Pozo, C. (2019) The Anti-Cancer Effect of Quercetin: Molecular Implications in Cancer Metabolism. International Journal of Molecular Sci-ences, 20, 3177. https://doi.org/10.3390/ijms20133177
- 27. Wang, Z.X., Ma, J., Li, X.Y., et al. (2021) Quercetin Induces p53-Independent Cancer Cell Death through Lysosome Activation by the Transcription Factor EB and Reactive Oxygen Species-Dependent Ferroptosis. British Journal of Pharmacology, 178, 1133-1148. https://doi.org/10.1111/bph.15350
- 28. Vinayak, M. and Maurya, A.K. (2019) Quercetin Loaded Nanoparticles in Targeting Cancer: Recent Development. Anti-Cancer Agents in Medicinal Chemistry, 19, 1560-1576. https://doi.org/10.2174/1871520619666190705150214
- 29. Guan, X., Gao, M., Xu, H., et al. (2016) Querce-tin-Loaded Poly(lactic-co-glycolic acid)-d-α-tocopheryl Polyethylene Glycol 1000 Succinate Nanoparticles for the Tar-geted Treatment of Liver Cancer. Drug Delivery, 23, 3307-3318. https://doi.org/10.1080/10717544.2016.1176087
- 30. Li, Y., Zhao, Z.G., Luo, Y., et al. (2020) Dual Targeting of Polo-Like Kinase 1 and Baculoviral Inhibitor of Apoptosis Repeat-Containing 5 in TP53-Mutated Hepatocellular Carci-noma. World Journal of Gastroenterology, 26, 4786-4801. https://doi.org/10.3748/wjg.v26.i32.4786
- 31. Li, F., Ambrosini, G., Chu, E.Y., et al. (1998) Control of Apoptosis and Mitotic Spindle Checkpoint by Survivin. Nature, 396, 580-584. https://doi.org/10.1038/25141
- 32. Lin, T.Y., Chan, H.H., Chen, S.H., et al. (2020) BIRC5/Survivin Is a Novel ATG12-ATG5 Conjugate Interactor and an Autopha-gy-Induced DNA Damage Suppressor in Human Cancer and Mouse Embryonic Fibroblast Cells. Autophagy, 16, 1296-1313. https://doi.org/10.1080/15548627.2019.1671643
- 33. Xu, L., Yu, W., Xiao, H. and Lin, K. (2021) BIRC5 Is a Prognostic Biomarker Associated with Tumor Immune Cell Infiltration. Scientific Reports, 11, Article No. 390. https://doi.org/10.1038/s41598-020-79736-7
- 34. Ausserlechner, M.J. and Hagenbuchner, J. (2015) Mito-chondrial Survivin—An Achilles’ Heel in Cancer Chemoresistance. Molecular & Cellular Oncology, 3, e1076589. https://doi.org/10.1080/23723556.2015.1076589
- 35. Li, F., Aljahdali, I. and Ling, X. (2019) Cancer Therapeutics Using Survivin BIRC5 as a Target: What Can We Do after over Two Decades of Study? Journal of Experimental & Clinical Cancer Research, 38, 368. https://doi.org/10.1186/s13046-019-1362-1
- 36. Renner, G., Janouskova, H., Noulet, F., et al. (2016) Integrin α5β1 and p53 Convergent Pathways in the Control of Anti-Apoptotic Proteins PEA-15 and Survivin in High-Grade Glioma. Cell Death & Differentiation, 23, 640-653. https://doi.org/10.1038/cdd.2015.131
- 37. Frazzi, R. (2021) BIRC3 and BIRC5: Multi-Faceted Inhibitors in Cancer. Cell & Bioscience, 11, 8. https://doi.org/10.1186/s13578-020-00521-0
NOTES
*通讯作者。
