Computer Science and Application
Vol.
13
No.
11
(
2023
), Article ID:
75899
,
9
pages
10.12677/CSA.2023.1311211
基于深度元学习的医学图像分类研究
董大山1,张仲荣1*,张其斌2*
1兰州交通大学数理学院,甘肃 兰州
2甘肃省科技厅高新技术创业服务中心,甘肃 兰州
收稿日期:2023年10月20日;录用日期:2023年11月17日;发布日期:2023年11月24日

摘要
本文针对医学图像分类问题,提出了一种基于深度元学习的多阶段小样本学习方法,该方法结合了元学习、迁移学习的思想。通过引入预训练的视觉变压器模型(vision transformer)作为特征提取模型,提升对医学图像特征的提取能力。使用额外的数据集对原型网络进行训练,以克服医疗图像数据量不足的问题。我们对于医学图像分类任务进行微调,以提高模型的针对性,使模型更易进行适应医学图像任务。我们在两个医学图像数据集(血液、病理学)上进行了实验,并与相关工作进行了比较。实验结果表明,我们的方法在血液数据集上3way 1-shot,5-shot,10-shot准确率分别为68.06%,91.55%,95.3%,在病理数据集上3way 1-shot,5-shot,10-shot准确率分别为78.50%,91.84%,94.93%,取得了领先的性能,具有可靠的识别率。
关键词
医学图像分类,元学习,小样本学习,迁移学习

Research on Medical Image Classification Based on Deep Meta-Learning
Dashan Dong1, Zhongrong Zhang1*, Qibin Zhang2*
1School of Mathematics and Physics, Lanzhou Jiaotong University, Lanzhou Gansu
2Gansu Provincial Department of Science and Technology High-Tech Entrepreneurship Service Center, Lanzhou Gansu
Received: Oct. 20th, 2023; accepted: Nov. 17th, 2023; published: Nov. 24th, 2023

ABSTRACT
This paper focuses on the problem of medical image classification and proposes a multi-stage few-shot learning method based on deep meta-learning, combining the ideas of meta-learning and transfer learning. By introducing a pre-trained vision transformer model as the feature extraction model, the capability of extracting features from medical images is enhanced. Additional datasets are used to train the prototype network to overcome the problem of insufficient medical image data. We fine-tune the model for medical image classification tasks to improve its specificity and make it more adaptable to medical image tasks. We conducted experiments on two medical image datasets (hematology and pathology) and compared the results with related works. The experimental results demonstrate that our method achieves leading performance with reliable recognition rates. For the hematology dataset, the accuracy rates of 3way 1-shot, 5-shot, and 10-shot are 68.06%, 91.55%, and 95.3% respectively, while for the pathology dataset, the accuracy rates of 3way 1-shot, 5-shot, and 10-shot are 78.50%, 91.84%, and 94.93% respectively.
Keywords:Medical Image Classification, Meta-Learning, Few-Shot Learning, Transfer Learning

Copyright © 2023 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
随着深度学习的迅猛发展,医学图像分析领域正逐渐探索并应用深度学习 [1] 技术,以应对医学图像分类所面临的挑战。这一技术的引入使得医院能够逐步推进智慧医疗,通过深度学习对医学图像进行分类 [2] ,从而提高就医效率并减轻医生的工作负担。准确可靠的医学图像分类问题正成为当前医学领域的研究热点。传统的医学图像分类方法往往依赖于手动设计的特征提取和分类器 [3] ,这些方法在复杂的医学图像中往往表现出有限的效果。而深度学习技术通过利用大规模标注数据和强大的计算能力,能够直接从原始图像数据中学习特征表示和分类模型。深度学习算法如卷积神经网络(CNN) [4] 和自编码器 [5] 等已经在医学图像分类任务中取得了显著的成果。然而,医学图像数据的获取和标注存在一些挑战,首先,由于医学图像的特殊性质,标注需要经验丰富的医学专家,因此标注的成本较高。其次,一些疾病的发病率很低,导致数据的收集变得更加困难。此外,由于医学图像具有隐私性,数据的分享和使用受到一定限制。这些问题导致医学图像数据的数量有限且分布不平衡。在设计深度学习模型时,需要考虑到医学图像数据的特点,并充分利用有限的数据进行医学图像分类任务。目前相关研究已经提出了一些有效的方法来解决这个问题,包括基于元学习(meta-learning) [6] 和基于迁移学习(transfer-learning) [7] 的方法,通过在大规模的医学图像数据集上进行元训练,模型可以学习到通用的特征表示和优化策略,从而在新的医学图像分类任务上快速适应和泛化。迁移学习可以利用在大规模数据集上预训练的模型作为初始模型,并在医学图像数据集上进行微调。尽管元学习和迁移学习在医学图像分类中具有潜力,但仍存在一些问题和挑战。首先,元学习需要大规模的医学图像数据集进行元训练,以学习共享的知识和经验。然而,由于医学图像数据的获取和标注成本较高,很多情况下可用的数据量有限。这可能限制了元学习方法的应用范围和性能。其次,医学图像数据的特殊性质和多样性使得迁移学习面临一些挑战。在不同医疗机构和设备之间,医学图像可能存在领域差异,导致预训练模型在新任务上的性能下降。此外,医学图像中的类别分布不平衡也可能影响迁移学习的效果,特别是在少数类别的分类上。另外一些研究工作探索了半监督学习(semi-supervised learning) [8] 和主动学习(active learning) [9] 等方法,以利用未标注数据和有限的标注数据来提升分类性能。这些方法可以通过利用未标注数据的信息来增强模型的泛化能力,并减少对标注数据的依赖。但是由于医学图像的特点,不平衡的数据分布会对半监督学习方法的性能产生影响,因为无标签样本的质量和数量会影响模型的训练和泛化能力,在主动学习中如何选择最具信息价值的样本进行标注是一个挑战,所以半监督学习和主动学习的效果并不理想。
我们提出了一个新的基于深度元学习的医疗图像深度学习模型。图1是我们模型的流程图。具体来说,我们使用视觉变压器模型(vision transformer) [10] [11] 作为一个特征提取器来提取图像的深度特征,视觉变压器模型具有强大的特征提取能力,然后使用Mini-Imagenet数据集来对原型网络 [12] 进行训练,Mini-Imagenet数据集中大量的数据训练使得原型网络可以克服样本分布不均衡的问题,接下来使用logistic分类器 [13] 来对医疗图像进行微调,以快速适应新的未知任务。这项工作的贡献可归纳如下:
1) 我们提出了一个有效的医学图像分类任务的深度元学习框架,基于预训练视觉变压器模型(vision transformer)和元学习两个阶段,可以解决医学图像学习样本稀缺造成的学习困境。
2) 我们引入了额外的Mini-Imagenet数据集来学习丰富的类别特征,以提高模型的鲁棒性和泛化性。
3) 基于两个公开可用的医学图像数据集,验证了两种医学图像(即血液、病理学)分类的准确性,验证了我们方法的有效性。
2. 算法模型
2.1. 网络结构
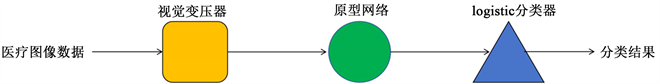
Figure 1. Medical image classification algorithm framework based on deep meta learning
图1. 基于深度元学习的医学图像分类算法框架
基于深度元学习的医学图像分类算法框架如图1所示,主要由特征提取器,原型网络,分类器,三部分组成,其中特征提取器使用预训练的视觉变压器(vision transforme)模型,原型网络(Prototypical Network)用于图片相似度度量以达到对医学图像的区分,logistic分类器则对每个图片进行分类,从而完成医学图像分类的任务。
在医学图像分类中,由于图像数据量较少,模型参数量过大,容易引起模型过拟合和训练成本过高,为了使模型能够更加适应当前分类任务。对分类模型进行优化,优化主要包括四个部分:1) 在元学习中使用数据增强,数据增强的方式包括颜色增强和平移增强,例如随机裁剪、颜色抖动、随机翻转,图像归一化等。2) 在特征提取网络视觉变压器(vision transforme)中使用预训练参数,并在之后的训练中冻结参数。3) 在原型网络中使用Mini-Imagenet数据集对参数进行更新。4) 使用参数逐步冻结来减少训练成本。下面将对各个部分进行阐述。
2.2. 数据增强
我们在训练模型时对原始数据进行一系列变换和扩充的技术来进行数据增强。主要目的是增加训练数据的多样性,提高模型的泛化能力。我们采用的数据增强包括随机裁剪、颜色抖动、随机翻转,图像归一化 [14] 等。需要注意的是,我们没有将所有图片都进行数据增强,而是选择以90%的概率对数据进行增强,这样也提高了模型的鲁棒性,减少了过拟合的风险。
2.3. 预训练的视觉变压器(Vision Transformer)
医学图像通常具有复杂的结构和多样的异常特征,例如肿瘤、病变、血管等。这使得对图像进行准确的标注变得具有挑战性。医学图像标注需要对医学知识有一定的理解和背景知识。标注人员需要了解不同病理学特征、解剖结构等,以便准确地标注图像。另外数据收集的困难、隐私保护的要求或其他因素导致医学图像数据难以大规模开发,医学图像数据量小的问题会限制我们对模型的训练和性能评估,造成过拟合问题,为了解决这个问题,我们为用于特征提取的视觉变压器(vision transformer)中使用了预训练参数,预训练模型在大规模数据上进行了预训练,学习到了丰富的通用特征表示。这些特征可以应用于各种下游任务,当应用于特定任务时,只需要使用相对较少的任务相关数据进行微调。这种迁移学习的方式使得在数据量有限的情况下,获得较好的性能表现,提高了模型的特征提取能力。我们使用了meta公司的dino预训练参数。
2.4. 元学习原型网络
原型网络(Prototypical Network)是一种简单,高效的小样本的学习方式,旨在通过学习一个适当的距离或相似度度量来衡量样本之间的相似性或差异性。目标是通过学习一个适当的度量函数,使得在度量空间中相似的样本距离更近,不相似的样本距离更远。通过学习合适的度量函数,可以更好地反映样本之间的相似性和差异性,从而在各种机器学习任务中获得更好的性能。原型网络的基本思路是对于每一个分类来创建一个原型表示。并且对于一个需要分类的查询,采用计算分类的原型向量和查询点的距离来进行确定。

Figure 2. Prototype network
图2. 原型网络
如图2所示,在小样本分类中,我们给出了N个示例 的小支持集,其中每个 是一个示例的D维向量, 是对应的标签, 表示标有K类的示例集,原型网络通过具有可学习的参数 的嵌入函数 来计算每一类的M维表示 ,也叫做原型,每个原型都是属于其类的嵌入式支撑点的平均向量。原型的计算如公式(1)所示:
(1)
给定距离函数 ,原型网络基于到嵌入空间中原型的距离上的softmax为查询点x生成类上的分布,如公式(2)所示:
(2)
由于医学图像数据集数据量较小,且在罕见的疾病中正负样本数量差别较大,我们使用Mini-ImageNet数据集对原型网络参数进行更新,Mini-ImageNet数据集包含了来自多个类别的图像样本,可以提供丰富的类别和样本多样性。通过使用这样的数据集进行参数更新,可以帮助原型网络更好地理解和表示不同类别之间的特征差异,提高模型的泛化能力,可以更好地适应数据有限的任务,并减少过拟合的可能性。注意的是通过Mini-ImageNet数据集更新的原型网络参数我们在后续的微调中会被冻结。
2.5. 模型参数冻结
医学图像数据集样本量普遍较少,而大多数模型参数量较多,很容易引起过拟合是医学领域常见的挑战之一。另外,由于模型参数量较多,还会导致训练时间过长。我们针对此问题的策略是采用模型参数冻结的方法。模型参数冻结的方法可以在有限的数据上利用已经训练好的模型参数,以减少过拟合并提高模型的泛化能力。我们通过添加一个logistic分类器层来对特定的医学图像进行模型微调,这种结构具有较少的参数,因此在有限的数据上进行微调不会引入过多的过拟合风险。具体结构如下图3所示。
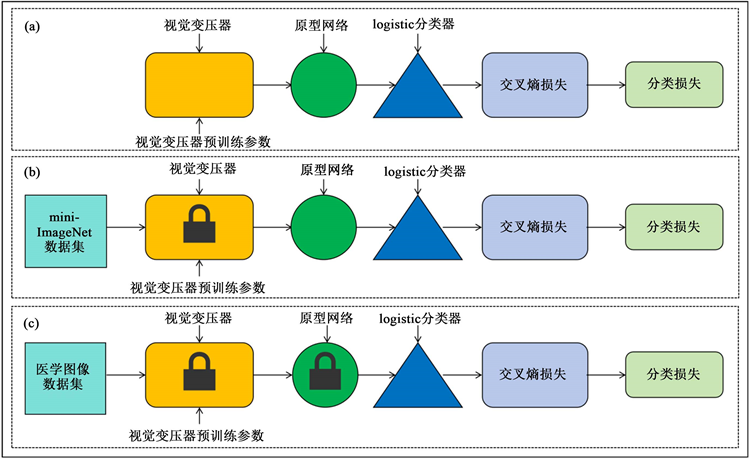
Figure 3. (a) shows the overall architecture of the model, where the parameters in the visual transformer are pre trained parameters using dino small [15] ; (b) freeze the parameters of the visual transformer in and use the Mini-Imagenet dataset to perform gradient updates on the parameters of the prototype network; (c) freeze the parameters of the visual transformer and prototype network in, and update the parameters of the logistic classifier using specific medical image datasets to achieve the goal of fine-tuning specific tasks
图3. (a) 是模型的整体架构,其中视觉变压器中的参数是使用dino small [15] 的预训练参数;(b) 中冻结视觉变压器的参数,使用Mini-ImageNet数据集,对原型网络的参数进行梯度更新;(c) 中冻结视觉变压器和原型网络的参数,使用特定的医学图像数据集对logistic分类器的参数进行更新,达到微调特定任务的目的。
如图3(a),我们首先在特征提取器视觉变压器模型(vision transformer)中使用视觉变压器预训练参数,在接下来的训练中,如图3(b)所示,我们冻结了特征提取器的参数,使其不参与后续的参数更新,然后我们使用Mini-ImageNet数据集,对原型网络的参数进行梯度更新,然后在接下来对医学图像数据集的分类中,如图3(c)所示,冻结了视觉变压器和原型网络的参数,使其反向传播的时候只更新logistic分类器的参数,达到对医学图像分类任务的微调。通过采用这种模型参数冻结的方法,我们可以在医学图像数据集样本量有限的情况下,利用预训练模型的特征表示能力,减少模型参数数量,并在分类器结构上的参数进行微调,以适应具体的医学图像分类任务。这种策略可以帮助提高模型性能,并降低过拟合的风险。
3. 实验结果与分析
3.1. 数据集介绍
本文使用的数据集是从两个公开可用的医学图像数据集中(即血液 [16] 、病理 [17] )每个数据集中的类别被分隔为元训练、元验证和元测试三个部分,血液是基于个体正常细胞的先前数据库构建的,这些细胞被组织成八类。在内部,未成熟粒细胞(IG)包含四个子类型(类型名称以“IG-”开头),它们被视为元验证类。血液中的其余七种类型,每种类型随机选择600张图像,用于元训练和元测试。病理学用来预测结肠直肠癌组织学载玻片的存活率,该载玻片是一个数据集(NCTCRC-HE-100K),具有来自染色组织学图像的非重叠图像斑块。该数据集由九种类型的组织组成,我们随机抽取每种类型的600张图像来构建医学图像分类场景。本文将元测试类中疾病类别的识别问题建模为医学图像分类问题,并分别在两个数据集上验证了我们的方法。在我们的实验中,基准图像大小为84 × 84。此外,在相关小样本学习工作中最广泛使用的非医学数据集Mini-ImageNet上也用于评估我们的方法。
3.2. 实验细节
我们在试验中对图像进行了数据增强,数据增强方式包括随机裁剪、颜色抖动、随机翻转、转换为张量和归一化。这些归一化方法在训练过程中以90%的概率随机应用于输入图像,以增加数据的多样性和模型的泛化能力。我们使用drop_path参数用于控制DropPath [18] 的概率,用于防止过拟合。它在训练过程中以一定的概率随机丢弃神经网络中的某些连接路径,从而减少网络的复杂性。对于两个医学数据集,我们考虑3类分类任务,从每个类中随机选择1,5或10幅图像作为训练样本,从其余的15幅图像作为测试样本。具体地说,我们为元学习构建K-shot (K = {1, 5, 10})任务的方式。我们使用两张GTX3090进行训练,我们在元训练阶段随机抽取最多5 × k张图片,在元验证和元测试的测试实验中随机抽取600张图片用于验证和元测试。我们使用元验证中准确率最高的模型用于元测试。对于视觉变压器(vision transformer),我们采用vit-small模型并采用meta公司的dinosmall作为预训练参数,在迁移学习阶段,由AdaBelief优化器 [19] 训练模型AdaBelief优化器的步数设置为40,总共执行100个轮次。学习速率初始化为5e−5,drop_path设置为0.1。在元学习阶段,对于N路K-shot任务,常规学习考虑通过AdamW优化器 [20] 优化,最后通过AdamW优化器优化最后的验证步骤。同时,AdamW的原始学习速率被设置为5e−5,没有周期性衰减。在迁移学习和元学习中,批量大小预设为1,训练一共进行100个轮次后停止。
3.3. 实验结果
Table 1. Accuracy of 3-way, 1-shot, 5-shot, and 10-shot classification on blood datasets (%)
表1. 血液数据集上的3way、1-shot、5-shot和10-shot分类准确度(%)
Table 2. Classification accuracy of 3-way, 1-shot, 5-shot, and 10-shot on pathological datasets (%)
表2. 病理数据集上的3way、1-shot、5-shot和10-shot分类准确度(%)
表1、表2给出了在血液、病理数据集上使用血液和病理数据集在其他算法模型的总体比较。报告了我们的方法的分类精度。这些评估结果是基于具有最高元验证准确性的模型的元测试结果。在这些表中,随着每个数据集上所有方法的shot数的增加,性能逐渐变好。在表1中,我们的方法分别实现了所有任务的最佳性能,远远优于比较方法,我们的方法有助于提高深度特征的提取,提高模型的性能。在表2中,我们给出了病理学结果。从这个表中,我们再次证实了我们的方法优于其他方法。另外,在每种少样本学习方法的基础上,从1-shot任务到5-shot任务的增加远远大于从5-shot任务到10-shot任务的增加。这表明,当shot数相对较小时,模型更需要数据。
Table 3. Classification accuracy of 5 way, 1-shot, 5-shot, and 10-shot on the Mini ImageNet dataset (%)
表3. 在Mini-ImageNet数据集上的5 way、1-shot、5-shot和10-shot分类准确度(%)
为了说明我们的方法在各种图像中的通用性,我们还在Mini-ImageNet数据集上测试了我们的方法。结果如表3所示,表明我们基于预训练视觉变压器(vision transformer)的方法优于比较方法。
4. 总结
针对医学图像分类任务,本文在现有的元学习原型网络算法的基础上,提出了一种结合预训练视觉变压器模型(vision transformer)和逐步冻结参数的医学图像分类算法。使用预训练模型提高对医学图像深度特征提取的能力,增强模型对医学图像不同类别之间差异的关注度;引入第三方Mini-ImageNet数据集更新原型网络的参数,使得模型具有多样性、适应小规模数据集、快速迭代和迁移学习等好处,这有助于提高原型网络的性能和泛化能力,并加速模型的开发和优化过程;最后利用模型各模块逐步冻结参数的方法,使得训练时间大大缩短,减少了模型过拟合,增加了模型的鲁棒性。实验证明,改进后的分类模型在血液数据集上1-shot,5-shot,10-shot准确率分别为68.06%,91.55%,95.3%,在病理数据集上1-shot,5-shot,10-shot准确率分别为78.50%,91.84%,94.93%,能够满足医学图像分类的任务需求。同时,我们的实验也为深度学习在智慧医疗中的应用提供了一定的借鉴作用。
文章引用
董大山,张仲荣,张其斌. 基于深度元学习的医学图像分类研究
Research on Medical Image Classification Based on Deep Meta-Learning[J]. 计算机科学与应用, 2023, 13(11): 2116-2124. https://doi.org/10.12677/CSA.2023.1311211
参考文献
- 1. LeCun, Y., Bengio, Y. and Hinton, G. (2015) Deep Learning. Nature, 521, 436-444. https://doi.org/10.1038/nature14539
- 2. He, K., Zhang, X., Ren, S., et al. (2016) Deep Residual Learning for Im-age Recognition. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Las Vegas, 27-30 June 2016, 770-778. https://doi.org/10.1109/CVPR.2016.90
- 3. Antonie, M.L., Zaiane, O.R. and Coman, A. (2001) Application of Data Mining Techniques for Medical Image Classification. Proceedings of the 2nd International Conference on Multime-dia Data Mining, San Francisco, 26 August 2001, 94-101.
- 4. Li, Q., Cai, W., Wang, X., et al. (2014) Medical Image Classification with Convolutional Neural Network. 2014 13th IEEE International Conference on Control Automation Robotics & Vision (ICARCV), Singapore, 10-12 December 2014, 844-848. https://doi.org/10.1109/ICARCV.2014.7064414
- 5. Gondara, L. (2016) Medical Image Denoising Using Convo-lutional Denoising Autoencoders. 2016 IEEE 16th International Conference on Data Mining Workshops (ICDMW), Barcelona, 12-15 December 2016, 241-246. https://doi.org/10.1109/ICDMW.2016.0041
- 6. Hu, S., Tomczak, J. and Welling, M. (2018) Meta-Learning for Medical Image Classification.
- 7. Yu, Y., Lin, H., Meng, J., et al. (2017) Deep Transfer Learning for Modality Classi-fication of Medical Images. Information, 8, Article No. 91. https://doi.org/10.3390/info8030091
- 8. Gyawali, P.K., Ghimire, S., Bajracharya, P., et al. (2020) Semi-Supervised Medical Image Classification with Global Latent Mixing. Medical Image Computing and Computer Assisted Intervention-MICCAI 2020: 23rd International Conference, Lima, 4-8 October 2020, 604-613. https://doi.org/10.1007/978-3-030-59710-8_59
- 9. Beluch, W.H., Genewein, T., Nürnberger, A., et al. (2018) The Power of Ensembles for Active Learning in Image Classification. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Salt Lake City, 18-22 June 2018, 9368-9377. https://doi.org/10.1109/CVPR.2018.00976
- 10. Dosovitskiy, A., Beyer, L., Kolesnikov, A., et al. (2020) An Image Is Worth 16x16 Words: Transformers for Image Recognition at Scale.
- 11. Vaswani, A., Shazeer, N., Parmar, N., et al. (2017) Attention Is All You Need. 31st Annual Conference on Neural Information Processing Systems (NIPS 2017), Long Beach, 4-9 December 2017, 5998-6008.
- 12. Snell, J., Swersky, K. and Zemel, R. (2017) Prototypical Networks for Few-Shot Learning. 31st Annual Conference on Neural Information Processing Systems (NIPS 2017), Long Beach, 4-9 December 2017, 4077-4087.
- 13. LaValley, M.P. (2008) Logistic Regression. Circulation, 117, 2395-2399. https://doi.org/10.1161/CIRCULATIONAHA.106.682658
- 14. Pei, S.C. and Lin, C.N. (1995) Image Normaliza-tion for Pattern Recognition. Image and Vision Computing, 13, 711-723. https://doi.org/10.1016/0262-8856(95)98753-G
- 15. Caron, M., Touvron, H., Misra, I., et al. (2021) Emerging Properties in Self-Supervised Vision Transformers. Proceedings of the IEEE/CVF International Conference on Comput-er Vision, 11-17 October 2021, 9650-9660. https://doi.org/10.1109/ICCV48922.2021.00951
- 16. Acevedo, A., et al. (2020) A Dataset of Microscopic Periph-eral Blood Cell Images for Development of Automatic Recognition Systems. Data in Brief, 30, Article ID: 105474. https://doi.org/10.1016/j.dib.2020.105474
- 17. Kather, J.N., Krisam, J., Charoentong, P., Luedde, T., Herpel, E., Weis, C.-A., Gaiser, T., Marx, A., Valous, N.A., Ferber, D., et al. (2019) Predicting Survival from Colorectal Cancer Histology Slides Using Deep Learning: A Retrospective Multicenter Study. PLOS Medicine, 16, e1002730. https://doi.org/10.1371/journal.pmed.1002730
- 18. Zheng, Q., Tian, X., Yang, M., et al. (2020) PAC-Bayesian Framework Based Drop-Path Method for 2D Discriminative Convolutional Network Pruning. Multidimensional Systems and Signal Processing, 31, 793-827. https://doi.org/10.1007/s11045-019-00686-z
- 19. Zhuang, J., Tang, T., Ding, Y., et al. (2020) Adabelief Optimizer: Adapting Stepsizes by the Belief in Observed Gradients. 34th International Conference on Neural Information Pro-cessing Systems, Vancouver, 6-12 December 2020, 18795-18806.
- 20. Kingma, D.P. and Ba, J. (2014) Adam: A Method for Stochastic Optimization.
- 21. Finn, C., Abbeel, P. and Levine, S. (2017) Model-Agnostic Meta-Learning for Fast Adaptation of Deep Networks. International Conference on Machine Learning, Sydney, 6-11 August 2017, 1126-1135.
- 22. Sung, F., Yang, Y., Zhang, L., Xiang, T., Torr, P.H. and Hospedales, T.M. (2018) Learning to Com-pare: Relation Network for Few-Shot Learning. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Salt Lake City, 18-22 June 2018, 1199-1208. https://doi.org/10.1109/CVPR.2018.00131
- 23. Gordon, J., Bronskill, J., Bauer, M., Nowozin, S. and Turner, R.E. (2018) VERSA: Versatile and Efficient Few-Shot Learning. 3rd Workshop on Bayesian Deep Learning, Montréal, 7 December 2018, 1-9.
- 24. Devos, A., Chatel, S. and Grossglauser, M. (2019) [Re]meta Learning with Differentiable Closed-Form Solvers. Tech. Rep.
- 25. Raghu, A., Raghu, M., Bengio, S. and Vinyals, O. (2019) Rapid Learning or Feature Reuse? Towards Understanding the Effective-ness of MAML.
- 26. Sun, Q., Liu, Y., Chen, Z., Chua, T.-S. and Schiele, B. (2020) Meta-Transfer Learning through Hard Tasks. IEEE Transactions on Pattern Analysis and Machine Intelligence, 44, 1443-1456.
- 27. Vinyals, O., Blun-dell, C., Lillicrap, T., Wierstra, D., et al. (2016) Matching Networks for One Shot Learning. NIPS’16: Proceedings of the 30th International Conference on Neural Information Processing Systems, Barcelona, 5-10 December 2016, 3630-3638.
- 28. Antoniou, A., Edwards, H. and Storkey, A. (2018) How to Train Your MAML.
- 29. Chen, Y., Wang, X., Liu, Z., Xu, H. and Darrell, T. (2020) A New Meta-Baseline for Few-Shot Learning.
- 30. Rusu, A.A., Rao, D., Sygnowski, J., Vinyals, O., Pascanu, R., Osindero, S. and Hadsell, R. (2018) Meta-Learning with Latent Embedding Optimization.
- 31. Yu, Z., Chen, L., Cheng, Z. and Luo, J. (2020) Transmatch: A Transfer-Learning Scheme for Semi-Supervised Few-Shot Learning. Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition, Seattle, 13-19 June 2020, 12856-12864. https://doi.org/10.1109/CVPR42600.2020.01287
NOTES
*通讯作者。
