Computer Science and Application
Vol.
11
No.
01
(
2021
), Article ID:
40094
,
13
pages
10.12677/CSA.2021.111016
肺结节图像的计算机自动检测研究进展
陈辰1,苗军1*,齐洪钢2,刘艳2,郭志军3,刘海涛3
1北京信息科技大学,网络文化与数字传播北京市重点实验室,北京
2中国科学院大学,计算机科学与技术学院,北京
3华北石油管理局总医院,河北 任丘

收稿日期:2020年12月26日;录用日期:2021年1月20日;发布日期:2021年1月28日

摘要
肺结节的早期筛查对肺癌的预防和治疗具有重要的意义。采用计算机断层扫描(CT)可以提高肺癌早期筛查中肺结节的检出率,是目前最为安全有效的肺癌筛查方式之一。随着CT数据日益增长,使用计算机辅助诊断(CAD)系统可以极大减少放射科医生的工作量以及降低漏检率。结合近年来肺结节检测的相关文献,对该领域的研究进展进行了综述。首先介绍了目前广泛应用的胸部CT数据集以及肺结节检测相关的评价指标。然后介绍并分析了肺结节检测框架中一些有效的方法,包括肺实质分割、候选结节检测以及假阳性剔除。最后,讨论了该领域存在的挑战以及未来发展的方向,为今后该领域的研究人员提供参考。
关键词
肺结节,检测,分割,假阳性剔除

Research Progress of Computer Automatic Detection of Pulmonary Nodules in Images
Chen Chen1, Jun Miao1*, Honggang Qi2, Yan Liu2, Zhijun Guo3, Haitao Liu3
1Beijing Key Laboratory of Internet Culture and Digital Dissemination Research, Beijing Information Science and Technology University, Beijing
2School of Computer Science and Technology, University of the Chinese Academy of Sciences, Beijing
3Huabei Petroleum General Hospital, Renqiu Hebei

Received: Dec. 26th, 2020; accepted: Jan. 20th, 2021; published: Jan. 28th, 2021

ABSTRACT
Early screening of pulmonary nodules is of great significance for the prevention and treatment of lung cancer. Computed tomography (CT) can improve the detection rate of pulmonary nodules in early lung cancer screening, which is one of the most effective lung cancer screening methods. With the increasing of CT data, the use of computer-aided diagnosis (CAD) system can greatly reduce the workload of radiologists and reduce the rate of missed detection. This paper reviews the research progress of pulmonary nodule detection in recent years. Firstly, this paper introduces the widely used chest CT open dataset and the evaluation metrics related to the detection of pulmonary nodule. Then it introduces and analyzes some effective methods in the framework of pulmonary nodule detection, including lung parenchyma segmentation, candidate nodule detection and false positive reduction after detection. Finally, the challenges and future development of this field are discussed, which can provide reference for future researchers in this field.
Keywords:Pulmonary Nodules, Detection, Segmentation, False Positive Reduction

Copyright © 2021 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
肺癌是目前发病率和死亡率最高的恶性肿瘤,2018年肺癌分别占全球癌症发病总数和死亡总数的11.6%和18.4% [1]。相较于其他恶性肿瘤患者,肺癌早期患者多无明显的不适症状,一经发现大多已处于肺癌中晚期。肺癌早期患者的临床表现一般为肺部存在恶性结节,尽早发现肺结节能够有效控制肺部癌变。肺结节的临床表现为单发或多发、圆形或不规则、边界清晰或不清晰的肺部病灶,直径一般在3毫米到30毫米。根据结节密度可以将其分为三类:实性结节(Solid Nodule)、部分实性结节(Part-Solid Nodule)和磨玻璃密度结节(Ground Glass Nodule)。不同密度的肺结节,其恶性概率不同,其中部分实性结节的恶性概率最高 [2]。
目前低剂量的计算机断层扫描(Computed Tomography, CT)被认为是最为有效且安全的肺结节筛查方式之一,与胸片检查相比,CT检查可以将肺癌死亡率降低20%以上 [3]。通常一次CT扫描会产生数百张切片图像,放射科医生需要观察每张切片上的肺部组织来筛查肺结节。由于肺结节大小形态各异且CT切片数量较大,通过人工筛查肺结节是件相当耗时且困难的任务。随着计算机技术的发展,采用计算机辅助诊断(Computer Aided Diagnosis, CAD)可以高效地实现肺结节检测。一般来说,肺结节检测框架主要包括三个阶段:肺实质分割、候选结节检测以及假阳性剔除。肺实质分割的目的是将肺结节的检测范围限制在肺部区域,以免检测到肺部外的假阳性。候选结节检测的是尽可能多地将肺结节检出,降低漏检的可能。假阳性剔除的目的是剔除候选结节中的假阳性结果,减少误检的情况。与一般的目标检测任务相比,由于多数肺结节尺寸较小、形态各异,且结节周边的肺内环境复杂,肺结节检测的难度更大。
本文结合近年来肺结节检测相关的文献方法,从数据集及评价指标、肺实质分割、候选结节检测和假阳性剔除四个方面,介绍了肺结节检测的研究进展,并对各检测方法的效果进行了比较分析。最后对该领域目前存在的难点以及未来的发展进行一定的讨论和总结。
2. 数据集及评价指标
2.1. LIDC-IDRI (The Lung Image Database Consortium and Image Database Resource Initiative)
LIDC-IDRI数据集是由美国国家癌症研究所发起,并由7个研究机构和8个医学影像公司共同建立的一个标准的数据集。该数据集提供了来自1010个患者的1018例临床胸部CT扫描图像以及相关的可扩展标记语言(Extensive Markup Language, XML)文件,其中CT图像的切片厚度平均为1.74毫米。XML文件中记录了四位放射科医生标注的结节特征、类型和位置等信息。每位放射科医生独立完成标注,将病灶分为三类:直径大于3毫米的结节、直径小于3毫米的结节以及非结节。LIDC-IDRI数据集共有7371个由至少一位放射科医生标注为结节的病灶,其中2669个病灶被至少一位放射科医生标注为直径大于3毫米的结节,有928个病灶被四位放射科医生均标注为大于3毫米的结节。LIDC-IDRI数据集提供一个重要的医学影像学研究资源,目前被肺结节研究领域广泛应用。
2.2. LUNA16 (Lung Nodule Analysis 2016)
LUNA16数据集是2016年为了大规模评估肺结节检测算法而推出的肺结节检测数据集,其数据来自LIDC-IDRI。为了使CT数据更为标准,LUNA16去除了LIDC-IDRI中切片厚度大于3 mm、切片间隔不一致以及部分切片缺失的CT,最后产生888张CT,保存为.mhd格式。在这888张CT中,共有36,378个来自LIDC-IDRI的标注。LUNA16只纳入其中直径大于3 mm的结节标注,并且合并两个中心距离小于半径和的结节(合并后的中心和半径是两个结节的均值)。经过上述处理,选取剩下至少由三位专家标注的1186个结节标注作为最后需要检测的区域。同时,LUNA16还提供了用于进行假阳性剔除的候选区域标注,候选区域的计算方式采用现有的三种候选检测算法 [4] [5] [6]。由于病灶可以被多个候选者检测到,因此将中心位置小于5 mm的候选区域合并。使用这种方法,在1186个结节中,有1120个结节被检测出551,065个候选区域,其中每个候选区域都被标注了类别标签,0代表非结节,1代表结节。
2.3. DSB2017 (Data Science Bowl 2017)
DSB2017是美国Kaggle公司举办的通过病人胸部CT诊断是否患有肺癌的竞赛,旨在召集数据科学和医学界开发有效的肺癌检测算法。该数据集是由美国国家癌症研究中心提供的高风险患者的DICOM格式的CT图像,其中包含切片厚度、切片间隔等信息。该数据集只提供了每个患者的二进制标签,表明此患者在之后一年内是否被诊断为肺癌。DSB数据集中含有许多直径超过40 mm的大结节,结节平均直径为13.68 mm。DSB数据分为两个阶段:stage 1和stage 2。stage 1为训练集和验证集,用于训练模型以及验证模型的性能,其中训练集1397个病例,验证集198个病例。stage 2为测试集,共506个病例,其数据质量要高于stage 1,通常扫描切片的厚度更薄。
2.4. 评价指标
2.4.1. 查准率(Precision)、查全率(Recall)以及特异度(Specificity)
查准率和查全率是信息检索中常用的评价检索或分类性能的方法 [7],目前通常用于定量分析肺结节检测和分类的实验结果。在计算时会用到四个变量:真阳性(True Positive, TP),实际为正样本,并被预测为正样本;假阳性(False Positive, FP),实际为负样本,但被预测为正样本;真阴性(True Negative, TN),实际为负样本,并被预测为负样本;假阴性(False Negative, FN),实际为正样本,但被预测为负样本。查准率又称精准率,表示当前被预测为正样本中正确分类的比例,如式(1)。查全率又称召回率或真阳性率(TPR),表示被预测为正的样本占所有真实的正样本的比例,如式(2)。特异度又称真阴性率(TNR),表示被预测为负的样本占所有真实的负样本的比例,如式(3)。表示F1-Score可以综合查准率和查全率的结果来评价预测的效果,如式(4)。
(1)
(2)
(3)
(4)
2.4.2. 常用的评价曲线
ROC (Receiver Operating Characteristic)曲线表示特异度和灵敏度(Sensitivity)之间的关系,其中灵敏度即为查全率,纵轴为灵敏度,横轴为假阳性率(1-特异度)。将模型检测时的置信度阈值设置多个值,每个阈值可以得到不同的特异度和灵敏度,从而可以绘制出ROC曲线。通常ROC曲线越趋近左上角,预测的假阳性和假阴性总数越少,检测性能越好。更为直观的比较方法是计算AUC (Area Under Curve)值,即ROC曲线下的面积,通常AUC值越大,模型的性能越好。FROC (Free-Response Operating Characteristic)曲线与ROC曲线相类似,纵轴为灵敏度,横轴为平均每次扫描的假阳性数(FPs/Scan),曲线越趋近左上角表示性能越好。在肺结节检测中假阳性数量通常较多,横轴采用平均每次扫描的假阳性数更方便对比不同模型的检测性能,因此在医学影像分析竞赛中大多采用FROC曲线。
2.4.3. 竞争性能指标(Competition Performance Metric, CPM)
CPM是指FROC曲线中定义的7个假阳性率的敏感度平均值:1/8,1/4,1/2,1,2,4和8 FPs/scan,如式(5)。CPM最早在2009年肺结节检测ANODE09 [8] 中提出,随后成为大多数肺部结节检测竞赛的评价指标。
(5)
3. 肺结节检测框架
肺结节检测框架一般包括三个部分:肺实质分割、候选结节检测以及假阳性剔除,如图1所示。肺实质分割是检测前十分重要的预处理步骤。在胸部CT中,通常会出现躯干、主气管以及床板等肺实质以外的图像信息,在检测时会出现将这些信息检测为肺结节的情况。为了避免这种情况,在进行肺结节检测之前,将肺实质区域从胸部CT中分割提取出来是保证检测有效性的必要前提。候选结节检测的目的是在保证一定精准率和召回率的情况下尽可能多的检出图像中疑似为结节的区域,肺结节检出的越多,漏检的可能越小,患者的生存率越高。为了保证候选结节检测时的高召回率,候选结节中通常含有大量的假阳性结果,因此需要进行假阳性剔除来进一步精确检测结果。通常采用有效的分类器对检测到的候选结节进行分类,进而剔除假阳性候选结节。
3.1. 肺实质分割
目前研究人员在肺实质分割领域做了大量的研究,采用不同的技术方法实现肺实质的自动分割,主要分为基于传统图像处理的分割方法和基于深度学习的分割方法。
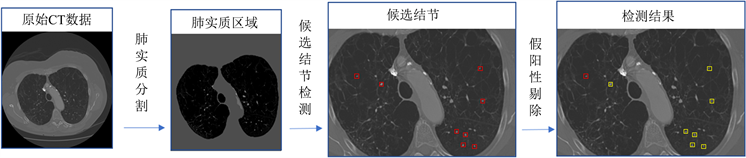
Figure 1. The framework of pulmonary nodule detection
图1. 肺结节检测框架
3.1.1. 基于传统图像处理的分割方法
基于传统图像处理的肺实质分割主要有 [9] 基于阈值的方法和基于区域的方法。基于阈值的方法是最基本的一种分割方法,由于肺实质区域内多为气体,其密度值小于周围组织,因此易于寻找阈值对肺实质进行分割。基于阈值的方法是假设CT图像中只包含两类体素:身体及胸壁结构的高密度体素和肺内气体以及体外空气的低密度体素。利用最优阈值法 [10] [11] [12] [13] 计算出合适的分割阈值,从而将肺实质区域进行初步的分割。一般初步分割后的结果并不准确,需要采用形态学方法对初步分割区域的组织进行分离,目的是得到分离的左右肺并移除主气管以及肺内的主支气管。最后对左右肺上由血管或结节形成的孔洞进行填充,并对其边界上的凹陷以及凸起进行平滑。基于阈值的分割方法比较直观,分割速度快且计算成本较低,结果一般具有可复现性。但由于阈值法通常不考虑分割目标的空间特征,因此不同成像条件下产生的噪声或成像伪影会对分割效果造成一定影响。
基于区域的方法是假设同一区域内的相邻体素值较为相似,其中应用最多的是区域生长法 [14] [15] [16],一般步骤是选取某个体素与其相邻体素进行比较,如果满足预设标准,则认为该体素与其一或多个相邻体素属于同一类。区域生长法的不足在于难以选取合适的预设标准,但相较于基于阈值的分割方法,区域生长法更为准确有效。分水岭算法 [13] [17] 也是基于区域的一种分割方法,主要思想是将图像看做拓扑地貌,图像中的灰度值作为该点海拔,区分“高山”与“湖泊”的界限即为分割线,分水岭算法对微弱边界的响应较强,但同时图像中的噪声及细微变化会导致过度分割的现象。文献 [17] 使用内部和外部标记对肺部区域精准标注,对标注后的图像与原始数据的梯度图合并后的图像应用分水岭算法,消除了一定过度分割的现象。除了上述两种方法,还有随机游走 [18] [19]、模糊连通性 [20] 等基于区域的方法。基于区域的分割方法利用了图像中的空间邻域信息,在应对成像伪影等问题时具有较强的鲁棒性。
3.1.2. 基于深度学习的分割方法
深度学习目前在图像处理和计算机视觉任务中的表现优于其他方法,继而出现了许多基于深度学习的肺实质分割方法。Harrison等 [21] 提出一种基于HNN [22] 的渐进式整体嵌套网络(progressive holistically-nested networks, P-HNNs)用于肺实质分割,P-HNNs在HNN的基础上增加了渐进的多分辨率路径连接,在不需要额外的网络参数的情况下,解决了HNN输出模糊以及全卷积网络分辨率粗化的问题。实验结果表明,该方法在929个胸部CT数据上的平均Dice系数 [23] 为0.985。
Ronneberger等 [24] 提出并实现了一种用于生物医学图像分割的U型全卷积网络结构—U-Net。如图2所示,U-Net包括一个用于收缩路径与其对称的扩展路径,在收缩路径与其对称扩展路径间有多条特征通道。由于U-Net的特殊结构,通常只需要很少的训练样本,便能在分割任务上提供了很好的性能 [24] [25]。此外,其端到端的训练方式可以在前向计算过程中处理整个图像并直接生成分割图。文献 [26] 采用11层卷积构成的U-Net进行了肺实质分割实验,研究人员将267张尺寸为128 × 128像素的肺部图像缩小为32 × 32像素,随后按7:3的比例划分训练集与测试集,模型在测试集上的分割精度为96.78%。但由于对原图像缩小比例过大,在高清CT图像(通常为512 × 512像素)上的分割效果并不理想,分割后的肺实质图像无法进行后续的检测工作。
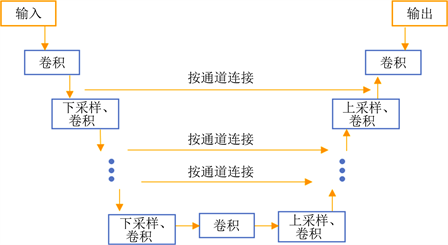
Figure 2. U-Net structure
图2. U-Net结构
Pang等 [27] 提出一种将U-Net与传统图像处理方法相结合的肺实质分割方法,该方法将预处理和分割作为一个迭代过程:首先预处理部分采用维纳滤波(Wiener Filter)对CT图像进行去噪,随后分割部分利用灰度共生矩阵生成初始分割图像,最后U-Net再对初始分割图像进一步的细化并输出分割图像,直至当前U-Net输出的分割图像与上一次输出相同时,得到最终的分割图像。文献 [25] 在U-Net的基础上结合ResNet [28] 和RCNN [29] 提出了两种实现分割任务的模型:RU-Net和R2U-Net。两个模型在包含267个图像的肺实质分割数据集 [30] 上训练与测试,Dice系数最高分别为0.9863和0.988。
3.2. 候选结节检测
早期的候选结节检测多采用一些传统方法,如阈值分割、形态学运算、聚类、区域生长等 [4] [5] [6] [31] [32]。这些传统的图像处理方法多是根据像素值以及图像的低层特征发展而来,由于结节的形状、大小以及纹理变化较大,传统方法很难提取到判别性特征。这会导致生成大量的假阳性候选结节,给后续的假阳性剔除工作带来很大的难度。随着深度学习的广泛应用,越来越多基于深度神经网络的检测算法被提出。目前,基于卷积神经网络(Convolutional Neural Network, CNN)的方法即能获取图像的低层特征,又能提取高层特征。常见的检测网络主要采用残差网络(Residual Network, ResNet) [28]、U-Net、特征金字塔网络(Feature Pyramid Network, FPN) [33]、区域卷积神经网络(Region Convolutional Neural Network, RCNN) [34] 等结构。
由于Faster RCNN [35] 出色的检测性能,因此被广泛应用于候选结节检测方向。Ding等 [36] 提出了一种基于2D Faster RCNN的候选结节检测方法,如图3所示,图像经过深度卷积神经网络(DCNN)特征提取,由区域建议网络(Region Proposal Network, RPN)给出感兴趣区域(Region Of Interest, ROI),随后经过ROI池化将感兴趣区域的特征图变为统一尺寸,并送入全连接网络对其分类和位置精修。相较于原始的Faster RCNN模型,该方法在RPN前加入反卷积层,恢复了更多的细粒度特征,使得检测灵敏度提升了12.9%。由于肺结节的尺寸要比自然图像中的检测目标更小,因此经过VGG-16 [37] 的五组卷积后不能清晰地描述结节特征,导致检测ROI的性能下降。此外,为了解决直接将原始CT的三维数据作为网络输入计算开销较大的问题,研究人员将待检测的切片与其相邻的两个切片按轴连接,并重新缩放为600 × 600 × 3像素的图像作为输入。但这种方式同时丢失了结节的部分空间特征,会生成较多假阳性候选结节。
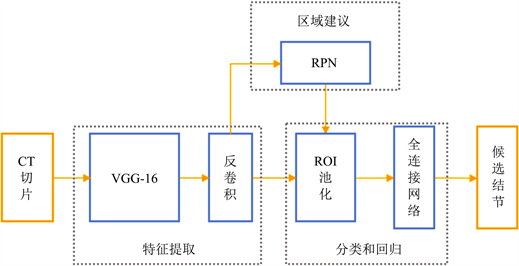
Figure 3. Faster R-CNN detection flow
图3. Faster RCNN检测流程
Liao等 [38] 提出一种利用3D RPN进行候选结节检测的方法。该模型由特征提取和定位两部分组成,特征提取部分采用残差块(Residual Block)与U型网络结构结合的3D DCNN,定位部分使用3D RPN直接输出结节的坐标与置信度。由于DCNN中加入了U型网络结构,其收缩与扩展两个路径使得RPN可以接受到更多细粒度特征,因此对于候选结节的定位更准确。文献 [39] 将双路径网络(DPN)与U型网络结构相结合,利用26个双路径块来学习三维数据更高层次的特征,随后将特征送入3D RPN得到候选结节区域。但由于候选结节的位置直接经过RPN进行输出,并未进行进一步的分类和回归,因此对于结节的检测具有一定的局限性。
Tang等 [40] 提出一种基于3D Faster RCNN的多任务端到端的模型,该模型将候选结节检测、假阳性剔除与结节分割整合到同一框架内。其中候选结节检测部分采用类似U-Net的网络结构对CT数据进行特征提取,随后将特征图送入两个平行的1 × 1 × 1的三维卷积层,分别生成特征图每个体素对应的置信度和对应锚点(Anchor) [35] 的六个回归项(深度、高度、宽度以及中心的三个轴坐标)以提取相应的ROI。由于使用耦合特征进行学习会降低网络性能 [41],因此用于分类和回归的ROI取自比RPN更浅层的特征图,使分类和回归能够学到与RPN不同的特征。实验结果表明,该方法在LIDC-IDRI数据集上的平均CPM为0.8727。
Ozdemir等 [42] 提出一种能够在任意尺寸CT上进行候选结节检测的模型,该模型是基于V-Net [43] 的3D全卷积网络,网络采用三个编码器与解码器的块对,除了输入层和输出层外加入对应的跨层连接。编码器使用两层卷积核尺寸为3的下采样卷积,并加入残差连接。与编码器基本相同,解码器采用两层上采样反卷积。首先根据LUNA16数据集提供的肺结节注释,制作其对应CT的结节掩码。由于结节相对于完整CT尺寸太小,将整个CT作为输入会导致网络不收敛,无法进行训练。研究人员在原始CT中随机采集结节附近64 × 64 × 64体素的立方数据,将其作为训练样本,使用交叉熵损失函数训练网络。通过对网络输出的体素阈值化,并基于每个体素的连通度来标记所有的独立区域,得到候选结节的区域。该方法成功地使用全卷积网络来进行候选结节检测的任务,候选结节的定位更加直观,但由于使用交叉熵损失函数,全卷积网络对于较小的结节不够敏感,容易造成漏识的情况。此外,对于不同成像条件下的CT图像,其网络输出的阈值需要进行一定的调整。
Masood等 [44] 基于云计算提出一种多层RPN的候选结节检测模型,通过使用多个RPN提取ROI增强特征提取的过程,使候选结节的定位更加准确。该模型通过VGG-16的五组卷积块和一层反卷积层后得到特征图,随后将特征图分别送入多个RPN进行给出区域建议。为了容纳不同尺寸的结节,研究人员为每个RPN设置了7组不同长宽比和尺寸的Anchor。采用多层RPN进行候选结节检测可以同时考虑结节的直径和体积,因此结节的检测效率显著提升。该模型在LDIC-IDRI数据集上的实验结果表明,使用多层RPN可以提升0.7%的召回率。
3.3. 假阳性剔除
传统的假阳性剔除工作通常利用人工设计的特征,将特征送入支持向量机、k近邻或Adaboost等分类器来降低假阳性 [4] [45] [46]。但由于结节形态、纹理和大小较为多变,因此针对一种结节设计的特征的表达能力较为有限,往往不适用于具有不同特征的其他类型结节。此外,由于肺内环境十分复杂,这些特征不具有较强的判别性来区分肺结节与其他组织,难以对大量的假阳性候选结节进行准确的剔除。近年来,CNN在医学图像分析领域取得了出色的效果,研究人员提出了大量卓有成效的基于CNN的假阳性剔除方法。
Zuo等 [49] 对HNN [22] 进行了知识转移(Knowledge Transfer),利用HNN能够从不同的深度的隐含层提取特征的能力,可以解决由于肺结节大小、形状不一致以及CT影像不均匀造成的特征表达困难。研究人员将以LUNA16中1186个标注结节的坐标中心生成的切片作为正样本,以肺实质区域随机生成坐标为中心的切片作为负样本。将候选结节经过不同深度的卷积与池化后的输出作为不同分支,每个分支可以得到不同层次和分辨率的特征,然后将这些特征裁切至相同尺寸后进行合并,合并后的特征经过全连接网络进行分类。实验结果表明,该方法在LUNA16数据集上进行假阳性剔除的准确率为97.33%。通过多分辨率的方式分析结节特征,该方法将一些不明显的结节正确识别,但由于完整的肺结节分布在多张切片上,使用2DCNN在获取切片间的上下文信息存在局限性。
Liu等 [47] 受到运动历史图(Motion History Image, MHI) [48] 的启发,提出一种位置历史图(Location History Image, LHI)算法进行假阳性剔除。首先通过3D FPN进行候选结节检测,将候选结节的立体数据生成LHI,随后将LHI送入一个由两层卷积和三层全连接组成的2D CNN中进行分类。由于真实结节在连续切片上的变化趋向于向轮廓外扩展或向中心递减,因此利用这一变化可以将结节切片间的上下文信息通过二维数据进行表达。实验结果表明,将这一方法应用在候选结节检测后,能够成功减少84.5%的假阳性。该方法采用2D CNN有效减少了计算成本,但由于LHI丢失了结节的密度、纹理等信息,对于假阳性剔除具有一定的局限性。
Dou等 [50] 提出了一种基于多尺度信息融合的3DCNN模型,首先以候选结节为中心提取尺寸分别为20 × 20 × 6,30 × 30 × 10和40 × 40 × 26的立方数据,三种尺寸的立方数据分别包含结节周围不同层次的背景信息。以结节的周围范围作为网络的感受野,通常感受野过大会导致冗余信息过多,造成网络性能下降,感受野太小会导致上下文信息不足,网络得不到充分训练,无法处理结节背景过大的变化。该模型由三个不同感受野大小的3D CNN组成,每个CNN结合候选结节周围的多层次背景信息给出各自的预测结果,随后将三组结果进行融合得出最后的分类结果。
Kim等 [51] 提出一种多尺度逐步积分3D CNN,通过逐步整合多尺度上下文信息有效的完成假阳性剔除。首先候选结节被处理为文献 [50] 中三种尺寸的立方数据,将其处理为同一尺寸,此时三种立方数据虽然对应同一结节坐标中心,但具有不同的分辨率。随后三种立方数据按照尺度变大和变小两种路径逐步进行特征提取。在放大路径上网络的视野逐渐变小,网络逐渐聚焦于结节区域,能够提取到更多结节的细节特征,同时缩小路径上网络的视野逐渐变大,网络逐渐聚焦于结节的周围区域,与周围区域的上下文相结合能够提取到结节的形态特征。通过两个路径得到的特征经过整合后送入后续的卷积层和全连接层,形成一个完整的端到端的网络。该方法在LUNA16数据集上的平均CPM为0.942,在假阳性剔除任务上取得很好的性能。
4. 结果比较与讨论
为了更好地展示各种检测方法的效果,表1中列举了所述文献中各方法所采用的网络结构,以及在不同数据集上的实验结果。其中两阶段的方法是指在肺结节检测过程中,候选结节检测与假阳性剔除两个模型分别完成各自的任务,表中两阶段的方法均为进行假阳性剔除后的实验结果。从目前提出的方法可以看到,部分研究人员选择采用两阶段方法完成肺结节检测,如文献 [36] [42] [44] [47],两阶段方法可以尽可能利用目前最先进目标检测算法和图像分类算法,并且在检测流程上更加直观。此外,当检测效果受到模型的限制时,采用两阶段方法可以更容易找到问题所在,从而有针对性对某一阶段模型进行调整,具有较高的灵活性。根据实验结果可以看出,采用两阶段方法在LUNA16数据集上进行肺结节检测的CPM均在0.89以上,相比于只进行候选结节检测的模型效果要更好。从长远的角度来看,在现有的候选结节检测模型基础上,结合更为有效的假阳性剔除模型,可以开发性能更好的肺结节检测框架。文献 [40] 采用端到端的方式,将候选结节检测和假阳性剔除两部分的损失作为模型的整体损失进行训练,模型的整体学习能力更强,因此在未来的研究工作中可以将两阶段方法改进为端到端训练的方式。
Table 1. Comparison of experimental results of different methods
表1. 各方法实验结果比较
从表1可以看到,当前肺结节检测模型都是基于CNN的变体,目前肺结节检测的发展趋势是对最先进的CNN模型进行调整,使其在肺结节检测和分类上达到最佳性能。Ding等将Faster RCNN引入肺结节检测领域,并在其基础上加入反卷积结构,在LUNA16数据集上的CPM达到0.891,取得了LUNA16挑战结节检测阶段的第一名。Liao则是将Faster RCNN拓展到三维,并将U-Net的结构运用在特征提取阶段,在DSB2017数据集上的CPM为0.8562,获得DSB2017肺癌检测竞赛的冠军。随着CNN的不断发展,更多的网络结构被应用在肺结节检测领域,目前在LUNA16数据集上取得最佳性能的模型是文献 [47] 提出的3D FPN和HS2Net,其CPM为0.957。
5. 总结与展望
本文着重介绍了肺实质分割的研究进展,相较于候选结节检测与假阳性剔除,肺实质分割作为一个重要的数据预处理步骤,与后续的检测工作关系十分紧密,选择不同的肺实质分割方法对肺结节检测的结果有着不同程度的影响。在本文所述文献中,利用深度学习方法进行肺实质分割取得了较好的效果,相较于利用传统图像处理技术的方法,深度学习能够以数据驱动的方式学习肺实质的特征,进而准确地分割肺实质区域。此外,由于采用传统图像处理方法进行肺实质分割需要进行迭代计算或连通区域计算等较为耗时的操作,采用深度学习方法能够更快地得到分割结果。但深度学习方法需要大量标注过的分割数据,同时需要较高要求的计算设备。因此,如果研究人员拥有较多带有金标准的肺实质分割数据集以及具备高标准的计算资源,建议采用基于深度学习的分割方法,否则建议采用基于传统图像处理的方法。
目前,由于CNN在计算机视觉的广泛应用,研究人员倾向于使用基于CNN的算法来代替传统的肺结节检测方法。在所述的文献中,具有优异性能的检测模型应用了大量先进的CNN网络结构,例如ResNet、U-Net、FPN等。在文献 [38] [39] [40] 中,采用ResNet的跨层连接结构解决了网络退化的问题,使网络深度可以进一步增加,提取更高层次的特征。在文献 [38] [39] [42] 中使用了类似U-Net的网络结构,由于U-Net中包括一个用于捕获上下文的收缩路径和一个支持精确定位的对称扩展路径,并在收缩路径与其对称扩展路径间增加了特征通道,使得收缩路径得到的高分辨率特征可以与扩展路径的高级语义特征相结合,其扩展路径对检测小尺寸结节有较大的帮助。文献 [47] 采用了FPN的网络结构,FPN可以对每一种尺度的图像进行特征提取,能够产生多尺度的特征表示,并将不同层次的特征图用连接的方式进行融合,使每个层次的特征图都具有较强的语义信息。通过整合多尺度信息,能够大幅度提升小尺寸结节的检测效果。可以看到,将目前计算机视觉领域较为先进的网络结构应用到肺结节检测模型上,可以进一步提升肺结节检测的效果。
虽然现有的肺结节检测方法达到了较为满意的效果,但当前仍存在一定的挑战。由于基于深度学习的肺结节检测方法依赖于大量高质量的金标准数据,因此高质量的金标准数据是进一步提升肺结节检测性能的关键。但现有的胸部CT公开数据集并没有进行有序的标记,大量数据集之间的注释信息较为混乱,因此金标准数据极为稀缺。数据缺乏一方面是因为胸部CT多为保密数据,医院通常出于保护病人隐私的需要,无法将数据提供给研究人员。另一方面是因为带有金标准的图像数据需要放射科医生花费大量的时间和精力进行标注,往往专业医生没有时间对数据进行精准的标注,而非专业人员的标注则容易出现误标或漏标。为了缓解这一问题,通常是采用数据扩增的技术,对现有的数据进行裁剪、旋转、翻转或者缩放的方法来增加训练样本的数量和多样性,但这种方法终归具有一定的局限性。目前,生成式对抗网络(Generative Adversarial Network, GAN) [55] 可以利用给定的训练集通过生成器和判别器的相互对抗生成新的数据样本,随着GAN的发展,若能采用GAN生成更多的CT图像以及结节样本用于训练,将大大降低数据获取的成本。当存在大量的未标记CT数据时,可以进一步研究基于半监督或自监督的学习方法,这样可以大大节省人工标注的时间和精力。目前多模态图像融合的方式在图像识别领域取得了显著的效果 [56],采用多流CNN可以充分利用多模态特征,包括低层图像特征和高层语义特征。在将来可以利用病人的更多信息,将多模态数据送入多流CNN框架,让模型学习不同的语义特征,从而提高结节检测的准确性。此外,还可以采用迁移学习的方法,将在其他医学影像上学习的模型应用在肺结节检测任务上,利用从不同的疾病模式中提取的特征进行学习,可以在一定程度上解决数据缺乏以及过拟合的问题。
基金项目
北京市自然科学基金项目(资助号:4202025),国家自然科学基金项目(资助号:61771341, 41776204),北京教委科技计划项目(资助号:KM201911232003),北京未来芯片技术高精尖创新中心科研基金(资助号:KYJJ2018004)。
文章引用
陈 辰,苗 军,齐洪钢,刘 艳,郭志军,刘海涛. 肺结节图像的计算机自动检测研究进展
Research Progress of Computer Automatic Detection of Pulmonary Nodules in Images[J]. 计算机科学与应用, 2021, 11(01): 143-155. https://doi.org/10.12677/CSA.2021.111016
参考文献
- 1. Bray, F., Ferlay, J., Soerjomataram, I., et al. (2018) Global Cancer Statistics 2018: GLOBOCA Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries. CA: A Cancer Journal for Clinicians, 68, 394-424. https://www.ncbi.nlm.nih.gov/pubmed/30207593https://doi.org/10.3322/caac.21492
- 2. 周清华, 范亚光, 王颖, 乔友林, 王贵齐, 黄云超, 王新允, 吴宁, 张国桢, 郑向鹏, 步宏. 中国肺部结节分类、诊断与治疗指南(2016年版) [J]. 中国肺癌杂志, 2016, 19(12): 793-798.
- 3. National Lung Screening Trial Research Team (2011) Reduced Lung-Cancer Mortality with Low-Dose Computed Tomographic Screening. New England Journal of Medicine, 365, 395-409. https://doi.org/10.1056/NEJMoa1102873
- 4. Messay, T., Hardie, R.C. and Rogers, S.K. (2010) A New Compu-tationally Efficient CAD System for Pulmonary Nodule Detection in CT Imagery. Medical Image Analysis, 14, 390-406. https://doi.org/10.1016/j.media.2010.02.004
- 5. Murphy, K., van Ginneken, B., Schilham, A.M.R., et al. (2009) A Large-Scale Evaluation of Automatic Pulmonary Nodule Detection in Chest CT Using Local Image Features and k-Nearest-Neighbour Classification. Medical Image Analysis, 13, 757-770. https://doi.org/10.1016/j.media.2009.07.001
- 6. Hardie, R.C., Rogers, S.K., Wilson, T., et al. (2008) Performance Analysis of a New Computer Aided Detection System for Identifying Lung Nodules on Chest Radiographs. Medical Image Analysis, 12, 240-258. https://doi.org/10.1016/j.media.2007.10.004
- 7. Lewis, D. (1991) Evaluating Text Categorization. In: Proceedings of Speech and Natural Language Workshop, Morgan Kaufmann, Burlington, 312-318. https://doi.org/10.3115/112405.112471
- 8. Van Ginneken, B., Armato III, S.G., de Hoop, B., et al. (2010) Com-paring and Combining Algorithms for Computer-Aided Detection of Pulmonary Nodules in Computed Tomography Scans: The ANODE09 Study. Medical Image Analysis, 14, 707-722. https://doi.org/10.1016/j.media.2010.05.005
- 9. Mansoor, A., Bagci, U., Foster, B., et al. (2015) Segmentation and Image Analysis of Abnormal Lungs at CT: Current Approaches, Challenges, and Future Trends. RadioGraphics, 35, 1056-1076. https://doi.org/10.1148/rg.2015140232
- 10. Hu, S., Hoffman, E.A. and Reinhardt, J.M. (2001) Auto-matic Lung Segmentation for Accurate Quantitation of Volumetric X-Ray CT Images. IEEE Transactions on Medical Imaging, 20, 490-498. https://doi.org/10.1109/42.929615
- 11. Shariaty, F., Hosseinlou, S. and Rud, V.Y. (2019) Automatic Lung Segmentation Method in Computed Tomography Scans. Journal of Physics: Conference Series, 1236, Article ID: 012028. https://doi.org/10.1088/1742-6596/1236/1/012028
- 12. Zhang, W., Zhang, X., Zhao, J., et al. (2017) A Segmenta-tion Method for Lung Nodule Image Sequences Based on Superpixels and Density-Based Spatial Clustering of Applica-tions with Noise. PLoS ONE, 12, e0184290. https://doi.org/10.1371/journal.pone.0184290
- 13. Vincent, L. and Soille, P. (1991) Watersheds in Digital Spaces: An Efficient Algorithm Based on Immersion Simulations. IEEE Transactions on Pattern Analysis & Machine Intelli-gence, 13, 583-598. https://doi.org/10.1109/34.87344
- 14. Adams, R. and Bischof, L. (1994) Seeded Region Growing. IEEE Transactions on Pattern Analysis and Machine Intelligence, 16, 641-647. https://doi.org/10.1109/34.295913
- 15. Wu, J., Poehlman, S., Noseworthy, M.D., et al. (2008) Texture Feature Based Automated Seeded Region Growing in Abdominal MRI Segmentation. 2008 International Conference on Bio-Medical Engineering and Informatics IEEE, Vol. 2, 263-267. https://doi.org/10.1109/BMEI.2008.352
- 16. Zhao, J., Ji, G., Han, X., et al. (2016) An Automated Pulmonary Parenchyma Segmentation Method Based on an Improved Re-gion Growing Algorithmin PET-CT Imaging. Frontiers of Computer Science, 10, 189-200. https://doi.org/10.1007/s11704-015-4543-x
- 17. Shojaii, R., Alirezaie, J. and Babyn, P. (2005) Automatic Lung Segmentation in CT Images Using Watershed Transform. IEEE International Conference on Image Processing, 2, II-1270. https://doi.org/10.1109/ICIP.2005.1530294
- 18. Grady, L. (2006) Random Walks for Image Segmentation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 28, 1768-1783. https://doi.org/10.1109/TPAMI.2006.233
- 19. Qin, C., Zhang, G., Zhou, Y., et al. (2014) Integration of the Sali-ency-Based Seed Extraction and Random Walks for Image Segmentation. Neurocomputing, 129, 378-391. https://doi.org/10.1016/j.neucom.2013.09.021
- 20. Udupa, J.K. and Samarasekera, S. (1996) Fuzzy Connectedness and Object Definition: Theory, Algorithms, and Applications in Image Segmentation. Graphical Models and Image Pro-cessing, 58, 246-261. https://doi.org/10.1006/gmip.1996.0021
- 21. Harrison, A.P., Xu, Z., George, K., et al. (2017) Progressive and Multi-Path Holistically Nested Neural Networks for Pathological Lung Segmentation from CT Images. International Conference on Medical Image Computing and Computer-Assisted Intervention, Springer, Cham, 621-629. https://doi.org/10.1007/978-3-319-66179-7_71
- 22. Xie, S. and Tu, Z. (2015) Holistically-Nested Edge Detection. Proceedings of the IEEE International Conference on Computer Vision, Santiago, 7-13 December 2015, 1395-1403. https://doi.org/10.1109/ICCV.2015.164
- 23. Dice, L.R. (1945) Measures of the Amount of Ecologic Association between Species. Ecology, 26, 297-302. https://doi.org/10.2307/1932409
- 24. Ronneberger, O., Fischer, P. and Brox, T. (2015) U-Net: Convolutional Networks for Biomedical Image Segmentation. International Conference on Medical Image Computing and Comput-er-Assisted Intervention, Springer, Cham, 234-241. https://doi.org/10.1007/978-3-319-24574-4_28
- 25. Alom, M.Z., Yakopcic, C., Hasan, M., et al. (2019) Recurrent Residual U-Net for Medical Image Segmentation. Journal of Medical Imaging, 6, Article ID: 014006. https://doi.org/10.1117/1.JMI.6.1.014006
- 26. Shaziya, H., Shyamala, K. and Zaheer, R. (2018) Automatic Lung Segmentation on Thoracic CT Scans Using u-Net Convolutional Network. 2018 International Conference on Communi-cation and Signal Processing (ICCSP) IEEE, Chennai, 3-5 April 2018, 643-647. https://doi.org/10.1109/ICCSP.2018.8524484
- 27. Pang, T., Guo, S., Zhang, X., et al. (2019) Automatic Lung Segmentation Based on Texture and Deep Features of HRCT Images with Interstitial Lung Disease. BioMed Research International, 2019, Article ID: 2045432. https://doi.org/10.1155/2019/2045432
- 28. He, K., Zhang, X., Ren, S., et al. (2016) Deep Residual Learning for Image Recognition. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Las Vegas, 27-30 June 2016, 770-778. https://doi.org/10.1109/CVPR.2016.90
- 29. Liang, M. and Hu, X. (2015) Recurrent Convolutional Neural Net-work for Object Recognition. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Boston, 7-12 June 2015, 3367-3375.
- 30. Lung Segmentation Dataset. https://www.kaggle.com/kmader/finding-lungs-in-ct-data/data
- 31. Han, H., Li, L., Han, F., et al. (2014) Fast and Adaptive Detection of Pulmonary Nodules in Thoracic CT Images Using a Hierarchical Vector Quantization Scheme. IEEE Journal of Biomedical and Health Informatics, 19, 648-659. https://doi.org/10.1109/JBHI.2014.2328870
- 32. Brown, M.S., Lo, P., Goldin, J.G., et al. (2014) Toward Clini-cally Usable CAD for Lung Cancer Screening with Computed Tomography. European Radiology, 24, 2719-2728. https://doi.org/10.1007/s00330-014-3329-0
- 33. Lin, T.Y., Dollár, P., Girshick, R., et al. (2017) Feature Pyramid Networks for Object Detection. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Hon-olulu, 21-26 July 2017, 2117-2125. https://doi.org/10.1109/CVPR.2017.106
- 34. Girshick, R., Donahue, J., Darrell, T., et al. (2014) Rich Feature Hi-erarchies for Accurate Object Detection and Semantic Segmentation. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Columbus, 23-28 June 2014, 580-587. https://doi.org/10.1109/CVPR.2014.81
- 35. Ren, S., He, K., Girshick, R., et al. (2016) Faster r-cnn: Towards Re-al-Time Object Detection with Region Proposal Networks. IEEE Transactions on Pattern Analysis and Machine Intelli-gence, 39, 1137-1149. https://doi.org/10.1109/TPAMI.2016.2577031
- 36. Ding, J., Li, A., Hu, Z., et al. (2017) Accurate Pulmonary Nodule Detection in Computed Tomography Images Using Deep Convolutional Neural Networks. In: International Conference on Medical Image Computing and Computer-Assisted Intervention, Springer, Cham, 559-567. https://doi.org/10.1007/978-3-319-66179-7_64
- 37. Simonyan, K. and Zisserman, A. (2014) Very Deep Convolu-tional Networks for Large-Scale Image Recognition.
- 38. Liao, F., Liang, M., Li, Z., et al. (2019) Evaluate the Malig-nancy of Pulmonary Nodules Using the 3-d Deep Leaky Noisy-Or Network. IEEE Transactions on Neural Networks and Learning Systems, 30, 3484-3495. https://doi.org/10.1109/TNNLS.2019.2892409
- 39. Zhu, W., Liu, C., Fan, W., et al. (2018) Deeplung: Deep 3d Dual Path Nets for Automated Pulmonary Nodule Detection and Classification. 2018 IEEE Winter Conference on Appli-cations of Computer Vision (WACV), Lake Tahoe, 12-15 March 2018, 673-681. https://doi.org/10.1109/WACV.2018.00079
- 40. Tang, H., Zhang, C. and Xie, X. (2019) Nodulenet: Decoupled False Positive Reduction for Pulmonary Nodule Detection and Segmentation. In: International Conference on Medical Image Computing and Computer-Assisted Intervention, Springer, Cham, 266-274. https://doi.org/10.1007/978-3-030-32226-7_30
- 41. Cheng, B., Wei, Y., Shi, H., et al. (2018) Revisiting rcnn: On Awakening the Classification Power of Faster rcnn. Proceedings of the European Conference on Computer Vision (ECCV), Munich, 8-14 September 2018, 453-468. https://doi.org/10.1007/978-3-030-01267-0_28
- 42. Ozdemir, O., Russell, R.L. and Berlin, A.A. (2019) A 3D Probabilistic Deep Learning System for Detection and Diagnosis of Lung Cancer Using Low-Dose CT Scans. IEEE Transactions on Medical Imaging, 39, 1419-1429. https://doi.org/10.1109/TMI.2019.2947595
- 43. Milletari, F., Navab, N. and Ahmadi, S.A. (2016) V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation. 2016 Fourth International Conference on 3D Vision (3DV) IEEE, Stanford, 25-28 October 2016, 565-571. https://doi.org/10.1109/3DV.2016.79
- 44. Masood, A., Yang, P., Sheng, B., et al. (2019) Cloud-Based Automated Clinical Decision Support System for Detection and Diagnosis of Lung Cancer in Chest CT. IEEE Journal of Transla-tional Engineering in Health and Medicine, 8, 1-13. https://doi.org/10.1109/JTEHM.2019.2955458
- 45. Jacobs, C., van Rikxoort, E.M., Twellmann, T., et al. (2014) Automatic Detection of Subsolid Pulmonary Nodules in Thoracic Computed Tomography Images. Medical Image Analysis, 18, 374-384. https://doi.org/10.1016/j.media.2013.12.001
- 46. Naqi, S.M., Sharif, M. and Lali, I.U. (2019) A 3D Nodule Can-didate Detection Method Supported by Hybrid Features to Reduce False Positives in Lung Nodule Detection. Multimedia Tools and Applications, 78, 26287-26311. https://doi.org/10.1007/s11042-019-07819-3
- 47. Liu, J., Cao, L., Akin, O., et al. (2019) Accurate and Robust Pulmonary Nodule Detection by 3D Feature Pyramid Network with Self-Supervised Feature Learning.
- 48. Davis, J.W. (2001) Hierarchical Motion History Images for Recognizing Human Motion. Proceedings IEEE Workshop on Detection and Recognition of Events in Video, Vancouver, 8 July 2001, 39-46.
- 49. Zuo, W., Zhou, F., Li, Z., et al. (2019) Mul-ti-Resolution CNN and Knowledge Transfer for Candidate Classification in Lung Nodule Detection. IEEE Access, 7, 32510-32521. https://doi.org/10.1109/ACCESS.2019.2903587
- 50. Dou, Q., Chen, H., Yu, L., et al. (2016) Mul-tilevel Contextual 3-D CNNs for False Positive Reduction in Pulmonary Nodule Detection. IEEE Transactions on Bio-medical Engineering, 64, 1558-1567. https://doi.org/10.1109/TBME.2016.2613502
- 51. Kim, B.C., Yoon, J.S., Choi, J.S., et al. (2019) Multi-Scale Gradual Integration CNN for False Positive Reduction in Pulmonary Nodule Detection. Neural Networks, 115, 1-10. https://doi.org/10.1016/j.neunet.2019.03.003
- 52. Armato III, S.G., Hadjiiski, L.M., Tourassi, G.D., et al. (2015) Special Section Guest Editorial: LUNGx Challenge for Computerized Lung Nodule Classification: Reflections and Les-sons Learned. Journal of Medical Imaging, 2, Article ID: 020103. https://doi.org/10.1117/1.JMI.2.2.020103
- 53. Dolejsi, M., Kybic, J., Polovincak, M., et al. (2009) The Lung Time: Annotated Lung Nodule Dataset and Nodule Detection Framework. Medical Imaging 2009: Computer-Aided Diagnosis, International Society for Optics and Photonics, Vol. 7260, 72601U. https://doi.org/10.1117/12.811645
- 54. Mak, R.H., Endres, M.G., Paik, J.H., et al. (2019) Use of Crowd Innovation to Develop an Artificial Intelligence-Based solu-tion for Radiation Therapy Targeting. JAMA Oncology, 5, 654-661. https://doi.org/10.1001/jamaoncol.2019.0159
- 55. Creswell, A., White, T., Dumoulin, V., et al. (2018) Generative Adversarial Networks: An Overview. IEEE Signal Processing Magazine, 35, 53-65. https://doi.org/10.1109/MSP.2017.2765202
- 56. Du, J., Li, W., Lu, K., et al. (2016) An Overview of Multi-Modal Medical Image Fusion. Neurocomputing, 215, 3-20. https://doi.org/10.1016/j.neucom.2015.07.160
NOTES
*通讯作者。
