Advances in Clinical Medicine
Vol.
13
No.
03
(
2023
), Article ID:
62799
,
11
pages
10.12677/ACM.2023.133580
胃源性柠檬双面神菌的耐药和毒力全基因组 分析
李志鹏1,2,王莉莉1,2,张庆龙1,2,3,董全江1,2*
1青岛大学附属青岛市市立医院中心实验室,山东 青岛
2青岛大学附属青岛市市立医院消化内科,山东 青岛
3山东第一医科大学(山东省医学科学院),山东 济南
收稿日期:2023年2月15日;录用日期:2023年3月11日;发布日期:2023年3月21日

摘要
【目的】从基因组水平上探讨胃源性柠檬双面神菌PA32株的耐药性和致病机制。【方法】采用耐酸实验测定PA32菌株的耐酸能力,采用纸片扩散法测定PA32菌株对4种抗菌药物的敏感性,利用三代测序平台对其进行全基因组测序和组装,利用glimmer进行基因预测,利用VFDB数据库、CARD数据库进行毒力和耐药基因功能注释。【结果】耐酸实验表明,PA32菌株能够耐受pH 2的酸性环境。药敏实验表明,PA32菌株对甲硝唑耐药,对克拉霉素、四环素、左氧氟沙星均敏感。该菌株的基因组包括一条完整的环状染色体序列,染色体长度为3,459,946 bp,鸟嘌呤–胞嘧啶(GC)含量为73.1%,编码3296个基因。PA32菌株含有黏附和(或)侵袭、分泌系统和无氧呼吸相关毒力基因,以及对氟喹诺酮类、氨基糖苷类、四环素类、大环内酯类、β-内酰胺类等多种抗生素的耐药基因。【结论】胃源性柠檬双面神菌耐酸能力强,从基因组学水平分析具备致病潜力,这与其携带的多种毒力基因和耐药基因相关。
关键词
柠檬双面神菌,全基因组,耐药性,毒力

Whole Genome Sequencing of Janibacter melonis from the Stomach and Virulence and Antibiotic-Resistant Gene Analysis
Zhipeng Li1,2, Lili Wang1,2, Qinglong Zhang1,2,3, Quanjiang Dong1,2*
1Central Laboratories, Qingdao Municipal Hospital, Qingdao University, Qingdao Shandong
2Department of Gastroenterology, Qingdao Municipal Hospital, Qingdao University, Qingdao Shandong
3Shandong First Medical University (Shandong Academy of Medical Science), Jinan Shandong
Received: Feb. 15th, 2023; accepted: Mar. 11th, 2023; published: Mar. 21st, 2023

ABSTRACT
[Objective] To investigate the drug resistance and pathogenic mechanism of a Janibacter melonis strain PA32 at the genome-wide level. [Method] The acid resistance capacity of strain PA32 was determined by acid resistance assay. The susceptibility of PA32 strain to 4 antimicrobial agents was determined by disk diffusion method. Whole genome sequencing and assembly of PA32 were conducted by the three-generation sequencing platform, and gene prediction was conducted using glimmer, and functional annotation of virulence and drug resistance genes was conducted using VFDB database and CARD database. [Result] The PA32 strain could withstand the acidic environment at pH 2. The PA32 strain was resistant to metronidazole, and susceptible to clarithromycin, tetracycline and levofloxacin. The complete genome sequence of PA32 comprises a 3,459,946 bp circular chromosome containing 3296 genes with the guanine-cytosine (GC) content of 73.1%. PA32 strain contained virulence genes related to adhesion, secretion system and anaerobic respiration, as well as drug resistance genes to fluoroquinolones, aminoglycosides, tetracycline, macrolides, β-lactam and other antibiotics. [Conclusion] Isolated from the stomach, J. melonis displays a strong ability of acid resistance. Comparative genomics indicate that J. melonis has pathogenic potential, which is associated with its multidrug resistance genes and virulence genes.
Keywords:Janibacter melonis, Whole Genome, Drug Resistance, Virulence

Copyright © 2023 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
双面神菌属(Janibacter)属于放线菌门,于1997年从腐烂的甜瓜中首次被分离鉴定 [1]。研究显示,双面神菌属可引起阴道感染、呼吸道感染,甚至严重的菌血症或败血症 [2] [3] [4]。同时,柠檬双面神菌(Janibacter melonis)曾从乳糜泻患者的十二指肠中分离出来,因此被认为与乳糜泻有关 [5]。对此十二指肠分离株的全基因组分析揭示了侵袭、黏附、生物膜形成、铁摄取和调控应激反应的关键毒力决定基因 [5]。这些发现证实双面神菌属在炎症性疾病中的致病作用。
胃菌群包含数百种细菌属,在胃生理过程的稳态维持中发挥重要作用。胃菌群失调会导致粘膜炎症和胃癌 [6]。除了引起胃癌的主要病原体幽门螺杆菌外,胃菌群中的其它细菌也会参与胃炎和胃癌的发生。研究显示,同时感染唾液链球菌(Streptococcus salivarius)与幽门螺杆菌的小鼠的胃黏膜病理分级明显高于单独感染幽门螺杆菌的小鼠 [7]。而可以产生脲酶且含有与幽门螺杆菌相似毒力基因的苍白杆菌(Ochrobactrum),被发现在早期胃癌中的相对丰度显著升高 [8]。因此长期以来,人们认为胃菌群的过度生长促进了癌变 [9]。对菌群的分子分析显示,双面神菌属的相对丰度在胃癌中增加,是慢性胃炎、肠化生和上皮内瘤变患者的数倍 [10]。因此,为研究胃源性柠檬双面神菌的耐药性和致病性,采用三代测序技术对分离自胃癌患者胃大切标本的柠檬双面神菌进行全基因组测序,从基因组水平对其耐药性和致病性进行分析,为进一步研究其致病性及与宿主相互作用提供理论依据。
2. 材料与方法
2.1. 菌株活化及菌悬液制备
青岛市市立医院临床分离株柠檬双面神菌PA32菌株为本实验室保存。挑取−80℃下冻干管保存的PA32菌株接种于BHI琼脂培养基,37℃活化两次后,取对数生长期菌液3000 × g室温离心20 min,得到菌体沉淀用等体积无菌0.1 mol/L PBS洗涤2次并重悬,测定并调整菌液浓度至OD600 ≈ 1.0并保存备用。
2.2. 体外实验
2.2.1. 耐酸实验
将上述浓度为OD600 ≈ 1.0的PA32菌液暴露于含有不同浓度盐酸(pH为2.0~6.0和7.4)的BHI液体培养基中。分别在培养0.5 h、1 h、2 h后测定不同pH值的BHI液体培养基的OD600。对照组为pH值为7.4的OD600。计算各组的存活率,公式如下:存活率(%) = (实验组OD600/对照组OD600) × 100。以存活率评价其耐酸性。本实验独立重复三次。
2.2.2. 药物敏感性实验
采用K-B纸片扩散法测定PA32菌株对甲硝唑、克拉霉素、四环素、左氧氟沙星等4种抗菌药物的药物敏感性。药敏纸片由英国OXOID公司生产,抗生素含量为:甲硝唑5 ug/片,克拉霉素15 ug/片,四环素30 ug/片,左氧氟沙星5 ug/片。在无菌BHI培养基上均匀涂布稀释到OD600 ≈ 0.1的细菌,将药敏纸片(OXOID,英国)放置在培养基表面,在37℃下培养18~24小时。使用游标卡尺测量抑菌圈直径,根据2017年美国临床和实验室标准协会(Clinical and Laboratory Standards Institute),CLSI抗生素敏感性试验标准 [11],判断细菌对抗生素的敏感性或耐药性。本实验独立重复三次。
2.3. 基因组学分析
2.3.1. 全基因组测序及组装
利用细菌基因组DNA提取试剂盒提取PA32菌株的基因组总DNA,利用三代测序平台Pacific Biosciences对其进行全基因组测序,然后基于三代测序的基因组从头组装技术,首先使用falcon进行原始数据的优化及基因组初步组装 [12],得到一致性序列(consensus),然后使用Genomic Consensus软件基于原始数据(subreads)再次优化一致性序列,最终使用sprai (single pass read accuracy improver)基于优化后的原始序列(corrected subreads)将组装出来的一致性序列(corrected consensus)进行circulator环化处理 [13],得到环化的细菌基因组(genome)。使用pbalign (BLASR, v0.4.1)分析基因组覆盖深度。该全基因组测序工作由北京奥维森基因科技有限公司完成。
2.3.2. 全基因组注释、分析及圈图绘制
使用Glimmer (v3.02)进行基因组注释 [14],通过RNAmmer (v1.2)鉴定核糖体RNA基因,通过tRNAscan-SE (v2.0)验证tRNA基因。通过CRISPRfinder (v20170509)预测全基因组中的正向重复序列和间隔区。通过PhiSpy (http://phaster.ca)预测基因组中存在的原噬菌体。通过IslandViewer 4 (http://www.pathogenomics.sfu.ca/islandviewer)预测基因组中存在的基因岛。对美国国立生物技术信息中心(NCBI)非冗余数据库NR、Swissprot数据库、代谢通路数据库KEGG (kyoto encyclopedia of genes and genomes)、蛋白质功能数据库COG (clusters of orthologous groups)和基因本体论数据库GO (Gene Ontology)进行BLAST全基因组搜索,然后利用diamond软件 [15] 进行比对,取e < 1e−5的注释,筛选具有最高序列相似性的蛋白质,得到功能注释信息。采用Circos软件绘制该基因组的圈图,在圈图上展示GC偏移、GC含量、COG基因注释、编码基因注释信息。
2.3.3. 耐药和毒力基因的注释
利用CARD抗生素耐药基因数据库(comprehensive antibiotic Resistance database, https://card.mcmaster.ca/)进行PA32菌株全基因组的耐药基因筛选 [16]。利用VFDB病原细菌毒力因子数据库(virulence factors database, http://www.mgc.ac.cn/VFs/main.htm)进行PA32菌株全基因组的毒力基因筛选 [17]。
3. 结果与分析
3.1. 耐酸实验结果
具有耐酸能力是细菌在人胃中生存的必要条件。为检测PA32菌株的耐酸能力,测定了其在酸性环境中的生存率(图1)。结果表明,暴露于酸性环境1 h内,PA32菌株的生存率迅速下降;然而,在暴露1 h后,生存率能够保持相对稳定。在pH = 2.0的条件下,PA32菌株的生存率在2 h后稳定在65.0%左右。表明柠檬双面神菌具有耐酸能力,能够在胃内高酸环境下生存。
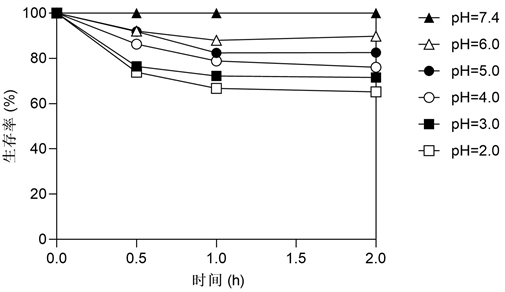
Figure 1. Survival rates of J. melonis PA32 exposed to acidic pH levels
图1. 暴露于酸性环境的PA32菌株的生存率
3.2. 药敏试验结果
经纸片扩散法法检测PA32菌株对4种药物的敏感性。结果显示,在甲硝唑作用下,培养基上无抑菌环出现;在克拉霉素、四环素、左氧氟沙星作用下,抑菌环直径分别为32.1 mm、31.7 mm和28.6 mm。上述结果表明,该菌株对甲硝唑耐药,对克拉霉素、四环素、左氧氟沙星均敏感(表1)。
Table 1. Drug susceptibility test results of the PA32 strain
表1. PA32菌株的药敏试验结果
3.3. 全基因组结构与分析
PA32菌株全基因组经三代Pacbio测序技术,共获得180,523条Reads,N50 Read Length为9855 bp,基因组测序深度为400.0X。经过组装和矫正,获得一条完整的基因组(染色体)序列(GenBank登录号:CP116501),未鉴定出质粒。基因组(染色体)序列大小为3,459,946 bp,鸟嘌呤–胞嘧啶(GC)含量为73.1%。测序得到的蛋白编码基因(CDs)数量有3296个,总长度为3,181,344 bp,占全基因组的91.95%;在非编码RNA (ncRNA)中含有tRNA基因48个,rRNA基因6个(其中5S rRNA基因2个,16S rRNA基因2个,23S rRNA基因2个);使用PhiSpy预测基因组中存在4个原噬菌体;经CRISPR finder预测发现菌株含有2组CRISPR相关序列;通过IslandViewer 4预测到PA32菌株基因组中有12个基因岛。使用Circos软件生成的基因组完成图如图2所示。
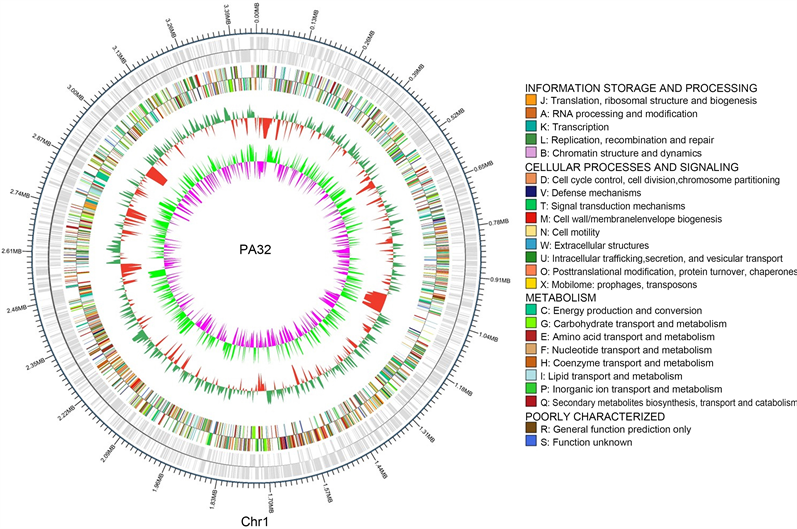 注:圈图从内到外依次为GC偏移(粉色:GC偏倚−;浅绿色:GC偏倚+)、GC含量(红色:GC含量−;绿色:GC含量+)、COG基因注释、编码基因注释。
注:圈图从内到外依次为GC偏移(粉色:GC偏倚−;浅绿色:GC偏倚+)、GC含量(红色:GC含量−;绿色:GC含量+)、COG基因注释、编码基因注释。
Figure 2. Circular genome map of the PA32 strain
图2. PA32菌株的全基因组圈图
3.4. COG数据库注释结果
结果显示,共注释到2459个蛋白,占全部基因的74.56%,分布于24个COG的条目中(图3)。其中,参与复制、重组和修复的基因有109个,占4.43%;参与翻译、核糖体结构和生物合成的基因有199个,占8.09%;与细胞壁、细胞膜、胞外被膜生物合成相关的基因有130个,占5.29%;参与氨基酸转运和代谢的基因有258个,占10.49%;与转录相关的基因有224个,占9.11%。
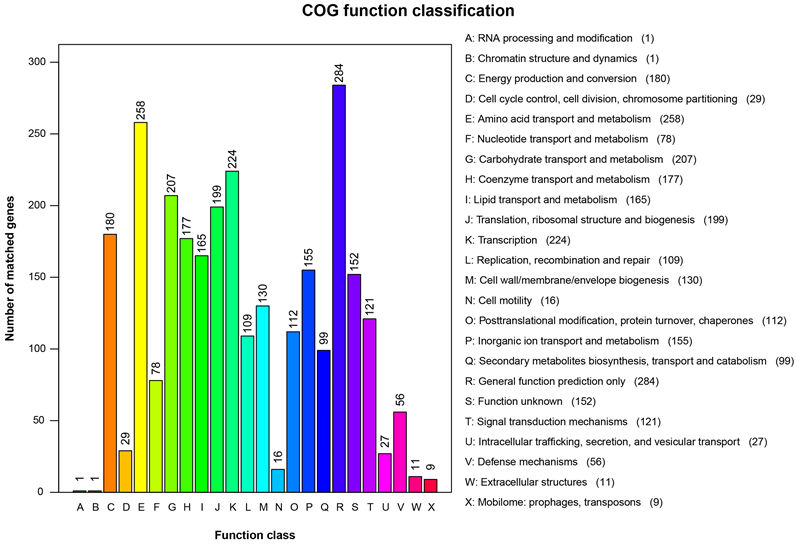
Figure 3. COG function classification of the PA32 strain
图3. PA32菌株的COG数据库功能分类图
3.5. 毒力基因注释结果
根据VFDB毒力因子数据库的注释,该菌株含有25个毒力相关基因,分布在17个类目中,其中有4个攻击性毒力基因,分别是3个黏附和(或)侵袭相关毒力基因和1个分泌系统相关毒力基因;和7个防御性毒力基因,分别是3个血清抗性和(或)免疫逃逸相关毒力基因和4个无氧呼吸相关毒力基因。攻击性和防御性毒力基因注释结果如表2所示。
Table 2. Virulence gene prediction of PA32 strain
表2. PA32菌株的毒力基因注释
3.6. 耐药基因注释结果
根据CARD综合抗生素研究数据库的注释,PA32菌株具有可介导对氨基糖苷类、大环内酯类、磺胺类、氟喹诺酮类、四环素类、β-内酰胺类等抗生素的耐药基因。耐药基因的作用机制包括抗生素靶点改变、抗生素外排、抗生素靶位保护、抗生素修饰导致失活、抗生素靶位替代和降低对抗生素的渗透性,PA32菌株的耐药基因及相对应抗菌药见表3。
Table 3. Resistance genes and corresponding antibiotics of PA32 strain
表3. PA32菌株的耐药基因及相对应抗菌药
4. 讨论
柠檬双面神菌PA32菌株是一株分离自青岛的胃癌临床分离株,这是从胃内分离出此细菌属的首次报道。本研究首先通过耐酸实验和药物敏感性实验,检测其耐酸能力和药物敏感性,然后利用三代测序技术平台Pacbio对该菌株高效并准确地进行全基因组测序,发现其含有1个环状染色体,对染色体基因组进行了基因注释分析,并对其毒力因子和耐药性进行了基因组层面分析。
生物体的生存取决于其适应不利环境条件的能力,耐酸、耐药和耐缺氧能力对胃内定植的细菌是至关重要的 [18]。本研究通过耐酸实验,证明了PA32菌株具有良好的耐酸能力,这为其在胃内高酸环境下生存提供了必要条件。药敏实验表明,PA32菌株具有对甲硝唑的耐药性,这可能有助于该菌株不被抗生素清除,从而在人体内长期定植。毒力因子分析表明,PA32菌株含有narG、narH、narI、narJ基因,它们在低氧条件时能够上调结核分枝杆菌(Mycobacterium tuberculosis)硝酸还原酶的表达水平,继而上调硝酸盐还原,在无氧代谢中发挥重要作用 [19]。以上结果说明PA32菌株具有在胃内生存的能力。
黏附在宿主细胞上有助于细菌从宿主获取营养物质,有利于定植,同时也是诱导宿主细胞损伤的途径之一,是一种关键的毒力决定因素。PA32菌株的全基因组毒力因子分析表明,它含有3个黏附和(或)侵袭相关的毒力因子,分别是EF-Tu、GroEL和plasmin receptor/GAPDH。其中,毒力因子EF-Tu,也称延长因子-Tu,被发现可以作为土拉弗朗西斯菌(Francisella tularensis)的配体与单核细胞样THP-1细胞表面核仁结合,并且可能促进细菌入侵宿主组织 [20] ;另一个毒力因子GroEL,被发现在艰难梭菌(Clostridium difficile)的肠道定植和中发挥重要作用 [21] ;而致病性血纤维蛋白溶酶受体/GAPDH可通过结合宿主咽部细胞的尿激酶纤溶酶原激活物受体,介导A组链球菌与人咽部细胞的黏附,进而在其定殖、内化和随后的增殖中发挥重要作用 [22]。这些黏附相关毒力因子有利于其在胃内的黏附和定植,提示柠檬双面神菌的潜在致病性。后续将进一步通过实验证实这些毒力因子的功能,探索其致病性及其机制。
全基因组耐药基因分析表明,同该菌种的其他菌株CD11-4和M714一样,PA32菌株具有对氨基糖苷类、大环内酯类、磺胺类、氟喹诺酮类、四环素类、β-内酰胺类等抗生素的耐药基因,且涉及抗生素靶点改变、抗生素外排和抗生素靶位保护等多种耐药机制 [5] [23]。然而,在药敏实验中PA32菌株却表现出了对大部分药物的敏感性,包括大环内酯类、四环素类和氟喹诺酮类。无独有偶,炎症性肠病肠道分离株CD11-4也对许多种类的抗生素敏感,包括氨基糖苷类、碳青霉烯类、青霉素类、氮酰胺类、氟喹诺酮类和黏菌素类等 [5]。柠檬双面神菌对大多数抗生素敏感可能是由于其较少从临床样本中报告,进而暴露于抗生素的机会较少,如果当它未来暴露于各种临床环境,它的遗传机制可能使其表现出耐药性。
5. 结论
通过全基因组三代测序和生物信息分析,本研究获得了具有较强耐酸能力的胃源性PA32菌株的全基因组序列和功能基因数据,并对PA32菌株的致病性和耐药性进行了基因组层面的分析,这为进一步探明其致病性和耐药性提供了重要的生物信息学依据。
基金项目
国家自然科学基金面上项目(基金编号:31870777)。
文章引用
李志鹏,王莉莉,张庆龙,董全江. 胃源性柠檬双面神菌的耐药和毒力全基因组分析
Whole Genome Sequencing of Janibacter melonis from the Stomach and Virulence and Antibiotic-Resistant Gene Analysis[J]. 临床医学进展, 2023, 13(03): 4037-4047. https://doi.org/10.12677/ACM.2023.133580
参考文献
- 1. Martin, K., Schumann, P., Rainey, F.A., et al. (1997) Janibacter limosus gen. nov., sp. nov., a New Actinomycete with Meso-Diaminopimelic Acid in the Cell Wall. International Journal of Systematic Bacteriology, 47, 529-534. https://doi.org/10.1099/00207713-47-2-529
- 2. Maaloum, M., Diop, K., Diop, A., et al. (2019) Description of Janibacter massiliensis sp. nov., Cultured from the Vaginal Discharge of a Patient with Bacterial Vaginosis. Antonie Van Leeuwenhoek, 112, 1147-1159. https://doi.org/10.1007/s10482-019-01247-x
- 3. Worodria, W. anderson, J., Cattamanchi, A., et al. (2011) The Role of Speciation in Positive Lowenstein-Jensen Culture Isolates from a High Tuberculosis Burden Country. PLOS ONE, 6, e27017. https://doi.org/10.1371/journal.pone.0027017
- 4. Fernández-Natal, M.I., Sáez-Nieto, J.A., Medina-Pascual, M.J., et al. (2015) First Report of Bacteremia by Janibacter terrae in Humans. Infection, 43, 103-106. https://doi.org/10.1007/s15010-014-0672-7
- 5. Chander, A.M., Kochhar, R., Dhawan, D.K., et al. (2018) Ge-nome Sequence and Comparative Genomic Analysis of a Clinically Important Strain CD11-4 of Janibacter melonis Iso-lated from Celiac Disease Patient. Gut Pathogens, 10, 2. https://doi.org/10.1186/s13099-018-0229-x
- 6. Lee, C.W., Rickman, B., Rogers, A.B., et al. (2008) Helicobacter pylori Eradication Prevents Progression of Gastric Cancer in Hypergastrinemic INS-GAS Mice. Cancer Research, 68, 3540-3548. https://doi.org/10.1158/0008-5472.CAN-07-6786
- 7. Shen, Z., Dzink-Fox, J., Feng, Y., et al (2022) Gastric Non-Helicobacter pylori Urease-Positive Staphylococcus epidermidis and Streptococcus salivarius Isolated from Hu-mans Have Contrasting Effects on H. pylori-Associated Gastric Pathology and Host Immune Responses in a Murine Model of Gastric Cancer. mSphere, 7, e0077221. https://doi.org/10.1128/msphere.00772-21
- 8. Wang, L., Xin, Y., Zhou, J., et al. (2020) Gastric Muco-sa-Associated Microbial Signatures of Early Gastric Cancer. Frontiers in Microbiology, 11, 1548. https://doi.org/10.3389/fmicb.2020.01548
- 9. Correa, P. (1992) Human Gastric Carcinogenesis: A Multistep and Multifactorial Process—First American Cancer Society Award Lecture on Cancer Epidemiology and Prevention. Cancer Research, 52, 6735-6740.
- 10. Wang, Z., Gao, X., Zeng, R., et al. (2020) Changes of the Gastric Mucosal Microbiome Associated with Histological Stages of Gastric Carcinogenesis. Frontiers in Microbiology, 11, 997. https://doi.org/10.3389/fmicb.2020.00997
- 11. Clinical and Laboratory Standards Institute (2018) Performance Standards for Antimicrobial Disk Susceptibility Testing. M02-A13.
- 12. Chin, C.S., Peluso, P., Sedlazeck, F.J., Nat-testad, M., et al. (2016) Phased Diploid Genome Assembly with Single-Molecule Real-Time Sequencing. Nature Meth-ods, 13, 1050-1054. https://doi.org/10.1038/nmeth.4035
- 13. Hunt, M., Silva, N.D., Otto, T.D., et al. (2015) Cir-clator: Automated Circularization of Genome Assemblies Using Long Sequencing Reads. Genome Biology, 16, 294. https://doi.org/10.1186/s13059-015-0849-0
- 14. Delcher, A.L., Bratke, K.A., Powers, E.C., et al. (2007) Identify-ing Bacterial Genes and Endosymbiont DNA with Glimmer. Bioinformatics, 23, 673-679. https://doi.org/10.1093/bioinformatics/btm009
- 15. Akhter, S., Aziz, R.K. and Edwards, R.A. (2012) PhiSpy: A Novel Algorithm for Finding Prophages in Bacterial Genomes That Combines Similarity and Composition-Based Strate-gies. Nucleic Acids Research, 40, e126. https://doi.org/10.1093/nar/gks406
- 16. Mistry, J., Finn, R.D., Eddy, S.R., et al. (2013) Challenges in Homology Search: HMMER3 and Convergent Evolution of Coiled-Coil Regions. Nucleic Acids Research, 41, e121. https://doi.org/10.1093/nar/gkt263
- 17. Chen, L., Yang, J., Yu, J., et al. (2005) VFDB: A Reference Database for Bacterial Virulence Factors. Nucleic Acids Research, 33, D325-D328. https://doi.org/10.1093/nar/gki008
- 18. Tanida, S., Joh, T., Itoh, K., et al. (2004) The Mechanism of Cleavage of EGFR Ligands Induced by Inflammatory Cytokines in Gastric Cancer Cells. Gastroenterology, 127, 559-569. https://doi.org/10.1053/j.gastro.2004.05.017
- 19. Smith, I. (2003) Mycobacterium tuberculosis Pathogenesis and Molecular Determinants of Virulence. Clinical Microbiology Reviews, 16, 463-496. https://doi.org/10.1128/CMR.16.3.463-496.2003
- 20. Barel, M., Hovanessian, A.G., Meibom, K., et al. (2008) A Novel Receptor-Ligand Pathway for Entry of Francisella tularensis in Monocyte-Like THP-1 Cells: Interaction between Surface Nucleolin and Bacterial Elongation Factor Tu. BMC Microbiology, 8, 145. https://doi.org/10.1186/1471-2180-8-145
- 21. Péchiné, S., Hennequin, C., Boursier, C., et al. (2013) Immunization Using GroEL Decreases Clostridium difficile Intestinal Colonization. PLOS ONE, 8, e81112. https://doi.org/10.1371/journal.pone.0081112
- 22. Seidler, K.A. and Seidler, N.W. (2013) Role of Extracellular GAPDH in Streptococcus pyogenes Virulence. Missouri Medicine, 110, 236-240.
- 23. Zhao, Q., Jin, M., Zhou, Z., et al. (2020) Complete Genome Sequence of Janibacter melonis M714, a Janus-Faced Bacterium with both Human Health Impact and Industrial Applications. Current Microbiology, 77, 1883-1889. https://doi.org/10.1007/s00284-020-01951-2
NOTES
*通讯作者。
