Statistics and Application
Vol.
09
No.
05
(
2020
), Article ID:
38415
,
9
pages
10.12677/SA.2020.95094
纵向数据下均值协方差模型的贝叶斯 统计诊断
徐登可1,赵远英2
1浙江农林大学统计系,浙江 杭州
2贵阳学院数学与信息科学学院,贵州 贵阳

收稿日期:2020年10月7日;录用日期:2020年10月22日;发布日期:2020年10月29日

摘要
研究了纵向数据下均值协方差模型的贝叶斯统计诊断。通过应用Gibbs抽样和Metropolis-Hastings (MH)算法相结合的混合算法获得模型贝叶斯数据删除影响诊断统计量来识别数据异常点。模拟研究和实例分析都表明所提出的诊断方法是可行有效的。
关键词
纵向数据,数据删除,Gibbs抽样,MH算法,贝叶斯诊断
Bayesian Statistical Diagnosis of Joint Mean and Covariance Models with Longitudinal Data
Dengke Xu1, Yuanying Zhao2
1Department of Statistics, Zhejiang Agriculture and Forestry University, Hangzhou Zhejiang
2College of Mathematics and Information Science, Guiyang University, Guiyang Guizhou
Received: Oct. 7th, 2020; accepted: Oct. 22nd, 2020; published: Oct. 29th, 2020

ABSTRACT
Bayesian statistical diagnosis of joint mean and covariance models with longitudinal data is studied. By combining the Gibbs sampler and Metropolis-Hastings algorithm, the Bayesian case deletion diagnosis statistic is obtained to identify data outliers. Simulation study and a real data analysis show that the proposed diagnosis method is feasible and effective.
Keywords:Longitudinal Data, Case Deletion, Gibbs Sampler, Metropolis-Hastings Algorithm, Bayesian Diagnosis

Copyright © 2020 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
纵向数据常常出现在经济学,生物学,环境科学,医学等领域。此类数据是指同一组受试个体在不同时间点上的重复观测数据,它具有组间独立,组内相关的特性。因此,在纵向数据分析中协方差矩阵的估计是很重要的,并且对协方差矩阵进行估计也是统计学家感兴趣的。众所周知,一个好的协方差矩阵估计是提高回归系数估计效率的常用方法。而对协方差矩阵进行估计的方法一般都是基于Cholesky分解建模展开的。目前已有很多作者基于此分解研究了协方差矩阵的估计问题。其中,Pourahmadi [1] [2] 对协方差矩阵进行改进的Cholesky分解且对分解后的部分进行广义线性建模,然后考虑了模型中的参数估计问题。这种建模的主要优点包括便于统计意义上的解释和参数估计中的方便计算。类似的研究还可见文献 [3] [4]。另外,Rothman等 [5] 提出了通过新的改进的Cholesky分解来参数化协方差矩阵自身,给出了分解后因子的新的回归解释,并且也确保了估计得到的协方差矩阵的正定性。进一步地,基于这种新的改进的Cholesky分解,Zhang和Leng [6] 针对联合均值协方差建模提出了一种有效的极大似然估计方法。Xu等 [7] 基于惩罚极大似然方法提出了一种有效的变量选择方法。
另一方面,异常点的识别是统计诊断的主要内容,而基于数据删除影响进行统计诊断是识别异常点的一个重要方法。自从Cook [8] 在这一领域做了开创性的工作之后,数据删除影响诊断方法,尤其是贝叶斯数据删除影响诊断方法引起越来越多的关注。例如,Cho等 [9] 在一般模型的框架下,以复杂生存数据模型为例提出一种简单可行的贝叶斯数据删除K-L距离计算公式。其它相关内容还可以参见文献 [10] [11] [12]。尽管已经有众多的作者研究贝叶斯数据删除影响诊断方法,也做出了卓有成效的工作。然而,很少有文献基于新的改进的Cholesky分解研究纵向数据下均值协方差模型的贝叶斯数据删除影响诊断问题。因此针对纵向数据下均值协方差模型,本文基于K-L距离研究贝叶斯数据删除影响诊断方法,以识别模型的异常点。
2. 模型与符号
2.1. 纵向数据下均值协方差模型
假设有n个独立样本和对第i个样本进行 次重复观测。具体地,记第i个个体在时间 的响应变量向量为 ,并且假设响应变量服从正态分布 ,其中 是一个( )向量和 是一个( )正定矩阵。
其中基于改进的Cholesky分解,Pourahmadi [1] 首先提出分解 为 ,其中 是下三角矩阵,对角线元素为1,下三角元素是自回归模型 中的自回归参数 的负数。 的对角元素是新息方差 。
而Rothman等 [5] 提出了新的分解思想,令 ,它是对角线元素为1的下三角矩阵,因此可以写成 。实际上也就是利用Rothman等 [5] 提出的改进的Cholesky分解来表示协方差矩阵,这样就可以用新的统计意义解释。 中的元素 可以解释为以下滑动平均模型的滑动平均系数: 。其中 和 ,且 。注意到这里的 和 是没有限制的。基于改进的Cholesky分解和受到Pourahmadi [1] [2],Ye和Pan [13] 的启发,无限制的参数 和 可以用以下线性模型来建模
(1)
其中
和 分别是
分别是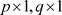 和
和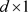 协变量向量。协变量
协变量向量。协变量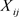 和
和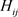 是回归分析中的一般协变量,
是回归分析中的一般协变量, 一般可以取成时间差
一般可以取成时间差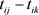 的多项式,即,
的多项式,即,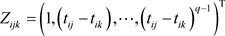 。另外,记
。另外,记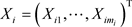 和
和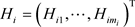 。进一步记
。进一步记 为滑动平均系数和
为滑动平均系数和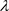 为新息系数。
为新息系数。
由模型(1),可以获得如下似然函数
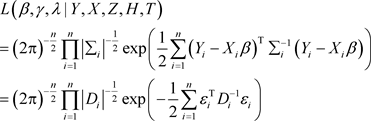 (2)
(2)
其中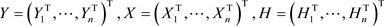 且
且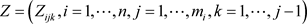 。
。
2.2. K-L距离
K-L距离也叫K-L信息,具有距离和信息的某些性质,在统计上以反映两个模型或分布的差异而著
称。根据韦博成 [14] 对K-L距离的介绍,密度函数为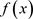 与
与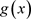 的两个分布的K-L距离
的两个分布的K-L距离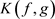 被定义为:
被定义为: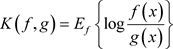 ,其中
,其中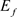 表示对
表示对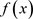 求期望。
求期望。
3. 贝叶斯统计诊断
3.1. 先验分布
为了应用贝叶斯方法来估计模型(1)中的未知参数,需要具体化未知参数的先验分布。为了简便,假
设 和
和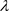 相互独立且具有正态先验分布,分别为
相互独立且具有正态先验分布,分别为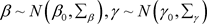 和
和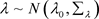 ,其中假设超参数
,其中假设超参数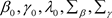 和
和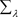 是已知的。
是已知的。
3.2. Gibbs抽样和条件分布
基于式子(2),我们可以按照以下过程用Gibbs抽样从后验分布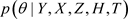 中进行抽样,其中
中进行抽样,其中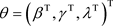 。
。
步骤1. 令参数的初值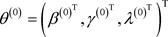 。
。
步骤2. 基于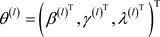 ,计算
,计算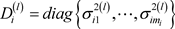 和
和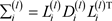 。
。
步骤3. 基于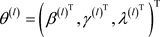 按照以下抽取
按照以下抽取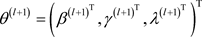 ;
;
· 抽样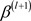 :
:
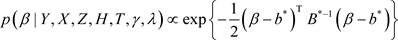 , (3)
, (3)
其中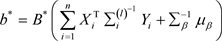 和
和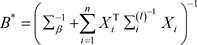 。
。
· 抽样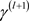 :
:
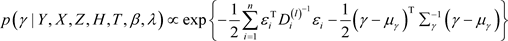 (4)
(4)
· 抽样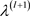 :
:
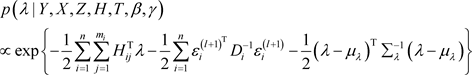 (5)
(5)
步骤4. 重复步骤2和3。
那么这样就通过以上算法产生了样本序列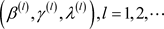 ,从(3)~(5)式中很容易发现,条件分布
,从(3)~(5)式中很容易发现,条件分布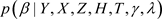 是熟悉的正态分布。从正态分布抽取随机数是比较容易的。但是条件分布
是熟悉的正态分布。从正态分布抽取随机数是比较容易的。但是条件分布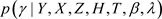 和
和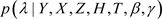 是一些不熟悉且相当复杂的分布,如何从这些分布中抽取随机数也变得相当困难。这样,MH算法就被应用来从这些分布中抽取随机数。选择正态分布
是一些不熟悉且相当复杂的分布,如何从这些分布中抽取随机数也变得相当困难。这样,MH算法就被应用来从这些分布中抽取随机数。选择正态分布 和
和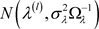 作为建议分布,其中通过选择
作为建议分布,其中通过选择 和
和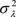 ,来使得接受概率在0.25与0.45之间,且取
,来使得接受概率在0.25与0.45之间,且取
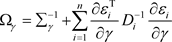 ,
,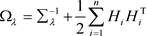 .
.
3.3. 贝叶斯估计
利用以上提出的计算过程来产生观测值来获得参数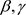 和
和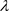 的贝叶斯估计。令
的贝叶斯估计。令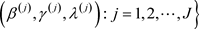
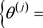 是通过上述混合算法从联合条件分布
是通过上述混合算法从联合条件分布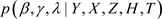 中产生的观测值,那么
中产生的观测值,那么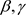 和
和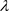 的贝叶斯估计为:
的贝叶斯估计为:
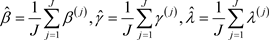
类似于Geyer [15] 中展示的一样,当J趋于无穷时,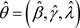 是对应后验均值向量的相合估计。
是对应后验均值向量的相合估计。
3.4. 贝叶斯数据删除影响诊断
在贝叶斯统计分析中,目前已存在许多诊断统计量用以评价个体观测对参数后验分布的影响,本文主要基于K-L距离研究贝叶斯数据删除影响的统计诊断方法。对任意的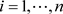 ,记
,记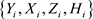 是第i个个体观测数据点,
是第i个个体观测数据点,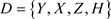 为完全数据集,
为完全数据集,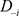 为完全数据集D删除第i个个体观测数据点得到的数据集,
为完全数据集D删除第i个个体观测数据点得到的数据集,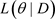 与
与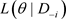 分别表示基于数据D与
分别表示基于数据D与 的似然函数,则
的似然函数,则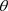 于数据D与
于数据D与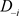 的后验分布分别为
的后验分布分别为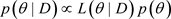 ,
,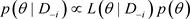 。根据Cho等 [9] 的讨论,定义K-L距离为:
。根据Cho等 [9] 的讨论,定义K-L距离为:
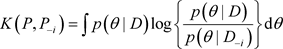 , (6)
, (6)
其中P与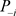 分别表示
分别表示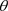 基于数据D与
基于数据D与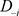 的后验分布,注意到K-L距离
的后验分布,注意到K-L距离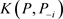 是完全数据集D删除第i个数据点前后对参数
是完全数据集D删除第i个数据点前后对参数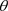 后验分布影响的一种很好的度量。经过简单的计算(6)式变为:
后验分布影响的一种很好的度量。经过简单的计算(6)式变为:
 , (7)
, (7)
其中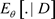 表示
表示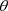 基于数据D的后验期望。由Gibbs抽样算法抽取的随机观测序
基于数据D的后验期望。由Gibbs抽样算法抽取的随机观测序 ,可以得到K-L距离
,可以得到K-L距离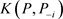 的估计为:
的估计为:
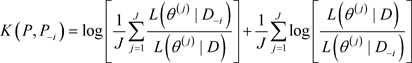 . (8)
. (8)
对任意的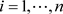 ,当
,当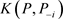 很大时,可以诊断第i个个体观测数据点为异常点。
很大时,可以诊断第i个个体观测数据点为异常点。
4. 模拟研究
在这部分通过模拟研究来说明前面提出的贝叶斯统计诊断方法的有效性。我们选择均值参数,滑动
平均参数和新息参数的真实值分别为 ,
,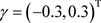 和
和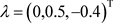 。在联合模型中
。在联合模型中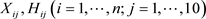 分别是
分别是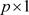 和
和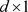 的协变量向量,其中的元素独立的产生于标准正态分布
的协变量向量,其中的元素独立的产生于标准正态分布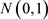 。选取
。选取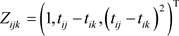 ,其中测量时间
,其中测量时间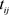 产生于均匀分布
产生于均匀分布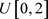 。利用这些值,均值
。利用这些值,均值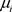 和协方差矩阵
和协方差矩阵 可以通过前面描述的改进的Cholesky分解来构建。响应变量
可以通过前面描述的改进的Cholesky分解来构建。响应变量 就可以从多元正态分布
就可以从多元正态分布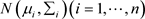 中产生。在这里取样本量
中产生。在这里取样本量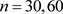 。
。
为了调查贝叶斯诊断方法对先验分布的敏感程度,考虑以下有关未知参数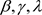 的先验分布中超参数值的设置的两种情形:
的先验分布中超参数值的设置的两种情形:
TypeI: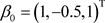 ,
,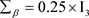 ,
,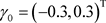 ,
, ,
,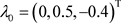 ,
,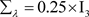 。这种设置具有很好的先验信息。
。这种设置具有很好的先验信息。
TypeII: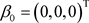 ,
,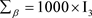 ,
,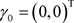 ,
, ,
,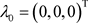 ,
, 。这些超参数值的设置代表的是没有先验信息的情况。
。这些超参数值的设置代表的是没有先验信息的情况。
为了在数据集中产生异常点,分别在第3和19个个体观测数据点的响应变量(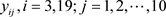 )都加1.2构成人工数据集D。然后对人工数据集D应用本文介绍的影响诊断方法来检测影响观测。其中MCMC算法的收敛性可以通过EPSR值来检验,并且发现在3000次迭代以后EPSR值都小于1.2。因此在计算中丢掉前3000次迭代以后再收集J = 2000个随机样本来通过(8)式计算K-L距离
)都加1.2构成人工数据集D。然后对人工数据集D应用本文介绍的影响诊断方法来检测影响观测。其中MCMC算法的收敛性可以通过EPSR值来检验,并且发现在3000次迭代以后EPSR值都小于1.2。因此在计算中丢掉前3000次迭代以后再收集J = 2000个随机样本来通过(8)式计算K-L距离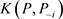 。图1和图2报道了相应的诊断结果。正如预期的一样,通过图1和图2很容易就发现,第3和19个个体观测数据点被诊断为异常点,且诊断方法对先验分布超参数取值的选取不敏感。
。图1和图2报道了相应的诊断结果。正如预期的一样,通过图1和图2很容易就发现,第3和19个个体观测数据点被诊断为异常点,且诊断方法对先验分布超参数取值的选取不敏感。
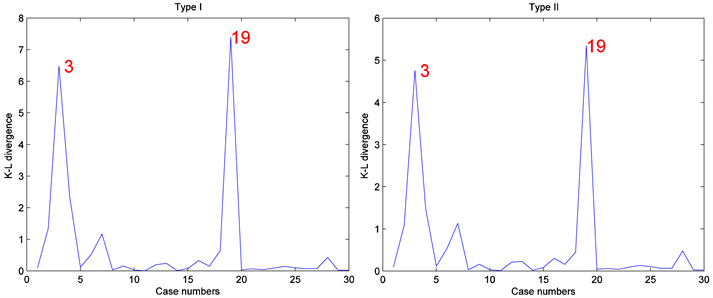
Figure 1. Results based on Bayesian case deletion diagnosis when n = 30
图1. 当n = 30时,贝叶斯数据删除影响诊断的数值结果
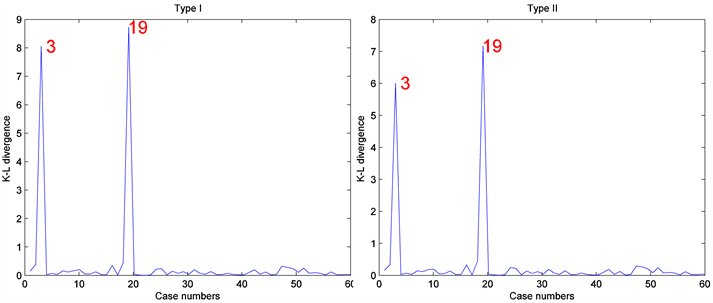
Figure 2. Results based on Bayesian case deletion diagnosis when n = 60
图2. 当n = 60时,贝叶斯数据删除影响诊断的数值结果
5. 实例分析
在这部分把提出的方法应用到牛生长数据。Kenward [16] 将牛实验随机的分为A,B两组,并分别记录它们的体重。组A中的30只动物接受A治疗,另外B组的30只动物接受B治疗。在133天的实验过程中,记录了11次动物的体重。采用Pourahmadi [1] [2] 中提出的联合建模模型,运用提出的贝叶斯方法分析A组的数据。图3显示了A组牛数据的曲线和多项式拟合曲线图。从图3中可观测在整个实验过程中,相应变量的均值(体重)都在增加,特别是在研究最初的几个星期,增长速度较快。从图3进一步可以看出,均值变量与时间变量不是线性的,因此牛群均值生长模型使用关于时间变量的二次或者三次模型更加合理。基于上述分析以及Pourahmadi [1] [2] 的研究,提出采用关于时间变量的三次模型分别对均值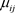 ,移动平均参数
,移动平均参数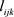 和对数新息方差
和对数新息方差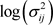 进行建模。具体如下:
进行建模。具体如下:
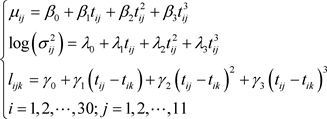
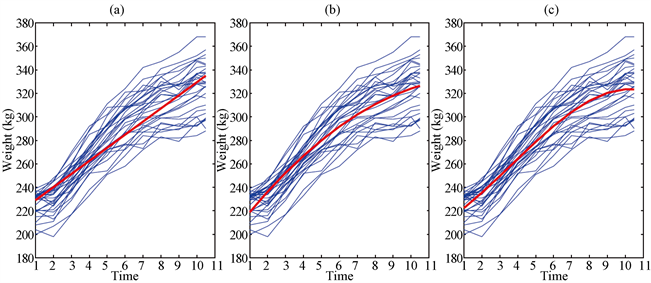
Figure 3. Plot for the cattle data and the thicker line is the polynomial fitted curve: (a) linear polynomial fitted curve; (b) quadratic polynomial fitted curve and (c) cubic polynomial fitted curve
图3. 牛数据和粗线表示多项式拟合曲线:(a) 线性多项式拟合曲线;(b) 二次多项式拟合曲线;(c)三次多项式拟合曲线
由于贝叶斯统计方法对超参数取值的选取并不敏感,因此在此选取无信息先验信息分布。在MH算法中,我们令建议分布中的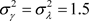 ,并且使得接受概率分别为29.60%和30.22%。为了测试算法的收敛性,画出了所有未知参数的EPSR值的图,具体在图4中展示,从图中也能看出3000次迭代以后所有参数的EPSR值都小于1.2,这也表示3000次迭代以后算法都收敛了。因此我们摒弃所有参数的前3000次迭代的迭代值,收集3000次迭代以后的2000个迭代样本,然后通过(8)估计贝叶斯诊断统计量K-L距离
,并且使得接受概率分别为29.60%和30.22%。为了测试算法的收敛性,画出了所有未知参数的EPSR值的图,具体在图4中展示,从图中也能看出3000次迭代以后所有参数的EPSR值都小于1.2,这也表示3000次迭代以后算法都收敛了。因此我们摒弃所有参数的前3000次迭代的迭代值,收集3000次迭代以后的2000个迭代样本,然后通过(8)估计贝叶斯诊断统计量K-L距离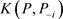 ,图5给出了相应的诊断结果。由图5知第7个和第25个个体观测数据点可诊断为异常点。
,图5给出了相应的诊断结果。由图5知第7个和第25个个体观测数据点可诊断为异常点。
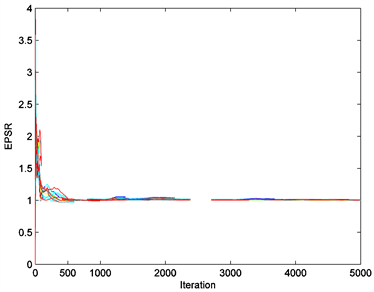
Figure 4. EPSR values of all parameters in the cattle data
图4. 牛数据中所有未知参数的EPSR值
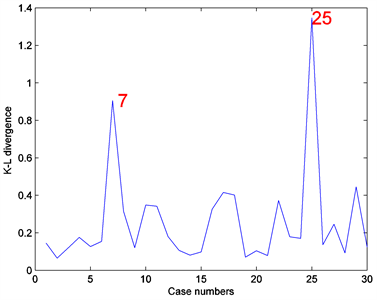
Figure 5. Statistical diagnosis results of the cattle data
图5. 牛数据统计诊断的数值结果
6. 结论
本文针对纵向数据下均值协方差模型,基于Gibbs抽样和MH算法相结合的混合算法,以及后验分布之间的K-L距离研究贝叶斯数据删除影响的统计诊断方法。模拟研究和实例分析都显示了模型与方法的可行性和有效性。
基金项目
浙江省高校重大人文社科攻关计划项目资助(2018QN037)。
文章引用
徐登可,赵远英. 纵向数据下均值协方差模型的贝叶斯统计诊断
Bayesian Statistical Diagnosis of Joint Mean and Covariance Models with Longitudinal Data[J]. 统计学与应用, 2020, 09(05): 900-908. https://doi.org/10.12677/SA.2020.95094
参考文献
- 1. Pourahmadi, M. (1999) Joint Mean-Covariance Models with Applications to Longitudinal Data: Unconstrained Parameterization. Biometrika, 86, 677-690.
https://doi.org/10.1093/biomet/86.3.677 - 2. Pourahmadi, M. (2000) Maximum Likelihood Estimation for Generalised Linear Models for Multivariate Normal Covariance Matrix. Biometrika, 87, 425-435.
https://doi.org/10.1093/biomet/87.2.425 - 3. Pan, J.X. and MacKenzie, G. (2003) On Modelling Mean-Covariance Structures in Longitudinal Studies. Biometrika, 90, 239-244.
https://doi.org/10.1093/biomet/90.1.239 - 4. Mao, J. and Zhu, Z.Y. (2011) Joint Semiparametric Mean-Covariance Model in Longitudinal Study. Science China Mathematics, 54, 145-164.
https://doi.org/10.1007/s11425-010-4078-4 - 5. Rothman, A.J., Levina, E. and Zhu, J. (2010) A New Approach to Cholesky-Based Covariance Regularization in High Dimensions. Biometrika, 97, 539-550.
https://doi.org/10.1093/biomet/asq022 - 6. Zhang, W.P. and Leng, C.L. (2012) A Moving Average Cholesky Factor Model in Covariance Modeling for Longitudinal Data. Biometrika, 99, 141-150.
https://doi.org/10.1093/biomet/asr068 - 7. Xu, D.K., Zhang, Z.Z. and Wu, L.C. (2013) Joint Variable Selection of Mean-Covariance Model for Longitudinal Data. Open Journal of Statistics, 3, 27-35.
https://doi.org/10.4236/ojs.2013.31004 - 8. Cook, R.D. (1977) Detection of Influential Observations in Linear Regression. Technometrics, 19, 15-18.
https://doi.org/10.1080/00401706.1977.10489493 - 9. Cho, H., Ibrahim, J.G., Sinha, D. and Zhu, H.T. (2009) Bayesian Case Influence Diagnostics for Survival Models. Biometrics, 65, 116-124.
https://doi.org/10.1111/j.1541-0420.2008.01037.x - 10. 赵远英, 徐登可, 庞一成. 联合均值与方差模型的Bayes分析[J]. 高校应用数学学报, 2018, 33(2): 241-252.
- 11. 戴琳, 陶治, 吴刘仓. 联合均值与方差模型的统计诊断[J]. 统计与信息论坛, 2017, 32(1): 14-19.
- 12. Tang, N.S. and Duan, X.D. (2012) A Semiparametric Bayesian Approach to Generalized Partial Linear Mixed Models for Longitudinal Data. Computational Statistics and Data Analysis, 56, 4348-4365.
https://doi.org/10.1016/j.csda.2012.03.018 - 13. Ye, H.J. and Pan, J.X. (2006) Modelling of Covariance Structures in Generalized Estimating Equations for Longitudinal Data. Biometrika, 93, 927-941.
https://doi.org/10.1093/biomet/93.4.927 - 14. 韦博成. 参数统计教程[M]. 北京: 高等教育出版社, 2006.
- 15. Geyer, C.J. (1992) Practical Markov Chain Monte Carlo. Statistical Science, 7, 473-511.
https://doi.org/10.1214/ss/1177011137 - 16. Kenward, M.G. (1987) A Method for Comparing Profiles of Repeated Measurements. Applied Statistics, 36, 296-308.
https://doi.org/10.2307/2347788