Hans Journal of Computational Biology
Vol.
12
No.
03
(
2022
), Article ID:
55513
,
8
pages
10.12677/HJCB.2022.123005
基于分子动力学模拟研究YTHDF3与m6A修饰RNA的相互作用动力学及关键位点
周文雪,韩中杰,吴志祥,李春华*
北京工业大学环境与生命学部,北京
收稿日期:2022年7月26日;录用日期:2022年8月26日;发布日期:2022年9月5日

摘要
YTHDF3 (YTH domain family protein 3)能够特异性识别N6-甲基腺嘌呤(N6-methylladenosine, m6A)修饰的RNA,参与调控mRNA加工代谢、正常生理和异常病理等重要生命进程,与许多癌症息息相关。研究YTHDF3与m6A修饰RNA的相互作用动力学及关键位点具有重要意义。在本工作中,采用分子动力学(Molecular dynamics, MD)模拟方法研究了YTHDF3单体及与甲基化RNA复合物的动力学性质。研究发现,甲基化RNA的结合使得YTHDF3结构更加紧凑,构象更加稳定。运动相关性分析揭示了YTHDF3上负责结合甲基化RNA的重要区域。最后,构造运动相关性加权的复杂网络,识别了参与m6A修饰特异性识别的关键残基,与实验结果高度吻合。另外,还识别了一些远离结合界面但对结构稳定发挥重要功能的残基。本研究有助于理解YTHDF3与m6A修饰RNA之间的相互作用和关键位点,可为靶向药物设计提供重要信息。
关键词
YTHDF3,分子动力学,关键残基

Study on the Interacting Dynamics and Key Residues between YTHDF3 and m6A-Mediated RNA Based on Molecular Dynamics Simulation
Wenxue Zhou, Zhongjie Han, Zhixiang Wu, Chunhua Li*
Faculty of Environmental and Life Sciences, Beijing University of Technology, Beijing
Received: Jul. 26th, 2022; accepted: Aug. 26th, 2022; published: Sep. 5th, 2022

ABSTRACT
YTHDF3 (YTH domain family protein 3) plays an important role in regulating mRNA processing, metabolism, normal physiology and abnormal pathology, which is associated with a variety of cancers. Investigating the interacting dynamics and key residues between YTHDF3 and m6A-mediated RNA is of important significance. Here, the molecular dynamics (MD) simulation is utilized to analyze the dynamic properties of YTHDF3 on apo and bound to methylated RNA states. The results reveal that upon RNA binding, the structure and the conformation of YTHDF3 become more compact and stable. The dynamical cross-correlated map analysis shows the important regions on YTHDF3 for the binding of methylated RNA. Finally, the covariance matrix is weighted to protein structure network for analyzing the key residues associated with specific interaction of m6A-modified RNA, which is consistent with the experimental information. In addition, we also find the residues which are far away from the binding interface but critical for the structural stability of YTHDF3. This study is helpful for the understanding of the interaction and key residues of YTHDF3 and m6A-mediated RNA, and can provide important information for the related drug design.
Keywords:YTHDF3 (YTH Family Protein 3), Molecular Dynamics, Key Residues

Copyright © 2022 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
m6A (N6-methylladenosine)是真核生物信使RNA(mRNA)上存在最为广泛的化学修饰,在调节大脑发育和免疫反应等许多生物过程中发挥重要作用。含有YT521-B(YTH)结构域的蛋白质能够特异性识别m6A,被称作Reader [1]。作为最后被结晶的YTH蛋白,YTHDF3拥有类似于其他成员特异性识别m6A的结合表面和芳香笼 [2]。然而,YTHDF3识别m6A的分子基础还未解释清楚。考虑到YTHDF3在癌症进展中的重要性,探索YTHDF3与m6A修饰RNA的结合动力学,并识别结合过程中发挥关键作用的残基,对于了解其分子机制和开发YTHDF3特异性抑制剂具有重要意义 [3]。
m6A修饰的RNA与YTHDF3中YTH结构域复合的2.7 Å分辨率晶体结构 [2],由4个α螺旋和4个β片层组成。β1与β2、β4平行,与β3反平行共同形成β桶形结构,周围4个α螺旋构成其疏水核心 [4]。RNA被紧紧结合在由α1、loop2、loop4、loop5和loop6形成的疏水性口袋中,其中loop5也被称为识别环(残基Lys480-Lys500) [2]。细节上,m6A位于由3个芳香残基组成的芳香笼中,分别由来自loop5的Trp492构成基底,来自loop2的Trp438和来自loop5的Trp497构成墙壁。Trp438和Trp492在YTH结构域蛋白家族中绝对保守,其丙氨酸突变完全破坏了YTHDF3与m6A修饰RNA的特异性结合 [5]。
YTH结构域蛋白受到了实验和理论研究者的广泛关注。在实验方面,Xu等人进行了等温滴注量热法分析,发现无论RNA长度如何,人类和酵母的YTH结构域家族蛋白都可以识别含m6A的RNA,这意味着YTH结构域是一个进化保守的依赖于m6A修饰的RNA结合结构域 [6]。此外,Wang等人利用液相色谱-串联质谱方法发现YTHDF3与甲基化RNA的结合亲和力比未甲基化RNA高5倍 [7]。在理论方面,Li等人在2020年通过对YTHDF1,YTHDF2和YTHDF3进行MD模拟,发现YTHDF3与YTHDF1和YTHDF2具有相似的内在可塑性和蛋白质动力学性质 [2]。同年,Li等人利用炼金术自由能模拟方法研究了甲基化对于YTHDC1特异性识别RNA的重要性 [8]。以上研究为YTH结构域对m6A修饰RNA的特异性识别提供了部分线索。然而,还未有人开展YTHDF3与m6A修饰RNA复合物的动力学研究,关于YTHDF3与RNA之间的分子识别、构象变化及其涉及的关键残基还需进一步研究。
本工作对YTHDF3在单体和其结合m6A修饰RNA的复合物分别进行分子动力学模拟,研究了结合m6A修饰RNA对YTHDF3动力学行为的影响,并结合复杂网络方法识别了YTHDF3中参与m6A修饰特异性识别及构象变化的关键残基。
2. 研究方法
2.1. 分子动力学模拟
从蛋白质数据库(Protein data bank, PDB)中获取人源YTHDF3与m6A修饰RNA结合的复合物晶体结构 [2] (PDB ID: 6ZOT),保留其核心结构域(Arg417-Phe553)。本文的MD模拟包含2个体系:移除RNA,保留蛋白质的apo_pro单体体系和保留蛋白质-RNA的m6A_com复合物体系。将两个模拟体系分别溶剂化,采用TIP3P水模型 [9] 并添加0.15 mol/L的NaCl保持整个体系呈电中性。采用最陡下降法约束模拟体系的重原子位置,在最大约束力达到1000 kJ∙mol−1∙nm−1后便停止优化。而后在恒温恒容(NVT)条件下进行1 ns的模拟。最后去除所有约束条件,将整个体系在恒温恒压(NPT)条件下进行500 ns的平衡分子动力学模拟。所有的MD模拟均采用GROMACS 2020软件包 [10] 完成,模拟温度保持在300 K,压强维持在1 bar。采用CHARMM36全原子力场 [11],为了参数化m6A,使用附加的CHARMM力场 [12]。范德华相互作用通过一个双截断半径(10 Å和12 Å)函数进行计算,长程静电相互作用采用particle-mesh Ewald (PME)方法计算 [13],在模拟过程中采用周期性边界条件,积分步长为2 fs,对所有键均用LINCS方法 [14] 约束。
2.2. 复杂网络模型
基于蛋白质-RNA结构,以残基为节点(Cα和C5’原子分别代表氨基酸和核苷酸)和连接节点的边构建加权的复杂网络。其中,两个节点之间的距离在截断距离内,则认为有相互作用用边连接,反之则不连 [15]。对于蛋白质、RNA以及界面节点间的截断距离分别设置为7、13和10 Å。节点i和节点j之间边的权重wij是由MD平衡轨迹的运动相关性关系Cij计算得到,其可以衡量信息通过该边的概率。
(1)
进而计算加权网络的特征路径长度(CPL)及衡量特征路径长度变化的Z-score:
(2)
其中,ΔCPLk是移除节点k后CPL的变化, 代表的是所有节点依次移除后CPL变化的平均值, 代表的是相应的标准差。CPL的改变指依次移除每个残基观察其对网络CPL变化的影响,它反映了该节点对网络通信的影响,因此Z-score值较大的为介导变构信号传导的功能性关键残基。
3. 结果与讨论
3.1. m6A修饰RNA结合对YTHDF3动力学行为的影响
根据系统的均方根偏差(root mean square deviation, RMSD)随时间的变化,可知50 ns后apo_pro和m6A_com系统均达到平衡状态,因此,所有分析均基于450 ns后的平衡轨迹。在平衡阶段,apo_pro体系有较大的波动RMSD = 1.90 ± 0.16 Å,而m6A_com中蛋白质波动则相对稳定RMSD = 2.23 ± 0.12 Å,这表明甲基化RNA结合使得YTHDF3结构更稳定。同时绘制了YTHDF3在结合甲基化RNA前后的残基均方根涨落,如图1(a)所示。从图1(a)中,可以发现甲基化RNA结合使得YTHDF3的loop1,loop4,识别环和loop6区域柔性明显下降,这表明RNA的结合限制了这些区域的灵活性,促使YTHDF3的构象更加稳定,与RMSD的分析具有一致性。此外,为了揭示YTHDF3在结合甲基化RNA前后结构的变化,计算能够反映分子紧凑程度的回转半径,可知相较于YTHDF3单体(14.15 ± 0.12 Å),甲基化RNA的结合使复合物(14.02 ± 0.08 Å)变得更紧凑,暗示着甲基化RNA结合导致YTHDF3的结合口袋产生闭合构象,进而可以解释m6A_com中蛋白的RMSD平均值高于apo_pro的原因。为了便于理解,分别从apo_pro和m6A_com的平衡轨迹中提取平均构象并进行叠落,如图1(b)所示。从图1(b)中,可以发现相较于YTHDF3的单体,甲基化RNA结合后使得YTHDF3上的loop1,识别环,loop4和loop6均向中间靠拢,结构更为紧凑,与回转半径的分析结果具有一致性。
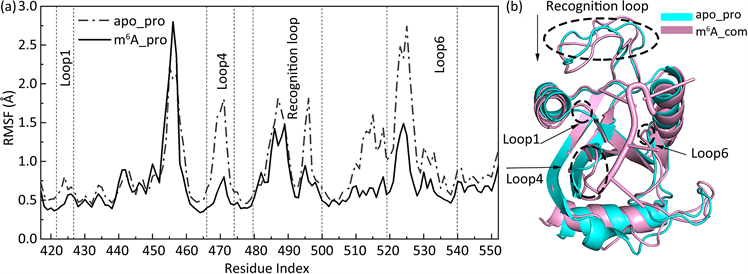
Figure 1. (a) Root mean square fluctuations (RMSFs) of YTHDF3 in apo (dotted line) and methylated RNA-bound (solid line) states. (b) Comparison of average conformation from the equilibrium trajectories of apo_pro (cyan) and m6A_com (pink) systems
图1. (a) YTHDF3在单体(虚线)和结合甲基化的RNA(实线)状态下的均方根涨落。(b) apo_pro (青色)和m6A_com (粉色)平衡轨迹中的平均构象的比较
3.2. 运动相关性分析
为了理解残基运动的关联性,采用m6A_com系统的平衡轨迹计算了残基涨落间的交叉相关系数,可以揭示与空间运动和相互作用相关的信息,如图2所示。从图2中可以看到,α螺旋与大多数β片层经历相似的相关位移(区域A),这些二级结构构成了YTHDF3的折叠核心,它们之间的强相关运动有助于维持结构的稳定性。Loop6的大部分与β片层和α2 (区域B)存在较强的负相关,其中的相互作用有利于约束loop6不发生明显的构象变化,导致RNA结合口袋的松散。相比之下,识别环与YTHDF3的其它部分没有显著的相关性,这有利于识别环进行自由的构象变化结合甲基化的RNA [16]。
而对于蛋白质-RNA之间的相关性,可以看到,loop1 (残基Tyr424-Ser425,区域C)、loop4 (残基Asn468-Gly471,区域E)、识别环(残基Gly490-Lys500,区域F)和loop6 (残基Thr530-Thr535,区域H)分别与甲基化RNA有一定程度的正相关,这些区域都位于甲基化RNA的结合表面,与RNA存在广泛的接触。其中,根据X-ray实验,发现位于loop4上的Gly469与RNA的U5形成氢键,位于loop6上的Asn524与RNA的C3形成氢键 [4],表明这些区域对于甲基化RNA的识别具有一定的作用 [17]。此外,还可
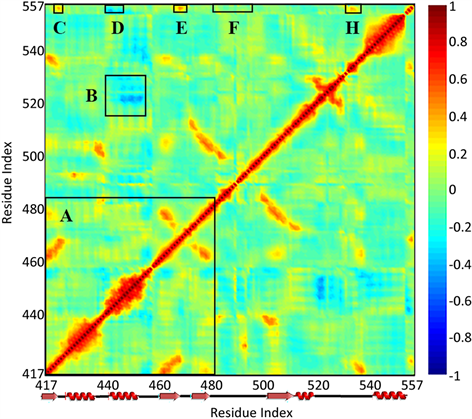
Figure 2. The cross-correlation of m6A_com system
图2. m6A_com体系的运动相关性
以看到loop2 (残基Ser436-Thr441,区域D)与甲基化RNA之间存在较强的负相关,这是由于该区域不仅能够提供构成芳香笼的疏水残基Tyr438,其中的Cys439还可与m6A形成氢键 [2]。总之,在复合物中,YTHDF3上的loop1、loop2、loop4、识别环和loop6与甲基化RNA分别有较强的运动相关性,表明它们有助于YTHDF3对甲基化RNA的特异性识别。
3.3. 基于复杂网络识别YTHDF3的关键位点
利用复杂网络中CPL的变化可以识别蛋白质变构相关的关键残基,因此计算了m6A_com的Z-score值,阈值设置为1.0。如图3(a)所示,共识别出10个残基簇,其中心残基分别映射到复合物的三级结构上,如图3(b)所示。簇10为m6A,扰动该节点会导致Z-score最大程度的变化,说明其在网络中对于介导信号传递起到重要作用,一致于前人的研究 [18]。甲基化有利于RNA被蛋白识别并进行后续的生理过程,同时也表明利用Z-score是识别关键残基的有效方法。
关键残基簇1在β1上,其中带正电的Arg417和Lys422位于RNA结合表面,负电性RNA与之存在广泛接触。Lys422在YTH蛋白中高度保守,Luo等人将酵母中YTH结构的对应残基Lys184进行突变,发现该突变完全阻断了YTH结构域与甲基化RNA的结合 [19]。簇2位于loop2上,其中Trp438参与构成识别m6A的芳香笼,在所有YTH蛋白中保守存在并与m6A形成稳定的π-π堆积相互作用 [20],Chang等人通过诱变实验发现将Trp438突变会导致YTHDF3与RNA的结合亲和性有很大程度的降低 [5]。同时,在YTHDF3的X-ray实验中发现,簇2中Cys439上的氧原子能够与m6A上发生甲基化的N6原子之间形成氢键 [2],有利于识别甲基化的RNA,并且该氢键在YTH蛋白中保守存在,可以有效地避免腺苷酸发生双甲基化 [18]。簇4,5和6均位于识别环上,其中Trp492和Trp497是构成芳香笼的重要残基,Trp492在YTH蛋白中高度保守。根据结晶实验发现Trp492与m6A上甲基形成阳离子-π相互作用,Trp497同Trp438一样与腺苷形成π-π堆叠相互作用 [21]。Govindaraju等人对恶性疟原虫pfYTH2蛋白的Trp46 (对应Trp492)进行定点突变实验和MD模拟 [22],发现该残基对于m6A的结合至关重要。

Figure 3. (a) The Z-score value for the difference in CPL. (b) Locations of the central residues for the 10 clusters of key residues
图3. (a) 特征路径长度变化的Z-score;(b) 10个中心残基簇在结构上的映射
簇8和9主要位于loop6上,其上具有带正电的残基Arg517,Arg520,Lys527和Arg533,与RNA的磷酸二酯主链有良好的静电相互作用,有利于结合RNA [4]。簇8中的Asn524和簇9中的Arg533和Asp534在YTH家族蛋白中具有高度保守性。根据YTHDC2的X-ray实验发现,Asn524对应的Asn1392以及Arg533对应的Arg1401均能与核苷酸C3形成稳定的氢键 [4]。对于簇9中的Asp534,YTHDF3的实验解析发现,一个保守的水分子介导了m6A的N7原子与Asp534侧链之间的氢键,进一步稳定了RNA的结合 [2]。另外残基Arg533的侧链胍基还能与核苷酸C3的碱基形成阳离子-π相互作用,Xu等人将YTHDC1中Arg475 (对应YTHDF3的Arg533)突变为苯丙氨酸和丙氨酸,发现对结合亲和力分别降低9
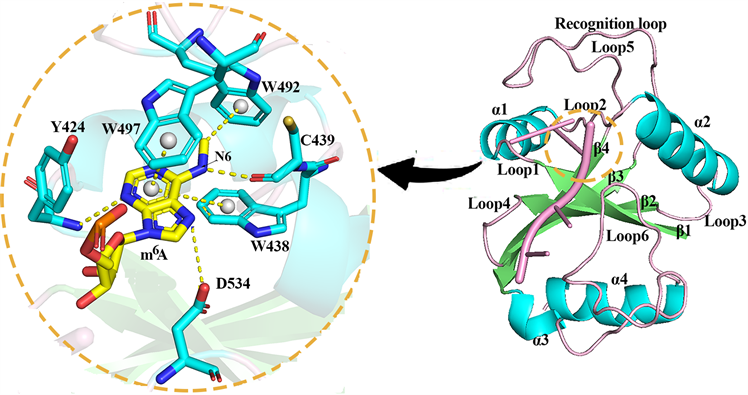
Figure 4. The diagram of interaction pattern (including H-bonds, π-π stacking and cation-π interactions) between residues in YTHDF3 and m6A.
图4. YTHDF3的残基与m6A的相互作用(包括氢键,π-π堆积相互作用和阳离子-π相互作用)模式图
倍或100倍以上,表明该残基对YTH蛋白结合RNA具有重要作用 [23]。为了方便理解,将氨基酸残基-m6A的相互作用进行细节显示,如图4所示。综上所述,可以知道YTHDF3的芳香笼残基Trp438、Trp492和Trp497在YTHDF1-2中保守存在,而在YTHDC1-2中,Trp497可为亮氨酸。因此可以推断YTHDF1-2与m6A的相互作用同YTHDF3具有相似性,而对于YTHDC1-2与m6A的相互作用,则会略有区别。
另外,对于识别到的簇3和簇7分别位于loop3和β4上,扰动簇3残基,会显著改变蛋白结构网络的信号传递,而且残基具有高柔性,因此我们认为簇3有利于进行构象转变并在结合RNA过程中起到传感的重要作用。对于簇7,不仅在RNA结合后RMSF明显降低,而且在相关性分析中发现其与β2和β3具有强的正相关,所以我们认为该处的残基能够以分子内部相互作用的形式稳定复合物的结构。
4. 结论
YTHDF3是一种RNA结合蛋白,能够特异性识别m6A修饰的RNA片段,在mRNA的翻译和降解中发挥着重要作用。首先,对YTHDF3单体和复合物分别进行了分子动力学模拟,发现甲基化的RNA结合能够使得YTHDF3的构象更加稳定,结构更为紧凑。计算复合物的运动相关性,发现甲基化的RNA主要与YTHDF3的loop1、lopp2,loop4、识别环和loop6具有一定的相关性,进而阐明这些loop区域对于甲基化RNA识别的重要性。最后基于加权的复杂网络,识别到了YTHDF3上与实验信息高度吻合的关键残基,另外所预测到的一些尚未被实验证实的关键位点可为未来实验提供重要信息。本工作有助于深入理解YTHDF3识别m6A修饰RNA的动力学机制,对于药物设计具有一定的指导意义。
基金项目
国家自然科学基金(31971180)资助项目。
文章引用
周文雪,韩中杰,吴志祥,李春华. 基于分子动力学模拟研究YTHDF3与m6A修饰RNA的相互作用动力学及关键位点
Study on the Interacting Dynamics and Key Residues between YTHDF3 and m6A-Mediated RNA Based on Molecular Dynamics Simulation[J]. 计算生物学, 2022, 12(03): 32-39. https://doi.org/10.12677/HJCB.2022.123005
参考文献
- 1. Liao, S., Sun, H. and Xu, C. (2018) YTH Domain: A Family of N6-methyladenosine (m6A) Readers. Genomics Prote-omics Bioinformatics, 16, 99-107. https://doi.org/10.1016/j.gpb.2018.04.002
- 2. Li, Y., Bedi, R.K., Moroz-Omori, E.V. and Caflisch, A. (2020) Structural and Dynamic Insights into Redundant Function of YTHDF Proteins. Journal of Chemical Information and Modeling, 60, 5932-5935. https://doi.org/10.1021/acs.jcim.0c01029
- 3. Patil, D.P., Pickering, B.F. and Jaffrey, S.R. (2018) Reading m6A in the Transcriptome: m6A-Binding Proteins. Trends in Cell Biology, 28, 113-127. https://doi.org/10.1016/j.tcb.2017.10.001
- 4. Ma, C., Liao, S. and Zhu, Z. (2019) Crystal Structure of Human YTHDC2 YTH Domain. Biochemical and Biophysical Research Communications, 518, 678-684. https://doi.org/10.1016/j.bbrc.2019.08.107
- 5. Chang, G., Shi, L., Ye, Y., Shi, H., Zeng, L., Tiwary, S., et al. (2020) YTHDF3 Induces the Translation of m6A-Enriched Gene Transcripts to Promote Breast Cancer Brain Metastasis. Cancer Cell, 38, 857-871.e7. https://doi.org/10.1016/j.ccell.2020.10.004
- 6. Xu, C., Liu, K., Ahmed, H., Loppnau, P., Schapira, M. and Min, J. (2015) Structural Basis for the Discriminative Recognition of N6-Methyladenosine RNA by the Human YT521-B Ho-mology Domain Family of Proteins. Journal of Biological Chemistry, 290, 24902-24913. https://doi.org/10.1074/jbc.M115.680389
- 7. Wang, X., Lu, Z., Gomez, A., Hon, G.C., Yue, Y., Han, D., et al. (2014) N6-Methyladenosine-Dependent Regulation of Messenger RNA Stability. Nature, 505, 117-120. https://doi.org/10.1038/nature12730
- 8. Li, Y., Bedi, R.K., Wiedmer, L., Sun, X., Huang, D. and Caflisch, A. (2021) Atomistic and Thermodynamic Analysis of N6-Methyladenosine (m6A) Recognition by the Reader Domain of YTHDC1. Journal of Chemical Theory and Computation, 17, 1240-1249. https://doi.org/10.1021/acs.jctc.0c01136
- 9. Jorgensen, W.L., Chandrasekhar, J. and Madura, J.D. (1983) Com-parison of Simple Potential Functions for Simulating Liquid Water. The Journal of Chemical Physics, 79, 926-935. https://doi.org/10.1063/1.445869
- 10. Pronk, S., Páll, S., Schulz, R., Larsson, P., Bjelkmar, P., et al. (2013) GROMACS 4.5: A High-Throughput and Highly Parallel Open Source Molecular Simulation Toolkit. Bioinformatics, 29, 845-854. https://doi.org/10.1093/bioinformatics/btt055
- 11. Huang, J. and MacKerell, A.J. (2013) CHARMM36 All-Atom Additive Protein Force Field: Validation Based on Comparison to NMR Data. Journal of Computational Chemistry, 34, 2135-2145. https://doi.org/10.1002/jcc.23354
- 12. Xu, Y., Vanommeslaeghe, K., Aleksandrov, A., MacKerell, A.D., MacKerell, J. and Nilsson, L. (2016) Additive CHARMM Force Field for Naturally Occurring Modified Ribonu-cleotides. Journal of Computational Chemistry, 37, 896-912. https://doi.org/10.1002/jcc.24307
- 13. Darden, T., York, D. and Pedersen, L. (1993) Particle Mesh Ewald: An Nlog(N) Method for Ewald Sums in Large Systems. Journal of Chemical Physics, 98, 10089-10092. https://doi.org/10.1063/1.464397
- 14. Hess, B. (2008) P-LINCS: A Paral-lel Linear Constraint Solver for Molecular Simulation. Journal of Chemical Theory and Computation, 4, 116-122. https://doi.org/10.1021/ct700200b
- 15. Shao, Q., Gong, W. and Li, C. (2020) A Study on Allosteric Communica-tion in U1A-snRNA Binding Interactions: Network Analysis Combined with Molecular Dynamics Data. Biophysical Chemistry, 264, Article ID: 106393. https://doi.org/10.1016/j.bpc.2020.106393
- 16. Krepl, M., Damberger, F.F., von Schroetter, C., Theler, D., Pokor-ná, P., Allain, F.H., et al. (2021) Recognition of N6-Methyladenosine by the YTHDC1 YTH Domain Studied by Molec-ular Dynamics and NMR Spectroscopy: The Role of Hydration. The Journal of Physical Chemistry B, 125, 7691-7705. https://doi.org/10.1021/acs.jpcb.1c03541
- 17. Luo, S. and Tong, L. (2014) Molecular Basis for the Recognition of Methylated Adenines in RNA by the Eukaryotic YTH Domain. Proceedings of the National Academy of Sciences of the United States of America, 111, 13834-13839. https://doi.org/10.1073/pnas.1412742111
- 18. Shi, R., Ying, S., Li, Y., Zhu, L., Wang, X. and Jin, H. (2021) Linking the YTH Domain to Cancer: The Importance of YTH Family Proteins in Epigenetics. Cell Death & Disease, 12, Article No. 346. https://doi.org/10.1038/s41419-021-03625-8
- 19. Zhang, Z., Theler, D., Kaminska, K.H., Hiller, M., de la Grange, P., Pudimat, R., et al. (2010) The YTH Domain Is a Novel RNA Binding Domain. Journal of Biological Chemistry, 285, 14701-14710. https://doi.org/10.1074/jbc.M110.104711
- 20. Govindaraju, G., Kadumuri, R.V., Sethumadhavan, D.V., Jabeena, C.A., Chavali, S. and Rajavelu, A. (2020) N6-Adenosine Methylation on mRNA Is Recognized by YTH2 Domain Pro-tein of Human Malaria Parasite Plasmodium falciparum. Epigenetics & Chromatin, 13, Article No. 33. https://doi.org/10.1186/s13072-020-00355-7
- 21. Xu, C., Wang, X., Liu, K., Roundtree, I.A., Tempel, W., Li, Y., et al. (2014) Structural Basis for Selective Binding of m6A RNA by the YTHDC1 YTH Domain. Nature Chemical Bi-ology, 10, 927-929. https://doi.org/10.1038/nchembio.1654
- 22. Xu, C., Wang, X., Liu, K., et al. (2014) Structural Basis for Se-lective Binding of m6A RNA by the YTHDC1 YTH Domain. Nature Chemical Biology, 10, 927-929. https://doi.org/10.1038/nchembio.1654
- 23. Luo, S. and Tong, L. (2014) Molecular Basis for the Recognition of Methylated Adenines in RNA by the Eukaryotic YTH Domain. Proceedings of the Na-tional Academy of Sciences of the United States of America (PNAS), 111, 13834-13839. https://doi.org/10.1073/pnas.1412742111