Botanical Research
Vol.
09
No.
06
(
2020
), Article ID:
38565
,
9
pages
10.12677/BR.2020.96066
沙田柚自交不亲和花柱数字基因表达谱及相关基因分析
张渝,李惠敏,刘玉洁,郭丹妮,刘华英,秦新民*
广西师范大学生命科学学院,广西 桂林

收稿日期:2020年10月13日;录用日期:2020年11月5日;发布日期:2020年11月12日

摘要
为探索沙田柚自交不亲和的分子机理,以沙田柚自交与异交1~3天的花柱为材料,利用数字基因表达谱技术对样品cDNA文库进行差异基因表达谱分析。结果表明:异交1 d/自交1 d花柱中,上调表达的基因有244个,下调表达的基因有27个;异交2 d/自交2 d花柱中,上调表达基因有265个,下调表达基因有244个;异交3 d/自交3 d花柱,上调表达的基因有193个,下调表达的基因有685个。在SY1/SZ1、SY2/SZ2和SY3/SZ3中共有的差异表达的基因为30个。差异表达基因的GO功能显著性富集分析结果表明,这些差异表达的基因主要参与应对各种化学物质、植物激素、内源和外源物质的刺激及信号转导过程。通过本研究,挖掘出了一些与沙田柚自交不亲和相关的基因,可为今后阐明沙田柚配子体自交不亲和的分子机理提供依据。
关键词
沙田柚,配子体自交不亲和,花柱,差异表达基因,数字基因表达谱

Digital Gene Expression Profiling Analysis of Self-Incompatibility Styles in Citrus grandis var. Shatinyu
Yu Zhang, Huimin Li, Yujie Liu, Danni Guo, Huaying Liu, Xinmin Qin*
College of Life Science, Guangxi Normal University, Guilin Guangxi

Received: Oct. 13th, 2020; accepted: Nov. 5th, 2020; published: Nov. 12th, 2020

ABSTRACT
This study aimed to understand the self-incompatibility mechanism in Citrus grandis var. Shatinyu. The one day, two days and three days styles of Shatinyu after self-pollination and cross- pollination were used to construct DNA libraries and the gene expression profiles were analyzed by Solexa sequencing technology. The results of solexa sequencing showed that a total of 271 different expression genes (DEGs) were obtained from the 1 d self-and cross-pollinated styles, in which 244 up-regulated expression and 27 down-regulated expression. 265 up-regulated expressed genes and 244 down-regulated expressed genes were in 2 d self-and cross-pollinated styles, 193 up-regulated expressed genes and 685 down-regulated expressed genes in comparing 3 d self-and cross-pollinated styles. Moreover, statistics of differentially expressed genes showed that there were 30 differentially expressed genes coexisted in SY1/SZ1, SY2/SZ2 and SY3/SZ3. Gene Ontology analysis showed that the DEGs were mainly involved in responding to all kinds of chemicals, plant hormones, the stimulation of endogenous and exogenous substances and signal transduction process. In sum, through this study some genes associated with self-incompatibility in Shatianyu were discovered. These results will provide a theoretical reference for further exploration of self-incompatibility molecular mechanism in Citrus grandis var. Shatinyu.
Keywords:Citrus grandis var. Shatinyu Hort, Gametophytic Self-Incompatibility, Styles, Differentially Expressed Genes, Digital Gene Expression Profile

Copyright © 2020 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
植物自交不亲和性(Self-incompatibility, SI)对于避免近亲繁殖有很重要的作用 [1] [2],其研究在植物生殖学和杂交优势利用等方面具有重要的理论和应用价值。
目前植物自交不亲和的研究主要集中在十字花科、茄科、蔷薇科、玄参科和罂粟科植物。沙田柚为芸香科、柑橘属植物,其自交不亲和的研究在细胞学、蛋白质化学等方面取得了一定进展 [3] - [9],但有关沙田柚配子体自交不亲和相关基因表达和鉴定方面的研究尚未见报道。
数字基因表达谱技术以高通量测序为基础,可用于检测特定细胞或组织在特定状态下不同样品中的差异表达基因的种类和丰度信息 [10],因而,近年来已被广泛应用于许多领域的研究中 [11] [12] [13]。
本研究以沙田柚自花授粉和异花授粉1 d、2 d、3 d后的花柱为实验材料,分别构建测序文库,进行了数字基因表达谱测序,试图挖掘与自交不亲和反应相关的差异表达的基因,进而为研究沙田柚配子体自交不亲和性机理提供分子依据。
2. 材料与方法
2.1. 材料处理与收集
实验材料为沙田柚(Citrus grandis var. Shatianyu Hort)花柱。在沙田柚的盛花期分别对沙田柚样树进行人工自花授粉(沙田柚 × 沙田柚)和异花授粉(酸柚 × 沙田柚),然后分别采集自交1~3d (标记为SZ1、SZ2、SZ3)和异交授粉1~3d (标记为SY1、SY2、SY3)后的花柱,立即放入液氮,保存于−80℃超低温冰箱中备用。
2.2. 总RNA的提取及文库构建
总RNA的提取采用TRIzol 试剂盒(Invitrogen 公司)并参照其操作手册进行。建库与测序由华大科技有限公司进行。
2.3. 测序数据分析及差异表达基因的筛选
测序数据采用RPM和RPKM [14] 值进行标准化处理。异交1d/自交1d、异交2d/自交2d和异交3d/自交3d花柱组合之间的差异表达基因采用DESeq方法 [15] 进行确定。差异基因筛选条件为qvalue < 0.005且log2 (fold change) > 1。
2.4. 差异基因基因功能注释和KEGG 代谢通路分析
沙田柚自交和异交授粉花柱差异表达基因进行GO功能富集显著性分析与KEGG代谢通路富集性分析分别采用采用GO数据库(http://www.Geneontology.org/)和KOBAS (2.0)软件进行。
2.5. 荧光定量RT-PCR检测
为检测测序结果的可靠程度,随机挑选6个基因进行RT-PCR测定。内参基因为GAPDH (表1)。荧光定量PCR的反应体系和程序参见Fermentas公司的SYBR Green master mix说明书,引物的退火温度为56℃~60℃,采用2−ΔΔCt法对表达量进行分析计算,实验设置3次重复。
Table 1. The primers of genes analysed by qPCR
表1. 差异表达基因qRT-PCR验证引物信息
3. 结果
3.1. 测序数据评估
文库测序得到的获得的Raw Data经处理分别得到Clean Reads,用比对软件SOAPaligner/SOAP2 [16] 将Clean Reads与参考基因组(甜橙)进行比对。结果表明SY1、SY2、SY3、SZ1、SZ2和SZ3获得的clean reads分别为11528402、11334516、11286963、11414971、11329671、11352636,6个样本中clean reads所占的比例在92.69%~95.07%。
3.2. 差异表达基因分析的GO分类与富集分析
3.2.1. 差异基因的筛选
以qvalue < 0.005 且log2 (fold change) > 1为标准对SY1/SZ1、SY2/SZ2、SY3/SZ3三个组合之间的差异基因进行筛选。结果表明SY1/SZ1中,差异表达基因总数为271 (上调表的基因244个,下调表达的基因27个)。SY2/SZ2的差异表达基因为509 (上调表达基因265个,下调表达基因244个)。SY3/SZ3的差异表达基因为878,其中上调表达基因193个,下调表达基因为685个(图1)。

Figure 1. The statistics of differentially expressed genes in self-pollinated styles and cross-pollinated styles
图1. 沙田柚自花授粉与异花授粉花柱差异表达基因的统计
3.2.2. 差异表达基因的GO功能显著性富集分析
在SY1/SZ1中,生物过程、细胞组分和分子功能三个功能富集分析的基因数分别为741个,425个,198个。其中,生物过程的741个基因分为19个功能基因群;细胞组分中的425个基因分为12个功能基因群;分子功能中的198个基因分为8个功能基因群。SY2/SZ2中生物过程、细胞组分、分子功能三个功能富集分析的基因个数分别为1199个,797个,384个。其中,生物过程的1199个基因分为21个功能基因群;细胞组分中的797个基因分为13个功能基因群;而分子功能中的384个基因则分为12个功能基因群。SY3/SZ3比对中,生物过程、细胞组分和分子功能富集分析的基因个数分别为2227个,1389个,715个。其中,生物过程中的2227个基因可分为21个功能基因群;细胞组分中的1389个基因分为14个功能基因群;分子功能中的715个基因分为10个功能基因群。
3.3. 差异表达基因的Pathway显著性富集分析
SY1/SZ1差异表达基因注释到146条KEGG代谢通路中(表2)。其中注释到淀粉和蔗糖代谢途径(ko00500)通路的基因数量最多,为10个(6.85%),其次是戊糖和葡萄糖醛酸酯互变途径(ko00040),以及聚糖降解(ko00511)途径,数量均为6 (4.11%)。
Table 2. Differentially expressed genes enriched of KEGG pathways in SY1-VS-SZ1
表2. SY1/SZ1差异表达基因Pathway显著性富集统计
SY2/SZ2差异表达基因注释到251个KEGG代谢通路中(表3)。其中注释代谢途径(ko01100)通路的基因数量最多,为73个(29.08%),其次为次生代谢产物的生物合成途径(ko01110)和植物激素信号转导途径(ko04075),基因数量分别为44个(17.53%)和26个 (10.36%)。
Table 3. Differentially expressed genes enriched of KEGG pathways in SY2-VS-SZ2
表3. SY2/SZ2差异表达基因Pathway显著性富集统计
SY3/SZ3差异表达基因注释到KEGG代谢通路为481条(表4)。与SY2/SZ2相同,注释到代谢途径(ko01100)通路的基因数量最多,为157 (32.64%),其次为次生代谢产物的生物合成途径(ko01110)和植物激素信号转导途径(ko04075),基因数量分别为118 (24.53%)和47 (9.77%)。
Table 4. Differentially expressed genes enriched of KEGG pathways in SY3-VS-SZ3
表4. SY3/SZ3差异表达基因Pathway显著性富集统计
3.4. 自交与异交花柱共有的差异表达基因
在SY1/SZ1、SY2/SZ2、SY3/SZ3组合差异表达基因分别统计的基础上,对在SY1/SZ1、SY2/SZ2和SY3/SZ3均达到差异表达的基因进行挖掘,结果表明共有30个基因在自交花柱与异交花柱之间达到差异表达水平,其表达量(Reads Per Kilobase per Million,RPKM)及功能注释见表5。在30个差异表达基因中目前G0、KEGG、NR数据库中未有功能注释(包括注释为hypothetical protein、uncharacterized protein或predicted protein)的基因为11个,其中无注释的基因4个。
Table 5. Differentially expressed genes coexisted in self- and cross-pollinated styles
表5. 自交与异交花柱之间差异表达基因
注:—示数据库中无任何注释。
3.5. qRT-PCR分析
分别提取自交和异交1~3d花柱总RNA,反转录后进行定量PCR验证。结果表明6个差异表达基因(EBA、SRA、UBP、RIB、RIH和CBP)在自交和异交1~3d花柱的相对表达量与表达谱分析的变化趋势一致(图2)。

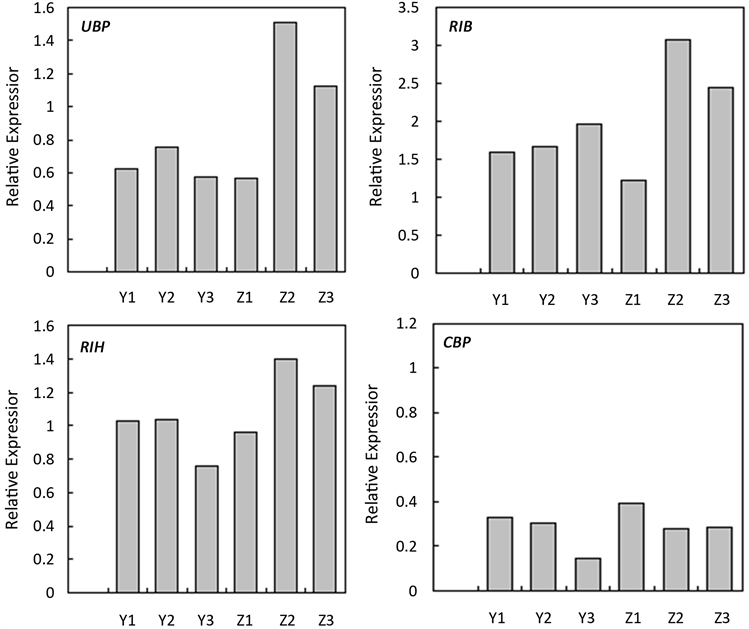
Figure 2. The relative expression between samples verified by qPCR
图2. 差异表达基因qPCR验证
4. 讨论与结论
数字基因表达谱技术能快速检测植物在不同组织细胞、不同发育阶段以及在众多逆境环境胁迫下基因的表达差异,在功能基因组学方面正在发挥着越来越重要的作用 [17] [18] [19]。本实验采用数字基因表达谱技术对沙田柚自交和异交1~3 d花柱之间的差异基因进行了分析。差异基因的Go功能注释结果表明在自交1 d与异交1 d的花柱中,差异表达的基因主要参与花粉管的伸长和发育的调控和植物细胞壁的修饰。在自交2 d/异交2 d的花柱和自交3 d/异交3 d的花柱,差异表达的基因主要参与应对化学物质、植物激素、内源和外源物质的刺激,信号转导。因此,沙田柚配子体自交不亲和反应是一个多基因协同作用的复杂过程。
KEEG显著性富集分析发现SY1-VS-SZ1、SY2-VS-SZ2、SY3-VS-SZ3三组差异表达基因的共有的通路为淀粉和蔗糖的代谢(ko00500)和戊糖和葡糖糖醛酸之间的互变(ko00040)。SY2-VS-SZ2和SY3-VS-SZ3所共有的显著性富集通路为木质素的生物合成(ko00940),苯丙氨酸代谢(ko00360),氰氨酸的代谢(ko00460),醚酯的代谢(ko00565),植物激素的信号转导(ko04075)、新陈代谢途径(ko01100)、黄酮和黄酮醇的生物合成(ko00944)、α-亚麻酸代谢(ko00592)。
自交与异交花柱共有的差异表达基因为30个,目前有注释的仅为15个,功能涉及到丝氨酸/苏氨酸激酶活性、α-岩藻糖苷酶、核糖核酸酶H、半乳糖醛酸酶、ATP结合蛋白(酶)、木葡聚糖半乳糖基转移酶、UDP-葡萄糖苷转移酶、乙酰乙基葡萄糖基转移酶、DNA结合蛋白,以及细胞壁修饰等,但其在沙田柚自交不亲和中的生理功能尚不明确。此外,15个差异表达基因目前数据库中尚无功能注释,其在沙田柚自交不亲和中的功能值得进一步深入研究。
基金项目
国家自然科学基金(31360477);广西教育厅项目(2013YB036)。
文章引用
张 渝,李惠敏,刘玉洁,郭丹妮,刘华英,秦新民. 沙田柚自交不亲和花柱数字基因表达谱及相关基因分析
Digital Gene Expression Profiling Analysis of Self-Incompatibility Styles in Citrus grandis var. Shatinyu[J]. 植物学研究, 2020, 09(06): 536-544. https://doi.org/10.12677/BR.2020.96066
参考文献
- 1. 孟金陵. 植物生殖遗传学[M]. 北京: 科学出版社, 1995.
- 2. Takayama, S. and Isogai, A. (2005) Self-Incompatibility in Plants. Annual Review of Plant Biology, 56, 467-489. https://doi.org/10.1146/annurev.arplant.56.032604.144249
- 3. 薛妙男, 陈腾土, 杨继华. 沙田柚自交和异交亲和性观察[J]. 园艺学报, 1995, 22(2): 127-132.
- 4. 杨继华, 李红艳, 薛妙男. 沙田柚花柱S-糖蛋白分离及鉴定[J]. 广西师范大学学报, 2000, 18(4): 66-70.
- 5. 杨继华, 尧桂荣, 薛妙男. 沙田柚花柱S-糖蛋白的纯化和N-端序列测定[J]. 广西师范大学学报, 2001, 19(1): 72-79.
- 6. 秦新民, 李惠敏, 薛妙男, 等. 沙田柚自交, 异交花粉管蛋白的双向电泳分析[J]. 广西植物, 2004, 24(6): 566-569.
- 7. 薛妙男, 杨继华. 沙田柚花粉管在花柱中的生长途径及其识别[J]. 广西师范大学学报, 2001, 19(2): 60-66.
- 8. 秦新民, 莫花浓, 万珊, 等. 沙田柚花粉管特异蛋白的免疫细胞化学研究[J]. 广西师范大学学报, 2008, 26(4): 113-115.
- 9. 秦新民, 莫花浓, 石菁萍, 等. 沙田柚花粉管S1-RNase免疫胶体金定位研究[J]. 广西农业科学, 2009, 40(5): 483-485.
- 10. Ness, R.W., Sisl, M. and Barrett, S.C. (2011) De Novo Sequence Assembly and Characterization of the Floral Transcriptome in Cross- and Self-Fertilizing Plants. BMC Genomics, 12, 298-310. https://doi.org/10.1186/1471-2164-12-298
- 11. Alagna, F., Torchia, L., Servili, M., et al. (2009) Comparative 454 Pyrosequencing of Transcripts from Two Olive Genotypes during Fruit Development. BMC Genomics, 10, 399-413. https://doi.org/10.1186/1471-2164-10-399
- 12. Severin, A.J., Woody, J.L., Bolon, Y.T., et al. (2010) RNA-Seq Atlas of Glycine Max: A Guide to the Soybean Transcriptome. BMC Plant Biology, 10, 160-165. https://doi.org/10.1186/1471-2229-10-160
- 13. Morrissy, A.S., Morin, R.D., Delanry, A., et al. (2009) Next-Generation Tag Sequencing for Cancer Gene Expression Profiling. Genome Research, 19, 1825-1835. https://doi.org/10.1101/gr.094482.109
- 14. Mortazavi, A., Williama, B.A., Schaeffer, L., et al. (2008) Mapping and Quantifying Mammalian Transcriptomes by RNA-Seq. Nature Methods, 5, 621-628. https://doi.org/10.1038/nmeth.1226
- 15. Anders, S. and Huber, W. (2010) Differential Expression Analysis for Sequence Count Data. Genome Biology, 11, 106-109. https://doi.org/10.1186/gb-2010-11-10-r106
- 16. Li, R., Yu, C., Li, Y., et al. (2009) SOAP2: An Improved Ultrafast Tool for Short Read Alignment. Bioinformatics, 25, 1966-1967. https://doi.org/10.1093/bioinformatics/btp336
- 17. 张渝, 刘玉洁, 郭丹妮, 等. 基于高通量测序的数字基因表达谱技术研究进展[J]. 北方园艺, 2015(10): 170-176.
- 18. 李穆, 程志远, 何丽莲, 等. 甘蔗印度种响应干旱胁迫的数字基因表达谱[J]. 分子植物育种, 2018, 16(7): 2099-2106.
- 19. 韩娟娟, 纪砚耘, 梁晓艳, 等. 干旱胁迫下丹参数字基因表达谱分析[J]. 基因组学与应用生物学, 2018, 37(10): 4439-4448.
