Advances in Clinical Medicine
Vol.
13
No.
12
(
2023
), Article ID:
78196
,
12
pages
10.12677/ACM.2023.13122819
骨关节炎差异表达基因和关键基因的生物信息学分析
杨春生,韩啸,王宝兰*
新疆医科大学第一附属医院康复医学科,新疆 乌鲁木齐
收稿日期:2023年11月27日;录用日期:2023年12月21日;发布日期:2023年12月28日

摘要
目的:通过对GEO数据库中骨关节炎相关基因芯片数据分析,探究骨关节炎相关的差异和关键基因、信号通路等。方法:采用生物信息学分析方法,从GEO数据库下载人OA相关数据集GSE16464、GSE169077,应用Perl语言、R语言、R软件和Rstudio软件,进行基因的差异表达分析、功能注释等,并应用GSE117999数据集进行验证。结果:共筛选出108个差异表达基因,其中34个上调,74个下调,ABCA6、AOX1为关键基因。通路水平主要与昼夜节律、ECM-受体相互作用、p53信号通路相关。结论:多种途径参与了骨关节炎发病过程,关键基因ABCA6、AOX1在骨关节炎发病过程中起重要作用。
关键词
骨关节炎,生物信息学,差异表达基因,关键基因

Bioinformatics Analysis of Differentially Expressed Genes and Key Genes in Osteoarthritis
Chunsheng Yang, Xiao Han, Baolan Wang*
Department of Rehabilitation Medicine, The First Affiliated Hospital of Xinjiang Medical University, Urumqi Xinjiang
Received: Nov. 27th, 2023; accepted: Dec. 21st, 2023; published: Dec. 28th, 2023

ABSTRACT
To explore the differences, key genes and signaling pathways related to osteoarthritis by analyzing the gene chip data related to osteoarthritis in GEO database. Methods: Bioinformatics analysis method was used to download human OA related datasets GSE16464 and GSE169077 from GEO database. Perl language, R language, R software and Rstudio software were used to analyze the differential expression of genes, functional annotation, etc., and the GSE117999 dataset was used for verification. Results: A total of 108 differentially expressed genes were screened, of which 34 were up-regulated and 74 were down-regulated. ABCA6 and AOX1 were key genes. The pathway level is mainly related to circadian rhythm, ECM-receptor interaction, and p53 signaling pathway. Conclusion : A variety of pathways are involved in the pathogenesis of osteoarthritis, and the key genes ABCA6 and AOX1 play an important role in the pathogenesis of osteoarthritis.
Keywords:Osteoarthritis, Bioinformatics, Differentially Expressed Genes, Key Genes

Copyright © 2023 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
骨关节炎(Osteoarthritis, OA)是一种严重危害人类身心健康的慢性致残性疾病,具有高患病率、高致残率的特点,是全球十大致残疾病之一。据统计,2020年全球40岁以上人群的患病率为22.9%,相当于全球6.541亿人 [1] 。除了个人负担外,骨关节炎的社会经济成本也是非常高的。在2010年全球疾病负担研究中,骨性关节炎在291种疾病中排名第11位 [2] 。因此,这是我们要面临的重大公共问题。OA的发病机制复杂,由多种因素引起,目前年龄的增长和肥胖症的流行是世界范围内骨性关节炎患病率稳步上升的主要原因 [3] [4] 。在目前保守治疗不能有效逆转病情,手术患者增加的情况下,OA已经引起广泛关注。近年来,随着该疾病的病理生理学和细胞分子生物学更深入的理解,多学科的交叉渗透,涉及OA信号通路中生物标志物的研究成为关注的重点、热点 [5] 。生物信息学是生物学与信息学的交叉科学,可将疾病的临床特征在基因、蛋白质水平相联系,进行功能富集、特征基因和主要通路识别,在生命科学中发挥着重要作用 [6] 。本研究用生物信息学分析的方法对GEO (Gene Expression Omnibus, GEO)数据库中OA患者的基因芯片数据进行差异富集分析,分析OA相关基因和信号通路以及作用,筛选关键基因,进一步探讨OA发病的分子机制。
2. 资料与方法
2.1. 检索GEO数据库
GEO数据库(http://www.ncbi.nlm.nih.gov/geo)是一个公共基因组数据库,储存芯片、二代测序以及其他高通量测序数据。我们以“osteoarthritis、arthritis”为检索词,下载人类基因表达数据集GSE16464、GSE169077和GSE117999,分别为GPL570、GPL96和GPL20844平台。OA组定义为实验组,非OA组为对照组。用于分析的样本总共为17个,OA软骨细胞和正常软骨细胞样本6个,OA患者相对正常和明显异常的膝骨关节软骨组织标本11个。数据集GSE117999为验证组,样本共24个,为行半月板部分切除术且没有任何OA证据患者和OA行膝关节置换患者的软骨组织。
2.2. 基因数据预处理
对下载的原始数据样本进行数据预处理,以减少原始数据的误差,增强进一步数据挖掘分析的信度。我们在分析过程中主要借助了GO、KEGG通路分析,以及基因/蛋白质作用关系检索工具等信息数据库,和R语言、Perl语言、Rstudio、Cytoscape分析软件及DAVID (https://david.ncifcrf.gov/home.jsp)等在线分析工具。使用Perl语言编辑软件对来自GPL570、GPL96的平台的数据集进行基因注释,将探针ID转化为基因名称。使用R语言编辑软件进行芯片数据预处理和分析,使样本之间归一化具有可比性,用R语言中的limma、sva包对注释后的两个数据集进行合并归一化处理,数据汇总后获取矫正后表达水平的标准化数据。
2.3. 筛选差异表达基因
应用Rstudio软件加载R语言中Limma、pheatmap包行差异表达基因筛选,分析条件为:① logFC = 1;② P < 0.05。
2.4. 基因富集分析
2.4.1. GO和KEGG富集分析
利用Rstudio软件,加载clusterProfiler、org.Hs.eg.db、enrichplot、ggplot2包,进行富集分析。GO分析主要在生物学过程、细胞成分和分子功能3个方面对基因及其产物的功能进行定义。KEGG分析则是通过对基因产物在细胞中的代谢途径以及这些基因产物功能的计算,对基因的功能进行注释和分析,筛选出与OA相关的通路。
2.4.2. DO和GSEA富集分析
利用Rstudio软件,加载clusterProfiler、org.Hs.eg.db、enrichplot、ggplot2、GSEABase、DOSE包,进行富集分析。DO分析差异基因和疾病的相关性。GSEA主要分析实验组和对照组中基因集的富集趋势。
2.5. 筛选特征基因和准确性验证
主要应用机器学习中LASSO回归和SVM-RFE的方法,加载glmnet、e1071、kernlab、caret包,对差异表达基因分别进行分析,将两种方法的结果取交集,结果为OA的特征基因。通过加载pROC包形成ROC曲线,与差异表达基因进行准确性比对。
2.6. 验证组差异分析和关键基因筛选
首先对验证组GSE117999数据集进行基因注释和分组,Rstudio软件加载ggpubr、limma、pROC包,然后将筛选的特征基因和验证组通过箱线图和ROC曲线的形式进行分析对比。
3. 结果
3.1. 差异表达基因
共筛选出108个差异表达基因,其中34个上调,74个下调,见图1。
3.2. 差异表达基因分析
3.2.1. GO和KEGG分析
GO富集分析主要在生物学过程、细胞组分、分子功能3方面进行。结果表明,OA在生物学过程方面与细胞对酸性物质和外界刺激的反应有关,细胞组分的变化在纤维胶原,分子功能与细胞外基质结构成分、抗张强度及血小板衍生生长因子相关。KEGG分析结果显示,OA与昼夜节律通路、ECM受体及p53信号通路相关,见表1。
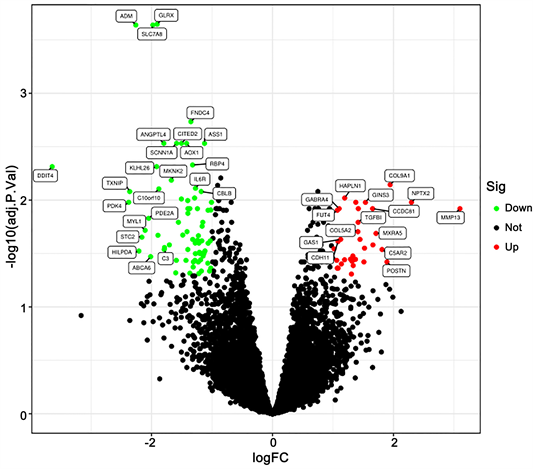 红点代表上调基因,绿点代表下调基因,黑点代表无差异基因。
红点代表上调基因,绿点代表下调基因,黑点代表无差异基因。
Figure 1. Volcano map of differentially expressed genes
图1. 差异表达基因火山图
Table 1. GO functional annotation and KEGG pathway enrichment analysis results
表1. GO功能注释和KEGG通路富集分析结果
3.2.2. DO和GSEA富集分析
 (A)
(A)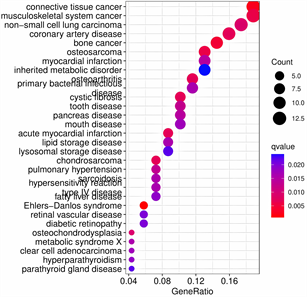 (B)(A) 柱状图横坐标是富集基因数目,纵坐标是富集疾病的名称,颜色越红代表富集越显著;(B) 气泡图横坐标是富集基因所占比例,纵坐标是富集疾病的名称,气泡大小代表富集基因数目,颜色越红代表富集越显著。
(B)(A) 柱状图横坐标是富集基因数目,纵坐标是富集疾病的名称,颜色越红代表富集越显著;(B) 气泡图横坐标是富集基因所占比例,纵坐标是富集疾病的名称,气泡大小代表富集基因数目,颜色越红代表富集越显著。
Figure 2. DO enrichment analysis: Diseases with major enrichment of differentially expressed genes
图2. DO富集分析:差异表达基因主要富集的疾病
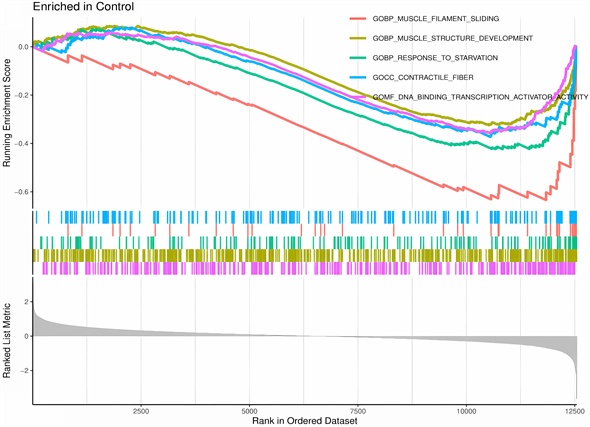 非OA对照组中基因多富集在排序基因的底部,与肌丝滑动、肌肉构成和发育、有机酸代谢、DNA结合转录激活因子活性有关。
非OA对照组中基因多富集在排序基因的底部,与肌丝滑动、肌肉构成和发育、有机酸代谢、DNA结合转录激活因子活性有关。
Figure 3. GSEA enrichment analysis: Analysis of the trend of genes in the gene set in the sorted genes
图3. GSEA富集分析:分析基因集中基因在排序基因中的变化趋势
 OA实验组的基因多富集在排序基因的顶部,与结构组织封装、细胞外基质胶原蛋白、内质网腔、细胞外基质的结构和化学成分有关。
OA实验组的基因多富集在排序基因的顶部,与结构组织封装、细胞外基质胶原蛋白、内质网腔、细胞外基质的结构和化学成分有关。
Figure 4. GSEA enrichment analysis: Analysis of the variation trend of genes in the gene set in the sorted genes
图4. GSEA富集分析:分析基因集中基因在排序基因中的变化趋势
通过DO分析差异表达基因,发现这些基因多富集在结缔组织,与肿瘤相关见,图2(A)、图2(B)。GSEA分析基因集中基因在排序基因中的变化趋势,总体上看,富集在非OA组中基因领头亚集位于排序基因底部,主要与肌丝滑动、肌肉构成和发育、有机酸代谢、DNA结合转录激活因子活性等有关,见图3;富集在OA组的基因领头亚集位于排序基因顶部,与结构组织封装、含有细胞外基质的胶原蛋白、内质网腔、细胞外基质的结构和化学成分等有关,见图4。
3.3. 特征基因和准确性验证
LASSO回归和SVM-RFE方法取交集分析后共得到11个特征基因ADM、SLC7A8、FNDC4、AOX1、HIST2H2BE、CD14、FUT4、MYL1、ENPP4、ABCA6、C8B,其中FUT4和C8B表达上调,其余表达下调,见图5(A)、图5(B)。特征基因在差异表达基因中ROC曲线验证提示,特征基因的曲线下面积均>0.9,准确性高,见表2。
Table 2. ROC curve analysis table of 11 characteristic genes
表2. 11个特征基因的ROC曲线分析表
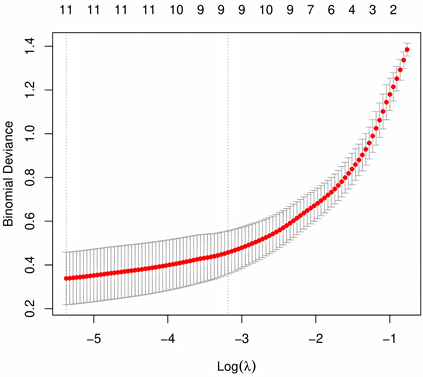 (A)
(A) (B)(A) LASSO回归筛选出差异表达基因11个;(B) SVM-REF筛选出差异表达基因108个。
(B)(A) LASSO回归筛选出差异表达基因11个;(B) SVM-REF筛选出差异表达基因108个。
Figure 5. Screening of characteristic genes
图5. 特征基因的筛选
3.4. 特征基因验证和筛选关键基因
将特征基因在GSE117999数据集进行验证,ABCA6在箱线图中P < 0.001,AOX1的P值 < 0.05,余均 > 0.05,见图6;在ROC曲线中,ABCA6的AUC > 0.9,AOX1的AUC > 0.7,其余基因AUG介于0.5~0.7之间见图7。结果表明,ABCA6和AOX1在非OA组、OA组中差异性最显著,与OA的关系最密切,可作为关键基因。
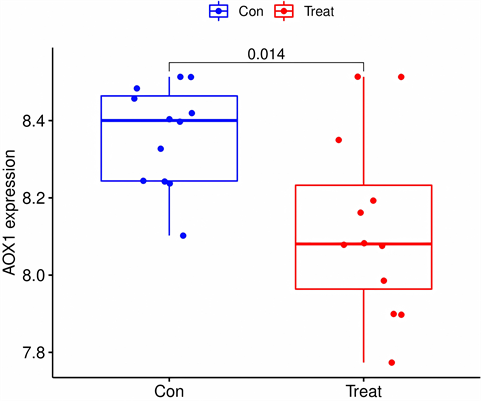
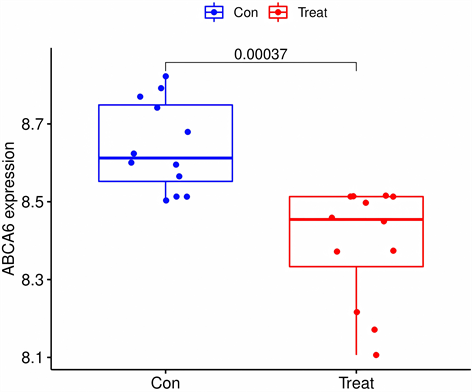 箱线图显示,ABCA6和AOX1在对照组和实验组中差异显著,P < 0.05。
箱线图显示,ABCA6和AOX1在对照组和实验组中差异显著,P < 0.05。
Figure 6. Data set GSE117999 verifies characteristic genes
图6. 数据集GSE117999验证特征基因
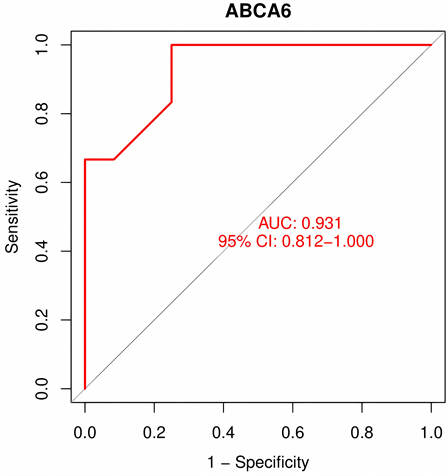 ROC曲线显示,ABCA6在对照组和实验组中差异最显著,AUC > 0.9,为关键基因。
ROC曲线显示,ABCA6在对照组和实验组中差异最显著,AUC > 0.9,为关键基因。
Figure 7. Data set GSE117999 verifies characteristic genes
图7. 数据集GSE117999验证特征基因
4. 讨论
骨性关节炎作为一种最常见的慢性和进行性关节疾病,其病因与软骨细胞、细胞外基质、软骨下骨降解和合成不平衡等有关 [7] [8] 。生理状态下,软骨细胞的合成代谢、分解代谢间保持平衡,OA时引起两种代谢调节失衡,导致了软骨基质成分的进行性丢失,软骨细胞的结构和功能破坏。而关节软骨细胞是关节软骨稳态的传感器,在维持关节软骨正常生理结构和功能方面起着重要作用。研究表明,关节软骨细胞稳态可被多种因素破坏,包括生物因素和机械损伤因素的异常刺激、衰老等。在分子水平,多种基因通过不同的信号通路在OA过程中发挥重要作用,包括基因表达、基质合成和降解、对内外环境刺激反应、细胞周期等 [9] [10] 。我们通过生物信息学方法,筛选出的108个差异表达基因中,34个表达上调,74个表达下调,在OA的过程中起重要作用。
GO富集分析中,OA在生物学过程方面,与细胞对酸性物质和外界刺激的反应有关。酸性物质可以破坏关节周围组织微环境,引起炎症因子释放,促使OA发生、进展。细胞组分的变化中,与OA相关的纤维胶原主要为胶原蛋白II和X。胶原蛋白II主要参与细胞外基质构成,由软骨细胞分泌;胶原X主要由肥大细胞分泌,是软骨受到损害的标志,二者与软骨基质降解密切相关。分子功能方面与细胞外基质结构成分、抗张强度及血小板衍生生长因子相关,软骨细胞外的基质破坏是OA发生的主要原因之一,因此细胞外基质的成分和抗张强度在保护关节免受体内外因素损伤方面尤为重要;此外,血小板衍生生长因子可促进损伤软骨修复和再生。KEGG分析结果中OA与昼夜节律通路、ECM受体及p53信号通路相关。昼夜节律通路是生物钟系统的重要组成部分,慢性昼夜节律紊乱会使大鼠体重显著增加,软骨严重磨损变薄、纤维样变、水肿,基质红染变浅且不均匀,出现OA样变。ECM是组织中细胞以外的部分,即细胞外基质,可由软骨细胞分泌。胞外基质为组织细胞正常生理活动提供支持,以及生存的微环境,参与细胞之间的信息传递,调节细胞免疫反应、生长因子和其它生物活性分子,参与、调控细胞生理生化行为,包括细胞的生长、分化、凋亡、修复、再生等。因此ECM受体在OA的发病中发挥重要作用。p53信号通路是机体非常重要的信号通路之一,参与体内多种生理活动。P53通路激活可以引起某些mRNA异常表达,通过影响细胞周期和凋亡等,引起细胞衰老死亡。P53可调节细胞周期,参与维持正常免疫应答、组织修复和器官功能。此外,乙酰化状态P53参与miR-34a对SIRT1/P53信号通路的直接调控,进而影响OA的发生发展。P53也可调控软骨细胞的凋亡和自噬,而软骨细胞衰老凋亡被认为在OA的发展中发挥重要作用,它的激活被认为是衰老调控的重要步骤,可能对于OA具有治疗作用。
DO分析主要是疾病相关分析,发现差异表达基因多富集在结缔组织,包括骨关节炎、骨肉瘤等,富集结果与预想一致。GSEA分析主要涉及组织细胞结构组分与功能,结果多与OA的发生相关。非OA组中基因领头亚集多富集于排序基因底部,主要与肌丝滑动、肌肉构成和发育、有机酸代谢、DNA结合转录激活因子活性等有关;OA组的基因领头亚集位于排序基因顶部,与结构组织封装、含有细胞外基质的胶原蛋白、内质网腔、细胞外基质的结构和化学成分等有关。
LASSO回归和SVM-RFE方法取交集分析后共得到11个特征基因,其中ABCA6和AOX1在非OA组、OA组中差异性最显著,与OA的关系最密切,可作为关键基因。ABC6A基因是一种蛋白质编码基因,该基因编码的膜相关蛋白是ATP结合盒(ABC)转运蛋白超家族成员,通过细胞外膜和细胞内膜运输各种分子。该基因可能在巨噬细胞脂质转运和体内平衡中发挥作用,促进脂质转运蛋白活性。ABC6A与胆固醇代谢关系密切,根据胆固醇反应性,可以预期ABCA6可能参与细胞内脂质转运过程 [10] [11] [12] 。研究表明,过量胆固醇与多种疾病的发病机制有关,包括肝脏疾病、糖尿病、骨质疏松症、骨关节炎等 [13] [14] [15] 。骨关节炎的存在和进展与动脉粥样硬化有关,可由膳食胆固醇升高诱导发生 [16] [17] 。而骨关节炎作为一种与代谢紊乱相关的疾病,软骨细胞中胆固醇代谢的CH25H-CYP7B1-RORα轴是骨关节炎发病的关键分解代谢调节剂 [16] 。AOX1又名醛氧化酶,是一种蛋白质编码基因,属于钼黄素家族。AOX1可作为一种解毒酶参与体内代谢过程,主要参与着氧化还原反应,而这一过程可产生过氧化氢,在一定条件下催化超氧化物的形成,ROS生成增多,从而参与细胞凋亡、增殖及分化功能 [18] 。此外,AOX1可能参与活性氧稳态的调节,是分子氧的单电子还原产生超氧化物的重要来源,还可以通过使用NADH或醛作为电子供体将亚硝酸盐还原为NO的产生,并在脂肪形成中发挥作用。因此,可通过各种途径直接或间接影响关节局部平衡,在OA发生、进展中发挥作用。
总之,多种途径参与了OA的发生发展过程,我们利用生物信息学分析方法,发现多个差异表达基因和相关通路,尤其是ABCA6、AOX1基因,可能在OA早期和晚期的进展中发挥重要作用,它们共同影响OA的发生、进展,为OA分子机制研究提供了另一途径。然而,目前的研究存在局限性,需要进一步细胞、动物实验和人体研究的证明,进一步了解引发骨关节炎的发病机制,为OA诊断和治疗提供潜在的策略。以便开发有针对性、个性化的治疗方法,帮助人们预防或减缓OA的发展。
文章引用
杨春生,韩 啸,王宝兰. 骨关节炎差异表达基因和关键基因的生物信息学分析
Bioinformatics Analysis of Differentially Expressed Genes and Key Genes in Osteoarthritis[J]. 临床医学进展, 2023, 13(12): 20013-20024. https://doi.org/10.12677/ACM.2023.13122819
参考文献
- 1. Oo, W.M. (2022) Prospects of Disease-Modifying Osteoarthritis Drugs. Clinics in Geriatric Medicine, 38, 397-432. https://doi.org/10.1016/j.cger.2021.11.010
- 2. Vos T, Flaxman AD, Naghavi M, et al. (2012) Years Lived with disability (YLDs) for 1160 Sequelae of 289 Diseases and Injuries 1990-2010: A Systematic Analysis for the Global Burden of Disease Study 2010. Lancet, 380, 2163-2196.
- 3. Liu, M.B., Jin, F., Yao, X.C. and Zhu, Z.X. (2022) Dis-ease Burden of Osteoarthritis of the Knee and Hip Due to a High Body Mass Index in China and the USA: 1990-2019 Findings from the Global Burden of Disease Study 2019. BMC Musculoskeletal Disorders, 23, Article No. 63. https://doi.org/10.1186/s12891-022-05027-z
- 4. Olsson, S., Akbarian, E., Lind, A., Razavian, A.S. and Gordon, M. (2021) Automating Classification of Osteoarthritis According to Kellgren-Lawrence in the Knee Using Deep Learning in an Unfiltered Adult Population. BMC Musculoskeletal Disorders, 22, Article No. 844. https://doi.org/10.1186/s12891-021-04722-7
- 5. Korostynski, M., Malek, N., Piechota, M. and Starowicz, K. (2018) Cell-Type-Specific Gene Expression Patterns in the Knee Cartilage in an Osteoarthritis Rat Model. Functional & Integrative Genomics, 18, 79-87. https://doi.org/10.1007/s10142-017-0576-6
- 6. Tapprich, W.E., Reichart, L., Simon, D.M., et al. (2021) An In-structional Definition and Assessment Rubric for Bioinformatics Instruction. Biochemistry and Molecular Biology Edu-cation, 49, 38-45. https://doi.org/10.1002/bmb.21361
- 7. Jang, S., Lee, K., Ju, J.H. (2021) Recent Updates of Diagnosis, Pathophysiology, and Treatment on Osteoarthritis of the Knee. International Journal of Molecular Sciences, 22, Article No. 2619. https://doi.org/10.3390/ijms22052619
- 8. Yunus, M., Nordin, A. and Kamal, H. (2020) Pathophysiological Perspective of Osteoarthritis. Medicina, 56, Article No. 614. https://doi.org/10.3390/medicina56110614
- 9. 王海明, 张驰, 魏向阳, 魏全, 何成奇. 膝骨关节炎软骨细胞中差异基因表达的生物信息学分析[J]. 华西医学, 2021, 36(5): 623-631.
- 10. Xu, B., Li, Y.Y., Ma, J. and Pei, F.X. (2016) Roles of microRNA and Signaling Pathway in Osteoarthritis Pathogenesis. Journal of Zhejiang Universi-ty-SCIENCE B, 17, 200-208. https://doi.org/10.1631/jzus.B1500267
- 11. van Leeuwen, E.M., Karssen, L.C., Deelen, J., et al. (2015) Genome of The Netherlands Population-Specific Imputations Identify an ABCA6 Variant Asso-ciated with Cholesterol Levels. Nature Communications, 6, Article No. 6065.
- 12. Papathanasiou I, Anastasopoulou L, Tsezou A. (2021) Cholesterol Metabolism Related Genes in Osteoarthritis. Bone, 152, Article ID: 116076. https://doi.org/10.1016/j.bone.2021.116076
- 13. Gierman, L.M., Kühnast, S., Koudijs, A., et al. (2014) Osteoar-thritis Development Is Induced by Increased Dietary Cholesterol and can be inhibited by atorvastatin in APOE*3Leiden.CETP Mice—A Translational Model for Atherosclerosis. Annals of the Rheumatic Diseases, 73, 921-927. https://doi.org/10.1136/annrheumdis-2013-203248
- 14. He, H., Lu, M., Shi, H., Yue, G. and Luo, H. (2021) Vaspin Regulated Cartilage Cholesterol Metabolism through miR155/LXRα and Participated in the Occurrence of Osteoarthritis in Rats. Life Sciences, 269, Article ID: 119096. https://doi.org/10.1016/j.lfs.2021.119096
- 15. Farnaghi, S., Crawford, R., Xiao, Y. and Prasadam, I. (2017) Cho-lesterol Metabolism in Pathogenesis of Osteoarthritis Disease. International Journal of Rheumatic Diseases, 20, 131-140. https://doi.org/10.1111/1756-185X.13061
- 16. Choi, W.S., Lee, G., Song, W.H., et al. (2019) The CH25H-CYP7B1-RORα Axis of Cholesterol Metabolism Regulates Osteoarthritis. Nature, 566, 254-258. https://doi.org/10.1038/s41586-019-0920-1
- 17. Kan, B., Zhao, Q., Wang, L., Xue, S., Cai, H. and Yang, S. (2021) Association between Lipid Biomarkers and Osteoporosis: A Cross-Sectional Study. BMC Musculoskeletal Disorders, 22, Article No. 759. https://doi.org/10.1186/s12891-021-04643-5
- 18. 张倩茹, 查晴, 陈辉, 张煜, 杨克, 刘艳. AOX1调控平滑肌细胞表型转换参与动脉钙化的研究[J]. 中国实验诊断学, 2023, 27(5): 572-577.
NOTES
*通讯作者。
