Advances in Clinical Medicine
Vol.
11
No.
07
(
2021
), Article ID:
43813
,
14
pages
10.12677/ACM.2021.117441
LncRNA XIST、miR-204及FN1在甲状腺乳头状癌中的表达及意义
孙茂林1,张文伟1,姜立伟1,臧传善1,秦作荣1,王效军1,孙彦1,刘明波2*,邱杰1*
1青岛大学附属医院,山东 青岛
2中国人民解放军总医院海南医院,海南 三亚

收稿日期:2021年6月7日;录用日期:2021年6月28日;发布日期:2021年7月12日

摘要
目的:探讨LncRNA XIST,miR-204及FN1在PTC组织和癌旁组织及LncRNA XIST在PTC细胞系中的表达及意义。方法:应用Real-time PCR技术检测20例甲状腺乳头状癌和其癌旁组织中LncRNA XIST,miR-204及FN1的表达情况,应用QPCR检测3株甲状腺癌细胞系(BCPAP, TPC-1, KTC-1) LncRNA XIST、miR-204-5p的表达水平,应用荧光原位杂交(FISH)检测PTC细胞系BCPAP中LncRNA XIST的表达定位,应用双荧光素酶报告基因检测人293T细胞的LncRNA XIST、miR-204及FN1相互作用。结果:与癌旁组相比,PTC癌组织组的LncRNA XIST的表达水平显著上调(p < 0.05),PTC癌组织组的FN1的表达水平极显著上调(p < 0.01),而PTC癌组织组的miR-204-5p的表达水平极显著下调(p < 0.01),差异均具有统计学意义;BCPAP细胞的LncRNA XIST的表达水平最高,且miR-204-5p的表达水平最低;LncRNA XIST主要在细胞质中表达;LncRNA XIST与miR-204有相互作用(p < 0.05),FN1与miR-204有相互作用(p < 0.05)。结论:LncRNA XIST在甲状腺乳头状癌中高表达,miR-204在甲状腺乳头状癌中低表达,FN1在甲状腺乳头状癌中高表达;LncRNA XIST,miR-204及FN1相互作用,参与甲状腺乳头状癌的发生发展,并可为甲状腺乳头状癌潜在诊断标志物和治疗靶点的寻找提供理论依据,LncRNA XIST可能是甲状腺乳头状癌诊断和治疗的一个新靶点。
关键词
LncRNA XIST基因,miR-204基因,FN1基因,甲状腺乳头状癌
Expression and Significance of LncRNA XIST, miR-204 and FN1 in Papillary Thyroid Carcinoma
Maolin Sun1, Wenwei Zhang1, Liwei Jiang1, Chuanshan Zang1, Zuorong Qin1, Xiaojun Wang1, Yan Sun1, Mingbo Liu2*, Jie Qiu1*
1Affiliated Hospital of Qingdao University, Qingdao Shandong
2Hainan Hospital of Chinese People’s Liberation Army General Hospital, Sanya Hainan

Received: Jun. 7th, 2021; accepted: Jun. 28th, 2021; published: Jul. 12th, 2021

ABSTRACT
Objective: To investigate the expression and significance of LncRNA XIST, miR-204 and FN1 in PTC tissues and adjacent tissues, and LncRNA XIST in PTC cell lines. Method: The expression of LncRNA XIST, miR-204 and FN1 in 20 cases of papillary thyroid carcinoma and its adjacent tissues were detected by Real-time PCR technology, and 3 thyroid cancer cell lines (BCPAP, TPC-1, KTC-1) were detected by QPCR, the expression levels of LncRNA XIST and miR-204-5p were detected by fluorescence in situ hybridization (FISH) to detect the expression and location of LncRNA XIST in the PTC cell line BCPAP, and double luciferase reporter gene was used to detect the interaction of LncRNA XIST, miR-204 and FN1 in human 293T cells. Result: The expression level of LncRNA XIST in the PTC cancer tissue group was significantly increased in comparison with the adjacent group (p < 0.05), the expression level of FN1 in the PTC cancer tissue group was extremely significantly up-regulated (p < 0.01), while the expression level of miR-204-5p in the PTC cancer tissue group was extremely significantly down-regulated (p < 0.01), the differences are statistically significant;the expression level of LncRNA XIST in BCPAP cells was the highest, and the expression level of miR-204-5p was the lowest; LncRNA XIST was mainly expressed in the cytoplasm; LncRNA XIST interacted with miR-204 (p < 0.05), and FN1 interacted with miR-204 (p < 0.05). Conclusion: LncRNA XIST is highly expressed in papillary thyroid carcinoma, miR-204 is low expressed in papillary thyroid carcinoma, and FN1 is highly expressed in papillary thyroid carcinoma; the interaction between LncRNA XIST, miR-204 and FN1 is involved in the occurrence and development of thyroid papillary carcinoma, and can provide a theoretical basis for the search of potential diagnostic markers and therapeutic targets for thyroid papillary carcinoma. LncRNA XIST may be a new target for the diagnosis and treatment of thyroid papillary carcinoma.
Keywords:LncRNA XIST Gene, miR-204 Gene, FN1 Gene, PTC

Copyright © 2021 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
甲状腺癌是最常见的甲状腺恶性肿瘤,全身恶性肿瘤中约3%是甲状腺癌,甲状腺癌的发病率近年来呈上升趋势 [1]。甲状腺癌按照病理分类可分为甲状腺乳头状癌(PTC)、未分化癌(ATC)、髓样癌(MTC)及滤泡状癌(FTC) [2] [3]。其中,PTC是最常见的甲状腺癌类型,占所有甲状腺癌病例的75%至85% [4],与其他病理类型相比,PTC分化好,生长缓慢,恶性程度低,因此总体预后相对较好,5年生存率可达96%以上 [5]。流行病学研究发现,相对男性而言,女性更容易罹患PTC,男女患病比例约为1:2.5 [6]。然而,PTC可能较早出现颈部淋巴结转移,因此,尽早诊断PTC并加以积极治疗非常重要。
临床上PTC的诊断一般通过病理切片证实。典型的PTC具有特征性核形态的乳头状结构,在病理切片中清晰可见。然而,仅仅通过病理学诊断,可能会将PTC与其他甲状腺良性病变,如结节性甲状腺肿、乳头状生长的甲状腺腺瘤或桥本氏甲状腺炎混淆,造成误诊及过度治疗。因此,近几年来逐渐发展出一些PTC和良性甲状腺肿瘤伴有乳头状增生的鉴别诊断的标志物,如CK19、Galectin-3及HBME1 [7]。但由于它们的敏感性和特异性相对较低,在临床应用上受到限制。有研究显示BrafV600E基因在甲状腺癌中的突变率很高,且特异性较高,可用于甲状腺癌的早期诊断 [8],但对于甲状腺癌具体分型的诊断仍需探索,因此,寻找新的PTC潜在诊断标志物,有利于PTC早期诊断,具有重要的临床意义。
非编码RNA (Non-coding RNAs)即不能翻译成蛋白质的功能性RNA分子,约占人类基因组的98%,广泛参与人体生理、病理活动,与多种肿瘤密切相关。根据RNA分子大小,非编码RNA可分为长链非编码RNA (long non-coding RNA, lncRNA)和短链非编码RNA (包括miRNA、siRNA和piRNA)两类 [9]。LncRNA是一类转录本长度超过200个核苷酸的功能性RNA分子。由于其缺少编码蛋白质的能力,一度被认为是“垃圾”RNA [10]。而随着研究的深入,研究人员逐渐发现,lncRNA是生命体中含有丰富信息的“暗物质”,这些信息仍有待探索。证据表明,lncRNA通过多种方式(如转录调控、转录后调控及表观遗传学等)调控基因的表达水平,从而在生物的发育、癌症、疼痛和炎症等过程发挥作用 [11]。MiRNA是短链非编码RNA,长度一般为18~22个核苷酸,主要通过其“种子序列”与靶基因3’端非翻译区域互补结合,裂解mRNA或抑制其翻译过程,从而参与肿瘤发生和发展 [12]。作为非编码RNA研究领域的热点,越来越多的研究表明曾被认为是基因组转录“噪音”的lncRNA逐步成为解释疾病发生、发展机制,作为诊断、预后判断和治疗靶点的新的候选分子。此研究旨在验证PTC中LncRNA XIST、miR-204-5p和FN1的表达、相互作用及细胞定位,寻找甲状腺乳头状癌潜在诊断标志物,并为临床靶向治疗PTC提供新资料、方法奠定基础。
2. 资料与方法
2.1. 研究对象
① 收集2019年7月至2020年8月期间在青岛大学附属医院行甲状腺切除患者的手术标本,所有患者均有完整临床资料。将所有患者分为甲癌和癌旁2组,甲状腺乳头状癌(甲癌组) 20例、配对癌旁正常组织(癌旁组) (癌旁组织取自距癌灶边缘 > 0.8 cm的位置) 20例,以上所有甲状腺标本均经我院病理科确诊,并且患者均签署实验知情同意书。② 3株甲状腺乳头状癌细胞系(BCPAP, TPC-1, KTC-1)。③ 人293T细胞。
2.2. 纳入标准
① 各项临床资料完整;② 无其他恶性肿瘤病史;③ 无桥本甲状腺炎病史;④ 无内分泌相关疾病病史;⑤ 术前无放疗、化疗治疗史及甲状腺手术史;⑥ 无严重的心脑血管疾病。
2.3. 研究方法
2.3.1. 仪器和试剂
RNA提取试剂等来自TAKARA公司,PCR反应膜来自ABI公司,DMEM培养基等来自Gibco公司;BCPAP、TPC-1、KTC-1细胞及293T细胞来自中科院细胞库,FISH探针等来自广州锐博生物,引物由上海生工合成,PAGE纯化。
2.3.2. 实验方法
1) PTC癌组织及其癌旁组织中LncRNA XIST,miR-204及FN1表达水平检测
将新鲜组织50~100 mg研磨至糜状,Trizol法提取组织总RNA,而后进行逆转录得到RNA总量,将产物cDNA取出后利用无酶水稀释至500 μl并保存待用,miRNA逆转录反应体系为10 μl,包含RT-Primer (10 μM) 3 μl、dNTP Mixture (10 mM each) 1 μl、RNA 500~1000 ng、RNase Free 水up to 10 μl,反应条件:65℃,5 min,冰上预冷。在上述反应管中配制下列反转录反应液,总量为20 μl,包括上述变性后反应液10 μl、5 × PrimeScript II Buffer 4 μl、RNase Inhibitor (40 U/μl) 0.5 μl (20 U)、PrimeScript II RTase (200 U/μl) 1 μl (200 U)、RNase Free水up to 20 μl,移液器轻轻混匀上述配制的反应液,短暂离心后按下列条件进行反转录反应:42℃,60 min,95℃,5 min (酶失活),将反转录产物放在冰上冷却备用。mRNA逆转录反应体系为20 μl,包括5 × primeScript RT Master Mix (perfect Real Time) 4 μl、Total RNA 0.5 μg、RNase Free水up to 20 μl,反应条件:37℃,60 min,85℃,5 s,将反转录产物放在冰上备用。而后进行qPCR检测,其中lncRNA XIST正向引物:5’-GACACAAGGCCAACGACCTA-3’,反向引物: 5’-TCGCTTGGGTCCTCTATCCA-3’;hsa-miR-204-5p正向引物: 5’-GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACaggcat-3’;hsa-miR-204-5p反向引物:GCGCGttccctttgtcatcct,引物由上海生工合成,PAGE纯化,PCR反应条件:温度50℃、2 min;95℃、2 min;95℃、15 s;60℃、60 s,共重复40个循环,每个样本重复测量3次取平均Ct值,相对定量采用2-ΔΔCt法,ΔCt = Ct (目的基因) − Ct (管家基因),ΔΔCt = 实验组ΔCt − 对照组ΔCt。
2) QPCR检测3株甲状腺癌细胞系(BCPAP, TPC-1, KTC-1) lncRNA XIST、miR-204-5p的表达水平
(1) RNA提取:① 将收集的细胞样本,加入1000 μl RNAiso Plus (Trizol)充分裂解,随后加入200 μl氯仿,振荡混匀后室温放置5 min。② 4℃,12000 g离心15 min,吸取上层水相,至另一离心管中。③ 加入0.5 ml异丙醇混匀,4℃,12000 g离心10 min,弃上清,RNA沉于管底。④ 加入1 ml 75%乙醇,温和颠倒离心管,4℃,7500 g离心5 min,尽量弃上清。⑤ 室温晾干10 min,用20 μl DEPC H2O溶解RNA沉淀。⑥ 检测RNA纯度和浓度:以DEPC H2O为对照(Blank),取2 μl RNA溶液于分光光度计检测。DEPC H2O为对照(Blank),取2 μl RNA溶液于酶标仪上检测样本浓度和质量。
(2) miRNA反转录:按照试剂盒说明书,配制如下10 μl体系:RT-Primer (10 μM),3 μl;dNTP Mixture (10 mM each),1 μl;RNA,500~1000 ng;RNase Free水,up to 10 μl。反应条件:65℃,5 min,冰上预冷。在上述反应管中配制下列反转录反应液,总量为20 μl:上述变性后反应液10 μl;5 × PrimeScript II Buffer,4 μl;RNase Inhibitor (40 U/μl),0.5 μl (20 U);PrimeScript II RTase (200 U/μl),1 μl (200 U);RNase Free水,up to 20 μl。移液器轻轻混匀上述配制的反应液,短暂离心后按下列条件进行反转录反应:42℃,60 min;95℃,5 min (酶失活),将反转录产物放在冰上冷却备用。
(3) mRNA反转录:按照试剂盒说明书,配制如下20 μl体系:5 × primeScript RT Master Mix (perfect Real Time),4 μl;Total RNA,0.5 μg;RNase Free水,up to 20 μl。反应条件:37℃,60 min;85℃,5 s,将反转录产物放在冰上备用。
(4) 荧光定量PCR扩增:按照试剂盒说明书,配制如下PCR扩增反应体系:SYBR Premix EX Taq (2×),10 μl;Forward primer,10 µM,1 μl;Reverse primer,10 µM,1 μl;cDNA,2 μl;ddH2O,up to 20 μl。用ABI ViiA7荧光定量PCR仪进行Real time PCR反应,程序如下:50.0℃,2 min;95.0℃,2 min;95.0℃ for 15 s;60.0℃ for 60 s,共重复40个循环。熔解条件:95.0℃ for 15 s;60.0℃ for 60 s;95.0℃ for 15 s。
引物信息
3) BCPAP细胞培养及荧光原位杂交(FISH)
BCPAP细胞培养:当细胞密度达80%~90%时进行传代。在超净工作台中吸弃细胞培养上清,用无菌1×PBS洗涤细胞1次,加入胰蛋白酶1~2 ml,轻轻晃动,保证胰蛋白酶充分接触细胞,在倒置显微镜下观察,细胞间隙增大开始脱落时,加入新鲜培养液终止消化,滴管吹打细胞使其全部脱落,转移细胞悬液于离心管中,1000 rpm离心5 min,然后弃去上清,加新鲜的培养液并吹打成细胞悬液,按1:2传代至新培养瓶中,补充培养液,继续培养。细胞培养基:1640 + 10% FBS + 1% PS。
荧光原位杂交(FISH)实验:(1) 细胞培养:将细胞爬片置于24孔板孔底,培养适量细胞(6 × 104/孔)。实验前使细胞融合度达到60%左右。(2) 细胞固定与通透:a. 用1× PBS清洗细胞5 min;b. 用4%多聚甲醛室温固定10 min;c. 用1× PBS清洗细胞5 min,共3次;d. 每孔加入1 ml预冷的通透液(含0.5% Triton X-100的PBS),4℃静置5 min;e. 弃去通透液后,加入1 × PBS清洗细胞5 min,3次。(3) 探针检测:a. 每孔加入200 μl预杂交液,37℃封闭30 min;b. 弃去每孔细胞中的预杂交液,加入100 μL含有探针的探针杂交液(避光条件下,把2.5 μl 20 μM FISH Probe Mix储存液加入到100 μl杂交液中),避光,37℃ 杂交过夜;c. 避光,42℃,杂交洗液I清洗每孔细胞3次,每次5 min;d. 避光,42℃,杂交洗液II清洗细胞1次;e. 避光,42℃,杂交洗液III清洗细胞1次;f. 避光,1× PBS清洗细胞,室温5 min。(4) 封片:避光条件下,从孔中小心取出细胞爬片,用封片剂(含DAPI)将其固定于载玻片上,荧光显微镜拍照检测。
4) 293T细胞复苏、传代及双荧光素酶报告基因检测
293T细胞复苏及传代:从液氮罐中取出293T细胞,迅速置于37℃水浴,并不断振荡使其融化,消毒冻存管表面后,在超净台中将细胞悬液转移至无菌离心管中,补加培养液5 ml,轻轻吹打成细胞悬液后,1000 r/min离心5 min,弃上清,加入培养液吹打,吸取悬液至25 ml培养瓶内,置于细胞培养箱中培养,次日换液,观察细胞贴壁情况。细胞培养液配制如下:45 ml DMEM + 5 ml FBS + 500 μl双抗。当细胞密度达80%~90%时进行传代。在超净工作台中吸弃细胞培养上清,用无菌1 × PBS洗涤细胞1次,加入胰蛋白酶1 ml,轻轻晃动,保证胰蛋白酶充分接触细胞,在倒置显微镜下观察,细胞间隙增大开始脱落时,加入新鲜培养液终止消化,滴管吹打细胞使其全部脱落,转移细胞悬液于离心管中,1000 rpm离心5 min,然后弃去上清,加新鲜的培养液并吹打成细胞悬液,按1:2传代至新培养瓶中,补充培养液,继续培养。
质粒构建及抽提:化学合成人LncRNA XIST序列,连接至psiCHECK2质粒中,构建psiCHECK2-lncRNA XIST-WT和psiCHECK2-lncRNA XIST-MUT质粒;
化学合成人FN1 3’UTR WT (野生型)及MUT (突变)序列,连接至pGL3-promoter质粒中,构建pGL3-FN1-WT和pGL3-FN1-MUT质粒。
摇菌:配制LB培养液:称取7.5 g LB Broth Medium,加入300 ml ddH2O,混匀后,高温高压消毒灭菌后室温放置。用无菌牙签或枪头挑穿刺菌落,至含有抗生素的LB液体培养基(15~30 ml)中,摇菌12~14 hr左右(220 rpm,37℃)。
质粒提取:具体步骤参见Omega试剂盒步骤:(1) 取10~15 ml的菌液,室温下4000g离心10分钟,收集菌体。(2) 弃培养液,往沉淀中加入500 μl Solution I/RNase A,涡旋振荡使细胞完全重新悬浮。(3) 将细胞悬液转移至新的2 ml离心管中。(4) 加入500 μl Soulution II,轻柔的上下颠倒混匀8~10次,室温静置2~3分钟,不应超过5分钟。(5) 加入250 μl预冷的N3 buffer,轻柔的上下颠倒混匀离心管数次,直至形成白色絮状沉淀。室温静置2分钟。(6) 室温13000g离心10分钟。(7) 收集上清到新的1.5 ml离心管中。(8) 加入0.1倍体积的ETR Solution,上下颠倒混匀10次,裂解液变浑浊,然后冰浴静置10分钟后将逐渐澄清。(9) 将上述裂解液于42℃水浴5分钟。裂解液将再次出现浑浊。(10) 室温12000g离心3分钟。ETR溶液将在底部形成蓝色分层。(11) 将上清转移至新的1.5 ml离心管中,加入0.5倍体积的无水乙醇,轻轻上下颠倒混匀6~7次,室温静置2分钟。(12) 把HiBind DNA MiniColumn II套入2 ml收集管中。(13) 转移步骤11得到的混合液700 μl到结合柱中,室温13000g离心1分钟,弃滤液。(14) 重复步骤13,直至步骤11所得混合液全部结合到结合柱中。(15) 加入500 μl HBC Buffer,室温13000g离心1分钟,弃滤液。(16) 加入700 μl DNA Wash Buffer,室温13000g离心1分钟,弃滤液。(17) 加入700 μl DNA Wash Buffer,室温13000 g离心1分钟,弃滤液。(18) 室温13000 g离心1分钟,空甩。(19) 把HiBind DNA MiniColumn II套入新的1.5 ml离心管,加入120 μl H2O,室温下静置3分钟,室温13000g离心1分钟。(20) 用紫外分光光度计测量质粒浓度及纯度。−20℃保存。
双荧光素酶报告基因检测:细胞种板:收集培养至对数生长期的293T细胞,用无PS的完全培养液重悬细胞,细胞计数,按每孔1 × 104个细胞,将细胞接种至96孔板中,放置细胞培养箱中静置培养过夜。转染:(1) 细胞换成无血清培养基,每孔500 μl (2) a1:Lipofectamin 3000 (0.2 μl/孔) + 无血清Opti-MEM培养基(10 μl/孔);b1:miRNA-204-5p mimic或NC (100 nM) + psiCHECK2-LncRNA XIST-WT或psiCHECK2-LncRNA XIST-MUT或psiCHECK2空载(0.4 μg/孔) + 无血清OMEM培养基(10 μl/孔);a2:Lipofectamin 3000 (0.2 μl/孔) + 无血清OMEM培养基(10 μl/孔);b2:miRNA-204-5p mimic或NC (100 nM) + pGL3-FN1 3’UTR WT或pGL3-FN1 3’UTR MUT或pGL3-Promoter空载(0.4 μg/孔) + pRL-TK (0.2 μg/孔) + 无血清OMEM培养基(10 μl/孔),室温静置5 min。(3) 将上述中的a1和b1混合或a2和b2混合,室温静置20 min。(4) 更换细胞培养基为无血清培养基,加入制备的转染混合液。6小时后换液,加入完全培养基常规培养48 hours,以未转染组为阴性对照组。收样、酶标仪检测:吸尽细胞培养液后,加入100 μl的报告基因细胞裂解液,充分裂解后,10,000~15,000g离心3~5分钟,取上清用于测定。融解萤火虫萤光素酶检测试剂和海肾萤光素酶检测缓冲液,并达到室温。海肾萤光素酶检测底物(100×)置于冰浴或冰盒上备用。按照每个样品需100 μl的量,取适量海肾萤光素酶检测缓冲液,按照1:100加入海肾萤光素酶检测底物(100×)配制成海肾萤光素酶检测工作液。按仪器操作说明书开启化学发光仪或具有检测化学发光功能的多功能酶标仪,将测定间隔设为2秒,测定时间设为10秒。每个样品测定时,取样品50 μl,加入100 μl萤火虫萤光素酶检测试剂,用枪打匀或用其它适当方式混匀后测定RLU (relative light unit)。在完成上述测定萤火虫萤光素酶步骤后,加入100微升海肾萤光素酶检测工作液,用枪打匀或用其它适当方式混匀后测定RLU (relative light unit)。
2.4. 统计学处理
作图软件为Graphpad prism 5 (Graphpad Software, San Diego, CA);荧光定量软件为Image J,数据分析软件Graphpad prism 5 (Graphpad Software, San Diego, CA);t-test分析LncRNA XIST,miR-204及FN1三者在PTC癌组织及癌旁组织的表达差异(表1),以单因素方差分析和TUKEY法进行事后检验研究LncRNA XIST,miR-204在BCPAP、TPC-1及KTC-1三组细胞系中的表达差异(表2),以双因素方差分析和TUKEY法进行事后检验研究人293T细胞中LncRNA XIST与miR-204、FN1与miR-204的关系(表3)。以P < 0.05和P < 0.01为显著性差异和极显著性差异的筛选标准。
3. 结果
① 甲状腺乳头状癌及癌旁组织中LncRNA XIST和miR-204-5p的表达比较:荧光定量PCR结果表明,与癌旁组相比,癌组织组的lncRNA XIST的表达水平显著上调(p < 0.05) (图1);与癌旁组相比,癌组织组的miR-204-5p的表达水平极显著下调(p < 0.01) (图2);与癌旁组相比,癌组织组的FN1的表达水平极显著上调(p < 0.01) (图3),差异均具有统计学意义。
② 荧光定量PCR结果表明,三组细胞系(BCPAP、TPC-1及KTC-1)中,lncRNA XIST的表达,BCPAP细胞表达水平比TPC-1细胞(p < 0.05)及KTC-1细胞(p < 0.01)显著上调,且miR-204-5p的表达,BCPAP细胞表达水平比TPC-1细胞(p < 0.05)及KTC-1细胞(p < 0.01)显著下调,差异均具有统计学意义。BCPAP细胞的lncRNA XIST的表达水平最高(图4),且miR-204-5p的表达水平最低(图5),故选择该细胞开展后续实验。
③ LncRNA XIST主要在细胞质中表达(图6~8)。
④ LncRNA XIST与miR-204有相互作用(p < 0.05),FN1与miR-204有相互作用(p < 0.05) (图10~图13)。
Table 1. LncRNA XIST, miR-204 and FN1 t-test
表1. LncRNA XIST,miR-204和FN1 t-test

Figure 1. Expression of LncRNA XIST in papillary thyroid carcinoma and adjacent tissues
图1. 甲状腺乳头状癌及癌旁组织中LncRNA XIST的表达

Figure 2. The expression of miR-204-5p in papillary thyroid carcinoma and adjacent tissues
图2. 甲状腺乳头状癌及癌旁组织中miR-204-5p的表达

Figure 3. Expression of FN1 in papillary thyroid carcinoma and adjacent tissues
图3. 甲状腺乳头状癌及癌旁组织中FN1的表达

Figure 4. The expression of LncRNA XIST in BCPAP, TPC-1 and KTC-1
图4. BCPAP、TPC-1及KTC-1中LncRNA XIST的表达
Table 2. LncRNA XIST, miR-204 one-way analysis of variance
表2. LncRNA XIST,miR-204单因素方差分析

Figure 5. The expression of miR-204-5p in BCPAP, TPC-1 and KTC-1
图5. BCPAP、TPC-1及KTC-1中miR-204-5p的表达

Figure 6. Results of in situ hybridization (FISH)—LncRNA XIST (300×)
图6. 原位杂交(FISH)结果——LncRNA XIST (300×)

Figure 7. Results of in situ hybridization (FISH)—DAPI (300×)
图7. 原位杂交(FISH)结果——DAPI (300×)

Figure 8. Results of in situ hybridization (FISH)—Merge (300×)
图8. 原位杂交(FISH)结果——Merge (300×)

Figure 9. LncRNA XIST indirectly regulates the expression of FN1 by competitively binding miR-204, and participates in the development of PTC
图9. LncRNA XIST通过竞争性结合miR-204,间接调控FN1的表达,参与PTC发生发展
Table 3. Two-way analysis of variance of LncRNA XIST, miR-204 and FN1
表3. LncRNA XIST、miR-204及FN1双因素方差分析
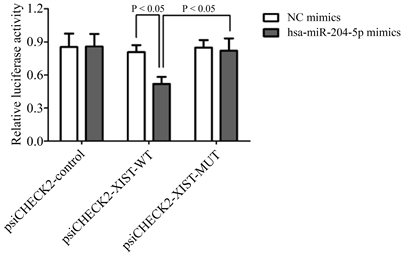
Figure 10. LncRNA XIST interacts with miR-204
图10. LncRNA XIST与miR-204有相互作用

Figure 11. FN1 interacts with miR-204
图11. FN1与miR-204有相互作用

Figure 12. LncRNA XIST and miR-204 dual luciferase reporter gene detection results
图12. LncRNA XIST与miR-204的双荧光素酶报告基因检测结果

Figure 13. FN1 and miR-204 dual luciferase reporter gene detection results
图13. FN1与miR-204的双荧光素酶报告基因检测结果
4. 讨论
LncRNA XIST位于X染色体上,是最早发现的非编码RNA之一。早在20年前,它就被确定为一个沉默因子。研究人员发现lncRNA XIST通过在X染色体上扩展来沉默基因 [13]。Engreitz等人研究发现Xist在诱导X染色体失活期间,通过利用X染色体的三维构象识别结合区域,在X染色体广泛结合,从而阻止X染色体上的基因转录 [14]。进一步的研究发现,lncRNA XIST通过与SHARP直接结合,招募SMRT,激活了HDAC3,将组蛋白去乙酰化以使RNA聚合酶II不能结合X染色体 [15]。因此,我们推测lncRNA XIST在PTC中异常高表达可能是PTC在女性患者中发病率显著高于男性的原因之一。
miR-204是定位于人类第9号染色体73424891~73425000位之间。在肿瘤组织中miR-204存在异常表达,miR-204与肿瘤增殖、转移、化疗抵抗及不良预后关系密切 [16] [17] [18] [19]。
FN1是在血浆中发现的细胞外基质可溶性蛋白质,在伤口愈合、胚胎发育等EMT相关过程中表达量均有增加。上皮–间质细胞转化(Epithelial-mesenchymal transition, EMT)是指上皮细胞获得特定的遗传变化及生物学特性,并逐步转变为间充质表型的过程。在胚胎发育、组织重建、慢性炎症以及癌症发生及转移等疾病中发挥了重要作用。在EMT期间,上皮细胞失去细胞极性并实现间充质表型,从而增加细胞侵袭和迁移能力 [20]。EMT主要特征有上皮细胞标志物表达量降低,如E-钙黏蛋白(E-cadherin),而间质细胞标志物表达量增加,如N-钙黏蛋白(N-cadherin),FN1、Snail、波形蛋白(Vimentin) [21]。除了我们所研究的PTC外,在乳腺癌、头颈部鳞状细胞癌、恶性外周神经鞘瘤以及鼻咽癌等等多种恶性肿瘤中均发现FN1的表达增高 [22] [23] [24] [25]。
大量文献报导,lncRNA XIST参与了多种癌症的发生发展 [26] [27]。研究发现,lncRNA可能通过诱导基因的表观遗传学修饰或作为miRNA“海绵”吸附miRNA,通过调控靶基因的表达的方式导致疾病发生,这种机制也被称为竞争性内源性RNA (competing endogenous RNA, ceRNA)机制 [27]。本实验结果提示,与癌旁组相比,癌组织组的lncRNA XIST和FN1的表达水平显著上调,而癌组织组的miR-204-5p的表达水平极显著下调,查阅文献发现,Ab Mutalib等人通过分析TCGA的数据,筛选出hsa-miR-204在PTC中显著下调 [28]。实验结果显示LncRNA XIST与miR-204及FN1与miR-204有相互作用,同时应用Starbase 2.0数据库,我们发现lncRNA XIST可以与miR-204结合,且XIST与miR-204结合的位点和miR-204与FN1结合位点有重叠部分,说明XIST与FN1可能存在竞争性结合miR-204的关系(图9),因此,我们推测lncRNA XIST在PTC中异常高表达可以吸附部分miR-204应答原件,从而导致PTC的发生发展。我们将在未来的研究中证明这些推测。同时,我们通过荧光原位杂交(FISH)实验证实,LncRNA XIST主要在细胞质中表达,这将有利于后续实验的开展,便于后续研究其通路,探究其对PTC细胞生物学功能的影响。
综上所述,实验证实LncRNA XIST和FN1在甲状腺乳头状癌中高表达,miR-204在甲状腺乳头状癌中低表达,lncRNA XIST主要在细胞质中表达,我们推测lncRNA XIST在PTC中异常高表达可以吸附部分miR-204,竞争性阻止miR-204对FN1的抑制作用,从而促进FN1的高表达,进而扰乱上皮–间质细胞转化等重要的癌症信号通路,最终导致了PTC的发生(图9)。lncRNA 通过多种方式调控miRNA的表达,从而导致肿瘤发生发展 [29],其中一种为lncRNA可发挥内源性 miRNA海绵(miRNA sponge) 的功能,抑制miRNA的表达进而影响肿瘤细胞的恶性生物学行为 [30]。因此,下调LncRNA XIST会降低甲状腺乳头状癌细胞的增殖及上调miR-204-5p [31],相反,我们在低表达PTC细胞中过表达LncRNA XIST可能会导致甲状腺乳头状癌的增殖以及侵袭迁移能力增强 [32]。另外,LncRNA XIST主要在细胞质中表达,便于后续研究其通路,探究其对细胞生物学功能的影响,沉默LncRNA XIST有望成为甲状腺乳头状癌治疗新的生物学靶点 [33] [34],LncRNA XIST可能与甲状腺乳头状癌的转移和恶化等有关 [35] [36] [37],其分子机制仍有待研究。LncRNA XIST,miR-204及FN1相互作用,参与甲状腺乳头状癌的发生发展,并可为甲状腺乳头状癌潜在诊断标志物和治疗靶点的寻找提供理论依据,并且LncRNA XIST可能是甲状腺乳头状癌诊断和治疗的一个新靶点。本实验研究样本量较少,在以后的研究将加大样本量研究LncRNA XIST等基因在PTC中的作用机制。
基金项目
海南省耳鼻咽喉头颈疾病临床医学研究中心项目(项目编号:HN01ENT07)青岛大学附属医院“临床医学 + X”科研项目(项目编号:3400)。
文章引用
孙茂林,张文伟,姜立伟,臧传善,秦作荣,王效军,孙 彦,刘明波,邱 杰. LncRNA XIST、miR-204及FN1在甲状腺乳头状癌中的表达及意义
Expression and Significance of LncRNA XIST, miR-204 and FN1 in Papillary Thyroid Carcinoma[J]. 临床医学进展, 2021, 11(07): 3033-3046. https://doi.org/10.12677/ACM.2021.117441
参考文献
- 1. Siraj, A.K., Hussain, A.R., Alrasheed, M., et al. (2011) Demethylation of TMS1 Gene Sensitizes Thyroid Cancer Cells to TRAIL-Induced Apoptosis. The Journal of Clinical Endocrinology and Metabolism, 96, E215-E224. https://doi.org/10.1210/jc.2010-0790
- 2. Kim, H.I., et al. (2018) Refining the Eighth Edition AJCC TNM Classification and Prognostic Groups for Papillary Thyroid Cancer with Lateral Nodal Metastasis. Oral Oncology, 78, 80-86. https://doi.org/10.1016/j.oraloncology.2018.01.021
- 3. Shteinshnaider, M., et al. (2018) Reassessment of Differentiated Thyroid Cancer Patients Using the Eighth TNM/AJCC Classification System: A Comparative Study. Thyroid, 28, 201-209. https://doi.org/10.1089/thy.2017.0265
- 4. Londero, S.C., et al. (2013) Papillary Thyroid Microcarcinoma in Denmark 1996-2008: A National Study of Epidemiology and Clinical Significance. Thyroid, 23, 1159-1164. https://doi.org/10.1089/thy.2012.0595
- 5. Gong, Y., et al. (2018) MiR-26a Inhibits Thyroid Cancer Cell Proliferation by Targeting ARPP19. American Journal of Cancer Research, 8, 1030-1039.
- 6. Vaisman, F., Corbo, R. and Vaisman, M. (2011) Thyroid Carcinoma in Children and Adolescents-Systematic Review of the Literature. Journal of Thyroid Research, 2011, Article ID: 845362. https://doi.org/10.4061/2011/845362
- 7. Wang, X., et al. (2018) A Potential Biomarker hsa-miR-200a-5p Distinguishing between Benign Thyroid Tumors with Papillary Hyperplasia and Papillary Thyroid Carcinoma. PLoS ONE, 13, e0200290. https://doi.org/10.1371/journal.pone.0200290
- 8. Kim, S.K., Lee, J.H., Woo, J.W., et al. (2015) Prediction Table and Nomogram as Tools for Diagnosis of Papillary Thyroid Carcinoma: Combined Analysis of Ultrasonography, Fine-Needle Aspiration Biopsy, and BRAF V600E Mutation. Medicine, 94, e760. https://doi.org/10.1097/MD.0000000000000760
- 9. Arjumand, W., Asiaf, A. and Ahmad, S.T. (2018) Noncoding RNAs in DNA Damage Response: Opportunities for Cancer Therapeutics. Methods in Molecular Biology, 1699, 3-21. https://doi.org/10.1007/978-1-4939-7435-1_1
- 10. 佘春晖. LncRNA XIST在原发性胆汁性胆管炎免疫细胞中的作用研究[D]: [硕士学位论文]. 青岛: 青岛大学, 2020.
- 11. Spizzo, R., et al. (2012) Long Non-Coding RNAs and Cancer: A New Frontier of Translational Research. Oncogene, 31, 4577-4587. https://doi.org/10.1038/onc.2011.621
- 12. 李墨林, 贾婷婷, 白晗, 李传刚, 李连宏. MicroRNA与甲状腺癌发生、发展关系的研究进展[J]. 临床与实验病理学杂志, 2015, 31(7): 794-798.
- 13. Plath, K., et al. (2002) Xist RNA and the Mechanism of X Chromosome Inactivation. Annual Review of Genetics, 36, 233-278. https://doi.org/10.1146/annurev.genet.36.042902.092433
- 14. Engreitz, J.M., et al. (2013) The Xist lncRNA Exploits Three-Dimensional Genome Architecture to Spread across the X Chromosome. Science, 341, Article ID: 1237973. https://doi.org/10.1126/science.1237973
- 15. McHugh, C.A., et al. (2015) The Xist lncRNA Interacts Directly with SHARP to Silence Transcription through HDAC3. Nature, 521, 232-236. https://doi.org/10.1038/nature14443
- 16. Ma, L., Deng, X., Wu, M., et al. (2014) Down-Regulation of miRNA-204 by LMP-1 Enhances CDC42 Activity and Facilitates Invasion of EBV-Associated Nasopharyngeal Carcinoma Cells. FEBS Letters, 588, 1562-1570. https://doi.org/10.1016/j.febslet.2014.02.039
- 17. Vimalraj, S., Miranda, P.J., Ramyakrishna, B., et al. (2013) Regulation of Breast Cancer and Bone Metastasis by MicroRNAs. Disease Markers, 35, 369-387. https://doi.org/10.1155/2013/451248
- 18. Ryan, J., Tivnan, A., Fay, J., et al. (2012) microRNA-204 Increases Sensitivity of Neuroblastoma Cells to Cisplatin and Is Associated with a Favourable Clinical Outcome. British Journal of Cancer, 107, 967-976. https://doi.org/10.1038/bjc.2012.356
- 19. Zeng, L., Yu, J., Huang, T., et al. (2012) Differential Combinatorial Regulatory Network Analysis Related to Venous Metastasis of Hepatocellular Carcinoma. BMC Genomics, 13, S14. https://doi.org/10.1186/1471-2164-13-S8-S14
- 20. Polyak, K. and Weinberg, R.A. (2009) Transitions between Epithelial and Mesenchymal States: Acquisition of Malignant and Stem Cell Traits. Nature Reviews Cancer, 9, 265-273. https://doi.org/10.1038/nrc2620
- 21. Sampayo, R.G., et al. (2018) Fibronectin Rescues Estrogen Receptor Alpha from Lysosomal Degradation in Breast Cancer Cells. Journal of Cell Biology, 217, 2777-2798. https://doi.org/10.1083/jcb.201703037
- 22. Ruiz-Garcia, E., et al. (2010) Gene Expression Profiling Identifies Fibronectin 1 and CXCL9 as Candidate Biomarkers for Breast Cancer Screening. British Journal of Cancer, 102, 462-468. https://doi.org/10.1038/sj.bjc.6605511
- 23. Jerhammar, F., et al. (2010) Fibronectin 1 Is a Potential Biomarker for Radioresistance in Head and Neck Squamous Cell Carcinoma. Cancer Biology & Therapy, 10, 1244-1251. https://doi.org/10.4161/cbt.10.12.13432
- 24. Karpinsky, G., et al. (2018) Tumor Expression of Survivin, p53, Cyclin D1, Osteopontin and Fibronectin in Predicting the Response to Neo-Adjuvant Chemotherapy in Children with Advanced Malignant Peripheral Nerve Sheath Tumor. Journal of Cancer Research and Clinical Oncology, 144, 519-529. https://doi.org/10.1007/s00432-018-2580-1
- 25. Wang, J., et al. (2017) High Expression of Fibronectin 1 Suppresses Apoptosis through the NF-kappaB Pathway and Is Associated with Migration in Nasopharyngeal Carcinoma. American Journal of Translational Research, 9, 4502-4511.
- 26. Sun, K., Jia, Z., Duan, R., et al. (2019) Long Non-Coding RNA XIST Regulates miR-106b-5p/P21 Axis to Suppress Tumor Progression in Renal Cell Carcinoma. Biochemical and Biophysical Research Communications, 510, 416-420. https://doi.org/10.1016/j.bbrc.2019.01.116
- 27. 邢益桓, 付斌, 夏鹰. lncRNA XIST介导的ceRNA调控网络在恶性肿瘤中作用的研究进展[J]. 中国肿瘤生物治疗杂志, 2020, 27(9): 1062-1067.
- 28. Wapinski, O. and Chang, H.Y. (2011) Long Noncoding RNAs and Human Disease. Trends in Cell Biology, 21, 354-361. https://doi.org/10.1016/j.tcb.2011.04.001
- 29. Ab Mutalib, N.S., et al. (2016) Integrated microRNA, Gene Expression and Transcription Factors Signature in Papillary Thyroid Cancer with Lymph Node Metastasis. PeerJ, 4, e2119. https://doi.org/10.7717/peerj.2119
- 30. 涂超峰, 武明花, 李桂源. lncRNA与miRNA相互调控作用及其与肿瘤的关系[J]. 中国生物化学与分子生物学报, 2013, 29(11): 1029-1034.
- 31. Wang, J.Y., Liu, X.F., Wu, H.C., Ni, P.H., Gu, Z.D., Qiao, Y.X., Chen, N., Sun, F.Y. and Fan, Q.S. (2010) CREB Up-Regulates Long Non-Coding RNA, HULC Expression through Interaction with microRNA-372 in Liver Cancer. Nucleic Acids Research, 38, 5366-5383. https://doi.org/10.1093/nar/gkq285
- 32. 王帅, 李文雅. lncRNA XIST调控miR-486-5p影响食管癌细胞增殖的实验研究[J]. 现代肿瘤医学, 2020(4): 539-543.
- 33. 刘颖. 过表达lncRNA GAS5对甲状腺乳头状癌侵袭和迁移能力的影响及机制研究[J]. 标记免疫分析与临床, 2020, 27(10): 1761-1766+1776.
- 34. 杨梅柳. LncRNA在甲状腺乳头状癌中的表达及功能分析[D]: [博士学位论文]. 石家庄: 河北医科大学, 2016.
- 35. 王科, 王应强, 叶静, 张雪, 钟玲, 杨力, 马世丽. LncRNA RP11-316M1.12在甲状腺乳头状癌中的表达及对细胞侵袭、迁移的影响[J]. 现代生物医学进展, 2020(14): 2646-2650+2622.
- 36. 林冲, 王霄. 基于竞争性内源RNA的甲状腺乳头状癌淋巴结转移差异性lnRNA-miRNA-mRNA差异网络的构建与分析[J]. 饮食保健, 2020, 7(22): 48-49.
- 37. 何亚丽, 毛玉娟, 杨锐, 江华, 谢青, 申进增, 伊琳, 顾远辉. lncRNA在甲状腺乳头状癌中的研究进展[J]. 世界最新医学信息文摘(连续型电子期刊), 2020, 20(68): 65-68.
NOTES
*通讯作者。
