World Journal of Cancer Research
Vol.
09
No.
03
(
2019
), Article ID:
31432
,
8
pages
10.12677/WJCR.2019.93013
CARM1 Regulates the Growth of Hepatocellular Carcinoma Cells via Regulation of Bax/Bcl-2 Level and Caspase-3 Activity
Meiling Liu, Honghai Hong*, Zheng Wang
Department of Clinical Laboratory, Third Affiliated Hospital of Guangzhou Medical University, Guangzhou Guangdong

Received: Jun. 30th, 2019; accepted: Jul. 19th, 2019; published: Jul. 26th, 2019

ABSTRACT
Objective: To investigate the effect and mechanism of CARM1 on the growth of HCC. Methods: qRT-PCR and western blot were used to detect the expression of CARM1. MTT assay and Trypan blue assay were used to detect the growth of hepatocellular carcinoma cells; Caspase-3 kit was used to detect the activity of Caspase-3; small interfering RNA interference CARM1 was used to study its role and molecular mechanism on HCC growth. Results: Small interfering RNA significantly knocked down the level of CARM1; interfering with CARM1 significantly, which inhibited the growth of hepatocellular carcinoma cells, down-regulated Bcl-2, up-regulated Bax expression, and activated Caspase-3 activity. Conclusion: CARM1 regulates the growth of hepatocellular carcinoma by regulating the expression of Bax/Bcl-2 and the activity of Caspase-3.
Keywords:HCC, CARM1, Bcl-2, Bax, Caspase-3
CARM1通过调控Bax/Bcl-2表达和Caspase-3活性进而调控肝癌生长作用
刘美玲,洪宏海*,王征
广州医科大学附属第三医院检验科,广东 广州

收稿日期:2019年6月30日;录用日期:2019年7月19日;发布日期:2019年7月26日

摘 要
目的:共激活因子相关精氨酸甲基转移酶1 (Coactivator-associated arginine methyltransferase 1, CARM1)在肝癌细胞中生长作用及机制研究。方法:qRT-PCR、western blot法检测肝癌细胞中CARM1表达;MTT法和台盼蓝实验检测肝癌细胞的生长情况;Caspase-3试剂盒检测Caspase-3活性;小分子干扰RNA干扰CARM1研究其作用和分子机制。结果:小分子干扰RNA明显敲低CARM1水平;干扰CARM1后明显抑制肝癌细胞生长,且下调抑凋亡蛋白Bcl-2和上调促凋亡蛋白Bax表达水平,同时激活Caspase-3活性。结论:CARM1通过调控Bax/Bcl-2表达和Caspase-3活性进而调控肝癌生长作用。
关键词 :肝癌,CARM1,Bcl-2,Bax,Caspase-3

Copyright © 2019 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
肿瘤的发生是由于促癌基因和抑癌基因的突变引起的。近些年发现,除促癌基因和抑癌基因的突变外,肿瘤表观遗传学也在肿瘤的发生发展中起着关键的作用 [1]。DNA甲基化和共价组蛋白修饰是表观遗传调控的重要组成部分。研究发现肿瘤基因调控区高甲基化可促进肿瘤发生和发展 [2]。因此,调控肿瘤表观遗传学的调控因子是潜在的肿瘤治疗靶点,目前几种DNA甲基转移酶和组蛋白脱乙酰基酶抑制剂已被批准用于癌症治疗 [3]。共激活因子相关精氨酸甲基转移酶1 (Coactivator-associated arginine methyltransferase 1, CARM1) CARM1是一种重要的甲基转移酶,也称为精氨酸甲基酶4 (protein arginine methyltransferase 4, PRMT4)。最初发现CARM1和核受体辅助因子p160结合调控生物学功能 [4]。后来研究发现CARM1与转录因子cAMP反应元件结合蛋白CBP、β-catenin和NF-κB的p65结合调控下游靶基因的转录 [5] [6] [7]。很多研究表明CARM1在肿瘤的发生、发展和转移过程发挥着重要作用。文献报道CARM1促进乳腺癌、胰腺癌、骨肉瘤增殖和转移 [8] [9] [10] [11]。在早期肝癌中,CARM1水平增高 [12]。研究发现SKP2通过调控细胞核内的AMPK-SKP2-CARM1 信号通路诱导自噬并影响肝癌进展 [13]。但是,CARM1在肝癌中直接作用尚不清楚。
在本篇论文中,我们采取小分子干扰片段抑制肝癌细胞中CARM1的表达,用MTT实验和台盼蓝实验检测肝癌的生长情况。同时检测相关凋亡蛋白Bax\Bcl-2和Caspase-3活性,初步阐明CARM1在肝癌中作用及分子机制。
2. 材料与方法
2.1. 细胞培养
肝母细胞瘤细胞HepG2和肝癌细胞Huh7购自ATCC。用DMEM + 10%的胎牛血清完全培养基无菌于37˚、5% CO2的细胞培养箱中培养。
2.2. 试剂和耗材
Caspase-3活性试剂盒购自Biovision公司。Real time PCR的SYBER Green和逆转录酶试剂盒购自Takara公司。RNA提取试剂盒购自康为世纪公司。siCARM1小分子干扰片段购自锐博公司。转染试剂Lipo2000购自Invtrogen公司。细胞培养液购Hyclone公司。细胞培养皿购自康宁公司。CARM1、Bax、Bcl-2、β-actin抗体购自CST公司。MTT和台盼蓝购自北京鼎国公司。
2.3. 方法
MTT法检测HepG2和Huh7细胞增殖能力:采用噻唑兰颜色反应法,即MTT法进行细胞增殖活力的测定 [14]。其原理为活细胞线粒体中的琥珀酸脱氢酶能使外源性MTT还原为水不溶性的蓝紫色晶体—甲缵(formazan),并沉淀在细胞中,而死细胞无此功能。HepG2和Huh7细胞种植于96孔板,处理24h后,每孔加入10 µl的MTT工作液(浓度为5 mg/ml),继续培养4小时;加入三联溶液,培养箱中过夜以使蓝色结晶颗粒(甲缵)完全溶解,用酶标仪测定OD570的光密度值。实验中每份样品设3个复孔,实验至少重复3次。
台盼蓝法检测HepG2和Huh7细胞活力:台盼蓝能使死细胞染色,而不能使活细胞染色。HepG2和Huh7细胞种植于96孔板,处理24 h后,胰酶消化细胞,离心,重悬细胞,吸10 μl细胞悬液和10 μl 0.4%的台盼蓝溶液混合,充入细胞计数板,显微镜下计数四个大方格中染色和非染色细胞总数。非染色细胞除以非染色细胞和染色细胞的总数的百分数即细胞活力。
qRT-PCR实验
qRT-PCR检测CARM1的mRNA水平。具体实验如下:收集处理后的HepG2和Huh7细胞RNA,逆转录为cDNA,用Takara公司的SYBER green试剂盒进行实时荧光PCR实验。PCR条件为:95℃ 30 s,1个循环;PCR反应,95℃ 5 s,60℃ 15 s,45个循环;溶解,95℃ 0 s,65℃ 20 s,95℃ 0 s,1个循环。PCR结束后,根据反应得到的Cp值,使用相对定量的分析方法,以标准曲线进行校正,最后计算出样品中各mRNA的相对浓度。进行qRT-PCR引物序列如表1。
Table 1. Real-time fluorescence quantitative PCR primers
表1. 实时荧光定量PCR引物
CARM1干扰实验
CARM1干扰小分子片段购自锐博公司。
CARM1干扰实验具体方法如下:HepG2和Huh7细胞种植于六孔板中,待其融合度为50%~60%时候,用Lipo2000转染试剂转染CARM1干扰小分子片段,24小时后收集细胞RNA和蛋白,检测CARM1转录和蛋白水平。干扰CARM1小分子序列如表2。
Table 2. HE4 interference sequence
表2. HE4干扰序列
Caspase-3活性实验
5×106 HepG2和Huh7细胞种植于6孔板中,干扰CARM1基因24 h后,将细胞重新悬浮在50 μl冷冻细胞溶解缓冲液中,冰上10分钟。在微型离心机(10,000 × g)中离心1分钟。将上清液(细胞溶质提取物)转移到新鲜的试管中,然后放在冰上。测定蛋白质浓度,将50 μg~200 μg蛋白质稀释到50 μl,添加50 μl 2X反应缓冲液和5 μl的Devd PNA (200 μm终浓度),在37˚培养1~2小时。分光光度计405 nm读数。
Western blot实验
Western blot [15] 实验检测CARM1蛋白水平,具体实验方案如下:收集处理好的HepG2和Huh7细胞蛋白,进行蛋白定量,跑胶。加入CARM1一抗,4度过夜,收集一抗,加入二抗,然后加入ECL显色,拍照。
2.4. 统计学分析
采用SPSS 17.0软件进行统计学处理。计量资料以均数±标准差表示。采用t检验。P < 0.05表示具有统计学意义。
3. 实验结果
3.1. 肝癌细胞中有效干扰CARM1基因
用小分子干扰片段siCARM1转染HepG2和Huh7细胞24小时后收集细胞RNA和蛋白。结果显示小分子干扰片段siCARM1明显敲低肝癌细胞的CARM1转录水平,沉默效果近80% (图1(A)和图1(C))。Western blot结果也显示小分子干扰片段siCARM1明显敲低CARM1蛋白水平(图1(B)和图1(D)),这些结果为进一步的功能实验提供基础。
 qRT-PCR和Western blot检测干扰CARM1的效果。干扰CARM1后,于24 h收集RNA和细胞蛋白分别进行qRT-PCR和Western blot实验。*** < 0.001。
qRT-PCR和Western blot检测干扰CARM1的效果。干扰CARM1后,于24 h收集RNA和细胞蛋白分别进行qRT-PCR和Western blot实验。*** < 0.001。
Figure 1. The effect of silencing of HE4by siRNA
图1. 小分子干扰片段siCARM1敲低CARM1水平效果
3.2. 干扰CARM1后抑制HepG2和Huh7细胞生长
用小分子干扰片段siCARM1转染HepG2和Huh7细胞24小时后,MTT实验和台盼蓝实验检测HepG2和Huh7细胞的生长能力。MTT实验结果显示沉默CARM1后明显抑制HepG2和Huh7细胞的生长(图2(A)和图2(C))。台盼蓝实验也表明敲低CARM1后明显抑制HepG2和Huh7细胞的生长(图2(B)和图2(D))。这些结果提示CARM1调控肝癌细胞生长过程。
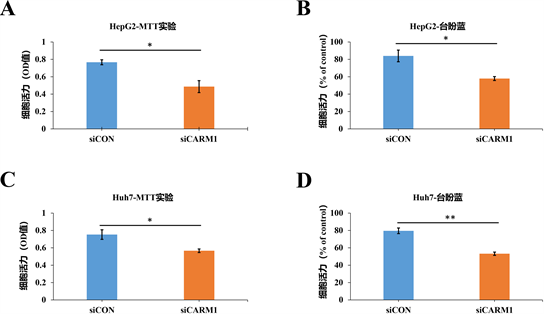 MTT法和台盼蓝实验检测肝癌细胞生长情况。干扰CARM1后,于24 h进行MTT实验和台盼蓝实验。* < 0.05, ** < 0.01。
MTT法和台盼蓝实验检测肝癌细胞生长情况。干扰CARM1后,于24 h进行MTT实验和台盼蓝实验。* < 0.05, ** < 0.01。
Figure 2. Silencing of CARM1 inhibits the growth of HepG2 and Huh7 cells
图2. 干扰CARM1后抑制HepG2和Huh7细胞生长
3.3. CARM1调控肝癌细胞Bax和Bcl-2蛋白表达
为了阐明CARM1调控肝癌细胞生长作用分子机制,我们首先检测凋亡相关蛋白Bax和Bcl-2表达。结果显示,沉默CARM1后明显上调促凋亡蛋白Bax表达,抑制抑凋亡蛋白Bcl-2表达(图3(A)和图3(C))。同时,也明显上调Bax/Bcl-2比例(图3(B)和图3(D))。这些结果表明阻断CARM1后可能促进肝癌细胞凋亡过程进而抑制肝癌细胞的生长。
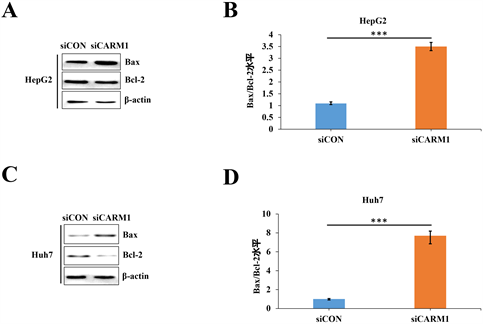 CARM1调控肝癌细胞Bax和Bcl-2蛋白表达。干扰HE4后,于24 h收集细胞蛋白检测Bax和Bcl-2表达。*** < 0.001。
CARM1调控肝癌细胞Bax和Bcl-2蛋白表达。干扰HE4后,于24 h收集细胞蛋白检测Bax和Bcl-2表达。*** < 0.001。
Figure 3. CARM1 regulates Bax and Bcl-2 expression
图3. CARM1调控肝癌细胞Bax和Bcl-2蛋白表达
3.4. CARM1调控肝癌细胞Caspase-3活性
为了进一步阐明CARM1通过调控肝癌细胞凋亡进而影响肝癌细胞生长作用的分子机制,我们检测细胞凋亡过程关键酶Caspase-3活性。结果显示,干扰CARM1后,Caspase-3活性明显升高(图4(A)和图4(B))。以上结果表明,CARM1通过调控凋亡相关蛋白Bax/Bcl-2和Caspase-3活性进而调控肝癌的凋亡过程,从而抑制肝癌的生长。
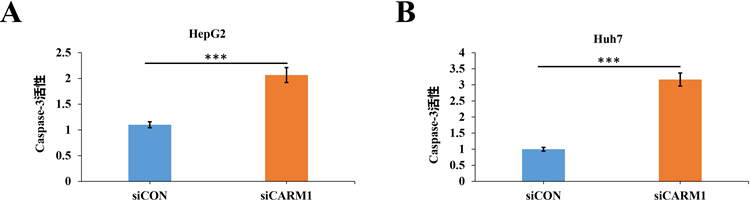 CARM1调控肝癌细胞Caspase-3活性。干扰HE4后,于24 h检测Caspase-3活性。*** < 0.001。
CARM1调控肝癌细胞Caspase-3活性。干扰HE4后,于24 h检测Caspase-3活性。*** < 0.001。
Figure 4. CARM1 regulates the activity of Caspase-3
图4. CARM1调控肝癌细胞Caspase-3活性
4. 讨论
肝癌是一个全球性的健康问题,其发病率越来越高,死亡率也越来越高 [16]。表观遗传的改变,如组蛋白修饰、DNA甲基化和小RNA介导过程,与多种癌症的增殖和转移密切相关 [17]。越来越多证据表明肝癌和表观遗传改变有关 [18]。共激活因子相关精氨酸甲基转移酶1 (Coactivator-associated arginine methyltransferase 1, CARM1)是蛋白质精氨酸甲基转移酶家族的一员,催化蛋白质底物中精氨酸(arg)残基甲基化,调控肿瘤表观遗传,进而改变基因转录过程 [19]。在这篇文章中,我们采用小分子干扰RNA敲低CARM1水平,研究其在肝癌中作用及分子机制。我们发现敲低CARM1后抑制肝癌细胞的生长,促进凋亡蛋白Bax表达,抑制抑凋亡蛋白Bcl-2水平,同时促进Caspase-3活性,表明CARM1通过调控肝癌细胞的凋亡过程进而调控肝癌细胞生长。
除了异常分化和增殖外,细胞凋亡也是肿瘤的一个重要特征 [20]。细胞凋亡主要由细胞凋亡蛋白Bcl-2家族调控。Bcl-2家族包括促凋亡蛋白Bak和Bax以及抑凋亡蛋白Bcl-2和Mcl-1 [21]。文献报道许多肝癌中Bcl-2表达增高,抑制肝癌凋亡促进其增殖 [22] ,同时Bcl-2高表达的病人比低表达病人的无病生存期明显缩短 [23]。在我们实验中发现沉默CARM1后明显上调促凋亡蛋白Bax水平,下调抑凋亡蛋白Bcl-2水平,明显升高Bax/Bcl-2比例,表明阻断CARM1后促进肝癌细胞凋亡过程进而抑制肝癌细胞生长,后者被MTT实验和台盼蓝实验所证明。我们结果与其他文献报道阻断CARM1后抑制乳腺癌、骨肉瘤和胰腺癌生长作用类似 [8] [9] [10] [11]。Caspase是调控细胞凋亡的重要酶。Caspase-3 (也称为cpp32、sca-1和cpp32b)是一种位于染色体4q34的casp-3基因编码的半胱氨酸蛋白酶 [24] ,在细胞凋亡中起着关键作用,其可切割和激活Caspase-6、Caspase-7和Caspase-9。我们实验结果发现阻断CARM1后促进Caspase-3活性,进一步表明CARM1通过调控肝癌细胞凋亡过程进而调控肝癌的生长。但是,CARM1调控肝癌的Caspase-3活性的分子机制尚未清楚,还值得我们进一步研究,这块也是这篇文章不足之处。
总之,在这篇论文中我们研究调控甲基化关键酶CARM1在肝癌细胞生长中作用及机制。发现沉默CARM1后抑制肝癌细胞生长,进一步研究发现阻断CARM1后上调促凋亡蛋白Bax水平,下调抑凋亡蛋白Bcl-水平,且促进Caspase-3活性,这些结果表明CARM1通过调控细胞凋亡过程进而调控肝癌细胞生长,提示着CARM1可能成为肝癌治疗新的靶点,具有一定的临床意义。
基金项目
广州医科大学博士启动基金(项目编号:2015C24);广州市卫生局一般引导项目(项目编号:20171A011312);广州市卫生局中西医结合项目(项目编号:20182A011020)广州医科大学大学生实验室开放项目(项目编号:B185004039);广东省教育厅项目(项目编号:2017KTSCX155);广东省自然科学基金(项目编号:2018A030310298)。
文章引用
刘美玲,洪宏海,王 征. CARM1通过调控Bax/Bcl-2表达和Caspase-3活性进而调控肝癌生长作用
CARM1 Regulates the Growth of Hepatocellular Carcinoma Cells via Regulation of Bax/Bcl-2 Level and Caspase-3 Activity[J]. 世界肿瘤研究, 2019, 09(03): 90-97. https://doi.org/10.12677/WJCR.2019.93013
参考文献
- 1. Rodriguez-Paredes, M. and Esteller, M. (2011) Cancer Epigenetics Reaches Mainstream Oncology. Nature Medicine, 17, 330-339.
https://doi.org/10.1038/nm.2305 - 2. Fraga, M.F., et al. (2005) Loss of Acetylation at Lys16 and Trimethylation at Lys20 of Histone H4 Is a Common Hallmark of Human Cancer. Nature Genetics, 37, 391-400.
https://doi.org/10.1038/ng1531 - 3. Kelly, T.K., De Carvalho, D.D. and Jones, P.A. (2010) Epigenetic Modifications as Therapeutic Targets. Nature Biotechnology, 28, 1069-1078.
https://doi.org/10.1038/nbt.1678 - 4. Suzuki, S., et al. (2004) Specific Differences in Gene Expression Profile Revealed by cDNA Microarray Analysis of Glutathione S-Transferase Placental form (GST-P) Immunohistochemically Positive Rat Liver Foci and Surrounding Tissue. Carcinogenesis, 25, 439-443.
https://doi.org/10.1093/carcin/bgh030 - 5. Xu, W., et al. (2001) A Transcriptional Switch Mediated by Cofactor Methylation. Science, 294, 2507-2511.
https://doi.org/10.1126/science.1065961 - 6. Koh, S.S., et al. (2002) Synergistic Coactivator Function by Coactivator-Associated Arginine Methyltransferase (CARM) 1 and β-Catenin with Two Different Classes of DNA-Binding Transcriptional Activators. The Journal of Biological Chemistry, 277, 26031-26035.
https://doi.org/10.1074/jbc.M110865200 - 7. Covic, M., et al. (2005) Arginine Methyltransferase CARM1 Is a Promoter-Specific Regulator of NF-κB-Dependent Gene Expression. The EMBO Journal, 24, 85-96.
https://doi.org/10.1038/sj.emboj.7600500 - 8. Wang, Y.P., et al. (2016) Arginine Methylation of MDH1 by CARM1 Inhibits Glutamine Metabolism and Suppresses Pancreatic Cancer. Molecular Cell, 64, 673-687.
https://doi.org/10.1016/j.molcel.2016.09.028 - 9. Frietze, S., et al. (2008) CARM1 Regulates Estrogen-Stimulated Breast Cancer Growth through Up-Regulation of E2F1. Cancer Research, 68, 301-306.
https://doi.org/10.1158/0008-5472.CAN-07-1983 - 10. Wang, L., et al. (2016) CARM1 Methylates Chromatin Remodeling Factor BAF155 to Enhance Tumor Progression and Metastasis. Cancer Cell, 30, 179-180.
https://doi.org/10.1016/j.ccell.2016.06.013 - 11. Li, S., et al. (2017) The Overexpression of CARM1 Promotes Human Osteosarcoma Cell Proliferation through the pGSK3β/β-Catenin/CyclinD1 Signaling Pathway. International Journal of Biological Sciences, 13, 976-984.
https://doi.org/10.7150/ijbs.19191 - 12. Osada, S., et al. (2006) Altered Gene Expression of Transcriptional Regulatory Factors in Tumor Marker-Positive Cells during Chemically Induced Hepatocarcinogenesis. Toxicology Letters, 167, 106-113.
https://doi.org/10.1016/j.toxlet.2006.08.014 - 13. Wei, X., et al. (2018) SKP2 Promotes Hepatocellular Carcinoma Progression through Nuclear AMPK-SKP2-CARM1 Signaling Transcriptionally Regulating Nutrient-Deprived Autophagy Induction. Cellular Physiology and Biochemistry, 47, 2484-2497.
https://doi.org/10.1159/000491622 - 14. Stockert, J.C., et al. (2012) MTT Assay for Cell Viability: Intracellular Localization of the Formazan Product Is in Lipid Droplets. Acta Histochemica, 114, 785-796.
https://doi.org/10.1016/j.acthis.2012.01.006 - 15. Jian, W., et al. (2019) A Novel Role of HuR in Epigallocatechin-3-Gallate (EGCG) Induces Tumour Cells Apoptosis. Journal of Cellular and Molecular Medicine, 23, 3767-3771.
https://doi.org/10.1111/jcmm.14249 - 16. Llovet, J.M., Burroughs, A. and Bruix, J. (2003) Hepatocellular Carcinoma. The Lancet, 362, 1907-1917.
https://doi.org/10.1016/S0140-6736(03)14964-1 - 17. Han, T.S., et al. (2018) The Epigenetic Regulation of HCC Metastasis. International Journal of Molecular Sciences, 19, 3978.
https://doi.org/10.3390/ijms19123978 - 18. Khan, F.S., et al. (2017) Epigenetic Mechanisms Regulating the Development of Hepatocellular Carcinoma and Their Promise for Therapeutics. Hepatology International, 11, 45-53.
https://doi.org/10.1007/s12072-016-9743-4 - 19. Bedford, M.T. and Clarke, S.G. (2009) Protein Arginine Methylation in Mammals: Who, What, and Why. Molecular Cell, 33, 1-13.
https://doi.org/10.1016/j.molcel.2008.12.013 - 20. Su, Z., et al. (2015) Apoptosis, Autophagy, Necroptosis, and Cancer Metastasis. Molecular Cancer, 14, 48.
https://doi.org/10.1186/s12943-015-0321-5 - 21. Adams, J.M. and Cory, S. (2007) The Bcl-2 Apoptotic Switch in Cancer Development and Therapy. Oncogene, 26, 1324-1337.
https://doi.org/10.1038/sj.onc.1210220 - 22. Shigematsu, S., et al. (2011) ZNF689 Suppresses Apoptosis of Hepatocellular Carcinoma Cells through the Down-Regulation of Bcl-2 Family Members. Experimental Cell Research, 317, 1851-1859.
https://doi.org/10.1016/j.yexcr.2011.05.012 - 23. Brito, A.F., et al. (2016) Targeting Hepatocellular Carcinoma: What Did We Discover So Far? Oncology Reviews, 10, 302.
https://doi.org/10.4081/oncol.2016.302 - 24. Khalilzadeh, B., et al. (2018) Advances in Nanomaterial Based Optical Biosensing and Bioimaging of Apoptosis via Caspase-3 Activity: A Review. Microchimica Acta, 185, 434.
https://doi.org/10.1007/s00604-018-2980-6
NOTES
*通讯作者。
