Botanical Research
Vol.
11
No.
06
(
2022
), Article ID:
57373
,
11
pages
10.12677/BR.2022.116077
小麦DUF642基因全基因组鉴定及 转录组分析
王志清1,夏鹏亮2,金莹莹1,杨艳1,李薇1
1长江大学农学院,湿地生态与农业利用教育部工程研究中心/主要粮食作物产业化湖北省协同创新中心,湖北 荆州
2湖北恩施州烟草公司,湖北 恩施
收稿日期:2022年9月8日;录用日期:2022年10月24日;发布日期:2022年10月31日

摘要
【目的】分析小麦细胞壁相关蛋白DUF642 (Domain of Unknown Function 642)基因家族的特点。【方法】在小麦全基因组内对DUF642家族成员进行鉴定,并且对其进化关系、基因结构、蛋白特性、保守基序、顺式作用元件和转录组信息进行系统分析。【结果】TaDUF642家族中共有34个家族成员,多物种系统进化树分析、基因结构和保守基序显示,TaDUF642家族成员在进化上具有较高的保守性;蛋白特征显示,大多数TaDUF642蛋白为酸性蛋白质和亲水性蛋白质;亚细胞定位预测显示,TaDUF642大多定位在叶绿体;转录组数据分析显示,TaDUF642基因广泛参与小麦的生长发育和非生物胁迫,并且与禾谷镰刀菌、条锈菌和白粉菌的侵染紧密相关。【结论】本研究精准鉴定了小麦TaDUF642基因家族成员及其表达模式,为小麦在抗逆方面的遗传改良提供了理论依据。
关键词
小麦,DUF642,全基因组鉴定,转录组分析

Genome-Wide Identification and Transcriptome Analysis of DUF642 Gene Family in Wheat
Zhiqing Wang1, Pengliang Xia2, Yingying Jin1, Yan Yang1, Wei Li1
1Hubei Collaborative Innovation Center for Grain Industry, Engineering Research Center of Wetland Ecology and Agricultural Use/Ministry of Education, College of Agriculture, Yangtze University, Jingzhou Hubei
2Enshi Tobacco Company of Hubei Province, Enshi Hubei
Received: Sep. 8th, 2022; accepted: Oct. 24th, 2022; published: Oct. 31st, 2022

ABSTRACT
[Objective] Further analyze the properties of DUF642 (Domain of Unknown Function 642) gene family related cell wall in wheat. [Method] The DUF642 family members were identified throughout the whole wheat genome, and their evolutionary relationships, gene structure, protein characteristics, conserved motifs, cis-acting elements and transcriptome information were systematically analyzed. [Result] There were 34 family members in TaDUF642 family. Phylogenetic tree analysis, gene structure and conserved motifs showed that TaDUF642 family members were highly conserved in evolution. Protein characteristics showed most of the TaDUF642 proteins were acidic and hydrophilic. Subcellular localization prediction indicated that TaDUF642 was mostly localized in chloroplast. The analysis of transcriptome data suggested that TaDUF642 genes is widely involved in the growth and development and abiotic stress of wheat, and is closely related to the infection of Fusarium graminis, stripe rust and powdery mildew. [Conclusion] Members of the TaDUF642 genes family and their expression patterns were accurately identified in wheat, which provided a theoretical basis for the genetic improvement of wheat resistance to stress.
Keywords:Wheat, DUF642, Genome-Wide Identification, Transcriptome Analysis

Copyright © 2022 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
【研究意义】小麦(Triticum aestivum)是世界第二大粮食作物,培育抗病、抗倒、节水、抗逆、广适、养分高效的小麦品种尤为重要。加强小麦重要性状功能基因克隆和功能标记开发,能够加快小麦遗传改良进程,缩短育种年限,提高育种效率 [1]。未知功能域蛋白家族DUF642 (Domain of Unknown Function 642, DUF642)是种子植物中特有的未知功能的细胞壁相关蛋白,含有1~2个高度保守的DUF642结构域 [2],它不仅参与细胞壁的形成,影响植物的生长发育 [3],而且在抵御逆境胁迫如干旱、盐和铝毒胁迫,病原微生物侵染等方面发挥着重要的功能,对提高小麦产量具有潜在的应用价值。
【前人研究进展】拟南芥(Arabidopsis thaliana) DUF642基因At4g32460被定位在细胞壁 [4],它参与了果胶甲基酯酶(Pectin methyl esterase, PME)在种子萌发和植株发育中的调控 [5]。拟南芥DUF642蛋白At2g41800在根、柱头和下胚轴中表达,其突变体能够使幼苗的下胚轴长度增加,说明该基因参与下胚轴的伸长 [6]。研究表明,DUF642蛋白家族基因积极响应植物的非生物胁迫。编码籽粒苋(Amaranthus hypochondriacus) DUF642蛋白的基因AhDGR2能够改变细胞壁的结构和组成,在干旱和盐胁迫下表达量发生显著变化,并且可以诱导转基因拟南芥对盐和脱落酸(Abscisic acid, ABA)的高敏感性 [7]。在对水稻(Oryza sativa) DUF642家族基因的研究中发现,OsDUF642基因可能参与ABA介导的非生物逆境响应,例如高温、干旱、盐胁迫等 [8]。在研究玉米(Zea mays) DUF642蛋白家族时,也发现了多个能够强烈响应干旱、盐和铝胁迫的基因 [9]。另外,拟南芥DUF642基因At5g25460和At3g08030可受细菌侵染诱导表达。在接种青枯雷尔氏菌(Ralstonia solanacearum)后At5g25460表达量下调 [10],在接种带化红球菌(Rhodococcus fascians)后At3g08030表达量上调 [11]。在陆地棉(Gossypium hirsutum) DUF642蛋白家族的研究中发现,6个GhDUF642基因受黄萎病菌诱导差异表达,并通过VIGS技术证明陆地棉DUF642基因参与了棉花抗黄萎病过程 [12]。研究从中国葡萄(Vitis. quinquangularis)丹凤2号中克隆的VqDUF642发现,VqDUF642参与植物果实发育及对白粉菌和灰霉菌的防御反应。VqDUF642定位于烟草表皮细胞细胞壁,在转基因番茄植株中过量表达VqDUF642加快植株生长,并且能够降低对灰霉菌的敏感性。过表达VqDUF642的转基因无核葡萄植株对白粉菌和灰霉病菌的抗性增强 [13]。
【本研究的切入点】基于DUF642蛋白家族在植物生命活动中的重要意义,本研究对小麦全基因组DUF642基因家族成员进行了鉴定,并对其进化关系、编码蛋白特征、保守性、染色体位置、启动子和转录组信息进行了分析。【拟解决的关键问题】为进一步研究小麦DUF642基因家族的功能和小麦的遗传改良提供了新的线索。
2. 材料与方法
2.1. TaDUF642基因鉴定和系统发育
首先,从Pfam (https://pfam.xfam.org) [14] 下载Pfam域为PF04862 (DUF642)的 [5] 种子序列,从EnsemblPlants数据库(http://plants.ensembl.org/index.html)下载拟南芥、水稻的DUF642蛋白序列,以上述序列作为查询序列进行BLASTp (E < 10-5)分析,删除不含有PF04862结构域的序列,再删除可变剪切和冗余序列,最终确定小麦DUF642基因家族成员。然后进行ClustalW2 [15] 比对,利用MEGA 7.0最大似然法(maximum likelihood, ML)生成系统发育树(bootstrap = 1000) [16],并使用ITOL工具(http://itol.embl.de)美化系统发育树。
2.2. TaDUF642基因蛋白特性预测
使用蛋白分析工具ExPASy (https://www.expasy.org) [17] 预测TaDUF642基因的蛋白特性,包括氨基酸长度(aa)、分子量(Mw)、等电点(pI)、稳定性和亲水性(GRAVY)。在小麦基因组注释信息GFF3文件中提取TaDUF642基因的染色体位置信息,使用MapInspect进行可视化分析。通过网站Plant-mPLo (http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/) [18] 进行亚细胞定位预测。
2.3. TaDUF642基因基序和基因结构分析
将TaDUF642蛋白序列上传到MEME v5.4.1 (http://meme-suite.org/index.html) [19] 搜索其保守序列,设置搜索Motif值为15,然后用TBtools绘制motif分析图。根据TaDUF642基因注释信息,通过TBtools绘制其基因结构图 [20]。
2.4. TaDUF642基因顺式作用元件鉴定
从小麦数据库提取TaDUF642基因上游序列1500 bp,使用PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/) [21] 搜索启动子序列中的顺式元件,然后用R软件包的“Pheatmap”展示预测结果。
2.5. TaDUF642基因转录组分析
从NCBI的SRA数据库收集小麦多条件转录组分析的原始RNA-seq,并将RNA-seq数据通过Hisat2 比对到小麦参考基因组。基因表达水平通过片段按每百万碱基对外显子每千克碱基数(FPKM)模型进行归一化 [22]。然后利用R软件包的“Pheatmap”绘制热图。
3. 结果与分析
3.1. TaDUF642基因鉴定和系统发育分析
经同源检索,在小麦中鉴定到34个DUF642基因家族成员。构建小麦、拟南芥和水稻的DUF642蛋白家族复合系统进化树(图1),然后根据其亲缘关系的远近进行命名。结果表明,DUF642家族基因可分为5组(group a、group b、group c、group d、group e),TaDUF642在各组中均有分布,其中,group e含有的DUF642基因最多(23个),group c含有的DUF642基因最少(2个)。与拟南芥和水稻相比,小麦DUF642家族基因数目较多,这可能是由于小麦经过两轮全基因组复制形成异源六倍体,从而使不同染色体组间具有了同源基因造成的 [23]。
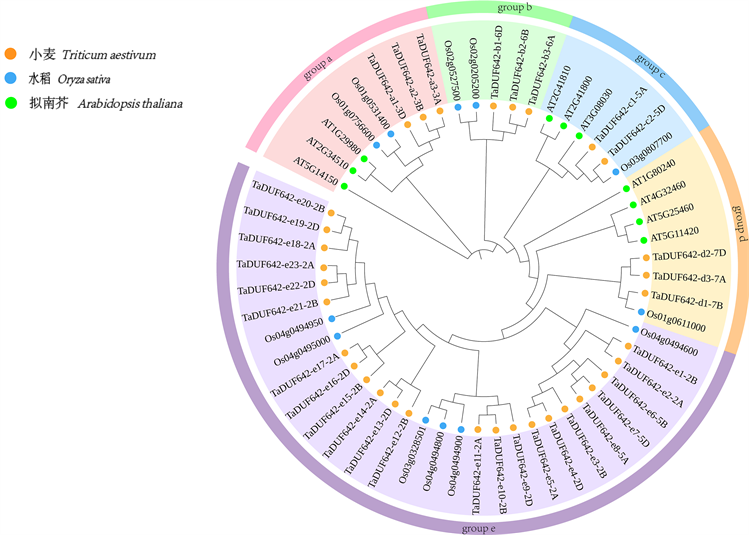
Figure 1. Phylogenetic relationships of DUF642 proteins in Triticum aestivum, Oryza sativa and Arabidopsis thaliana
图1. 小麦,水稻和拟南芥DUF642蛋白的进化关系
3.2. TaDUF642基因蛋白特性预测
用ExPASy Server 10预测的34个小麦DUF642基因的蛋白特性(表1)显示,TaDUF642的蛋白质平均长度为377 aa (307~432);平均等电点为6.8 (4.54~9.24),大多为酸性蛋白质;不稳定指数值为38.47 (31.1~47.51),蛋白质相对稳定(指数小于40);脂溶性指数值为84.02 (74.3~92.89);亲水性指数为−0.032 (−0.21~0.115),大部分为亲水性蛋白质(指数小于0)。TaDUF642家族基因在A、B和D三个染色体组上均有分布且具有相似性,分布在chr2A、chr2B和chr2D上的数量较多,分别为7、7和6个。亚细胞定位预测结果显示,大多数TaDUF642蛋白位于叶绿体上。
Table 1. Sequence characteristics of the 34 TaDUF642 genes identified
表1. 34个TaDUF642基因的序列特征
3.3. TaDUF642基因保守性分析
将搜索到的15个Motif命名为motif1~motif15,基序的宽度从6到42不等,绘制34个TaDUF642蛋白序列的基序分布(图2)。结果显示,同一亚组的TaDUF642蛋白具有相似的基序组成,例如,TaDUF642-a3-3A、TaDUF642-a2-3B和TaDUF642-a1-3D的氨基酸序列具有高度相似性。另外,除TaDUF642-b1-6D外,在所有的TaDUF642序列中都识别到了motif4。基因结构分析显示,同一亚组的TaDUF642蛋白基因结构也具有相似性,且大部分的TaDUF642序列两端都具有UTR非编码区(图2)。TaDUF642家族保守基序和基因结构具有相似性,说明TaDUF642家族的在进化上较为保守。
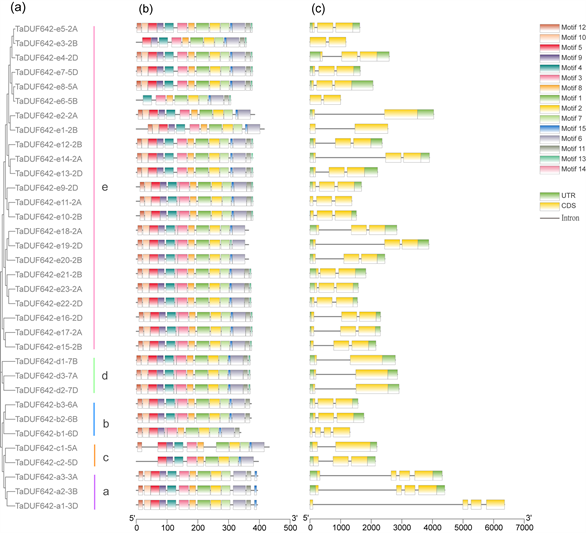
Figure 2. Conserved gene sequence and gene structure of TaDUF642 genes. (a) Phylogenetic tree of TaDUF642 family; (b) Motif analysis of TaDUF642 family; (c) Gene structure of TaDUF642 family
图2. TaDUF642基因的保守序列和基因结构。(a) TaDUF642家族系统发育树;(b) TaDUF642家族motif分析;(c) TaDUF642家族基因结构
3.4. TaDUF642基因启动子顺式作用元件鉴定
对PlantCARE预测的顺式元件进行可视化分析(图3),可将鉴定到的791个顺式元分为三类:生长发育、激素应答和生物/非生物胁迫,其数量分别为192 (24.3%)、287 (36.3%)和312 (39.4%)个,相应的调控因子种类分别为14、8和25种。其中,有较多的光反应元件(G-box、Sp1、Box II、GATA-motif、I-box、GT1-motif、Box 4、AE-box、TCCC-motif、ATCT-motif、TCT-motif、GATT-motif、GTGGC-motif、ATC-motifchs-CMA2a、Gap-box、GA-motif、ACE、chs-CMA1a、L-box、MRE)。分布最广的顺式作用元件是光反应元件G-box (88.2%),其次是脱落酸反应元件ABRE (82.3%),茉莉酸甲酯反应元件CGTCA-motif (76.5%)和TGACG-motif (76.5%)分布也较为广泛。启动子分析显示光反应元件广泛分布,这与亚细胞定位预测反映的大多数基因定位在叶绿体相一致。
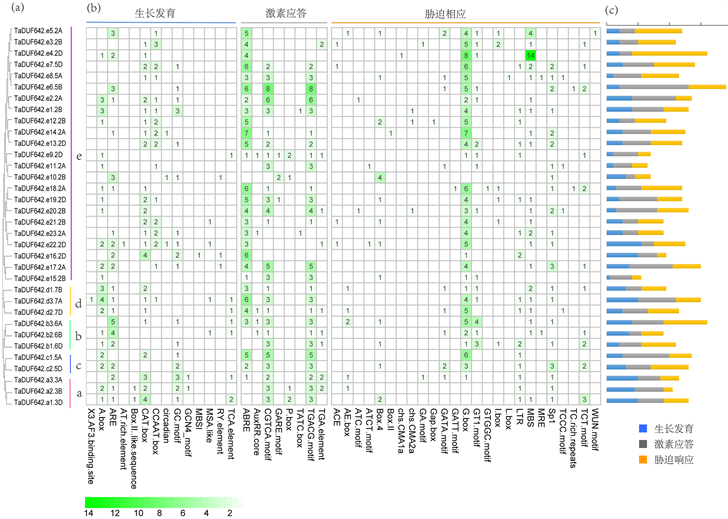
Figure 3. Cis-element analysis of TaDUF642 genes. (a) Phylogenetic tree of TaDUF642 family; (b) The cis-acting element of TaDUF642 genes; (c) Statistical map of cis-elements of TaDUF642 genes
图3. TaDUF642基因的顺式元件分析。(a) TaDUF642家族系统发育树;(b) TaDUF642基因顺式作用元件;(c) TaDUF642基因顺式作用元件
3.5. TaDUF642基因多条件转录组分析
从NCBI下载的转录组数据分为三类:生长发育、非生物胁迫和生物胁迫。不同组别中的基因表达量明显不同。在生长发育阶段(图4),除TaDUF642-e2-2A、TaDUF642-e6-5B外,其余基因在各时期均有表达,且大多数基因在苗期、三叶期和分蘖期的根中都有表达。TaDUF642-a1-3D、TaDUF642-a2-3B、TaDUF642-a3-3A、TaDUF642-c2-5D、TaDUF642-d1-7B、TaDUF642-d2-7D和TaDUF642-d3-7A基因在苗期、三叶期和分蘖期中高表达。group a和group d中的基因在胚芽鞘、叶片、茎分生组织和根分生组织中的表达量相对更高。
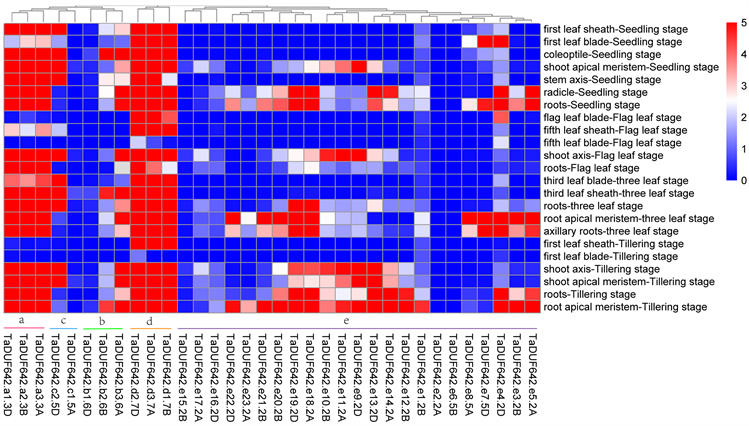
Figure 4. Transcriptome analysis of TaDUF642 genes under growth and development
图4. TaDUF642基因在生长发育阶段的转录组分析
对于非生物胁迫(图5),group d中的基因在多种胁迫处理下有较高的表达量。在PEG6000处理下,group d中三个基因表达良好,与对照组相比,Gemmiza 10品种小麦上调表达,TAM107则下调。在干旱和热胁迫下,与对照组相比,group d中的基因大多下调表达,TaDUF642-e7-5D和TaDUF642-e8-5A基因表达量上调。在寒冷胁迫下,与对照组相比,group d组中三个基因表达量上调,TaDUF642-e4-2D基因表达量下调。在磷饥饿处理下,与对照组相比,group a和group d中的基因表达量相对较高,且有明显差异。
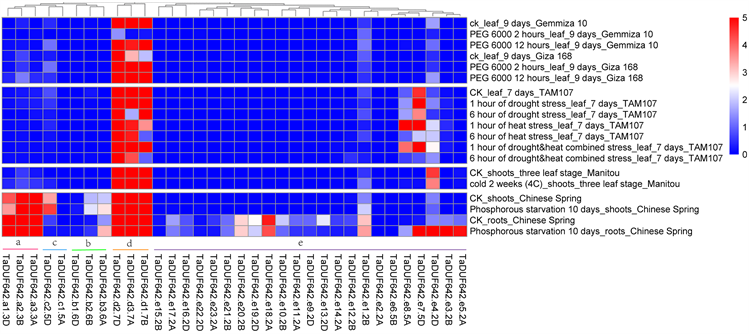
Figure 5. Transcriptome analysis of TaDUF642 genes under abiotic stress
图5. TaDUF642基因在非生物胁迫下的转录组分析
对于生物胁迫(图6),在禾谷镰刀菌处理下,group a和group d中的基因、TaDUF642-b3-6A和TaDUF642-c2-5D表达量较高,且与对照组相比差异明显,说明其功能与禾谷镰刀菌的侵染有关。在条锈菌处理下,group a和group d中的基因和TaDUF642-e4-2D表达良好,且与对照组相比具有一定差异,说明这些基因参与了小麦响应条锈菌侵染过程的调控。在白粉菌处理下,与对照组相比,group a和group d中的基因大多表达量下调,说明其功能可能与白粉菌E09侵染有关。
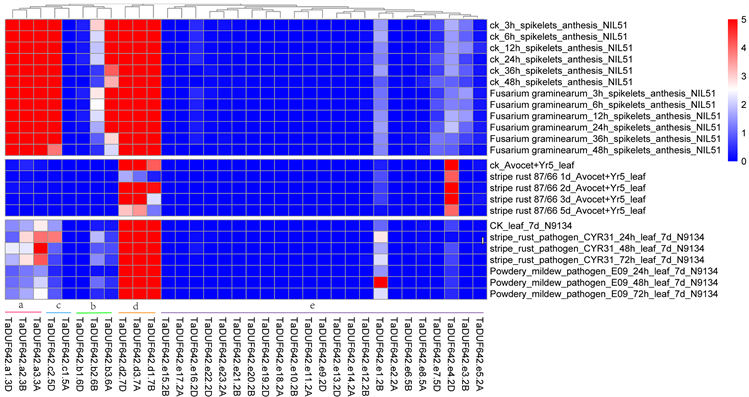
Figure 6. Transcriptome analysis of TaDUF642 genes under biological stress
图6. TaDUF642基因在生物胁迫下的转录组分析
4. 讨论
通过生物信息学技术鉴定得到34个TaDUF642基因,保守性较高,且大多定位在叶绿体上。在启动子顺式作用元件分析中,TaDUF642-e4-2D基因启动子中含有较多的顺式元件MBS (14个),表明该基因可能与干旱胁迫的调控有着很大关系。另外,干旱诱导元件MBS在玉米DUF642家族基因中也分布广泛,表明TaDUF642基因家族成员可能在小麦响应非生物胁迫中具有潜在功能。
关于TaDUF642基因的表达模式,不同组别的基因表达水平有所差异。在生长发育阶段,group a和group d中的基因在胚芽鞘、叶、茎和根中均有较高的表达量,其功能可能与小麦多个组织器官的生长发育相关。在拟南芥的研究中发现,At4g32460和At5g11420基因编码的DUF642蛋白在植物的生长发育过程中对PME的活性起到调控作用,At4g32460基因对细胞内PME活性起到正向调控租用,可能对种子的萌发有重要影响 [5]。在TaDUF642基因家族中,group d中三个基因与At4g32460和At5g11420有较高的同源性,它们可能与果胶的去甲基化有关。
对group d中的基因在非生物胁迫下的表达量进行分析,该组基因可能参与小麦抵抗干旱、寒冷和磷饥饿等多种功能。在水稻的研究中发现,Os04g0494900可能是野生型水稻参与抗铝毒活动较为重要的基因 [24],并且研究发现玉米DUF642家族中也存在与抗铝毒胁迫相关的基因 [13]。在TaDUF642基因家族中,TaDUF642-e11-2A、TaDUF642-e10-2B和TaDUF642-e9-2D三个基因与Os04g0494900同源性较高,它们可能与小麦抗铝毒胁迫相关。
另外,group a和group d中的基因在禾谷镰刀菌、条锈菌和白粉菌处理下均具有较高的表达量,且与对照组相比存在差异,这些基因在小麦抵御生物胁迫的过程中发挥着重要的影响。在拟南芥的研究中发现,At5g25460和At3g08030基因受细菌侵染和害虫取食诱导表达 [8],而group d中三个基因与At5g25460同源性较高,TaDUF642-c2-5D与At3g08030同源性较高,这些基因在生物胁迫下都表现出对植物较大的影响。At1g80240和At5g25460参与了细胞壁的形成,对细胞生长和植物发育有重要作用 [25],而group d中三个基因与At1g80240和At5g25460同源性较高,这与转录组分析显示group d中三个基因在小麦生长发育多个时期有较高的表达量相一致。
5. 结论
本研究通过生物信息学方法对小麦DUF642基因家族进行全面的研究。共得到34个TaDUF642基因,而且小麦DUF642基因在非生物和生物胁迫中发挥着重要的作用,为进一步挖掘DUF642基因的功能提供了参考依据。
基金项目
中国烟草总公司科技项目(110202102040)和湖北省烟草公司科技项目(027Y2020-006)。
文章引用
王志清,夏鹏亮,金莹莹,杨 艳,李 薇. 小麦DUF642基因全基因组鉴定及转录组分析
Genome-Wide Identification and Transcriptome Analysis of DUF642 Gene Family in Wheat[J]. 植物学研究, 2022, 11(06): 630-640. https://doi.org/10.12677/BR.2022.116077
参考文献
- 1. 刘志勇, 王道文, 张爱民, 等. 小麦育种行业创新现状与发展趋势[J]. 植物遗传资源学报, 2018, 19(3): 430-434.
- 2. Vázquez-Lobo, A., Roujol, D., Zuñiga-Sánchez, E., et al. (2012) The Highly Conserved Spermatophyte Cell Wall DUF642 Protein Family: Phylogeny and First Evidence of Interaction with Cell Wall Polysaccharides in Vitro. Molecular Phylogenetics and Evolution, 63, 510-520. https://doi.org/10.1016/j.ympev.2012.02.001
- 3. Cruz-Valderrama, J.E., Gómez-Maqueo, X., Salazar-Iribe, A., et al. (2019) Overview of the Role of Cell Wall DUF642 Proteins in Plant Development. International Journal of Molecular Sciences, 20, Article No. 3333. https://doi.org/10.3390/ijms20133333
- 4. Salazar-Irib, A., Zúñiga-Sánchez, E., Gamboa-deBuen, A., et al. (2017) Cell Wall Localization of Two DUF642 Proteins, BIIDXI and TEEBE, during Meloidogyne incognita Early Inoculation. The Plant Pathology Journal, 33, 614-618. https://doi.org/10.5423/PPJ.NT.05.2017.0101
- 5. Zúñiga-Sánchez, E., Soriano, D., Martínez-Barajas, E., et al. (2014) BIIDXI, the At4g32460 DUF642 Gene, Is Involved in Pectin Methyl Esterase Regulation during Arabidopsis thaliana Seed Germination and Plant Development. BMC Plant Biology, 14, Article No. 338. https://doi.org/10.1186/s12870-014-0338-8
- 6. Salazar-Iribe, A., Agredano-Moreno, L.T., Zúñiga-Sánchez, E., et al. (2016) The Cell Wall DUF642 At2g41800 (TEB) Protein Is Involved in Hypocotyl Cell Elongation. Plant Science, 253, 206-214. https://doi.org/10.1016/j.plantsci.2016.10.007
- 7. Palmeros-Suárez, P.A., Massange-Sánchez, J.A., Sán-chez-Segura, L., et al. (2017) AhDGR2, an Amaranth Abiotic Stress-Induced DUF642 Protein Gene, Modifies Cell Wall Structure and Composition and Causes Salt and ABA Hyper-Sensibility in Transgenic Arabidopsis. Planta, 245, 623-640. https://doi.org/10.1007/s00425-016-2635-y
- 8. 王晓睿, 胡琴, 杜雪竹, 等. 水稻DUF642家族基因的鉴定及在非生物逆境中的表达分析[J]. 湖北大学学报(自然科学版), 2022, 44(1): 14-23.
- 9. 刘天宇, 杨永娟, 赵卓, 等. 玉米DUF642基因家族的鉴定和分析[J]. 分子植物育种, 2018, 16(21): 6888-6898.
- 10. Hu, J., Barlet, X., Deslandes, L., et al. (2008) Transcriptional Responses of Arabidopsis thaliana during Wilt Disease Caused by the Soil-Borne Phytopathogenic Bacterium, Ralstonia solanacearum. PLOS ONE, 3, e2589. https://doi.org/10.1371/journal.pone.0002589
- 11. Depuydt, S., Trenkamp, S., Elftieh, S., et al. (2009) An Integrated Genomics Approach to Define Niche Establishment by Rhodococcus fascians. Plant Physiology, 149, 1366-1386. https://doi.org/10.1104/pp.108.131805
- 12. 解美霞, 杨君, 王国宁, 等. 基于表达谱分析陆地棉DUF642基因家族抗逆功能[J]. 棉花学报, 2019, 31(6): 493-504.
- 13. Xie, X.Q. and Wang, Y.J. (2016) VqDUF642, a Gene Isolated from the Chinese Grape Vitisquinquangularis, Is Involved in Berry Development and Pathogen Resistance. Planta, 244, 1075-1094. https://doi.org/10.1007/s00425-016-2569-4
- 14. Punta, M., Coggill, P.C., Eberhardt, R.Y., et al. (2012) The Pfam Protein Families Database. Nucleic Acids Research, 40, 290-301. https://doi.org/10.1093/nar/gkr1065
- 15. Thompson, J.D., Higgins, D.G. and Gibson, T.J. (1994) CLUSTAL W: Improving the Sensitivity of Progressive Multiple Sequence Alignment through Sequence Weighting, Position-Specific Gap Penalties and Weight Matrix Choice. Nucleic Acids Research, 22, 4673-4680. https://doi.org/10.1093/nar/22.22.4673
- 16. Kumar, S., Stecher, G. and Tamura, K. (2016) MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets. Molecular Biology and Evolution, 33, 1870-1874. https://doi.org/10.1093/molbev/msw054
- 17. Wilkins, M.R., Gasteiger, E., Bairoch, A., et al. (1999) Protein Identification and Analysis Tools in the ExPASyserver. Methods in Molecular Biology (Clifton, N.J.), 112, 531-552.
- 18. Chou, K.C. and Shen, H.B. (2010) Cell-PLoc 2.0: An Improved Package of Web-Servers for Predicting Subcellular Localization of Proteins in Various Organisms. Natural Science, 2, 1090-1103. https://doi.org/10.4236/ns.2010.210136
- 19. Bailey, T.L., Boden, M., Buske, F.A., et al. (2009) MEME SUITE: Tools for Motif Discovery and Searching. Nucleic Acids Research, 37, W202-W208. https://doi.org/10.1093/nar/gkp335
- 20. Chen, C.J., Chen, H., Zhang, Y., et al. (2020) TBtools: An Integrative Toolkit Developed for Interactive Analyses of Big Biological Data. Molecular Plant, 13, 1194-1202. https://doi.org/10.1016/j.molp.2020.06.009
- 21. Zhu, Y.X., Yang, L., Liu, N., et al. (2019) Genome-Wide Identification, Structure Characterization, and Expression Pattern Profiling of Aquaporin Gene Family in Cucumber. BMC Plant Biology, 19, Article No. 345. https://doi.org/10.1186/s12870-019-1953-1
- 22. Trapnell, C., Roberts, A., Goff, L., et al. (2012) Differential Gene and Transcript Expression Analysis of RNA-seq Experiments with TopHat and Cufflinks. Nature Protocols, 7, 562-578. https://doi.org/10.1038/nprot.2012.016
- 23. IWGSC, Rudi, A., Kelly, E., et al. (2018) Shifting the Limits in Wheat Research and Breeding Using a Fully Annotated Reference Genome. Science, 361, eaar7191.
- 24. Yamaji, N., Huang, C.F., Nagao, S., et al. (2009) A Zinc Finger Transcription Factor ART1 Regulates Multiple Genes Implicated in Aluminum Tolerance in Rice. The Plant Cell, 21, 3339-3349. https://doi.org/10.1105/tpc.109.070771
- 25. Gao, Y.S., Badejo, A.A., Sawa, Y., et al. (2012) Analysis of Two L-Galactono-1,4-lactone-responsive Genes with Complementary Expression During the Development of Arabidopsis thaliana. Plant & Cell Physiology, 53, 592-601. https://doi.org/10.1093/pcp/pcs014
