Advances in Clinical Medicine
Vol.
10
No.
08
(
2020
), Article ID:
37339
,
9
pages
10.12677/ACM.2020.108268
Bioinformatics Analysis of Gene Expression and Signaling Pathways in Basal Cell Carcinoma
Da Wang, Lixin Wang, Guanzhi Chen*
Department of Dermatology, Affiliated Hospital of Qingdao University, Qingdao Shandong

Received: Aug. 3rd, 2020; accepted: Aug. 20th, 2020; published: Aug. 27th, 2020

ABSTRACT
Objective: To determine abnormally expressed genes in basal cell carcinoma (BCC), examine the function of the differentially expressed genes and the signaling pathways in which they are involved, and identify functional hub genes using gene expression analysis, and to investigate the molecular mechanism of BCC pathogenesis. Methods: The gene expression data of GSE125285 were downloaded from the Gene Expression Omnibus (GEO) database. Differentially expressed genes were analyzed by Gene body (GO) analysis and Kyoto Encyclopedia of Genes (KEGG) pathway analysis, and the protein interaction network of differential Gene products was constructed by Cytoscape3.8.0 to identify the functional hub genes. Result: Of the 653 screened in BCC, 387 genes were up-regulated and 266 genes were down-regulated. GO analysis showed that the differentially expressed genes were mainly involved in biological processes of collagen breakdown, composition of extracellular matrix and the process of oxidation-reduction. KEGG pathway analysis revealed that the differentially expressed genes were mainly enriched in the digestibility of protein, the pathway of Hedgehog signal transduction and peroxisome proliferators’ receptor activation. MYC, IL6, PPARG, FOS, LEP, EGR1, COL1A1, NTRK3, SPP1 and ADIPOQ were identified as the top 10 hub genes by the protein interaction network. Conclusion: Abnormalities in protein metabolism, REDOX reactions and Hedgehog signal transduction by polygenic variation may be involved in the development and progression of BCC.
Keywords:Basal Cell Carcinoma, Skin, Differentially Expressed Genes, Bioinformatics

基于生物信息学分析基底细胞癌的基因表达及信号传导通路
王达,王丽欣,陈官芝*
青岛大学附属医院皮肤科,山东 青岛

收稿日期:2020年8月3日;录用日期:2020年8月20日;发布日期:2020年8月27日

摘 要
目的:通过生物信息学的方法分析皮肤基底细胞癌(BCC)的差异基因和关键通路,明确差异基因的功能、参与的信号传导通路,筛选出枢纽基因,从分子水平为研究BCC的发病机制提供候选基因。方法:从Gene Expression Omnibus (GEO)数据库中下载GSE125285的基因表达数据,利用DAVID数据库及R语言的Limma包对差异基因进行基因本体分析(GO分析)和基因组数据库的通路分析(KEGG分析),并应用Cytoscape3.8.0软件构建差异基因相互作用网络,筛选枢纽基因。结果:总共筛选获得了653个差异基因,其中表达上调的基因387个,表达下调的基因266个。GO本体分析表明差异基因主要参与了胶原蛋白的分解代谢过程、细胞外基质的组成以及氧化还原过程,KEGG信号通路富集分析主要包括了蛋白质的消化和吸收、Hedgehog信号通路及过氧化物酶体增殖物激活受体通路等。筛选获得了前10位关键基因MYC,IL6,PPARG,FOS,LEP,EGR1,COL1A1,NTRK3,SPP1及ADIPOQ。结论:多基因变异所导致的蛋白质代谢及氧化还原反应等多通路异常可能参与BCC的发生及疾病进展。
关键词 :基底细胞癌,皮肤,差异表达基因,生物信息学

Copyright © 2020 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
基底细胞癌是一种常发生在表皮或皮肤附属器的恶性肿瘤,起源于上皮干细胞,位居皮肤肿瘤发病率的第一位 [1]。尽管发病率高,但病死率低,据估计,540万确诊为非黑色素瘤皮肤癌(NMSCs)的患者中,有330万患基底细胞癌,转移率仅有0.0028%~0.5%,经年龄调整后的死亡率仅有0.12‰ [2]。研究显示,Hedgehog信号通路的异常可导致BCC的发生。Vismodegib等小分子抑制剂可阻断这一信号通路,抑制肿瘤生长 [3] [4]。然而肿瘤的发生通常存在着多条细胞信号通路的异常,且各通路之间存在着互相交叉的关系,现发病机制仍不是十分明确。因此,全面探究基底细胞癌潜在的候选基因具有重要的意义。本研究从GEO数据库中下载其原始数据(GSE125285)进行分析,通过比较基底细胞癌和癌旁正常细胞的基因表达谱筛选出差异基因(DEGs),然后对DEGs进行基因功能注释分析和信号传导通路富集分析,进而从分子水平研究皮肤基底细胞癌的发生机制,为靶向治疗寻找有意义的治疗靶点。
2. 材料和方法
2.1. 获取基因芯片数据
从GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)下载皮肤基底细胞癌相关基因表达谱芯片(编号GSE125285),该研究是基于GPL1154平台(Illumina HiSeq 2000 (Homo sapiens))的芯片。该基因表达谱中有25个人皮肤基底细胞癌组织样本,及25个癌旁正常皮肤组织样本。
2.2. 差异基因筛选
利用R语言软件(https://www.r-project.org/)的Limma包运行已处理的基因矩阵文件筛选出差异表达基因,差异表达基因(Differentially expressed genes, DEGs)的筛选条件为差异倍数(Fold change, FC)对数值的绝对值|Log2(FC)| ≥ 2,并且P < 0.05为差异具有统计学意义,并用火山图体现。
2.3. 差异基因的GO分析和KEGG分析
基因本体(GO)分析(Gene ontology analysis, GO)是一种常用于注释基因和基因产物以及鉴别高通量基因组或转录组数据特有生物学属性的方法 [5];京都基因和基因组数据库(Kyoto encyclopedia of genes and genomes, KEGG)可用于系统分析基因功能和有高通量功能信息的相耦合基因组 [6]。DAVID数据库(http://david.abcc.ncifcrf.gov/)是一套完整的基因功能注释在线分析工具,供研究者理解大量基因背后的生物学意义。本研究将筛选出上调及下调的DEGs分别输入DAVID数据库进行GO及KEGG分析,以P < 0.01为标准进行筛选。
2.4. 蛋白相互作用网络分析
通过STRING数据库及Cytoscape3.8.0软件对DEGs所编码的蛋白构建蛋白–蛋白相互作用网络。进一步筛选得到相关度最高的10个蛋白,其对应的基因即为枢纽基因。
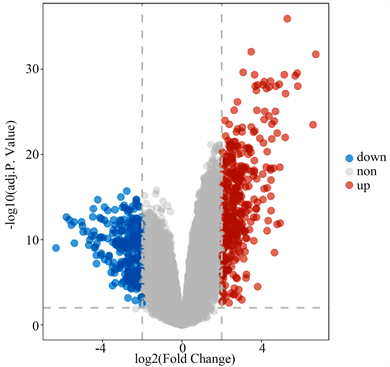 注:火山图展示了基因表达情况的分布状况,横轴表示差异倍数,纵轴表示P值,蓝色的点表示下调基因,红色的点表示上调基因。
注:火山图展示了基因表达情况的分布状况,横轴表示差异倍数,纵轴表示P值,蓝色的点表示下调基因,红色的点表示上调基因。
Figure 1. Volcanic plot of differential genes expression
图1. 基底细胞癌差异基因表达谱的火山图
3. 结果
3.1. 筛选差异表达基因
利用R语言软件的Limma包分析GSE125285芯片的基因表达谱,共筛选出653个差异表达基因,其中387个上调基因,266个下调基因。差异表达基因以火山图表示(见图1)。
3.2. GO本体分析
GO分析显示上调基因主要富集的生物进程(BP):胶原纤维的形成、细胞外基质的组成、胶原蛋白分解代谢的过程、皮肤发育、调控B细胞活化、吞噬作用;下调基因主要富集的BP:氧化还原过程、脂质代谢过程、亚油酸代谢过程、药物反应。上调基因主要富集的分子功能(MF):细胞外基质结构成分、结合免疫球蛋白受体、抗原结合;下调基因主要富集的MF:结构分子活性。上调基因主要富集的细胞成分(CC):蛋白质性细胞外基质、胞外区、胶原三聚体、细胞外隙;下调基因主要富集的CC:角蛋白丝、中间丝状体、脂质颗粒、细胞外隙、内质网膜。以P值从小到大进行排列,展示GO分析中的结果,见表1。
Table 1. GO analysis of the differential genes
表1. BCC差异基因的GO分析结果
3.3. KEGG信号通路分析
对DEGs进行KEGG富集分析后显示,上调基因主要富集的信号通路包括:蛋白质的消化和吸收、基底细胞癌的形成和Hedgehog信号通路(图2);下调基因仅有一条信号通路为:过氧化物酶体增殖物激活受体。以P值从小到大进行排列,展示KEGG分析中的结果,见表2。
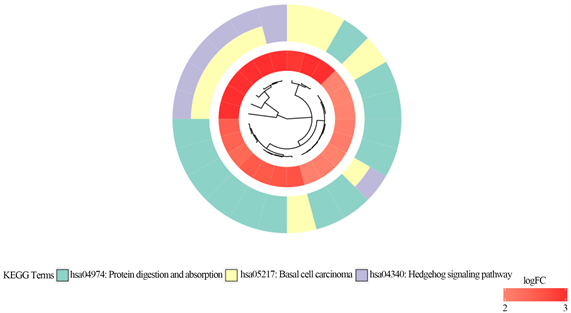
Figure 2. KEGG enrichment analysis of up-regulated genes
图2. 上调基因KEGG富集分析结果
Table 2. KEGG analysis of the differential genes
表2. KEGG通路富集分析结果
3.4. 构建蛋白–蛋白相互作用网络及筛选核心基因
通过STRING数据库及Cytoscape3.8.0软件对DEGs所编码的蛋白构建蛋白–蛋白相互作用网络(图3)。进一步筛选得到相关度最高的10个蛋白,其对应的基因即为枢纽基因。枢纽基因的成员包括:MYC,IL6,PPARG,FOS,LEP,EGR1,COL1A1,NTRK3,SPP1及ADIPOQ。

Figure 3. Protein-protein interaction network diagram of differentially expressed genes
图3. 差异表达基因的蛋白相互作网络
4. 讨论
皮肤基底细胞癌(BCC)是最常见的非黑色素瘤性皮肤癌,由Kropecher于1903年首次发现并报道,好发于中老年人(≥40岁),其发生的主要原因是由紫外线照射引起编码DNA发生基因突变 [7]。皮肤基底细胞癌临床表现多样,起病隐匿,早期自觉症状轻微或无自觉症状。治疗方式有多种,目前手术扩大切除是彻底治愈基底细胞癌的首选方案,光动力疗法可直接杀伤肿瘤细胞并造成血管损伤,作为非手术疗法治疗基底细胞癌已得到广泛应用,其有效性已经被证实,可使手术范围缩小,有益于伤口愈合及减少瘢痕,增加手术的美观度 [8]。基底细胞癌发病率逐年上升且病情诊断易延误,具体发病机制尚不明确,故探索其潜在的致病机制已成为近年来国内外研究的热点。
本研究应用生物信息学分析方法确定了BCC和正常对照组间具有表达差异的基因653个,包括387个上调基因和266个下调基因,应用GO分析和KEGG分析方法探讨了DEGs的功能。GO分析显示,上调的DEGs富集的细胞成分主要是胞外区,具体包括了细胞外基质结构成分的形成;下调的DEGs主要参与外泌体的形成。KEGG分析显示上调基因主要富集的通路为:Hedgehog信号通路,Hedgehog信号通路失调与基底细胞癌的形成密切相关。我们的研究与先前的研究基本一致,显示出该通路是基底细胞癌发生的核心通路,Hedgehog信号通路是一个经典的控制胚胎发育的信号转导途径,控制细胞的生长与增殖 [9]。Notch、RAS/MAPK、Wnt/β-catenin等通路在既往的研究中也发现与基底细胞癌的形成有关 [10]。
本研究对DEGs进行分析,筛选出10个最具代表性的枢纽基因:MYC,IL6,PPARG,FOS,LEP,EGR1,COL1A1,NTRK3,SPP1及ADIPOQ。分析显示,MYC是连通度最高的枢纽基因,说明该基因与BCC的发生及进展关系最为密切,可能是本病潜在的生物标志物 [11]。已知C-myc与肿瘤发生与转归密切相关,N-myc对肿瘤的预后判断有重要意义,L-myc与肿瘤的易患性和预后在不同的肿瘤中表现不一样。研究表明,与正常皮肤相比,基底细胞癌中MYC水平较低,这与先前大多数研究结果一致,即MYC表达的上调可以使人角质形成细胞快速增殖,MYC表达随角质形成细胞终末分化的发生而下调 [4]。第二个枢纽基因是IL-6,IL-6广泛存在于多种细胞中,IL-6膜受体形成可溶性受体,经信号传导和转录激活因子发挥生物效应 [12]。IL-6在不同类型的人类肿瘤中的作用已被广泛研究,研究表明它可能以自分泌或旁分泌的方式,通过改变肿瘤环境直接或间接影响肿瘤的生长,与恶性肿瘤的发病、预后及治疗存在密切的关系 [13]。IL-6多作为上路基因参与多条信号传导通路,在包括BCC在内的多种癌症中均过表达 [14]。我们可以考虑通过监测IL-6水平变化来合理治疗,同时IL-6的检测对恶性肿瘤的诊断也有重要价值。第三个枢纽基因为PPARG,PPAR(也称为过氧化物酶体增殖激活受体)是核激素受体亚家族配体激活转录因子的成员,调节皮肤生理的多个方面,包括皮脂细胞分化,角质形成细胞增殖,基底细胞存活,脂肪细胞生物学和炎症性皮肤反应,并在人类多种癌症中差异表达 [15]。目前的研究数据和临床经验表明,PPARAG可以直接介导抗肿瘤和免疫调节作用,以及广泛的基质调节活性,包括抗血管生成、抗炎和免疫增强作用 [16]。PPARG配体也证实了其抗增殖和诱导凋亡的作用 [16]。然而,对BCC发生及进展的影响尚不明确,有待进一步研究。第四个枢纽基因是FOS。有研究表明,FOS基因通过增加某些蛋白酶的产生,破坏基底膜和周围组织,使癌细胞获得更高的侵袭能力 [17]。除此之外,试验证明浸润型BCC中FOS阳性的细胞数量大于局限型BCC [18]。因此,浸润型BCC相较于局限性BCC有更高的侵袭性及复发性。第五个枢纽基因是LEP。LEP (也称为瘦素)是一种主要由脂肪组织产生的激素,LEP的分泌与体重呈正相关,因此,肥胖伴随着较高水平的LEP [19]。有证据表明,肥胖引起的炎症与紫外线辐射引起的炎症相互作用,加速皮肤癌变过程。例如,肥胖小鼠的暴露于UVB的皮肤中炎症因子TNF-α,IL-6和IL-1b的水平较高,表明肥胖与UVB诱导的炎症之间存在正相关 [20]。在这些高水平的炎症因子的作用下,可发生各种炎症性皮肤病 [21],在持续高水平的炎症因子的作用下,可能导致皮肤癌风险增加 [22] [23]。第六个枢纽基因是EGR-1。该基因为即刻早期反应基因家族中最为重要的一员,多种因素均可使EGR-1转录激活,活化的EGR-1可与靶基因启动子特异性结合,从而调控下游靶基因的转录和表达。目前研究发现,EGR-1在组织床上修复在上以及肿瘤性疾病的发生、发展中扮演重要作用。研究表明EGR-1在BCC中低表达,可在体外抑制良性及恶性表皮细胞的生长,并可同时抑制Cdc25A表达和Cdk2去磷酸化 [24]。因此,EGR-1基因表达谱可以区分表皮过度增生性疾病,并表明EGR-1可防止表皮细胞的非正常生长。第七个枢纽基因是COL1A1。I型胶原是细胞外质的主要成分,也是器官纤维化时病变部位沉积的主要细胞外质成分。I型胶原由两条α1链和一条α2链组成。这两种多肽链分别由COLI1A1和COLI1A2基因编码,通常以2:1的比例合成,形成三螺旋结构。有研究证明,COL1A1与基地细胞痣综合征(KCOTs)的发生、进展有关 [25]。COL1A1高表达具有更大的成骨潜能,因此肿瘤的分化较高、侵袭性较低。第八个枢纽基因是NTRK3。该基因编码神经营养性酪氨酸受体激酶(NTRK)家族的一个成员。这种激酶是一种膜结合受体,通过神经营养素结合,参与自身磷酸化和丝裂原活化蛋白激酶(MAPK)通路。黑素细胞与神经系统有共同的神经外胚层起源,酪氨酸受体在正常黑素细胞和黑色素瘤细胞系中均表达 [26]。研究发现NTRK3融合是黑素细胞瘤中的癌基因,在进展过程的早期出现,与其他激活MAPK的黑色素瘤癌基因相互排斥 [27]。目前尚无研究证明NTRK3对BCC的发生发展有密切关系,有待进一步的研究。第九个枢纽基因是SPP1。SPP1 (也称为骨桥蛋白)通过参与NF-κB信号通路和激活整合素αvβ3 [28] [29],在黑色素瘤的进展中发挥作用 [30]。SPP1的高表达与肿瘤厚度增加、Clark分级增加和核分裂象增多有关 [31]。此外,SPP1表达可显著预测系统性红斑狼疮淋巴结转移发生率及数量 [32]。对皮肤基底细胞癌的影响有待进一步研究。最后一个枢纽基因是ADIPOQ。研究表明,ADIPOQ (也称为脂联素)水平与葡萄糖、脂肪代谢和胰岛素敏感性有关,并影响与癌症发生密切相关的炎症发展 [33];其次,ADIPOQ的缺乏会抑制AMPK的激活,从而导致直接参与细胞增殖的mTOR通路的激活,从而导致癌症的发生;第三,ADIPOQ可以显著影响免疫反应,显著激活几种抗炎细胞因子,抑制NF-JB依赖的TNF和IFN-c等炎症因子的产生 [34]。对皮肤基底细胞癌的影响上不明确,有待进一步研究。
5. 结论
综上所述,本研究对BCC的基因表达差异进行了全面的生物信息学分析,对明确诊断、靶向治疗及预测预后有非常重要的意义。目前,现有研究对基底细胞癌的认识尚不全面,需要进一步发掘相关基因及治疗靶点,以期为基底细胞癌患者实施精准的靶向治疗药物提供新的理论依据。
文章引用
王 达,王丽欣,陈官芝. 基于生物信息学分析基底细胞癌的基因表达及信号传导通路
Bioinformatics Analysis of Gene Expression and Signaling Pathways in Basal Cell Carcinoma[J]. 临床医学进展, 2020, 10(08): 1782-1790. https://doi.org/10.12677/ACM.2020.108268
参考文献
- 1. Lear, J.T. and Smith, A.G. (1997) Basal Cell Carcinoma. Postgraduate Medical Journal, 73, 538-542. https://doi.org/10.1136/pgmj.73.863.538
- 2. Lomas, A., Leonardi-Bee, J. and Bath-Hextall, F. (2012) A Systematic Review of Worldwide Incidence of Nonmelanoma Skin Cancer. British Journal of Dermatology, 166, 1069-1080. https://doi.org/10.1111/j.1365-2133.2012.10830.x
- 3. Besselink-Lobanova, A., Maandag, N.J.G., Voermans, N.C., et al. (2010) Trachea Rupture in Tenascin-Xdeficient Type Ehlers-Danlos Syndrome. Anesthesiology, 113, 746-749. https://doi.org/10.1111/j.1365-2133.2012.10830.x
- 4. Wan, C. and Li, Y. (2020) Integrative Analysis of mRNA-miRNA-TFs Reveals the Key Regulatory Connections Involved in Basal Cell Carcinoma. Archives of Dermatological Research, 312, 133-143. https://doi.org/10.1007/s00403-019-02002-y
- 5. Ashburner, M., Ball, C.A., Blake, J.A., et al. (2000) Gene Ontology: Tool for the Unification of Biology. The Gene Ontology Consortium. Nature Genetics, 25, 25-29. https://doi.org/10.1038/75556
- 6. Ogata, H., Goto, S., Sato, K., et al. (1999) KEGG: Kyoto Encyclopedia of Genes and Genomes. Nucleic Acids Research, 27, 29-34. https://doi.org/10.1093/nar/27.1.29
- 7. Marzuka, A.G. and Book, S.E. (2015) Basal Cell Carcinoma: Pathogenesis, Epidemiology, Clinical Features, Diagnosis, Histopathology, and Management. The Yale Journal of Biology and Medicine, 88, 167-179.
- 8. 邢天娇, 李东霞. 基底细胞癌诊治的研究进展[J]. 医学综述, 2019(1): 60-64.
- 9. 潘思文, 白云, 阎超, 等. 浅谈Hedgehog信号通路与肿瘤的相关性[J]. 中国药师, 2015(7): 1203-1206.
- 10. 乔琪, 张国惠. 紫外线所致基底细胞癌的分子生物学机制研究进展[J]. 世界最新医学信息文摘, 2019(98): 78-79 + 84.
- 11. Kim, D.P., Kus, K.J.B. and Ruiz, E. (2019) Basal Cell Carcinoma Review. Hematology/Oncology Clinics of North America, 33, 13-24. https://doi.org/10.1016/j.hoc.2018.09.004
- 12. Sławińska, M., Zabłotna, M., Gleń, J., et al. (2019) STAT3 Polymorphisms and IL-6 Polymorphism Are Associated with the Risk of Basal Cell Carcinoma in Patients from Northern Poland. Archives of Dermatological Research, 311, 697-704. https://doi.org/10.1007/s00403-019-01952-7
- 13. Brennan-Crispi, D.M., Overmiller, A.M., Tamayo-Orrego, L., et al. (2019) Overexpression of Desmoglein 2 in a Mouse Model of Gorlin Syndrome Enhances Spontaneous Basal Cell Carcinoma Formation through STAT3-Mediated Gli1 Expression. Journal of Investigative Dermatology, 139, 300-307. https://doi.org/10.1016/j.jid.2018.09.009
- 14. Taher, M.Y., Davies, D.M. and Maher, J. (2018) The Role of the Interleukin (IL)-6/IL-6 Receptor Axis in Cancer. Biochemical Society Transactions, 46, 1449-1462. https://doi.org/10.1042/BST20180136
- 15. Heller, E.R., Gor, A., Wang, D., et al. (2013) Molecular Signatures of Basal Cell Carcinoma Susceptibility and Pathogenesis: A Genomic Approach. International Journal of Oncology, 42, 583-596. https://doi.org/10.3892/ijo.2012.1725
- 16. Ogino, S., Shima, K., Baba, Y., et al. (2009) Colorectal Cancer Expression of Peroxisome Proliferator-Activated Receptor γ (PPARG, PPARgamma) Is Associated with Good Prognosis. Gastroenterology, 136, 1242-1250. https://doi.org/10.1053/j.gastro.2008.12.048
- 17. Urabe, A., Nakayama, J., Taniguchi, S., et al. (1994) Expression of the fos Oncogene in Basal Cell Carcinoma. Journal of Dermatological Science, 8, 50-53. https://doi.org/10.1016/0923-1811(94)90321-2
- 18. Lacroix, M. and Body, J.J. (1997) Regulation of c-fos and c-jun Expression by Calcitonin in Human Breast Cancer Cells. Calcified Tissue International, 60, 513-519. https://doi.org/10.1007/s002239900273
- 19. Strong, A.L., Ohlstein, J.F., Biagas, B.A., et al. (2015) Leptin Produced by Obese Adipose Stromal/Stem Cells Enhances Proliferation and Metastasis of Estrogen Receptor Positive Breast Cancers. Breast Cancer Research, 17, 112. https://doi.org/10.1186/s13058-015-0622-z
- 20. Farag, A.G.A., Elnaidany, N.F. and El-Dien, M.M.S. (2016) Immunohistochemical Expression of Leptin in Non Melanoma Skin Cancer. Journal of Clinical and Diagnostic Research, 10, WC08-WC12. https://doi.org/10.7860/JCDR/2016/19755.8385
- 21. Sharma, S.D. and Katiyar, S.K. (2010) Leptin Deficiency-Induced Obesity Exacerbates Ultraviolet B Radiation-Induced Cyclooxygenase-2 Expression and Cell Survival Signals in Ultraviolet B-Irradiated Mouse Skin. Toxicology and Applied Pharmacology, 244, 328-335. https://doi.org/10.1016/j.taap.2010.01.010
- 22. Mukhtar, H. and Elmets, C.A. (1996) Photocarcinogenesis: Mechanisms, Models and Human Health Implications. Photochemistry and Photobiology, 63, 356-357. https://doi.org/10.1111/j.1751-1097.1996.tb03040.x
- 23. Scott, K.A., Moore, R.J., Arnott, C.H., et al. (2003) An Anti-Tumor Necrosis Factor-α Antibody Inhibits the Development of Experimental Skin Tumors. Molecular Cancer Therapeutics, 2, 445-451.
- 24. Fang, M., Wee, S.A., Ronski, K., et al. (2007) Evidence of EGR1 as a Differentially Expressed Gene among Proliferative Skin Diseases. Genomic Medicine, 1, 75-85. https://doi.org/10.1007/s11568-007-9010-9
- 25. Hong, Y.Y., Yu, F.Y., Qu, J.F., et al. (2014) Fibroblasts Regulate Variable Aggressiveness of Syndromic Keratocystic and Non-Syndromic Odontogenic Tumors. Journal of Dental Research, 93, 904-910. https://doi.org/10.1177/0022034514542108
- 26. Tallegas, M., Fraitag, S., Binet, A., et al. (2019) Novel KHDRBS1-NTRK3 Rearrangement in a Congenital Pediatric CD34-Positive Skin Tumor: A Case Report. Virchows Archiv, 474, 111-115. https://doi.org/10.1007/s00428-018-2415-0
- 27. Yeh, I., Tee, M.K., Botton, T., et al. (2016) NTRK3 Kinase Fusions in Spitz Tumours. Journal of Pathology, 240, 282-290. https://doi.org/10.1002/path.4775
- 28. Naor, D., Sionov, R.V. and Ish-Shalom, D. (1997) CD44: Structure, Function, and Association with the Malignant Process. Advances in Cancer Research, 71, 241-319. https://doi.org/10.1016/S0065-230X(08)60101-3
- 29. Liaw, L., Birk, D.E., Ballas, C.B., et al. (1998) Altered Wound Healing in Mice Lacking a Functional Osteopontin Gene (spp1). Journal of Clinical Investigation, 101, 1468-1478. https://doi.org/10.1172/JCI1122
- 30. Crawford, H.C., Matrisian, L.M. and Liaw, L. (1998) Distinct Roles of Osteopontin in Host Defense Activity and Tumor Survival during Squamous Cell Carcinoma Progression in Vivo. Cancer Research, 58, 5206-5215.
- 31. Rangel, J., Nosrati, M., Torabian, S., et al. (2008) Osteopontin as a Molecular Prognostic Marker for Melanoma. Cancer, 112, 144-150. https://doi.org/10.1002/cncr.23147
- 32. Kashani-Sabet, M., Nosrati, M., Miller, J.R., et al. (2017) Prospective Validation of Molecular Prognostic Markers in Cutaneous Melanoma: A Correlative Analysis of E1690. Clinical Cancer Research, 23, 6888-6892. https://doi.org/10.1158/1078-0432.CCR-17-1317
- 33. Xu, Y., He, B., Pan, Y., et al. (2013) The Roles of ADIPOQ Genetic Variations in Cancer Risk: Evidence from Published Studies. Molecular Biology Reports, 40, 1135-1144. https://doi.org/10.1007/s11033-012-2154-2
- 34. Tilg, H. and Moschen, A.R. (2006) Adipocytokines: Mediators Linking Adipose Tissue, Inflammation and Immunity. Nature Reviews Immunology, 6, 772-783. https://doi.org/10.1038/nri1937
NOTES
*通讯作者。
