Open Journal of Fisheries Research
Vol.02 No.04(2015), Article ID:16601,7
pages
10.12677/OJFR.2015.24008
Comparison of Mitochondrial D-Loop and Cyt b Sequences of Hypophthalmichthys molitrix
Weijun Wu, Min Peng, Dapeng Wang, Jun Shi, Yusen Li, Yaoquan Han, Jianjun Lei, Anyou He*
*通讯作者。
Guangxi Key Laboratory of Aquatic Genetic Breeding and Healthy Aquaculture, Guangxi Institute of Fisheries, Nanning Guangxi

Received: Dec. 5th, 2015; accepted: Dec. 18th, 2015; published: Dec. 24th, 2015
Copyright © 2015 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



ABSTRACT
Sequences of mitochondrial D-loop and Cyt b gene of 24 individuals of Hypophthalmichthys molitrix which were sampled from three populations in Hongshui River were determined. Using PCR amplification and sequencing, 820 bp of D-loop partial sequences and 1117 bp of Cyt b partial sequences were obtained. In D-loop sequences, a total of 74 polymorphic sites were defined; 20 distinct haplotypes, 19 transitions, 3 transversions and 52 insert/lacuna loci were found. In Cyt b sequences, 48 polymorphic sites and 9 haplotypes were defined; 13 transitions, 1 transversion and 34 insert/lacuna loci were found. Two-sequence analysis showed that, within groups and between groups, the genetic distance is small, and the genetic variation of molecular variation level is not significant. Upper, middle and lower reaches are shared haplotypes, and the genetic differentiation among three populations is little.
Keywords:Hypophthalmichthys molitrix, Hongshui River, D-Loop, Cyt b, Genetic Diversity

红水河鲢线粒体D-Loop和Cyt b基因序列分析
吴伟军,彭敏,王大鹏,施军,李育森,韩耀全,雷建军,何安尤*
广西水产科学研究院/广西水产遗传育种与健康养殖重点实验室,广西 南宁

收稿日期:2015年12月5日;录用日期:2015年12月18日;发布日期:2015年12月24日

摘 要
研究了红水河野生鲢Hypophthalmichthys molitrix上游、中游和下游3个群体24个个体的线粒体控制区(D-loop)和细胞色素b (Cyt b)基因序列,通过PCR扩增与测序,分别获得了长度为820 bp和1117 bp的D-loop和Cyt b基因片段。分析表明,D-loop序列共定义了20个单倍型,存在74个多态位点,发生转换19次、颠换3次、插入/缺失52次。Cyt b序列共定义了9个单倍型,存在48个多态性位点,发生转换13次、颠换1次、插入/缺失34次。两序列分析结果表明,群体间和群体内遗传距离较小,遗传变异的分子变异等级不显著。上、中、下游均有共享单倍型,3个群体间遗传分化不大。
关键词 :鲢,红水河,D-Loop,Cyt b,遗传变异

1. 引言
鲢(Hypophthalmichthys molitrix)属鲤形目,鲤科,鲢亚科,鲢属。又名白鲢、跳鲢、鲢子。体侧扁、稍高,呈纺锤形,头大吻短,口宽、眼小,鳞细,体呈银白色,腹部有肉棱。鲢滤食浮游植物,生长速度快、产量高,在广西一般做为生态养殖品种。鲢属中上层鱼。春夏秋三季,绝大多数时间在水域的中上层游动觅食,冬季则潜至深水越冬。秋除浮游生物外,还大量地吃腐屑类饵料。其食欲与水温成正比,夏季水位越低,其摄食量越大:冬季则少吃少动。最适宜的水温为23℃~32℃。鲢性情活泼,喜欢跳跃,有逆流而上的习性。
线粒体D-环区(displacement loop region, D-loop),是线粒体DNA中的一段非编码区,位于tRNA Pro和tRNA Phe基因之间,是线粒体DNA序列和长度变异最大的区域,也是线粒体基因中进化最快的部分,适用于种群水平差异的检测,也可用于种间分析[1] -[3] 。细胞色素b基因(Cytochrome b gene, Cyt b)是线粒体DNA上的蛋白质编码基因,其进化速度适中,适合种群水平差异的检测,也是探讨种内群体遗传分化程度的良好标记 [4] [5] 。对鲢线粒体DNA的研究多见于长江流域,如李思发等对长江中下游四大家鱼线粒体DNA多样性进行了分析 [6] 。张四明等对长江中游汉江和湘江水系鲢和草鱼群体的mtDNA遗传变异进行了对比研究 [7] 。朱日财对赣江赣州江段四大家鱼遗传多样性进行了研究 [8] 。珠江流域,尤其是广西境内区域尚未见相关研究报道,本文对红水河流域3个相互分隔的鲢群体进行遗传多样性状况和群体遗传结构分析,为红水河流域物种的多样性保护提供必要的遗传背景资料和数据支持。
2. 材料与方法
2.1. 材料
采集3个鲢群体共27条,分别为上游岩滩水库群体(shang)、中游百龙滩水库群体(zhong)和下游来宾市区江段群体(xia),三个群体采样区域有大坝分隔,大坝无过鱼设施,可认为群体间无遗传交流。每尾个体取0.1 g背部肌肉新鲜样品,于无水乙醇中保存。
2.2. 测序方法
通过液氮研磨、裂解液裂解、有机溶剂抽提,使进入水相的蛋白质与核酸分层,在RNA酶的作用下,降解RNA,得纯度较高的DNA样品并进行PCR测序。D-loop引物序列为:DL1 (5′-ACCCCTGGCTCCCAA AGC-3′),DH2 (5′-ATCTTAGCATCTTCAGTG-3′) [9] 。Cyt b引物序列为:L14724 (5′--GACTTGAAAAACC ACCGTTG-3′),H15915 (5′--CTCCGATCTCCGGATTACAAG AC-3′) [5] 。反应总体积50 μL,其中10 × Taq Buffer 5 μL,Taq Polymer-ase 0.5 μL (5 U∙μL−1),dNTPs (2.5 mmol∙L−1) 4 μL,Mg2+ (25 mmol∙L−1) 2.5 μL,引物1和2 (10 umol∙L−1)各μL,模板DNA 50 ng,无离子超纯水35.5 μL。扩增条件为:94℃预变性2 min后,再进行35个循环,每一循环包括:94℃ 45 s,58℃ 45 s,72℃ 1 min;最后72℃延伸7 min。每次PCR反应均设不含模板DNA的空白对照。扩增产物经W = 1.5% TBE琼脂糖凝胶电泳分离,EB染色,紫外灯光下检测、拍照。
2.3. 分析方法
使用SeqMan II软件拼接获得序列并人工校对,用CLUSTAL-X程序比对,DAMBE统计单倍型。用ARLEQUIN统计软件计算获得单倍型多样性(Haplotype diversity, H)和核苷酸多样性(Nucleotide diversity, π),群体间的分化指数(F-statistics, Fst) [10] ,群体遗传变异的分子方差分析(AMOVA) [11] 。群体间的基因流由公式Nm = [(1/Fst) − 1]/2 [12] 计算得出,采用MEGA4.1软件统计DNA序列的碱基组成[13] ,转换/颠换、插入/缺失位点数,单突变位点,简约信息位点;个体间、群体内和群体间的遗传变异率利用Kimura双参数模型计算;用NJ法进行聚类分析,构建聚类关系树。利用Network4610软件构建单倍型网络关系图。
3. 结果
3.1. D-Loop和Cyt b基因序列信息
由表1可见,在27个820 bp的D-loop基因序列中共检测出20个单倍型。检测到4个单突变位点,70个简约信息位点数;在74个突变位点中:转换位点19个,颠换位点3个,插入/缺失位点52个,转换高于颠换。基因片段中,A + T的含量(67.3%)明显高于C + G的含量(32.7%)。29个1117 bp的Cyt b基因序列中共检测出9个单倍型,检测到1个单突变位点,47个简约信息位点;在48个突变位点中:转换位点13个,颠换位点1个,插入/缺失位点34个,A + T的含量(58.3%)高于C + G的含量(41.6%)。对比分析可知:Cyt b单突变位点、多态位点百分比和简约信息位点百分比分别为4.30%、4.21%和0.09%,均小于D-loop,后者分别为9.02%,8.54%和0.48%。
3.2. D-Loop和Cyt b基因序列的遗传变异分析
由表2可见,D-loop序列中,红水河3个鲢群体内的遗传距离分别为0.0322、0.0246和0.0150。上游群体与中游群体间遗传距离为0.0284,上游群体与下游群体间遗传距离为0.0246,中游群体和下游群体间遗传距离为0.0199。Cytb基因序列中,群体内的遗传距离分别为0.0164、0.0111、0.0036。上游群体与中游群体间遗传距离为0.0137,上游群体与下游群体间遗传距离为0.0107,中游群体和下游群体间遗传距离为0.0074。两序列的群体内遗传距离均为上游高于中游,中游又高于下游。
3个群体的D-loop和Cyt b序列的单倍型多样性H、平均核苷酸变异数K、和核苷酸多样性π见表3,下游群体的D-loop和Cyt b序列单倍型多样性高于上游和中游,可能与其采集到的样品较少,检测到的单倍型数也较少有关,可以作为例证的是,其平均核苷酸变异数K和核苷酸多样性π低于上游和中游。对比D-loop和Cyt b序列,D-loop的平均核苷酸变异数和核苷酸多样性在上、中下游均高于Cyt b序列。
Table 1. Composition and variability of the analyzed mtDNA of H. molitrix
表1. 鲢线粒体D-loop及Cyt b碱基组成及变异分析
Table 2. The intrapopulation, interpopulation and overall mean genetic distances of H. molitrix in Hongshui River based on D-loop and Cyt b gene
表2. 红水河鲢D-loop和Cyt b基因序列群体内和群体间遗传距离
注:上面的代表D-loop,下面的代表Cyt b (下同)。
Table 3. Indexes of genetic diversity in the 3 populations of H. molitrix
表3. 3个鲢群体相关遗传多样性指标
表4和表5分别列出群体间的Fst、Nm值和群体内群体间的遗传变异的分子变异等级分析(AMOVA),从分析结果可见,3个群体间D-loop和Cyt b序列遗传差异均不显著(Fst = −0.01379或−0.03160,p > 0.05)。
3.3. 分子系统树
以鲢D-loop和Cyt b基因序列为外群,用NJ法构建分子系统树,其拓扑结构见图1。可见,3个群体的个体混杂在一起,群体间未表现出聚类关系。
4. 讨论
单倍型多样性(H)、平均核苷酸差异数(K)和核苷酸多样性(π)是反映物种遗传变异水平的3个重要参数,3个群体遗传多样性差别不大,群体内遗传距离表现为上游高于中游,中游高于下游。遗传变异的分子变异等级分析结果为不显著。24个样本的D-loop序列分属20个单倍型,而Cyt b序列分属9个单倍型。上、中、下游均有共享单倍型,NJ系统树也显示3个群体并未聚集为独立的分支。由于线粒体DNA为母系遗传,一般不发生重组,表明3个群体间遗传分化不大。
Table 4. Three population pairwise Fst and Nm values
表4. 3个鲢群体间的Fst和Nm
Table 5. Analysis of molecular variance (AMOVA) of 3 populations
表5. 3个鲢群体间的遗传变异的分子变异等级分析(AMOVA)
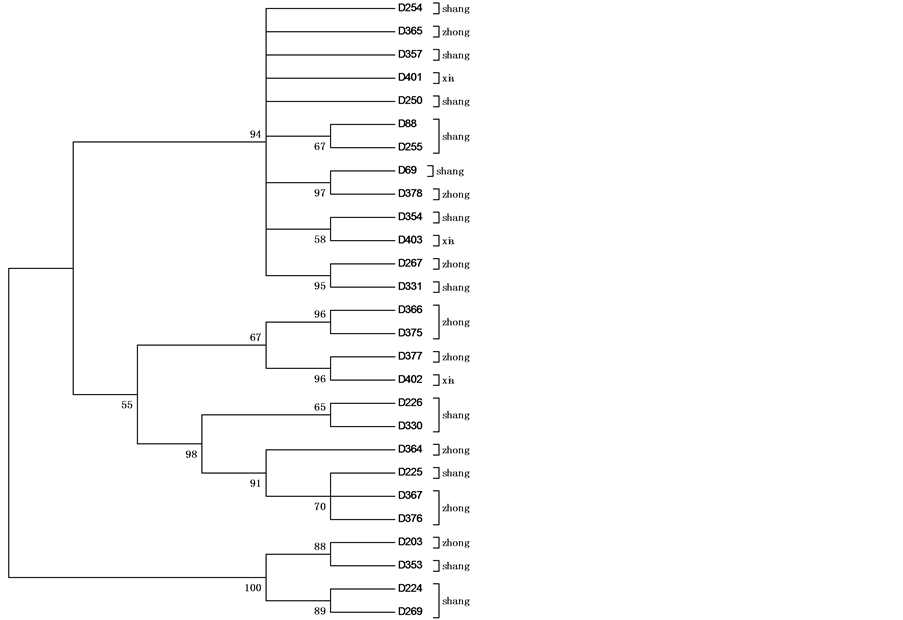
Figure 1. The molecular phylogenetic tree of H. molitrix in Hongshui River by NJ method based on D-loop (left) and Cyt b (right)
图1. 红水河3个鲢群体D-loop (左)和Cyt b (右)基因序列的NJ分子系统树
红水河综合利用规划实施后,红水河流域形成了9个首尾衔接的梯级水库,流水生境绝大部分已经萎缩,天然河道和库区流水江段大为缩短,多数流水性鱼类,特别是产漂流性卵的鱼类,在库区已不具备形成自然种群条件 [14] ,近十年来,针对产漂流性卵鱼类资源下降的问题,各地政府大力开展了增殖放流活动,由于其他土著性鱼类人工繁育技术成熟度相对较低,苗种获得不易,四大家鱼的放流数量占了各地放流比例的90%以上。而鲢由于其食性为滤食性,在红水河流域各水库普遍具有富营养化趋势的前提下,在增殖放流中所占比例较大,更是生态网箱养殖和围栏养殖的主要品种。鲢的3个群体间遗传分化不大,可能是流域开发时间仅三十年左右,原本连通的群体分隔后尚未产生显著遗传分化。也可能是各地增殖放流和生态养殖的鲢,亲本来源较为接近,流域原有的群体因繁殖环境丧失,已经完全被放流群体取代。由于缺乏红水河流域梯级开发前,鲢的遗传多样性分析数据,无法确定是何种原因导致。笔者所在的研究团队拟开展红水河流域鲢捕捞群体和各地放流群体间的遗传多样性对比分析,以明确红水河鲢遗传多样性特征的形成机理。
5. 总结
对红水河野生鲢上游、中游和下游3个群体的24个个体的D-loop和Cyt b基因序列分析表明,D-loop序列的单倍型数、多态位点数均高于Cyt b序列。红水河鲢群体间和群体内遗传距离较小,遗传变异的分子变异等级不显著。上、中、下游均有共享单倍型,3个群体间遗传分化不大。可能是原本连通的群体分隔后尚未产生显著遗传分化,也可能是来自近缘亲本的增殖放流群体已经取代了野生群体。
基金项目
社会公益研究项目(CXIF-2014-006);自治区直属公益性科研院所基本科研业务专项资金项目(编号:GXIF-2014-006)。
文章引用
吴伟军,彭 敏,王大鹏,施 军,李育森,韩耀全,雷建军,雷建军,何安尤. 红水河鲢线粒体D-Loop和Cyt b基因序列分析
Comparison of Mitochondrial D-Loop and Cyt b Sequences of Hypophthalmichthys molitrix[J]. 水产研究, 2015, 02(04): 67-73. http://dx.doi.org/10.12677/OJFR.2015.24008
参考文献 (References)
- 1. 郭新红, 刘少军, 刘巧, 等. 鱼类线粒体DNA研究新进展[J]. 遗传学报, 2004, 31(9): 983-990.
- 2. Lee, W.J. and Kocher, T.D. (1996) Complete Sequence of a Sea Lamprey (Petromyzoa marinus) Mitochondrial Genome: Early Es-tablishment of the Vertebrate Genome Organization. Genetics, 139, 873-887.
- 3. 肖武汉, 张亚平. 鱼类线粒体DNA的遗传与进化[J]. 水生生物学报, 2000, 24(4): 384-391.
- 4. Mommsen, H. (1993) Biochemistry and Molecular Biology of Fishes. Elsevier Science Publishers, London, 1-38.
- 5. Xiao, W.H., Zhang, Y.P. and Liu, H.Z. (2001) Molecular Systematics of Xenocyprinae (Telestei: Cyprinidae): Taxonomy, Biogeography, and Evolution of a Special Group Restricted in East Asia. Molecular Phylogenetics and Evolution, 18, 163-173. http://dx.doi.org/10.1006/mpev.2000.0879
- 6. 李思发, 吕国庆, Louis, B., 等. 长江中下游鲢鳙草青四大家鱼线粒体DNA多样性分析[J]. 动物学报, 1998, 44(1): 82-93.
- 7. 张四明, 汪登, 强邓怀, 等. 长江中游水系鲢和草鱼群体mtDNA遗传变异的研究[J]. 水生生物学报, 2002, 26(2): 142-147.
- 8. 朱日财. 赣江赣州江段四大家鱼生物学特性及其遗传多样性的研究[D]: [硕士学位论文]. 江西: 南昌大学, 2010: 23-40.
- 9. 唐琼英, 刘焕章, 杨秀平, 等. 沙鳅亚科鱼类线粒体DNA控制区结构分析及系统发育关系的研究[J]. 水生生物学报, 2005, 29(6): 645-653.
- 10. Wright, S. (1965) The Interpretation of Population Structure by F2 Statistics with Special Regard to Systems of Mating. Evolution, 19, 395-420. http://dx.doi.org/10.2307/2406450
- 11. Excoffier, L., Smouse, P.E. and Quatfro, J.M. (1992) Analysis of Molecular Variance Inferred from Metric Distances among DNA Heliotypes: Application to Human Mitochondrial DNA Restriction Data. Genetics, 131, 479-497.
- 12. Nei, M. (1982) Evolution of Human Races at the Gene Level. In: Bonne, T.B., Cohen, T., Goodman, R.M., et al., Eds., Human Genetics, Part A: The Unfolding Genome, Alan R Liss, New York, 167-181.
- 13. Kumar, S., Tamura, K., Jakobseni, B., et al. (2001) MEGA2.0: Molecular Evolutionary Genetics Analysis Software. Arizona State University, Tempe.
- 14. 王崇, 谢山, 谢文星, 等. 红水河干流梯级运行后鱼类资源调查[J]. 淡水渔业, 2015, 45(2): 30-36.
