Botanical Research
Vol.
10
No.
06
(
2021
), Article ID:
46656
,
11
pages
10.12677/BR.2021.106096
利用转录组数据分析水稻植株包穗的分子机制
龙晨洁,张蕾*
武汉大学杂交水稻国家重点实验室,湖北 武汉
收稿日期:2021年10月23日;录用日期:2021年11月15日;发布日期:2021年11月23日

摘要
包穗会引起水稻不能正常授粉,进而降低水稻的产量和经济效益。研究水稻抽穗的调控机理具有非常重要的科学意义和应用价值。CRKs (CDPK-related kinases)是一种植物特有的钙依赖性蛋白激酶相关激酶。水稻CRK5-RNAi植株在抽穗期部分或全部稻穗被顶叶鞘包裹。本文以RNAi植株和野生型水稻抽穗期间的倒一节部位为实验材料,利用RNA-Seq技术探索研究水稻植株包穗的机制。结果显示,RNAi植株中2581个基因表达上调,2208个下调;GO分析和KEGG分析表明DEGs主要富集在植物激素信号转导、代谢过程、催化活性等。qRT-PCR实验表明ABA应答基因、GA信号通路基因、转录因子和叶绿体合成相关基因等10个DEGs表达趋势与RNA-Seq一致,表明CRK5基因在抽穗期间主要在植物激素信号转导通路中发挥功能。
关键词
水稻,抽穗期,包穗,转录组,植物激素信号转导

Transcriptome Analysis Identifies Genes Involved in Rice Panicle Enclosure
Chenjie Long, Lei Zhang*
State Key Laboratory of Hybrid Rice, Wuhan University, Wuhan Hubei
Received: Oct. 23rd, 2021; accepted: Nov. 15th, 2021; published: Nov. 23rd, 2021

ABSTRACT
Panicle enclosure can cause the failure of rice pollination, and then reduce the yield and economic benefit of rice. It is of great scientific significance and application value to study the regulation mechanism of rice heading. CRKs (CDPK-related kinases) is a plant-specific calcium-dependent protein kinase-related kinase. In rice CRK5-RNAi plants, part or all of the panicles are covered by apical leaf sheaths at the heading stage. In this study, RNAi plants and the inverted portion of wild-type rice during heading were used as experimental materials to explore the mechanism of rice panicle enclosure by RNA-Seq technology. The results showed that 2581 genes were up-regulated and 2208 genes down-regulated in RNAi plants. GO analysis and KEGG analysis showed that DEGs were mainly enriched in plant hormone signal transduction, metabolic process and catalytic activity. qRT-PCR showed that the expression trends of 10 DEGs, including ABA response genes, GA signaling pathway genes, transcription factors and chloroplast synthesis genes, were consistent with RNA-Seq, indicating that CRK5 gene mainly plays a role in plant hormone signal transduction pathway during heading.
Keywords:Rice, Heading Stage, Panicle Enclosure, Transcriptome, Plant Hormone Signal Transduction

Copyright © 2021 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
在水稻抽穗期间,由于倒一节间伸长不足而致小穗不能完全抽出,被顶叶鞘部分或全部包裹的现象称为包穗。尹昌喜阐述了细胞质雄性不育株系中花粉败育导致生长素亏缺,进而致使倒一节间中活性赤霉素合成受阻,抑制倒一节间伸长而产生包穗的分子机理 [1]。水稻包穗会影响正常授粉,直接造成水稻产量降低。因此,研究水稻抽穗的调控机理具有重要的科学意义和应用价值。
本实验室主要对水稻中的CRKs进行研究。与典型的CDPKs (Calcium-dependent protein kinases)相比,CRKs的连接区具有钙调素结合结构域,可以结合钙调素;并且CRKs的EF手型结构退化,不受钙离子的直接调控,因此CRKs的激酶活性受Ca2+和CaM的协同调控 [2] [3] [4]。与CDPKs相比,对CRKs的研究较少。
实验室前期已通过RNA沉默技术获得了水稻CRK5基因的RNAi转基因植株,通过对转基因植株的表型观察发现:与野生型相比,CRK5-RNAi转基因植株在开花授粉结束,甚至种子成熟时,花穗仍有部分或全部被顶叶鞘包裹(图1)。为进一步探索研究CRK5在抽穗期的功能,笔者选取抽穗期的倒一节部位进行转录组测序分析。

Figure 1. Comparison of phenotypes at heading stage
图1. 抽穗期表型对比
本研究拟通过Illumina平台的高通量RNA-Seq技术对水稻抽穗后的倒一节间中基因表达情况进行分析,筛选出抑制(或延迟)水稻开花的关键基因。通过GO分析和KEGG代谢通路分析,找出参与水稻抽穗期间的代谢途径以及次生代谢产物合成的相关基因,为进一步探究水稻CRK5基因的功能提供参考依据。
2. 材料与方法
2.1. 水稻材料种植
实验中所用到的野生型水稻品种为“Hejiang19”,CRK5-RNAi转基因植株的遗传背景是Hejiang19。
水稻种子用5%次氯酸钠消毒后转移至含有MS培养基的育苗盒中,在28℃光照培养箱中生长一周后,转移到水培液中继续培养约20天,再将其移植到植物温室的泥土盆中生长,条件为光照时间16 h,黑暗时间8 h,温度为25℃~32℃。
2.2. 转录组测序材料选取
待水稻材料生长至五叶期(生长26天)时,取水稻叶片提取RNA,反转录为cDNA,对CRK5基因表达量进行qRT-PCR检测,qRT-PCR结果证实CRK5-RNAi转基因植株中CRK5基因表达量只有野生型中的20% (图2)。
当水稻生长至抽穗期(生长60天),此时Hejiang19植株的小穗已完全抽出,而CRK5-RNAi转基因植株的小穗仍部分被顶叶鞘包裹。分别取野生型和转基因植株的倒一节部位,取材后立即放入液氮中冷冻。
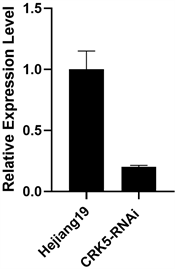
Figure 2. qRT-PCR results
图2. qRT-PCR结果
实时荧光定量PCR分析Hejiang19和CRK5-RNAi叶片中CRK5基因的表达。
2.3. 总RNA提取
材料由北京擎科生物科技有限公司使用植物总RNA提取试剂盒进行RNA提取,对总RNA的量与纯度进行质控检测。经检测,样品总RNA浓度 > 50 ng/μL,RIN值 > 7.0,OD260/280 > 1.8,Total RNA > 1 μg满足建库测序要求,随后进行文库构建及转录组测序。
2.4. Illumina测序文库的构建及测序数据分析
利用mRNA含有PolyA尾的特征,使用oligo(dT)磁珠通过两轮的纯化对mRNA进行特异性捕获。将捕获到的mRNA进行片段化并合成cDNA,同时在其中掺入dUTP Solution。通过PCR使其形成片段大小为300 bp ± 50 bp的文库。进行双端测序。下机原始数据格式为fastq,进行处理后得到CleanData,然后对基因或转录本进行初组装,合并。之后对基因或转录本进行FPKM定量。对样本之间进行显著差异分析,将差异倍数FoldChange > 2倍且P-value < 0.01的基因定义为差异基因,并对其进行GO和KEGG富集分析。
2.5. 差异表达基因的qRT-PCR验证
取同时期的Hejiang19及CRK5-RNAi转基因植株的倒一节部位,液氮速冻,使用TRIzol试剂提取总RNA,合成cDNA。依据待分析基因的CDS序列设计qRT-PCR的特异性引物,所有引物均由北京擎科生物科技有限公司进行合成。使用2 × SYBR Green qPCR Master Mix对10个待检测基因进行qRT-PCR分析,以Actin作为内参基因。PCR反应体系总体积为10 μL,其中2 × SYBR Green qPCR Master Mix使用量为5 μL,上下游引物各0.25 μL,cDNA模板2 μL,无菌水2.5 μL,每个样品进行3次技术重复和生物学重复。PCR反应程序为:95℃预变性5 min,之后95℃ 15 s,60℃ 30 s,重复40个循环,最后每5 s升高0.5℃ (从65℃至95℃)绘制溶解曲线,以相对表达量ΔΔCq计算。
3. 结果与分析
3.1. 测序质量分析
材料进行RNA提取后进行转录组测序,测序共产出17.41 G Raw data,经过测序质量控制,共过滤得到16.76 G Clean Data,各样品Q30碱基百分比均不小于95.45% (表1),说明测序质量很好,可以做进一步分析探究。
Table 1. Statistical table of transcriptome sequencing data
表1. 转录组测序数据统计表
3.2. 差异表达基因筛选及统计
在差异表达基因检测过程中,将差异倍数(Fold Change) ≥ 2且错误发现率(False Discovery Rate, FDR) < 0.01作为筛选标准。经过分析统计(图3),共筛选出4789个差异表达基因,其中有2581个基因表达水平在CRK5-RNAi植株中的表达量高于Hejiang19,2208个基因表达下调。其中|log2FC| ≤ 10的差异表达基因占比99.62%;|log2FC| > 10的差异表达基因有18个,其中13个上调,5个下调。从表2可以看出,表达上调的基因主要与植物激素信号转导因子(脱落酸ABA、生长素)、氧化酶等相关;表达下调的基因与渗调蛋白、还原酶、细胞色素等相关。
Table 2. |log2FC| > 10 DEGs
表2. |log2FC| > 10的显著差异表达基因
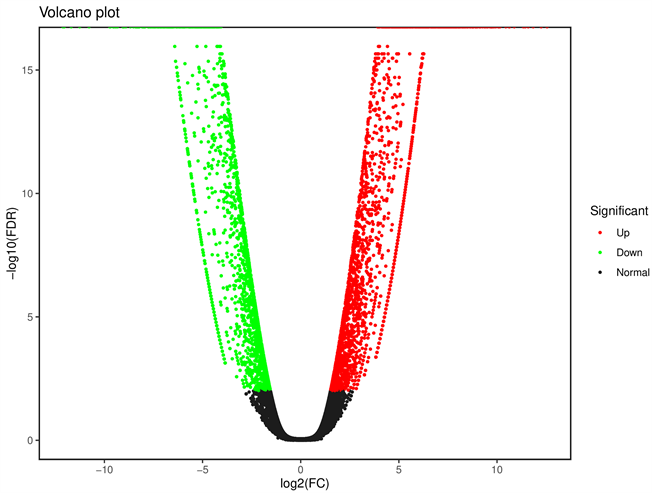
Figure 3. Volcano map of differentially expressed genes
图3. 差异表达基因火山图
3.3. 差异表达基因GO分析
对筛选得到的差异表达基因进行GO分析 [5],绘制分类统计图(图4)。GO分析表明,在抽穗期间从CRK5-RNAi植株与Hejiang19植株中筛选得到的差异表达基因中,参与生物过程(BP)的差异表达基因占31.60%,细胞组分(CC)占34.50%,分子功能(MF)占33.90%。说明多数差异表达基因与其生物学功能显著相关。BP中主要富集到生物学过程(GO:0008150,581个)、代谢过程(GO:0008152,411个)、有机物代谢过程(GO:0071704,316个)、氮化合物代谢过程(GO:0006807,241个)、细胞大分子代谢过程(GO:0044260,183个)、高分子代谢过程(GO:0043170,226个)、有机氮化合物的代谢过程(GO:1901564,148个)等7个方面;在CC中主要富集在细胞内膜结合的细胞器(GO:0043231,464个)、膜固有成分(GO:0031224,322个)、细胞质(GO:0005737,163个)等3个方面;在MF中主要富集在结合(GO:0005488,430个)、催化活性(GO:0003824,404个)等2个方面。这表明在抽穗期间,CRK5-RNAi植株与野生型植株的代谢过程差异较大,主要发生在细胞内膜结合的细胞器如质体、叶绿体类囊体等,涉及结合与催化活性等生物学功能方面的基因较为活跃。
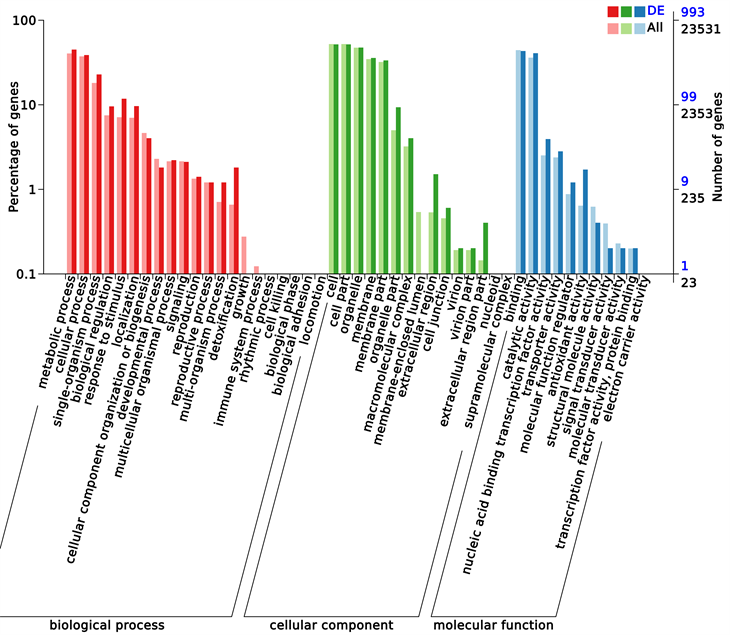 横坐标为GO分类,纵坐标左边为基因数目所占百分比,右边为基因数目。
横坐标为GO分类,纵坐标左边为基因数目所占百分比,右边为基因数目。
Figure 4. GO annotation classification statistical map of DEGs
图4. 差异表达基因GO注释分类统计图
3.4. 差异表达基因KEGG代谢路径分析
为探索抽穗期间CRK5-RNAi植株与Hejiang19植株代谢途径的变化,进一步揭示差异表达基因的生物学功能,对转录组测序得到的DEGs进行KEGG代谢途径富集分析 [6],绘制差异表达基因KEGG通路富集散点图(图5)。结果表明DEGs共富集到94条代谢通路上,主要富集在植物激素信号转导(Plant hormone signal transduction, ko04075)、碳代谢(Carbon metabolism, ko01200)、苯丙素的生物合成(Phenylpropanoid biosynthesis, ko00940)、MAPK信号通路(MAPK signaling pathway, ko04016)、光合作用(Photosynthesis, ko00195)、氨基酸生物合成(Biosynthesis of amino acids, ko01230)、乙醛酸和二羧酸代谢(Glyoxylate and dicarboxylate metabolism, ko00630)、淀粉和蔗糖代谢(Starch and sucrose metabolism, ko00500)、核糖体(Ribosome, ko03010)等通路上,其中植物激素信号转导通路富集的差异表达基因最多。
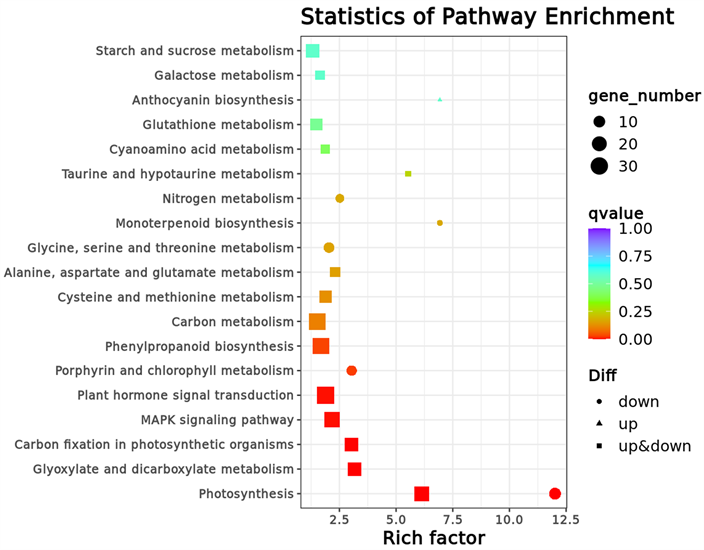
Figure 5. Rich hub map of KEGG pathway of DEGs
图5. 差异表达基因KEGG通路富集散点图
3.5. 差异表达基因的qRT-PCR验证
为了验证上述RNA-Seq实验结果,笔者随机选取了包括ABA应答基因、GA信号通路基因、转录因子和叶绿体合成相关基因等10个差异表达基因,对其抽穗期间转录水平的表达量进行qRT-PCR进行验证。
实验结果表明(图6),绝大部分基因的qRT-PCR结果与RNA-Seq变化趋势一致,其中6个DEGs在CRK5-RNAi植株中的表达量高于野生型,分别为Os11g0453900 (ABA响应基因)、Os11g0454300 (ABA应答因子基因)、Os02g0810200 (激酶相关蛋白,调节细胞分裂和植物发育)、Os04g0465300 (赤霉素刺激转录基因)、Os06g0341300 (胚胎发育晚期丰富蛋白)和Os12g0541300 (烟酰胺腺嘌呤二核苷酸磷酸氧化酶基因);有4个DEGs在CRK5-RNAi植株中的表达量低于在野生型中的表达量,分别为Os02g0610500 (水稻开花抑制因子)、Os01g0227100 (叶绿素b还原酶基因)、Os10g0419500 (双加氧酶蛋白)和Os10g0567400 (叶绿素a加氧酶基因)。
虽然差异表达基因的qRT-PCR结果与RNA-Seq结果的表达差异倍数不尽相同,但其表达趋势一致,再次验证了本次转录组测序实验结果的可靠性。植物激素信号转导通路相关基因在水稻抽穗期间表达量发生变化,很有可能就是造成两种植株抽穗期间表型变化的原因。
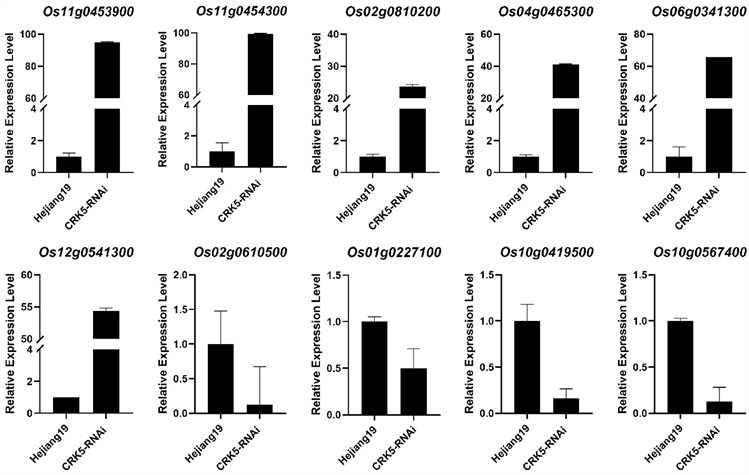
Figure 6. Verified DEGs expression by qRT-PCR
图6. qRT-PCR验证DEGs表达量
4. 讨论
随着转录组测序技术的普及应用,科研人员可以有效利用这一新兴技术来探究不同特定时空环境下某个基因的功能,通过基因表达量分析、差异表达分析、GO注释及分析、KEGG代谢通路分析等可以推断未知基因的功能,或者研究某个基因在生物体信号通过中的大致位置,为进一步深入研究提供了生物学信息基础。目前对于水稻中钙依赖性蛋白激酶(CDPK)的研究较多,但关于钙依赖性蛋白激酶相关激酶(CDPK-related kinases, CRKs)的研究不多,笔者观察到实验室前期获得的CRK5-RNAi植株与野生型在抽穗期间存在差异,基于这一现象,笔者对同种环境下生长的两种植株进行转录组测序分析。
本研究使用Illumina平台的高通量测序对CRK5-RNAi和Hejiang19在抽穗期间的倒一节部位进行全转录组测序分析,比较了两者的差异表达基因。结果表明,在抽穗期间,与Hejiang19相比,CRK5-RNAi植株表达上调的DEGs有2581个,比下调的DEGs多373个;GO功能分类表明DEGs主要富集在生物学过程、代谢过程、有机物代谢过程、氮化合物代谢过程、细胞大分子代谢过程、高分子代谢过程、有机氮化合物的代谢过程等7个生物过程;在细胞组分分类中主要富集在细胞内膜结合的细胞器、膜固有成分、细胞质等3个方面;在分子功能分类中主要富集在结合和催化活性等2个方面;KEGG代谢通路分析表明DEGs主要富集在代谢过程、催化活性、植物激素信号转导、碳代谢、MAPK信号通路、核糖体等方面,这表明在抽穗期间,CRK5-RNAi植株与野生型植株的代谢过程差异较大,主要发生在细胞内膜结合的细胞器如质体、叶绿体类囊体等,涉及结合与催化活性等生物学功能方面的基因较为活跃,主要通过植物激素信号转导途径参与调控植物生长。
水稻生长发育过程主要分为五个阶段:幼苗期、分蘖期、拔节期、抽穗期和结实期等 [7],其中水稻抽穗期直接影响到植株产量,该阶段的生长发育受光照、温度、激素等多种因素共同影响。如在抽穗期间,外源施加活性GA可促进倒一节间伸长,解除包穗表型 [8],因此在农业生产上,常用施加赤霉素的方法促进倒一节的伸长生长进而解决水稻包穗问题。本研究检测并验证了3个ABA信号和GA信号通路基因Os11g0453900、Os11g0454300和Os04g0465300在CRK5-RNAi植株中的表达量高于在野生型中的表达量,表明这两种植株在抽穗期间的差异有可能是由于激素信号转导不同造成的,暗示了CRK5基因也在信号转导过程中发挥作用。Os02g0610500基因属于包含B-box结构域的锌指蛋白家族,BBX蛋白在植物的生长发育过程中发挥重要作用,如幼苗的光形态发生、开花的光周期调节、避荫和对生物或非生物胁迫的响应等 [9] [10] [11] [12],已有研究发现,BBX蛋白Hd1和OsCO3是水稻中的CO同源物,可调节抽穗期 [13] [14]。OsBBX14编码一个带有两个B-box且没有CCT基序的BBX转录因子,作为Ehd1的间接抑制因子而延迟抽穗 [15]。qRT-PCR验证表明BBX家族蛋白(Os02g0610500)在CRK5-RNAi植株中的表达量降低,与RNA-Seq结果一致。
5. 结论
本研究基于观察到的水稻抽穗期间表型差异,采用RNA-Seq技术对决定抽穗长度的水稻倒一节部分进行转录组分析,经过GO分析和KEGG分析,确定了两种植株的DEGs主要富集在代谢过程、催化活性、植物激素信号转导等方面,这表明在抽穗期间,CRK5-RNAi植株与野生型植株的代谢过程差异较大。同时,采用qRT-PCR对10个DEGs的表达水平进行验证,结果与RNA-Seq的表达趋势变化一致,说明RNA-Seq结果可靠。本研究为进一步探究水稻CRK5基因的功能以及揭示水稻抽穗的分子机理提供理论依据。
致谢
衷心感谢张蕾副教授在我研究生求学过程中给予我的鼓励和帮助,感谢张老师对本篇论文的认真指导,感谢国家自然科学基金(31570288)对本论文的资助。感谢左世文先生、李子慧、王子颖、范兆宇等同学对我的支持和帮助。
文章引用
龙晨洁,张 蕾. 利用转录组数据分析水稻植株包穗的分子机制
Transcriptome Analysis Identifies Genes Involved in Rice Panicle Enclosure[J]. 植物学研究, 2021, 10(06): 770-780. https://doi.org/10.12677/BR.2021.106096
参考文献
- 1. 尹昌喜. CMS水稻包穗及不良开花习性的激素调控[M]. 北京: 化学工业出版社, 2018: 1-3.
- 2. Wang, Y., Liang, S., Xie, Q.G. and Lu, Y.T. (2004) Characterization of a Calmodulin-Regulated Ca2+-Dependent- Pro-tein-Kinase-Related Protein Kinase, AtCRK1, from Arabidopsis. Biochemical Journal, 383, 73-81. https://doi.org/10.1042/BJ20031907
- 3. Popescu, S.C., Popescu, G.V., Bachan, S., Zhang, Z., Seay, M., Gerstein, M., Snyder, M. and Dinesh-Kumar, S.P. (2007) Differential Binding of Calmodulin-Related Proteins to Their Targets Revealed through High-Density Arabidopsis Protein Microarrays. Proceedings of the National Academy of Sciences of the United States of America, 104, 4730-4735. https://doi.org/10.1073/pnas.0611615104
- 4. Rigó, G., Ayaydin, F., Tietz, O., Zsigmond, L., Kovács, H., Páy, A., Salchert, K., Darula, Z., Medzihradszky, K.F., Szabados, L., Palme, K., Koncz, C, and Cséplo, A. (2013) Inactivation of Plasma Membrane-Localized CDPK- RELATED KINASE5 Decelerates PIN2 Exocytosis and Root Gravitropic Response in Arabidopsis. Plant Cell, 25, 1592-1608. https://doi.org/10.1105/tpc.113.110452
- 5. Ashburner, M., Ball, C.A., Blake, J.A., Botstein, D., Butler, H., Cherry, J.M., et al. (2000) Gene Ontology: Tool for the Unification of Biology. The Gene Ontology Consortium. Nature Genetics, 25, 25-29. https://doi.org/10.1038/75556
- 6. Kanehisa, M., Goto, S., Kawashima, S., Okuno, Y. and Hattori, M. (2004) The KEGG Resource for Deciphering the Genome. Nucleic Acids Research, 32, D277-D280. https://doi.org/10.1093/nar/gkh063
- 7. Du, H., Wang, L. and Xiong, L.Z. (2018) Phenotypic Observations of Rice Critical Development Stages. Bio-101, Article ID: e1010178. https://doi.org/10.21769/BioProtoc.1010178
- 8. Nagai, K., Mori, Y., Ishikawa, S., Furuta, T., Gamuyao, R., Niimi, Y., et al. (2020) Antagonistic Regulation of the Gibberellic Acid Response during Stem Growth in Rice. Nature, 584, 109-114. https://doi.org/10.1038/s41586-020-2501-8
- 9. Cheng, X.F. and Wang, Z.Y. (2005) Overexpression of COL9, a CONSTANS-LIKE Gene, Delays Flowering by Reducing expression of CO and FT in Arabidopsis thaliana. The Plant Journal, 43, 758-768. https://doi.org/10.1111/j.1365-313X.2005.02491.x
- 10. Crocco, C.D., Locascio, A., Escudero, C.M., Alabadí, D., Blázquez, M.A. and Botto, J.F. (2015) The Transcriptional Regulator BBX24 impairs DELLA Activity to Promote Shade avoidance in Arabidopsis Thaliana. Nature Communications, 6, Article No. 6202. https://doi.org/10.1038/ncomms7202
- 11. Gangappa, S.N., Crocco, C.D., Johansson, H., Datta, S., Hettiarachchi, C., Holm, M. and Botto, J.F. (2013) The Arabidopsis B-BOX Protein BBX25 Interacts with HY5, Negatively Regulating BBX22 Expression to Suppress Seedling Photomorphogenesis. Plant Cell, 25, 1243-1257. https://doi.org/10.1105/tpc.113.109751
- 12. Kumagai, T., Ito, S., Nakamichi, N., Niwa, Y., Murakami, M., Yamashino, T. and Mizuno, T. (2008) The Common Function of a Novel Subfamily of B-Box Zinc Finger Proteins with Reference to Circadian-Associated Events in Arabidopsis Thaliana. Bioscience, Biotechnology, and Biochemistry, 72, 1539-1549. https://doi.org/10.1271/bbb.80041
- 13. Kim, S.K., Yun, C.H., Lee, J.H., Jang, Y.H., Park, H.Y. and Kim, J.K. (2008) OsCO3, a CONSTANS-LIKE Gene, Controls Flowering by Negatively Regulating the Expression of FT-Like Genes under SD Conditions in Rice. Planta, 228, Article No. 355. https://doi.org/10.1007/s00425-008-0742-0
- 14. Yano, M., Katayose, Y., Ashikari, M., Yamanouchi, U., Monna, L., Fuse, T., et al. (2000) Hd1, a Major Photoperiod Sensitivity Quantitative Trait Locus in Rice, Is Closely Related to the Arabidopsis Flowering Time Gene CONSTANS. Plant Cell, 12, 2473-2484. https://doi.org/10.1105/tpc.12.12.2473
- 15. Bai, B., Zhao, J., Li, Y., Zhang, F., Zhou, J., Chen, F. and Xie, X. (2016) OsBBX14 Delays Heading Date by Repressing Florigen Gene Expression under Long and Short-Day Condi-tions in Rice. Plant Science, 247, 25-34. https://doi.org/10.1016/j.plantsci.2016.02.017
附录
Table A1. The primer sequence for qRT-PCR
附表1. qRT-PCR用引物序列
NOTES
*通讯作者。
