Advances in Microbiology
Vol.
07
No.
03
(
2018
), Article ID:
26936
,
9
pages
10.12677/AMB.2018.73014
Distribution Characteristics of Repeat Sequences in the Genome of the Yeast Recombinant Han0458 Obtained by Low-Energy Ion Beam Implantation
Hanyu Zhang1, Chao Tang1, Ting Wang2, Guangwen Feng1, Weidong Qian2, Peihong Mao1,3*, Changlong Cai3*
1Research Center of Ion Beam Biotechnology, College of Physics Science and Technology, Xinjiang University, Urumqi Xinjiang
2School of Food and Biological Engineering, Shaanxi University of Science & Technology, Xi’an Shaanxi
3Research Center of Ion Beam Biotechnology and Biodiversity, Xi’an Technological University, Xi’an Shaanxi

Received: Sep. 5th, 2018; accepted: Sep. 18th, 2018; published: Sep. 25th, 2018

ABSTRACT
Repeat sequence is an important part of the genome and play an important role in the biological evolution, genetics, and regulation of gene expression. In order to further understand the biological effects of low-energy ion implantation on the yeast genome structure, in this study, we used bioinformatics methods to study the distribution characteristics of the genomic repeat sequences on the basis of de novo sequencing of the Han0458 genome. The results shows that the length of tandem repeats of the recombinant Han0458 genome is 227,825 bp and interspersed repeats is 107,439 bp, which account for 2.46% of the total genome length, and the frequency of repeats in the genome is 0.25 per Kb. The copy number of repeat units in microsatellite DNA are mostly less than 15 times, the dominant base type is trinucleotide repeats, and the number of microsatellite sequences with repeat unit motif AAC is the largest; the copy number of repeat units in minisatellite DNA is smaller than that of microsatellite DNA. The number of minisatellite DNA with a repeat unit greater than 15bp is decreasing with the increase of the repeat unit length. The number of long-terminal repeats (LTRs) is the largest in interspersed repeats, and the average length of rolling coils (RCs) is the longest. The results of this study provide molecular evidence for low-energy ion implantation-mediated yeast genome mutation and evolution.
Keywords:Ion Beam Recombinant Yeast, Genome, Minisatellite DNA, Microsatellite DNA, Interspersed Repeats
离子束重组酵母菌Han0458基因组重复序列的分布特征
张寒玉1,唐朝1,王婷2,冯光文1,钱卫东2,毛培宏1,3*,蔡长龙3*
1新疆大学,物理科学与技术学院,离子束生物技术中心,新疆 乌鲁木齐
2陕西科技大学,食品与生物工程学院,陕西 西安
3西安工业大学,离子束生物工程与生物多样性研究中心,陕西 西安

收稿日期:2018年9月5日;录用日期:2018年9月18日;发布日期:2018年9月25日

摘 要
重复序列是基因组的重要组成部分,对生物的进化、遗传和基因的表达调控有重要作用。为了进一步认识低能离子注入对酵母菌基因组结构的生物学效应,本研究利用生物信息学方法,在离子束重组酵母菌株Han0458全基因组de novo测序的基础上,对其基因组重复序列的分布特征进行了研究。结果表明,重组菌株Han0458基因组串联重复序列227,825 bp,散在重复序列107,439 bp,共占基因组长度的2.46%,重复序列在基因组中出现的频率为0.25个/Kb。微卫星DNA中重复单元拷贝数大多低于15次,其优势碱基类型为三碱基重复,重复单元基序为AAC的微卫星序列数目最多;小卫星DNA重复单元拷贝数小于微卫星DNA,主要分布1~3次,重复单元大于15 bp的小卫星序列数目随着重复单位长度的增加呈下降趋势。散在重复序列中长末端重复序列(LTR)数目最多,滚环(RC)平均长度最长。本研究结果为低能离子注入介导的酵母菌基因组突变与进化提供了分子证据。
关键词 :离子束重组酵母菌,基因组,微卫星DNA,小卫星DNA,散在重复序列

Copyright © 2018 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
重复序列是真核生物基因组中重要的组成部分,按其在基因组中的分布方式,分为串联重复序列(Tandem Repeat Sequences)和散在重复序列(Interspersed Repeat Sequences) [1] 。串联重复序列又可根据其重复单元长度划分为卫星DNA (Satellite DNA) [2] 、小卫星DNA (Minisatellite DNA) [3] 和微卫星DNA (Microsatellite DNA) [4] 。其中微卫星DNA又称为短串联重复序列(Short Tandom Repeat, STR)或简单重复序列(Simple Sequence Repeats, SSRs),随机分布于生物体整个基因组中,微卫星标记作为理想的分子遗传标记,被广泛地用于目的基因筛选、基因诊断多样性分析及遗传连锁图谱构建等工作中。而散在重复序列(又称转座子元件,Transposable Element, TE)分为RNA介导的转座元件(又称RNA转座子)和DNA介导的转座元件(又称DNA转座子),不仅可以影响基因组的大小,还能直接或间接促成基因组重排,并可影响基因表达水平、改写基因调控网络。因此TEs对基因组的进化中具有重要意义 [5] 。本研究在离子束重组酵母菌株Han0458全基因组de novo测序的基础上,利用生物信息学方法分析其基因组中各种重复序列的类型及分布特点,以期深入了解低能离子注入对酵母菌基因组结构特征的影响,并为重组菌的分子育种及SSR分子标记的开发提供理论依据。
2. 材料与方法
2.1. 菌株重复序列的获取
低能N+注入介导外源基因转化异常汉逊酵母,经选择性筛选获得重组菌株Han0458 [6] [7] 。应用PacBio单分子测序技术对其进行基因组De nove测序,获得的基因组DNA序列,作为本研究的基本数据。
2.2. 生物信息学方法
应用TRF (Tandem Repeat Finder)方法 (http://tandem.bu.edu/trf/trf404.linux64.download.html)获取重组菌株Han0458基因组DNA序列中的串联重复序列,最大的重复单元bp数设置为2000 bp。
对TRF获取的结果进行细分,设置微卫星DNA序列重复单位为2~6 bp,小卫星DNA序列重复单位为10~60 bp。
使用RepeatMasker方法(http://www.repeatmasker.org/RMDownload.html)获取重组菌株Han0458基因组DNA序列中的散在重复序列。
3. 结果与分析
3.1. 微卫星DNA序列的分布特征
利用TRF方法在重组菌株Han0458基因组重复序列中发现了175个SSR,总长为8310 bp,占基因组序列总长度的0.06%,平均每77.98 Kb就能检测到一个SSR。
SSR在三碱基(Trinucleotide)模体中的数目最多,为97条,占重复序列总数的55.43%;其次是六碱基(Hexanucleotide)模体,为47条,占26.86%;五碱基(Pentanucleotide)和四碱基(Tetranucleotide)模体数目相对较少,分别在13~16条之间,占比约为7.43%~9.14%;二碱基(Dinucleotide)模体的重复序列最少,仅有3条,占1.71% (图1)。
碱基类型的重复基序分析结果(表1)表明,在4种两碱基类型重复中,仅有AT重复基序。
三碱基类型重复基序有10种,其中数量较多的碱基类型依次是AAC (51条,52.58%)、ACT (13条,13.40%)、ATG (12条,12.37%)。累积长度最长的依然是上述三个类型的重复序列:AAC (2176 bp)、ACT (756 bp)、ATG (595 bp)。
四碱基类型重复序列中含有AAAT、AACT、ATTA、GTTA和TTCA重复类型,且前两种类型数量较多,占四碱基重复序列数目的76.92%,长度较长,约占四碱基重复序列累积长度的66.42%;
五碱基类型重复序列共有15条,其中AATTC和TATAC重复单元的序列各有2条,其余各类型基序重复序列均只有1条。
六碱基类型重复序列共47条,其每种重复单元基序数目,均为1~2个。
各种重复类型的拷贝数分析结果(表2)表明,微卫星序列均在低拷贝区出现频率较高,拷贝数低于15次的微卫星序列占比73.14%;拷贝数在15~27之间的微卫星序列,占比18.29%;拷贝数在27~39之间的占比7.43%;拷贝数大于39次的微卫星序列最少,仅占1.14%。
从图2可以看出,拷贝数越大,微卫星序列数目越少,微卫星平均拷贝数随着重复单位长度的增加而减少。

Figure 1. Distribution of the SSR of the recombinant yeast strain Han0458 genome in different motif types
图1. 重组酵母菌株Han0458基因组SSR在不同模体类型中的分布
Table 1. Repeat motif distribution of Microsatellite DNA in the genome of yeast recombinant Han0458
表1. 重组菌株Han0458基因组微卫星DNA的重复基序分布
Table 2. Copy number distribution of different motifs of Microsatellite DNA in the genome of yeast recombinant Han0458
表2. 重组菌株Han0458基因组微卫星DNA不同模体的拷贝数分布
注:Dinucleotide:两碱基;Trinucleotide:三碱基;Tetranucleotide:四碱基;Pentanucleotide:五碱基;Hexanucleotide:六碱基。
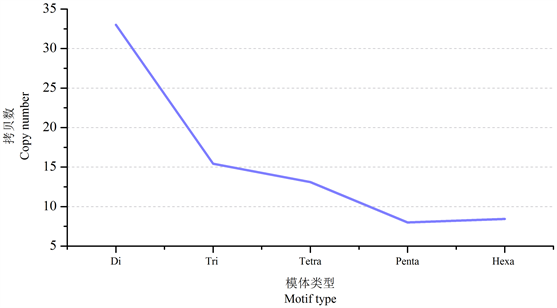
Figure 2. Relationship between repeat unit length and copy number of Microsatellite DNA in the genome of yeast recombinant Han0458
图2. 重组菌株Han0458基因组微卫星DNA的重复单元长度与拷贝数关系
3.2. 小卫星DNA序列的分布特征
利用TRF方法在重组菌株Han0458基因组的串联重复序列中发现了1361条小卫星DNA序列,总长分别为74,362bp,占串联重复序列72.71%,占基因组序列总长的0.54%,平均每10 Kb出现一个小卫星序列。
小卫星DNA序列集中分布于25 bp~74 bp,占小卫星DNA序列总数量的78.55%,其中长度为30 bp的小卫星序列数目最多,为76条;重复单位为15 bp的序列数目最多,高达192条,占小卫星序列总数的14.11%;以18 bp为重复单元的序列长度最长,8258 bp,占小卫星序列总长的11.11%;各重复单元的拷贝数范围为1.9~20.7,平均拷贝数为2.6的重复序列数目最多。小卫星DNA序列的重复类型数目、序列长度及拷贝数如图3所示。
小卫星DNA序列数目与重复单位长度有一定关系,随着重复单位长度的增加呈下降趋势,这一特征在重复单元大于15 bp的小卫星序列中尤为显著;与微卫星DNA类似,小卫星DNA序列重复单位拷贝数较低,主要分布在1~3次;重复单元拷贝数与小卫星DNA序列之间无显著相关关系(图4)。
3.3. 散在重复序列分布特征
运用RepeatMasker方法,获得了重组菌株Han0458的多种散在重复序列(表3),其在基因组中占比很小,仅为0.79%左右。其中长末端重复序列(LTR)数目最多,为690条,占总数的46.34%;其次是DNA转座子,为440个;长散在重复序列(LINE)共315条;而短散在重复序列(SINE)只有25条;滚环(RC) 13个。

Figure 3. Relationship between period side and quantity of minisatellite DNA sequence in the genome of yeast recombinant Han0458
图3. 重组菌株Han0458基因组小卫星DNA序列的重复单元长度与其数量关系

Figure 4. Relationship between period side and copy number of minisatellite DNA sequence in the genome of yeast recombinant Han0458
图4. 重组菌株Han0458基因组小卫星DNA序列的重复单元长度与其拷贝数关系
Table 3. Distribution of interspersed repeats in the genome of the yeast recombinant Han0458
表3. 重组菌株Han0458基因组中散在重复序列的分布
分析结果表明,各类型散在重复的总长度分布与数目分布保持一致,其长度大小关系为LTR > DNA > LINE > SINE > RC,各占散在重复序列总长的48.27%、29.58%、25.01%、1.34%和1.33%。值得注意的是,虽然RC的重复序列数目较少,但其平均长度约为SINE的两倍。
4. 讨论与结论
重复序列是基因组的重要组成部分,对生物的进化、遗传和基因的表达与调控有重要作用。重复序列的出现,说明基因组中的遗传物质在不断地进行自我复制,并进行水平交换和垂直交换,对丰富生物的遗传信息具有重要作用 [8] 。生物体中许多关键基因是单拷贝的,重复序列的存在能保护这些重要的基因结构不受破坏,同时也是新基因产生的物质基础,是驱动生物进化的重要因素之一 [9] 。
本研究利用生物信息学方法分析了来源于异常汉逊酵母菌的离子束重组菌株Han0458基因组重复序列在其基因组中的分布及特征,结果表明,重组酵母菌株Han0458基因组微卫星DNA序列在其基因组中的占比不到千分之一,重复单元的拷贝数大多低于15个,重复单位长度与其拷贝数间存在着负相关;优势重复类型为三碱基重复,AAC为所有微卫星DNA类型中数目最多的基序;两碱基重复序列数目最少,且仅有AT重复。在微卫星DNA和小卫星DNA中,AT含量均大于50%。这与Edwards等人的研究结果一致,AT类型重复的在植物、酵母和真菌类串联重复序列中的频率最高 [10] 。
重组酵母菌株Han0458基因组重复序列在基因组中含量较少,约为2.5%左右,对其转座元件的分析发现,RNA转座子的数目与长度均高于DNA转座子,与其他真菌的研究结果一致 [11] 。
三碱基类型重复和六碱基类型重复是重组酵母菌株Han0458基因组微卫星DNA序列的优势碱基类型,因此,在设计SSR引物时,可优先考虑从这两种重复类型入手。
低能离子注入对微生物的系统发育与进化具有重要的驱动作用 [12] ,本研究结果为进一步认识低能离子注入对酵母菌基因组结构的生物学效应提供了素材,同时为低能离子注入介导的酵母菌基因组突变与进化提供了分子证据。
致谢
本研究由国家自然科学基金(11575149和31760016)和陕西科技大学博士科研项目(126021759)资助。北京诺禾致源生物信息科技股份有限公司微生物事业部协助进行离子束重组酵母菌株Han0458全基因组de novo测序。
文章引用
张寒玉,唐 朝,王 婷,冯光文,钱卫东,毛培宏,蔡长龙. 离子束重组酵母菌Han0458基因组重复序列的分布特征
Distribution Characteristics of Repeat Sequences in the Genome of the Yeast Recombinant Han0458 Obtained by Low-Energy Ion Beam Implantation[J]. 微生物前沿, 2018, 07(03): 115-123. https://doi.org/10.12677/AMB.2018.73014
参考文献
- 1. Li, X., Kahveci, T. and Settles, A.M. (2007) A Novel Genome-Scale Repeat Finder Geared towards Transposons. Bioinformatics, 24, 468-476. https://doi.org/10.1093/bioinformatics/btm613
- 2. Férandon, C., Peuchant, O., Renaudin, H., et al. (2013) Diversity of Mycoplasma hominis Clinical Isolates from Bordeaux, France, as Assessed by Multiple-Locus Variable-Number Tandem Repeat Analysis. BMC Microbiology, 13, 120. https://doi.org/10.1186/1471-2180-13-120
- 3. 杨惠. 条斑紫菜功能基因组及重复序列特征研究[D]: [博士学位论文]. 青岛: 中国海洋大学, 2011: 10.
- 4. Sukumaran, S. and Grant, A. (2013) Effects of Genotoxicity and Its Consequences at the Population Level in Sexual and Asexual Artemia Assessed by Analysis of Inter-Simple Sequence Repeats (ISSR). Mutation Research/Genetic Toxicology and Environmental Mutagenesis, 757, 8-14. https://doi.org/10.1016/j.mrgentox.2013.03.015
- 5. 韩丽娟. 比较研究啮齿类动物基因组中的散在重复序列[J]. 科技通报, 2012, 28(12): 30-31.
- 6. Lü, J., Jin, X., Mao, P.H., et al. (2009) Transfer of Ephedra Genomic DNA to Yeasts by Ion Implantation. Applied Biochemistry and Biotechnology, 158, 571-581. https://doi.org/10.1007/s12010-009-8597-9
- 7. 毛培宏, 吕杰. 离子束重组酵母菌Ar_Han0458的RAPD与SSH的初步研究[J]. 基因组学与应用生物学, 2015, 34(3): 449-453.
- 8. Eichler, E.E. and Sankoff, D. (2003) Structural Dynamics of Eukaryotic Chromosome Evolution. Science, 301, 793-797. https://doi.org/10.1126/science.1086132
- 9. 艾对元. 基因组中重复序列的意义[J]. 生命的化学, 2008, 28(3): 343-345.
- 10. Edwards, K.J., Barker, J.H., Daly, A., et al. (1996) Microsatellite Libraries Enriched for Several Microsatellite Sequences in Plants. Biotechniques, 20, 758-760. https://doi.org/10.2144/96205bm04
- 11. Kim, J.M., Vanguri, S., Boeke, J.D., et al. (1998) Transposable Elements and Genome Organization: A Comprehensive survey of Retrotransposons Revealed by the Complete Saccharomyces cerevisiae Genome Sequence. Genome Research, 8, 464-478. https://doi.org/10.1101/gr.8.5.464
- 12. 蔡长龙, 吕杰, 毛培宏, 等. 低能氮离子注入驱动DOB的16S rRNA基因突变与进化[J]. 西安工业大学学报, 2017, 27(11): 288-293.
NOTES
*通讯作者。
