Modeling and Simulation
Vol.
13
No.
01
(
2024
), Article ID:
79760
,
9
pages
10.12677/MOS.2024.131057
基于3D CMFF深度网络模型的肺结节分类
刘一展
上海理工大学出版印刷与艺术设计学院,上海
收稿日期:2023年12月2日;录用日期:2023年12月25日;发布日期:2024年1月24日

摘要
当今基于深度学习的肺小结节分类成为了研究热点。然而,现有的网络模型大都精度过低,且只能处理2D肺结节切片,无法在肺部CT中学习到3D特征。为了应对这些挑战,提出了一个具有高精度的多尺度特征融合网络模型3DCMFF。3DCMFF的主干基于3DECABlock,可以在提高网络的特征表达能力。模型中的3D PSA模块,能够提取特征信息更丰富的3D精细化特征图。多尺度特征融合将阶段2~4层输出的特征图进行融合,在几乎不增加计算量的同时提升精度。在LUNA16数据集上进行综合消融实验和对比实验中,3DCMFF取得了93.58的精度。实验结果表明,我们的方法对分类精度有着可观的提升,并且和现有的先进模型相比较,3DCMFF具有精度高,鲁棒性优秀的特点,并能够给医生提供辅助诊断。
关键词
肺癌,肺结节,深度学习,多尺度特征融合

Classification of Pulmonary Nodules Based on 3D CMFF Deep Network Model
Yizhan Liu
College of Communication and Art Design, University of Shanghai for Science and Technology, Shanghai
Received: Dec. 2nd, 2023; accepted: Dec. 25th, 2023 published: Jan. 24th, 2024

ABSTRACT
Nowadays, deep learning based classification of pulmonary nodules has become a research hotspot. However, most existing network models have low accuracy, as they can only handle 2D lung nodule slices and cannot learn 3D features in lung CT. To address these challenges, a high-precision multi-scale feature fusion network model 3D CMFF has been proposed. The backbone of 3D CMFF is based on 3D ECA Block, which can improve the feature expression ability of the network. The 3D PSA module in the model can extract 3D refined feature maps with richer feature information. Multi scale feature fusion integrates feature maps output from stages 2~4, improving accuracy with almost no increase in computational complexity. In the comprehensive ablation and comparative experiments conducted on the LUNA16 dataset, 3D CMFF achieved an accuracy of 93.58. The experimental results show that our method has significantly improved classification accuracy, and compared with existing advanced models, 3D CMFF has the characteristics of high accuracy and excellent robustness, which can provide auxiliary diagnosis for doctors.
Keywords:Lung Cancer, Lung Nodules, Deep Learning, Multi-Scale Feature Fusion

Copyright © 2024 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
自2010年开始,肺癌的死亡率每年大约以4%的比例减少 [1] 。这主要得益于CAD系统带来的早期诊断。但肺癌仍作为死亡率最高和发病率最高的癌症,每年夺去百万条的生命。为了进一步降低肺癌的死亡率,肺部良恶性结节的分类在肺癌的早期诊断中显得尤为重要。随着LIDC-IDRI (The Lung Image Database Consortium) [2] 的出现,基于肺CT图像的自动肺结节良恶性分类有重要的研究价值,成为了当前的研究热点。
肺结节分类方法主要分为传统机器学习方法和深度学习方法。
基于肺CT图像的肺小结节良恶性分类研究早期基于传统机器学习方法。最初在肺图像数据集和算力不足的情况下,研究人员使用传统机器学习通过手工特征对肺结节CT图像进行分类。Po-Whei Huang等人 [3] 在使用SVM支持向量机的基础上添加了使用分形维度(FD)的分形纹理特征来区分良恶性结节。Ayman El-Baz等人 [4] 将肺结节的形状复杂度用形状指数来表示,并将它用K-近邻算法进行良恶性分类。QiMao等人 [5] 提出了智能免疫克隆优化算法来分类肺结节。设计了一种新的高斯–柯西混合变异算子,结合了高斯变异算子(GMO)和大尺度柯西变异算子(CMO)的优点。该方法解决了免疫算法初始种群的单一性问题,具有全局收敛和局部收敛的特点。Hao Chen等人 [6] 提出进的窗口自适应灰度级共现矩阵,能够对实性肺结节纹理特征提取对肺结节分类有较好的作用,但是他们使用的数据集样本量太小,可能存在偏差。虽然传统的机器学习也可以完成肺结节分类任务,但是普遍需要耗费大量时间去制作手工特征,并且很大程度依赖于研究人员的主观判断,因此不能实现自动化的高精度分类。
当前主要运用深度学习对肺CT图像的肺结节良恶性分类展开研究。深度学习可以直接从数据中获取高级特征,而省去了传统机器学习对每一个特征手工制作特征提取器的任务,并且在肺结节分类任务中,可以实现高鲁棒性的计算机自动诊断。Yohei Momoki等人 [7] 针对CAD系统需要大量标记数据的问题,提出从患者放射学报告中进行标记,生成伪标签来构建肺结节图像分类器。他们针对不同数据集,分别设计了基于层次注意网络(HAN)的文本分类器,和基于VGG的图像分类器。Meenakshi Bharti等人 [8] 为解决原始CT的计算量过大的问题,提出在预处理后将CT图片切割成小块,再使用3DResNet进行结节分类。Amrita Naik等人 [9] 肺结节图像因CT机的硬件上限,通常分辨率过低,导致难以被卷积网络训练的问题,提出使用Fractalnet作为网络主干,同时在增加数据集和数据扩充的情况下表现得更好。Astaraki, M等人 [10] 提出双路径的深度CNN,双路径不但可以获取结节的纹理特征,同时可以捕捉在结节附近的可以代表病变的背景与组织。在最后添加了随机森林模型用来生成分类结果。双路径CNN比普通单路径CNN有着更好的特征辨别能力,并且性能也可以超过单路径CNN,但是在将来,还可以将深度学习抽象特征和手工制作的特征相结合。Ahmed Shaffie等人 [11] 提出一种新的肺结节诊断系统,结合了纹理和轮廓特征来预测结节生长速度,能够作为标准的临床诊断。他们使用深度神经网络增强这两个特征用以分类肺结节。Sakshiwala等人 [12] 利用迁移学习提取多尺度特征和权重初始化,同时提出一种新的多尺度卷积神经网络结构用于结节二值分类。Muhammad Muzamml等人 [13] 为解决个人学习方法容易陷入局部最小值导致结果不佳的问题,提出对AlexNet、VGG-16和VGG-19三个分布网络模型和的特征和多个DCNN学习器进行基于MAX-VOTE的融合。Apostolopoulos, Ioannis D.等人 [14] 提出三维成像相比于二维成像在深度学习中更能捕获孤立性肺结节(SPN)的深层特征,同时提出运用半监督方法生成弱标记SPN,但是因此数据库中存在大量不确定的数据,缺少鲁棒性。Xiaohang Fu等人 [15] 为解决现有方法对每张肺结节三维切片一视同仁,没有考虑相关性和异质性,提出了MTL模型,把多个注意力模块和CNN结合在一起,在一整个结节卷中用注意模块过滤掉不关键的切片,并得出了得出了每张切片可解释的权重。Alejandra Moreno [16] 等人提出肺结节的恶性程度和组织学和几何形态有关,提出一种多尺度自注意(MSA)网络从多尺度分解中挖掘非局部结节之间关系,同时精确生成结节的可解释纹理图。2022年Rukhmini Roy等人 [17] 提出attribute-driven Generative Adversarial Network (ADGAN)来解释肺结节图像的合成和多类分类。设计了自我注意属性模块(SaAM)来根据给定的肺结节属性来生成肺结节图像。Hong Huang等人 [18] 提出深度特征优化框架(DFOF)解决了传统CNN模型单输入策略特征提取能力不足,多输入CNN会造成维数诅咒的问题,采用双流网络提取结节周围和内部的高维特征。并设计了流形优化方法来增强嵌入特征的类内紧致性和类间可分离性。Weilin He等人 [19] 为解决高性能模型结构复杂,可解释性差的问题,提出一种基于ISHAP (modified SHapley Additive explained)可解释模型。该方法将利用医学先验知识对肺CT提取的特征进行引导得出最关键的特征,在临床中帮助医生识别使结节恶性的因素。Yaowei Li等人 [20] 提出使用DeepLabv3+作为重要性估计网络,将不相关的特征进行销毁,并可以为每一张肺结节图像生成视觉解释诊断。同时将ResNet作为分类网络。Hanliang Jiang等人 [21] 提出了NAS-lung模型,提出在肺结节分类上使用NAS搜索,在保持高精度的同时,使用了相较于其他模型1/40的参数。在他们的模型中,还结合了CBAM注意力机制,并能生成表示高低注意力值的肺结节注意力图。但此注意力图并不能确切的表示肺癌诊断中应该被高度关注的部分,这是造成分类精度难以提升的根本原因,同时不能作为临床诊断依据。
综上所述现有的研究虽然都取得了显著的成果但仍存在一些问题,研究人员们大都使用二维的肺结节图像作为训练,但是肺结节是三维的,二维图像不能训练得出三维特征,造成良恶性分类精度有待进一步提高,为了应对挑战本文构建了一个多特征融合的网络模型。我们的主要贡献如下:1) 提出总体框架结构3DCMFF,使用3DECABlock作为框架的主干,通过将通道维度上的3D特征进行自适应加权,来增强有用的特征,抑制无用的特征,能够在提高网络的表示能力。2) 提出3D高效金字塔注意力分割模块(3D PSA)能够建立通道关系,获取3D精细化特征图。3) 提出使用多尺度特征融合,将不同阶段网络层的输出结果进行拼接,使后面的网络层能获取网络模型前几层的特征,在不增加计算量的同时获得精度的提升。了应对挑战本文构建了一个多特征融合的网络模型。我们的主要贡献如下:1) 提出总体框架结构3DCMFF,使用3DECABlock作为框架的主干,通过将通道维度上的3D特征进行自适应加权,来增强有用的特征,抑制无用的特征,能够在提高网络的表示能力。2) 提出3D高效金字塔注意力分割模块(3D PSA)能够建立通道关系,获取3D精细化特征图。3) 提出使用多尺度特征融合,将不同阶段网络层的输出结果进行拼接,使后面的网络层能获取网络模型前几层的特征,在不增加计算量的同时获得精度的提升。
2. 本文方法
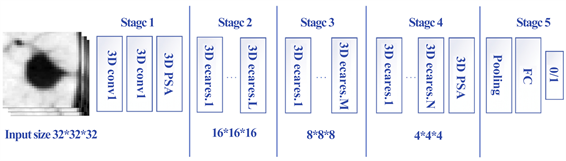
Figure 1. The architecture of 3DCMFF model
图1. 3DCMFF模型架构
如图1所示,三维多尺度特征融合网络模型(3DCMFF)主要分为五个阶段,将32*32*32的肺结节图像作为模型的输入。
阶段1有两个3D卷积块和一个3DPSA注意模块,同时可以生成可解释注意力图;阶段2~4有三个3DECA残差块,其中每个阶段的3DECA残差块数分别为L,M,N,其中阶段四的3DECA残差后有一个3DPSA注意力模块;在阶段1~4中,每个阶段的末尾都通过一个卷积层来进行降采样并升高通道数。阶段5使用全局平均池和全连接(FC)层生成最终预测。FC层输出一个二进制标签,表示输入结节是良性还是恶性。
2.1. 3DECA模块
ResNet [22] 提出跳跃连接,成功解决了梯度消失,网络退化的问题,同时可以保留原始特征,增加网络泛化能力。但是由于肺结节图像中结节形状,大小,纹理各不相同,ResNet难以从中提出最有效的特征。因此在我们的方法中引入3DECA模块 [23] 来代替普通的3D模块来作为网络框架的主干。
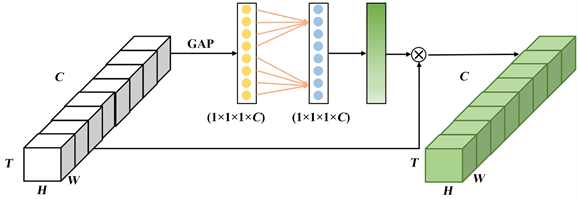
Figure 2. 3DECA block
图2. 3DECA模块
在3DCMFF中的阶段2~4中,我们引入3D高效通道注意力3DECA模块作为它们的主要构建,如图2所示。
3DECA模块能够通过增加少部分的参数,达到性能增益。该模块避免了降维,并且可以捕捉跨通道交互,如图所示适当的跨通道交互能够在降低模型参数的同时提升网络性能,3DECA模块在通过GAP模块后,再通过大小为k的快速一维卷积实现跨通道交互,见公式(1):
(1)
Odd代表取最小的奇数,在所有3D的ECA模块中, 和b分别被设置为2和1,通过非线性映射 ,高纬度的通道有较大的相互作用,相反低纬度的通道的相互作用较小。
3D ECA ResNet将不降维的局部跨通道交互策略与选择一维卷积核大小的自适应方法相结合。它实现了复杂的注意力模块,提高了分类性能。通过动态有效地学习通道特征之间的相关性,极大地提高了网络的特征表达能力,能够从肺结节图像中获取最关键的特征。
2.2. 3DPSA模块
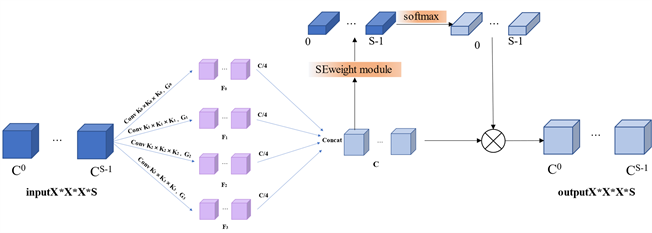
Figure 3. The architecture of 3DPSA block
图3. 3DPSA框架结构图
3DCMFF在2,4阶段都使用了3DPSA模块。3D金字塔挤压注意(PSA)模块如图3所示。该处理过程分为三个步骤:1) 32*32*32的图像通过挤压与连接SPC模块,获取通道上的3D多尺度特征图;2) 利用SEWeight模块提取不同比例特征图的3D注意向量,再利用Softmax函数对注意向量重新校准;3) 将3D多尺度特征图和重新校准的权值进行相乘,得到多尺度特征信息更丰富的3D精细化特征图作为输出。
3D卷积多尺度特征图获取
基于3D卷积多尺度特征图生成:为了能够在不增加过多参数的同时处理不同卷积核大小的输入张量,如图3所示,引入了3D群卷积,群的大小根据卷积核的大小自适应调整。生成多尺度特征图的函数如公式(2):
(2)
其中k是卷积核大小,G是群大小,如公式(3)、(4):
(3)
(4)
多尺度特征图拼接:将四个不同尺度,通道为C/4的特征图拼接得到融合多尺度特征图,如公式(5):
(5)
SEweight模块能够从不同尺度的输入特征图中获得不同注意力权重,再通过Softmax损失函数重新校准权重得到C × 1 × 1 × 1的atti,可以实现全局注意力的交互,在通过拼接,得到全部通道的注意力向量,如公式(6):
(6)
将不用尺度的注意力权重atti和相应尺度的特征图进行相乘,得到不同尺度通道关注权重的特征图Yi,如公式(7):
(7)
将不同尺度的特征图Yi进行拼接,得到最后的输出结果,能够完整地表示原始特征图的信息,如公式(8):
(8)
2.3. 多尺度特征融合
如图4所示,多尺度特征融合采用两个阶段(阶段1,阶段2)的特征图(16 × 16 × 16, 8 × 8 × 8),分别通过两个和一个平均池化层,得到两个不同的4 × 4 × 4的特征图,如公式(9)、(10):
(9)
(10)
因为肺结节的大小,形状都存在较大差异,很难找到一个统一固定的感受野。多尺度特征融合能够捕获在不同卷积层的特征,增强空间信息,增加模型的鲁棒性。将它们和同样为4×4×4,stage4的特征图进行通道上的拼接,再通过1 × 1 × 1的卷积层,获取三个不同感受野的信息,完成多尺度的特征融合,并把结果作为stage5的输入,如公式(11):
(11)
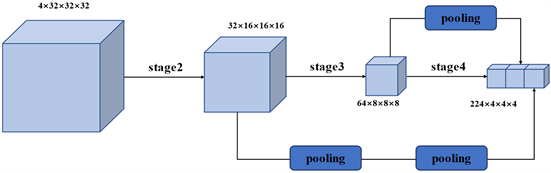
Figure 4. Multi-scale feature fusion
图4. 多尺度特征融合
3. 实验结果与分析
3.1. 数据集
在实验中,LIDC-IDRI数据集和LUNA16的设置被采用,LIDC-IDRI它包括1018个低剂量的肺部CT影像。LIDC-IDRI删除了切片厚度大于3 mm和肺结节小于3 mm的CT影像,得到LUNA16数据集,包含888个低剂量肺部CT影像数据,含有1185个肺结节。在LUNA16数据集中删除切片间距不一致和切片缺失的CT数据,还剩下1005个结节。结节的注释信息由四位思维经验丰富的放射科专家评定,包括肺结节的三维坐标恶性程度评分(1~5分),其中评分越高代表恶性程度越高,评分在1~2被标记为良性结节,评分在4~5的被标记为恶性结节。在我们的工作中,我们去除了恶性程度评分在3左右的结节,还剩下933个结节作为我们的训练数据集。在模型训练中我们使用LUNA16的十倍交叉验证进行训练。
3.2. 数据预处理
将Luna16的原始数据被裁切为[−1200, 600],再将其通过线性变化变为[0, 1]。根据Luna16的地表真值标签信息从CT图像中裁切出32 × 32 × 32的结节立方块,填充成36 × 36 × 36,再随机裁剪成32 ×32 × 32。我们将结节水平翻转,垂直翻转和Z轴翻转进行数据增强。
3.3. 评价指标
为了评价肺结节的分类表现,四个广泛使用的指标,准确率,敏感性,特异性,F1分数如公式(12)、(13)、(14)、(15):
(12)
(13)
(14)
(15)
以上公式中,TP、FN、FP、TN依次为真阳性、假阴性、假阳性、真阴性。这些标准的值越大表明性能越好。F1评分评估敏感性和特异性之间的权衡。一般来说,F1分数越高,性能越好。
3.4. 与现有工作的比较
为了证明3DCMFF的优越性,与现有的先进方法进行比较,包括deeplung,NASlung,ADGAN。在这里,我们在表中报告了3DCMFF的性能。
Table 1. Comparison of classification indicators of different models
表1. 不同模型分类指标的对比
从表1中,3DCMFF达到了最高的精确度和F1评分,分别为93.53%和90.05%,精确度相比ADGAN高了0.73%。我们的方法同时能够在敏感性和特异性之间取得了很好的平衡,达到了90.20%和95.73%。换句话说,3DCMFF可以正确分类大多数结节,也很大程度上解决了假阳性的问题,这将大大减轻医生的负担。所有这些结果表明,3DCMFF通过使用ECAblock,3DPSA,和特征融合方法能够优于现有的先进模型。
3.5. 消融实验
Table 2. Results of ablation experiment
表2. 消融实验结果
我们使用消融实验来分析我们所提出的技术的影响,即3DECAblock,3DPSA和特征融合策略。我们首先从总体架构中依次去除特征融合策略,3DPSA block和ECAblock,来构建3个模型变体。表2报告了模型变体的准确度,敏感度,特异度和F1分数。结果表明3DPSA,ECAblock和特征融合在总体上,都提高了原始模型的准确性和鲁棒性。
4. 结论
在本文中,我们通过使用3DECAblock,3DPSA和特征融合策略,能够从肺结节图像中提取3D特征,并拥有良好准确的深度学习肺结节分类模型。本文中所有已提出使用的技术都被证明有效,并且模型的效果能够优于现有的优秀模型。
在未来的工作中,可以将网络模型可解释化,能够在保持高精度的同时,在临床中可以帮助医生识别到使结节恶性的因素。
文章引用
刘一展. 基于3D CMFF深度网络模型的肺结节分类
Classification of Pulmonary Nodules Based on 3D CMFF Deep Network Model[J]. 建模与仿真, 2024, 13(01): 588-596. https://doi.org/10.12677/MOS.2024.131057
参考文献
- 1. Siegel, R.L., Miller, K.D., Fuchs, H.E., et al. (2021) Cancer Statistics, 2021 (vol 71, pg 7, 2021). CA: A Cancer Journal for Clinicians, 71, 359-359.
- 2. Armato, S.G., Mclennan, G., Bidaut, L., et al. (2011) The Lung Image Database Con-sortium (LIDC) and Image Database Resource Initiative (IDRI): A Completed Reference Database of Lung Nodules on CT Scans. Medical Physics, 38, 915-931. https://doi.org/10.1118/1.3528204
- 3. Huang, P.W., Lin, P.L., Lee, C.H., et al. (2013) A Classification System of Lung Nodules in CT Images Based on Fractional Brownian Motion Model. IEEE International Conference on System Science and Engineering (ICSSE), Budapest, 4-6 July 2013, 37-40. https://doi.org/10.1109/ICSSE.2013.6614710
- 4. El-Baz, A., Nitzken, M., Khalifa, F., et al. (2011) 3D Shape Analysis for Early Diagnosis of Malignant Lung Nodules. 22nd International Conference on Information Processing in Medical Imaging (IPMI), Kloster Irsee, 3-8 July 2011, 772-783. https://doi.org/10.1007/978-3-642-22092-0_63
- 5. Mao, Q., Zhao, S.G., Ren, L.J., et al. (2021) Intelligent Im-mune Clonal Optimization Algorithm for Pulmonary Nodule Classification. Mathematical Biosciences and Engineering, 18, 4146-4161. https://doi.org/10.3934/mbe.2021208
- 6. Chen, H., Li, W. and Zhu, Y.Y. (2021) Improved Win-dow Adaptive Gray Level Co-Occurrence Matrix for Extraction and Analysis of Texture Characteristics of Pulmonary Nodules. Computer Methods and Programs in Biomedicine, 208, Article ID: 106263. https://doi.org/10.1016/j.cmpb.2021.106263
- 7. Momoki, Y., Ichinose, A., Shigeto, Y., et al. (2022) Characteri-zation of Pulmonary Nodules in Computed Tomography Images Based on Pseudo-Labeling Using Radiology Reports. IEEE Transactions on Circuits and Systems for Video Technology, 32, 2582-2591. https://doi.org/10.1109/TCSVT.2021.3073021
- 8. Bharti, M., Choudhary, J. and Singh, D.P. (2022) Detection and Classification of Pulmonary Lung Nodules in CT Images Using 3D Convolutional Neural Networks. 8th Interna-tional Conference on Advanced Computing and Communication Systems, ICACCS 2022, Coimbatore, 25-26 March 2022, 1319-1324. https://doi.org/10.1109/ICACCS54159.2022.9785134
- 9. Naik, A., Edla, D.R. and Kuppili, V. (2021) Lung Nodule Classification on Computed Tomography Images Using Fractalnet. Wireless Personal Communications, 119, 1209-1229. https://doi.org/10.1007/s11277-021-08258-w
- 10. Astaraki, M., Zakko, Y., Dasu, I.T., et al. (2021) Benign-Malignant Pulmonary Nodule Classification in Low-Dose CT with Convolutional Features. Physica Medi-ca-European Journal of Medical Physics, 83, 146-153. https://doi.org/10.1016/j.ejmp.2021.03.013
- 11. Shaffie, A., Soliman, A., Khalifeh, H.A., et al. (2020) A Compre-hensive Framework for Accurate Classification of Pulmonary Nodules. 2020 IEEE International Conference on Image Processing, ICIP 2020, Abu Dhabi, 25-28 September 2020, 408-412. https://doi.org/10.1109/ICIP40778.2020.9190909
- 12. Sakshiwala and Singh, M.P. (2022) A New Framework for Multi-Scale CNN-Based Malignancy Classification of Pulmonary Lung Nodules. Journal of Ambient Intelligence and Humanized Computing, 14, 4675-4683. https://doi.org/10.1007/s12652-022-04368-w
- 13. Muzammil, M., Ali, I., Haq, I.U., et al. (2021) Pulmonary Nod-ule Classification Using Feature and Ensemble Learning-Based Fusion Techniques. IEEE Access, 9, 113415-113427. https://doi.org/10.1109/ACCESS.2021.3102707
- 14. Apostolopoulos, I.D., Apostolopoulos, D.J. and Panayiotakis, G.S. (2022) Solitary Pulmonary Nodule Malignancy Classification Utilising 3D Features and Semi-Supervised Deep Learning. 13th International Conference on Information, Intelligence, Systems and Applications, IISA 2022, Corfu, 18-20 July 2022, 1-6. https://doi.org/10.1109/IISA56318.2022.9904334
- 15. Fu, X.H., Bi, L., Kumar, A., et al. (2022) An Atten-tion-Enhanced Cross-Task Network to Analyze Lung Nodule Attributes in CT Images. Pattern Recognition, 126, Article ID: 108576. https://doi.org/10.1016/j.patcog.2022.108576
- 16. Moreno, A., Rueda, A. and Martinez, F. (2022) A Multi-Scale Self-Attention Network to Discriminate Pulmonary Nodules. 19th IEEE International Symposium on Bio-medical Imaging, ISBI 2022, Kolkata, 28-31 March 2022, 1-4. https://doi.org/10.1109/ISBI52829.2022.9761574
- 17. Roy, R., Mazumdar, S. and Chowdhury, A.S. (2022) ADGAN: Attribute-Driven Generative Adversarial Network for Synthesis and Multiclass Classification of Pulmonary Nodules. IEEE Transactions on Neural Networks and Learning Systems.
- 18. Huang, H., Li, Y., Wu, R.Y., et al. (2022) Benign-Malignant Classification of Pulmonary Nodule with Deep Feature Optimization Framework. Biomedical Signal Processing and Control, 76, Article ID: 103701. https://doi.org/10.1016/j.bspc.2022.103701
- 19. He, W.L., Li, B., Liao, R.Q., et al. (2022) An ISHAP-Based In-terpretation-Model-Guided Classification Method for Malignant Pulmonary Nodule. Knowledge-Based Systems, 237, Article ID: 107778. https://doi.org/10.1016/j.knosys.2021.107778
- 20. Bonavita, I., Rafael-Palou, X., Ceresa, M., et al. (2020) Integra-tion of Convolutional Neural Networks for Pulmonary Nodule Malignancy Assessment in a Lung Cancer Classification Pipeline. Computer Methods and Programs in Biomedicine, 185, Article ID: 105172. https://doi.org/10.1016/j.cmpb.2019.105172
- 21. Jiang, H.L., Shen, F.H., Gao, F., et al. (2021) Learning Efficient, Explainable and Discriminative Representations for Pulmonary Nodules Classification. Pattern Recognition, 113, Article ID: 107825. https://doi.org/10.1016/j.patcog.2021.107825
- 22. He, K.M., Zhang, X.Y., Ren, S.Q., et al. (2016) Deep Residual Learning for Image Recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), Las Ve-gas, 27-30 June 2016, 770-778. https://doi.org/10.1109/CVPR.2016.90
- 23. Wang, Q.L., et al. (2019) ECA-Net: Efficient Channel Attention for Deep Convolutional Neural Networks.
- 24. Zhu, W.T., Liu, C.C., Fan, W., et al. (2018) DeepLung: Deep 3D Dual Path Nets for Automated Pulmonary Nodule Detection and Classification. 18th IEEE Winter Conference on Applications of Computer Vision (WACV), Lake Tahoe, 12-15 March 2018, 673-681. https://doi.org/10.1109/WACV.2018.00079
