Hans Journal of Medicinal Chemistry
Vol.
11
No.
04
(
2023
), Article ID:
75785
,
11
pages
10.12677/HJMCe.2023.114031
自噬–溶酶体系统靶向降解蛋白质技术 研究进展
沈钦旭*,马陇君#
兰州交通大学化学化工学院,甘肃 兰州
收稿日期:2023年10月22日;录用日期:2023年10月31日;发布日期:2023年11月22日


摘要
在靶向蛋白降解(TPD)药物开发领域,利用泛素–蛋白酶体系统的蛋白质水解靶向嵌合体(PROTACs)得到了广泛的研究。然而,泛素–蛋白酶体系统仅限于降解可溶性蛋白和膜蛋白的降解,不包括聚集蛋白/细胞外蛋白和功能失调的细胞器。溶酶体作为一种替代的蛋白质降解途径,既可通过自噬–溶酶体途径,降解细胞内靶点:如可溶性蛋白和聚集蛋白;又能通过核内体–溶酶体途径,降解细胞外靶点:如膜蛋白和分泌的细胞外蛋白。本文中,我们重点介绍新兴的溶酶体介导的靶向降解技术,如AUTAC、ATTEC、AUTOTAC、LYTAC和MoDE-A。
关键词
溶酶体,自噬,靶向降解,蛋白质

Research Progress in Targeted Protein Degradation Technology of Autophagy-Lysosome System
Qinxu Shen*, Longjun Ma#
School of Chemistry and Chemical Engineering, Lanzhou Jiaotong University, Lanzhou Gansu
Received: Oct. 22nd, 2023; accepted: Oct. 31st, 2023; published: Nov. 22nd, 2023

ABSTRACT
In the field of Targeted Protein Degradation (TPD) drug development, Protein Targeting Chimeras (PROTACs) utilizing the ubiquitin-proteasome system have been extensively studied. However, the ubiquitin-proteasome system is limited to the degradation of soluble and membrane proteins and does not include aggregated proteins/extracellular proteins and dysfunctional organelles. Lysosomes, as an alternative protein degradation pathway, can degrade intracellular targets through the autophagy-lysosome pathway, such as soluble proteins and aggregated proteins, and can also degrade extracellular targets through the endolysosome-lysosome pathway, such as membrane proteins and secreted extracellular proteins. In this article, we focus on emerging lysosome-mediated targeted degradation techniques, such as AUTAC, ATTEC, AUTOTAC, LYTAC, and MoDE-A.
Keywords:Lysosome, Autophagy, Targeted Degradation, Proteins

Copyright © 2023 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
泛素–蛋白酶体系统(UPS)和溶酶体系统是蛋白质降解的两个主要且互补的途径。UPS途径降解胞内、可溶性和寿命短的蛋白质,近十年来,基于UPS途径的PROTACS药物研发取得了优异的成绩 [1] [2] 。然而,与UPS相关的小分子基础的PROTAC仍然存在一些限制,例如,只能降解细胞内、可溶性和寿命短的蛋白质,从而限制了它们在由细胞外蛋白质和蛋白质聚集引起的许多疾病中的应用,如亨廷顿病和阿尔茨海默病 [3] [4] 。相反,溶酶体系统可以降解许多完全折叠、寿命长的聚集蛋白质、细胞外蛋白质、核酸、脂质、受损的细胞器以及细菌和病毒等传染性生物体。因此,溶酶体降解生物分子可以极大地丰富靶向蛋白质降解(TPD)的工具箱,并扩展其在人类疾病中的应用 [5] 。如图1所示。
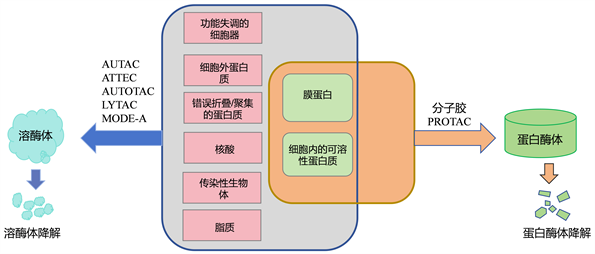
Figure 1. Two pathways of targeted protein degradation
图1. 靶向蛋白降解的两种途径
基于溶酶体的降解途径可分为以下两种:一是通过自噬被称为自噬–溶酶体途径的方式来降解细胞质蛋白质和受损细胞器;二是通过内吞作用被称为内吞–溶酶体途径的方式来降解细胞外蛋白质 [6] ,本文将对自噬–溶酶体途径原理及一些以溶酶体为基础的靶向蛋白降解的技术进展进行综述。
2. 自噬–溶酶体途径
自噬是一种保守的分解过程,用于细胞质组分的周转和再循环,已在酵母中鉴定出40多个自噬相关基因及其编码蛋白质,其中大多数也存在于哺乳动物中,这表明自噬是一种进化保守的过程 [7] 。自噬在维持细胞的稳态和能量平衡方面发挥着至关重要的作用。根据被降解组分的具体降解机制,自噬又分为三种类型:微自噬 [8] 、分子伴侣介导的自噬(CMA) [9] 和巨自噬 [10] 。微自噬是将细胞质组分直接包裹在溶酶体内进行降解;而分子伴侣介导的自噬则是降解含有KFERQ样序列的特定蛋白质,该序列可以被分子伴侣HSPA8/HSC70识别,将蛋白质引导到溶酶体表面蛋白LAMP2A中进行溶酶体包裹。巨自噬(以下简称自噬)涉及自噬体——一个双膜囊泡的形成,被定向降解的物质被困在自噬体内,随后通过自噬体和溶酶体之间的膜融合传递到溶酶体中被降解,自噬过程的机制受到自噬相关蛋白质的高度调控 [11] 。
2.1. 自噬的机制和调控
自噬是一种复杂的细胞过程,涉及多个与自噬相关的(Atg)蛋白质,可以分为三个主要阶段:
首先,自噬的诱导阶段是由细胞应激情况触发的,例如养分匮乏、氧化应激和mTORC1抑制 [12] 。这个阶段涉及ULK1/2复合物的激活,以及其他蛋白质如Atg13、Atg101和FIP200的参与。其次,自噬体的核聚合成熟阶段。该阶段需要类III磷脂酰肌醇3-激酶(PI3K)复合物的协助,包括Beclin1、VPS34、VPS15和Atg14L。这一阶段还包括将磷脂酰肌醇(PI)磷酸化为磷脂酰肌醇3-磷酸(PI3P),以确保正确定位Atg蛋白质,如Atg18和Atg2,以促使自噬小体膜的形成 [13] 。最后,与溶酶体的融合和降解是自噬的终阶段。这个阶段涉及Rab-SNARE蛋白质的参与,实现自噬小体与溶酶体的融合,形成自溶小体。自溶小体释放其内部成分,包括内膜和组分,以供溶酶体水解酶进行降解。此外,自噬还依赖于类似泛素的共轭系统,包括LC3-II和Atg12-Atg5-Atg16L。这些系统在伸长/成熟阶段发挥作用,其中LC3-I转化为LC3-II,并与自噬小体膜共价结合,而Atg12则与Atg5和Atg16L结合,促进磷脂醇乙醇胺(PE)的共轭。Atg4B在LC3-II的回收中发挥作用,将其转化回LC3-I的形式,这被称为去脂化。LC3-II还充当自噬受体(如p62和NBR1)的结合平台,将自噬小体运送到溶酶体以进行降解 [14] [15] 。如图2所示。
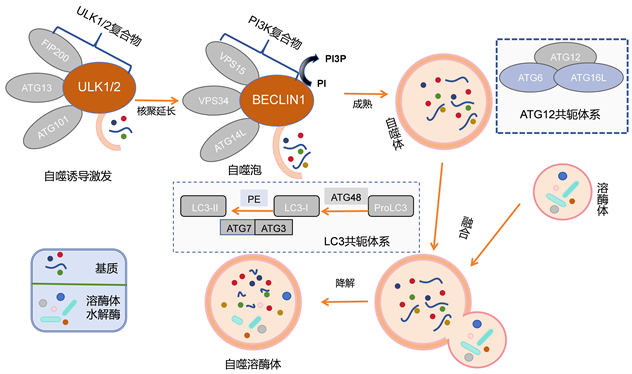
Figure 2. Schematic diagram of autophagy mechanism
图2. 自噬机制示意图
2.2. 自噬体的选择性
尽管最初认为自噬是非选择性的,但最近的研究揭示了某些类型的巨自噬是有选择性的 [16] 。自噬的选择性普遍由载体受体蛋白负责控制,它们可以识别载体并与分布在隔离膜和吞噬体上的LC3蛋白质结合 [17] 。而细胞器主要是通过承担载体角色不同的细胞器的类型来确定自身的自噬选择性。例如,蛋白质聚集体(聚集体自噬)、病原体(异体自噬)、铁蛋白(铁蛋白自噬)、线粒体(线粒体自噬)、核糖体(核糖自噬)和脂质体(脂肪自噬) [18] - [23] 。
哺乳动物中选择性自噬的一般机制途径由自噬受体(例如p62、NBR1、NDP52、TAX1BP1、OPTN和CCT2)调控 [24] [25] 。在选择性自噬过程中,自噬受体经历翻译后的结构修饰,如泛素化、磷酸化、乙酰化和寡聚化。在泛素依赖的载体选择中,错误折叠的蛋白质标记为多泛素链,这些链通过它们的泛素结合域(UBD)被受体识别并结合,最终送入自噬体。相反,泛素不依赖的途径则是涉及特定载体的识别,如蛋白质、脂类或基于糖的信号由专门的自噬受体识别。例如,Mancias等人发现铁蛋白的选择性自噬(即,铁蛋白吞噬)是由泛素非依赖受体NCOA4.5介导的 [26] 。
p62/SQSTM1是一种存在于后生动物中的特定自噬受体蛋白。p62在泛素化蛋白聚集体的选择性自噬降解中发挥关键作用,并包含了用于结合各种底物的多个保守结构域。p62的结构域通过与UBD结合促进泛素化的载体降解,通过PB1结构域自我寡聚化,然后通过与自噬体膜蛋白LC3的LIR (LC3相互作用区域)相互作用,将蛋白聚集体送入自噬体 [27] [28] 。除此之外,p62的ZZ结构域还通过对N-识别蛋白位点的选择性调节和N-末端降解物的结合(包括N-末端精氨酸(NT-R)在内)从而在诱导自噬的过程中发挥作用。其中,p62的ZZ结构域可与精氨酸化底物(NT-R)有效结合,这种结合会促进LC3与二硫键连接的p62的自聚集体进行相互作用,最后促使p62及其载体进入自噬过程 [28] 。
3. 基于溶酶体的靶向蛋白降解的进展
一些基于自噬机制的新兴技术,如含有cGMP的降解标签的自噬靶向嵌合物(Autophagy-Targeting Chimera, AUTAC)、自噬体系连接化合物(Autophagosome-Tethering Compounds, ATTECs)、溶酶体靶向嵌合物(Lysosometargeting Chimera, LYTAC)和含有p62配体的自噬靶向嵌合物(Autophagy-Targeting Chimera, AUTOTAC)正在开发中。
3.1. AUTACs/AUTOTACs
自噬靶向嵌合体(Autophagy-Targeting Chimera)是A组链球菌(GAS)细菌通过8-nitro-cgmp的S-鸟苷化,促进了K63-链多泛素化,最终信号传导到自噬体,随后进行有选择性的运输和降解 [29] 。日本东北大学Arimoto小组充分利用了GAS细菌的选择性降解过程,开发了一种具有靶向蛋白质降解能力的嵌合分子,命名为自噬靶向嵌合体(AUTACs),也称为第一代AUTACs [30] [31] [32] [33] 。AUTACs分子由三部分组成:一个基于cGMP的降解标签、一个linker、一个靶向蛋白(POI)或细胞器的小分子配体。由于Fumagillin能与甲硫氨酸氨肽酶2 (MetAP2)进行有效共价结合,所以Arimoto等人开发的AUTAC1,选择了甲硫氨肽酶2 (MetAP2)蛋白结合物fumagillin作为其弹头,并且在细胞实验中,成功地以1 μM浓度降解了MetAP2。类似地,AUTAC2被设计用于降解FK506结合蛋白(FKBP12),FKBP的非共价合成配体(SLF)已被用于通过AUTAC系统以10 μM浓度降解。AUTAC3也被设计用于靶向核蛋白BRD4,但其降解效果不如细胞质蛋白MetAP2和FKBP12。最重要的是,AUTAC4被设计用于有选择性地降解线粒体。AUTAC4包含一苯基吲哚基团,可结合到位于外线粒体膜(OMM)上的线粒体转运蛋白(TSPO),并可以有选择性地移除功能失调的线粒体。
表1中四种AUTAC分子通过引入不同的靶向蛋白,最终都能对不同目标蛋白产生有效的降解,但是相比于AUTAC2和AUTAC3,AUTAC1对靶向蛋白MetAP2的降解效果是最好的,只需要1 μM。对比前三种AUTAC分子,AUTAC4则是利用了其结构上的苯基吲哚部分可以于外线粒体膜(OMM)上内源性的线粒体转运蛋白(TSPO)进行非共价结合的特性,使其对线粒体的降解更有选择性。
总之,AUTAC分子的一端可以选择性地结合到靶蛋白(POIs),另一端含有S-鸟嘌呤基团,有助于诱导K63多泛素化。自噬受体如p62识别K63多泛素化的蛋白货物,并将其运送到自噬体以进行后续降解。如表1所示。
Table 1. Summary of AUTAC molecules
表1. AUTAC分子汇总
2022年,韩国首尔大学Yong Tae Kwon团队开发了第二代自噬靶向嵌合体称为AUTOTACs (AUTOphagy-Targeting Chimera) [34] ,不同于AUTAC分子的泛素依赖降解机制,AUTOTACs可以直接将受体p62绑在靶向降解组分上并诱导选择性自噬降解。AUTOTACs也是一种双功能分子,包含一个自噬靶向配体(ATL)以及一个靶向蛋白配体(TBL)。自噬靶向配体(ATL)与p62结合遵循N-末端规则途径,ATL结合到自噬载体受体p62-ZZ结构域上,使非激活状态的p62激活为自噬兼容形式,p62暴露其PB1结构域,用于与TBL结合的蛋白进行自体聚合,并暴露其LIR结构域,用于与自噬膜上的LC3相互作用,诱导了自噬体的形成和靶蛋白降解。
Yong Tae Kwon团队的工作中,选择了雌激素受体β (ERβ)、雄激素受体(AR)和蛋氨酸氨肽酶-2 (MetAP2)三种作为靶向蛋白,使用能和它们靶向结合的配体PHTPP-1304、Vinclozolin M2和Fumagillin作为靶向配体(TBLs)设计了三种AUTOTACs分子,经过测试,这三种分子都可以实现纳摩尔级别降解其相应的靶向蛋白质。虽然ATL和TBL也可以对单独对这些靶蛋白进行降解,但是其效果对比三种AUTOTACs分子的降解效率是远远不如的。AUTOTAC还可以用于自噬清除广泛的细胞内靶向蛋白质。它不仅可以靶向单体蛋白质,还可以靶向具有持续功效的聚合寡聚体蛋白质。AUTOTAC的另一个特点是,与PROTAC不同,它的效力不严重依赖于连接子长度,因此能够进行直接的AUTOTAC设计。如表2所示。
Table 2. Summary of AUTOTACs molecules
表2. AUTOTACs分子汇总
3.2. ATTECs
自噬体系连接化合物(Autophagosome-Tethering Compounds, ATTECs)。2019年,复旦大学鲁伯埙小组开发了一种名为自噬体拴系化合物(ATTECs)的靶向蛋白质降解的新方法 [35] 。ATTECs是一种双功能的嵌合化合物,将靶标蛋白(POI)连接到特定的蛋白质降解机制(PDM)组分,例如自噬体膜蛋白LC3。这些化合物对特定蛋白质具有等位基因选择性。鲁伯埙小组还设计了一个小分子微阵列筛选,用于筛选与LC3和导致亨廷顿疾病的突变亨廷顿蛋白(mHTT)相互作用的化合物,突变亨廷顿蛋白(mHTT)具有扩展的多谷氨酸(PolyQ)区域 [36] 。通过微阵列筛选,该团队鉴定出四种等位基因选择性化合物(10O5、8F20、AN1和AN2),它们只能与mHTT相互作用,而不能与野生型(WT)HTT相互作用。这些化合物还导致了一些其他含多谷氨酸(PolyQ)结构扩展域蛋白质的降解,如ATXN3。一旦ATTEC分子通过LC3结构域将具有扩展的多谷氨酸(PolyQ)区域的蛋白质连接到自噬体中,它就会被定向进行后续降解 [35] 。如表3所示。
Table 3. ATTECs for selective degradation of mHTT
表3. 选择性降解mHTT的ATTECs
鲁伯埙小组也将他们的ATTEC策略应用于降解非蛋白质生物分子,如脂滴(LDs),并开发了一类称为LD-ATTECs的新分子。鲁伯埙团队将选择性的LD检测探针(例如苏丹染料)与LC3结合分子相连接。这些分子在细胞中形成三元复合物,促使LD和自噬体的接近,从而导致自噬降解。在这个机制中,只有由油酸诱导的成纤维细胞中的LD和分化的脂肪细胞中的内源性LD被降解,其他含脂质的膜没有受到影响。该研究为对抗因脂滴积累引发的疾病,如肥胖、心血管疾病或脂肪肝病,提供了一种新方法。尽管ATTEC技术在许多细胞类型中可能有效,但LC3中的结合位点尚不为人所知 [37] 。
2021年,四川大学欧阳亮和王冠团队也发展了一种以LC3为靶点的自噬嵌合分子,可以通过自噬途径成功降解BRD4蛋白质。他们将可逆的BET溴域抑制剂JQ1和LC3结合物GW5074连接起来,成功地降解了BRD4蛋白 [38] 。2022年,第二军医大盛春泉团队应用ATTEC原理,用柔性链将NAMPT抑制剂和LC3结合物Ispinesib连接起来,开发了第一代尼克酸磷酸核糖转移酶(NAMPT)的自噬降解剂NAMPT-ATTECs,并证实了NAMPT的降解是通过自噬–溶酶体途径发生的 [39] 。如表4所示。
Table 4. ATTECs for selective degradation of NAMPT
表4. 选择性降解NAMPT的ATTECs
3.3. LYTACs和MoDE-As
溶酶体靶向嵌合体技术,称为LYTACs,主要由两部分组成,一部分是与靶点蛋白结合的小分子或者抗体,另一部分是能与溶酶体靶向受体如无钙依赖的甘露糖6-磷酸受体(CI-M6PR)或缺乏醛基糖蛋白受体(ASGPR)的结合的聚糖(多肽)配体,也称为糖标签,LYTACs可降解胞外分泌蛋白质和与细胞膜相关的蛋白质 [40] 。该项技术2020年由斯坦福大学Bertozzi团队开发,首先Bertozzi小组在生物素化的糖多肽配体上连接了荧光标记蛋白NeutrAvidin-647 (NA-647,与生物素结合),确认LYTACs体系能够有效地将靶标组分运输到溶酶体中 [40] 。其次,他们通过CRISPR干扰(CRISPRi)遗传筛选,发现LYTACs降解途径依赖于CI-M6PR,内涵体酸化、囊泡运输、内涵体-溶酶体融合等。Bertozzi小组也证明了带有poly(M6Pn)的糖多肽偶联到抗体能够快速地将特异性结合的细胞外物质导向溶酶体,并将该技术进一步应用到载脂蛋白E4、表皮生长因子受体(EGFR)、CD71和程序化死亡配体1 (PD-L1)的降解,证明了LYTACs技术的特异性和普适性 [41] 。如表5所示。
Table 5. LYTACs for selective degradation of NA-647
表5. 选择性降解NA-647的LYTACs
2021年,威斯康星大学麦迪逊分校,Tang课题组还开发了一种三价N-乙酰半乳糖(tri-GalNAc)为ASGPR配体(tri-GalNAc),连接到生物素和抗体上,生成了一类新的LYTACs,可以内化和降解整合素和EGFR [42] 。如表6所示。
与单独抑制相比,GalNAc-LYTACs降解EGFR并诱导下游激酶信号通路的破坏作用更明显。总而言之,GalNAc-LYTACs代表了一种受体介导的细胞特异性降解策略,可应用于与肝脏疾病(如HCC)相关的不依赖于细胞外显率或载体降解的膜蛋白。
Table 6. LYTACs for selective degradation of NA-650
表6. 选择性降解NA-650的LYTACs
借鉴类似的概念,Spiegel小组开发了通过无酸性糖蛋白受体(ASGPR)去降解细胞外蛋白的分子去除剂(Molecular Degraders of Extracellular Proteins through the Asialoglycoprotein Receptor (ASGPR), MoDE-As) [43] ,这是以ASGPR为靶点的LYTACs的小分子版本。MoDE-As分子可以招募并诱导α-DNP抗体和细胞因子MIF蛋白的降解。ASGPR的降解依赖于ASGPR通过克拉亭介导的内吞作用–溶酶体系统。经过LYTACs处理的细胞中,溶酶体维持稳态稳定性的能力没有受到影响,这表明这种模式在细胞水平上可能是安全的。LYTACs的成功是由于针对目标蛋白的内源性蛋白质转运和降解动力学。然而,LYTACs不能用于靶向降解细胞内蛋白。作为小分子,MoDE-As可能能够实现比基于抗体的LYTACs更深的组织渗透。
与LYTACs相比,MoDE-A化合物具有几个优势:它们的尺寸相对较小,单分散且基于非蛋白质。此外,MoDE-A化合物还能通过将蛋白质引导至肝细胞,通过肝细胞对内吞蛋白进行降解,这是LYTACs分子所不具有的。此外,有证据表明,ASGPR在免疫学上具有耐受性,这将最大限度地减少对靶蛋白诱导自身免疫反应的可能性。如表7所示。
Table 7. MoDE-As for selective degradation of MIF
表7. 选择性降解MIF的MoDE-As
4. 总结与展望
定向蛋白质降解(TPD)是一种新兴的治疗方法,对于细胞内蛋白质水平的调控至关重要。异常或致病的蛋白质通常需要被降解以维持细胞健康。尽管PROTACs已经被广泛研究,但PROTACs在降解不溶性或复杂的蛋白质聚集体和功能障碍的细胞器和线粒体方面存在局限。
为了解决这些问题,已经探索了一些新颖的TPD方法,包括AUTAC、AUTOTAC、ATTEC、和LYTAC,它们基于溶酶体介导的降解途径。这些方法能够选择性地识别和运送目标蛋白质和细胞器至溶酶体进行降解,有望克服泛素–蛋白酶体介导降解的局限性。然而,这些基于溶酶体的TPD技术仍然存在一些未解之谜和挑战。例如,AUTAC如何通过S-鸟苷化诱导K63多泛素化的机制尚不完全清楚,AUTOTAC激活了自噬货物受体如p62,但需要进一步的研究来更好地理解它们的机制,ATTEC分子与LC3的结合机制仍然未知。此外,与PROTAC相比,基于溶酶体的降解剂往往具有较低的效力,通常在微摩尔或亚微摩尔范围内。这种差异可能归因于泛素–蛋白酶体系统(UPS)和自噬溶酶体系统(ALS)之间的降解动力学差异。
总之,溶酶体介导的靶向蛋白质降解代表了一种有潜力的PROTAC替代技术,基于溶酶体的降解剂应主要集中在PROTAC无法高效降解的目标上,例如蛋白质的错误折叠、聚集体和功能失调的细胞器。
文章引用
沈钦旭,马陇君. 自噬–溶酶体系统靶向降解蛋白质技术研究进展
Research Progress in Targeted Protein Degradation Technology of Autophagy-Lysosome System[J]. 药物化学, 2023, 11(04): 262-272. https://doi.org/10.12677/HJMCe.2023.114031
参考文献
- 1. Ciechanover, A. (2005) Intracellular Protein Degradation: From a Vague Idea, through the Lysosome and the Ubiqui-tin-Proteasome System, and onto Human Diseases and Drug Targeting (Nobel Lecture). Angewandte Chemie Interna-tional Edition, 44, 5944-5967. https://doi.org/10.1002/anie.200501428
- 2. Dikic, I. (2017) Proteasomal and Au-tophagic Degradation Systems. Annual Review of Biochemistry, 86, 193-224. https://doi.org/10.1146/annurev-biochem-061516-044908
- 3. Rousseau, A. and Bertolotti, A. (2018) Regulation of Proteasome Assembly and Activity in Health and Disease. Nature Reviews Molecular Cell Biology, 19, 697-712. https://doi.org/10.1038/s41580-018-0040-z
- 4. Varshavsky, A. (2005) Regulated Protein Degradation. Trends in Biochemical Sciences, 30, 283-286. https://doi.org/10.1016/j.tibs.2005.04.005
- 5. Lilienbaum, A. (2013) Relationship between the Proteasomal Sys-tem and Autophagy. International Journal of Biochemistry and Molecular Biology, 4, 1-26.
- 6. Wang, F., Gómez-Sintes, R. and Boya, P. (2018) Lysosomal Membrane Permeabilization and Cell Death. Traffic, 19, 918-931. https://doi.org/10.1111/tra.12613
- 7. Li, X., He, S. and Ma, B. (2020) Autophagy and Autophagy-Related Pro-teins in Cancer. Molecular Cancer, 19, Article No. 12. https://doi.org/10.1186/s12943-020-1138-4
- 8. Mizushima, N., Levine, B., Cuervo, A.M. and Klionsky, D.J. (2008) Autophagy Fights Disease through Cellular Self-Digestion. Nature, 451, 1069-1075. https://doi.org/10.1038/nature06639
- 9. Kaushik, S. and Cuervo, A.M. (2012) Chaper-one-Mediated Autophagy: A Unique Way to Enter the Lysosome World. Trends in Cell Biology, 22, 407-417. https://doi.org/10.1016/j.tcb.2012.05.006
- 10. Mizushima, N. and Yoshimori, T. (2007) How to Interpret LC3 Immunoblotting. Autophagy, 3, 542-545. https://doi.org/10.4161/auto.4600
- 11. Yorimitsu, T. and Klionsky, D.J. (2005) Autophagy: Molecular Machinery for Self-Eating. Cell Death & Differentiation, 12, 1542-1552. https://doi.org/10.1038/sj.cdd.4401765
- 12. Kroemer, G., Mariño, G. and Levine, B. (2010) Autophagy and the In-tegrated Stress Response. Molecular Cell, 40, 280-293. https://doi.org/10.1016/j.molcel.2010.09.023
- 13. Russell, R.C., Tian, Y., Yuan, H., Park, H.W., Chang, Y.Y., Kim, J., Kim, H., Neufeld, T.P., Dillin, A. and Guan, K.L. (2013) ULK1 Induces Autophagy by Phosphorylating Beclin-1 and Activating VPS34 Lipid Kinase. Nature Cell Biology, 15, 741-750. https://doi.org/10.1038/ncb2757
- 14. Tanida, I., Ueno, T. and Kominami, E. (2004) LC3 Conjugation System in Mammalian Autophagy. The International Journal of Biochemistry & Cell Biology, 36, 2503-2518. https://doi.org/10.1016/j.biocel.2004.05.009
- 15. Tsuboyama, K., Koyama-Honda, I., Sakamaki, Y., Koike, M., Morishita, H. and Mizushima, N. (2016) The ATG Conjugation Systems Are Important for Degradation of the Inner Autophagosomal Membrane. Science, 354, 1036-1041. https://doi.org/10.1126/science.aaf6136
- 16. Faruk, M.O., Ichimura, Y. and Komatsu, M. (2021) Selective Au-tophagy. Cancer Science, 112, 3972-3978. https://doi.org/10.1111/cas.15112
- 17. Zaffagnini, G. and Martens, S. (2016) Mechanisms of Selective Autophagy. Journal of Molecular Biology, 428, 1714-1724. https://doi.org/10.1016/j.jmb.2016.02.004
- 18. Lamark, T. and Johansen, T. (2012) Aggrephagy: Selective Dispos-al of Protein Aggregates by Macroautophagy. International Journal of Cell Biology, 2012, Article ID: 736905. https://doi.org/10.1155/2012/736905
- 19. Lemasters, J.J. (2005) Selective Mitochondrial Autophagy, or Mitophagy, as a Targeted Defense against Oxidative Stress, Mitochondrial Dysfunction, and Aging. Rejuvenation Research, 8, 3-5. https://doi.org/10.1089/rej.2005.8.3
- 20. Bauckman, K.A., Owusu-Boaitey, N. and Mysorekar, I.U. (2015) Selec-tive Autophagy: Xenophagy. Methods, 75, 120-127. https://doi.org/10.1016/j.ymeth.2014.12.005
- 21. Wyant, G.A., Abu-Remaileh, M., Frenkel, E.M., Laqtom, N.N., Dharamdasani, V., Lewis, C.A., Chan, S.H., Heinze, I., Ori, A. and Sabatini, D.M. (2018) NUFIP1 Is a Ribosome Re-ceptor for Starvation-Induced Ribophagy. Science, 360, 751-758. https://doi.org/10.1126/science.aar2663
- 22. Cebollero, E., Reggiori, F. and Kraft, C. (2012) Reticulophagy and Ribophagy: Regulated Degradation of Protein Production Factories. International Journal of Cell Biology, 2012, Article ID: 182834. https://doi.org/10.1155/2012/182834
- 23. Wilson, C.J., Myer, V.E., Porter, J.A., Bussiere, D.E., Finan, P.M., La-bow, M.A., Mao, X., Hamann, L.G., Manning, B.D., Valdez, R.A., Nicholson, T., Schirle, M., Knapp, M.S., Keaney, E.P. and Murphy, L.O. (2014) Selective VPS34 Inhibitor Blocks Autophagy and Uncovers a Role for NCOA4 in Ferri-tin Degradation and Iron Homeostasis in Vivo. Nature Cell Biology, 16, 1069-1079. https://doi.org/10.1038/ncb3053
- 24. Johansen, T. and Lamark, T. (2020) Selective Autophagy: ATG8 Family Proteins, LIR Motifs and Cargo Receptors. Journal of Molecular Biology, 432, 80-103. https://doi.org/10.1016/j.jmb.2019.07.016
- 25. Ma, X., Lu, C., Chen, Y., Li, S., Ma, N. and Tao, X. (2022) CCT2 Is an Aggrephagy Receptor for Clearance of Solid Protein Aggregates. Cell, 185, 1325-1345.E22. https://doi.org/10.1016/j.cell.2022.03.005
- 26. Mancias, J.D., Wang, X., Gygi, S.P., Harper, J.W. and Kimmelman, A.C. (2014) Quantitative Proteomics Identifies NCOA4 as the Cargo Receptor Mediating Ferritinophagy. Nature, 509, 105-109. https://doi.org/10.1038/nature13148
- 27. Kumsta, C., Chang, J.T., Lee, R., Tan, E.P., Yang, Y., Loureiro, R., Choy, E.H., Lim, S.H.Y., Saez, I., Springhorn, A., Hoppe, T., Vilchez, D. and Hansen, M. (2019) The Autophagy Receptor P62/SQST-1 Pr
NOTES
*第一作者。
#通讯作者。
