Advances in Clinical Medicine
Vol.
11
No.
06
(
2021
), Article ID:
43614
,
9
pages
10.12677/ACM.2021.116419
幽门螺旋杆菌相关性胃病与HP基因分型的关系研究
杨慧君,王学红*
青海大学附属医院消化内科,青海 西宁

收稿日期:2021年5月28日;录用日期:2021年6月19日;发布日期:2021年6月30日

摘要
目的:通过检测青海地区慢性非萎缩性胃炎(CSG)、慢性萎缩性胃炎(CAG)及胃腺癌(GC)等患者胃粘膜组织中的幽门螺旋杆菌(H. pylori)菌株的基因分型,探讨各基因型与三组疾病之间的关系。方法:收集2019年12月至2020年12月在青海大学附属医院就诊的满足排标准的,并经13C或14C尿素呼气试验检测证实存在H. pylori感染的并行胃镜及病理组织学检查诊断为CAG、CAG、GC的患者共78例为研究对象,通过胃镜活检提取胃粘膜组织并分离培养获得H. pylori菌株,将上述获得的H. pylori菌株提取DNA,采用PCR扩增方法检测上述样本中的CagA、VacA (s1、s2、m1、m2)、UreA、UreB,babA等基因型分布。结果:1) 本实验共扩增出78例H. pylori菌株,其中CSG组24例,CAG组36例,GC组18例。CagA基因共检测出72例,总体检出率为92.31% (P > 0.05)。BabA、UreA、UreB基因检出率分别为64.1%、64.10%、79.49% (P > 0.05)。VacA基因亚型VacAs1、VacA s2、VacA m1、VacA m2基因检出率分别为75.64%、15.38%、42.31%、53.85% (P > 0.05)。2) H. pylori各基因型在三组疾病间的分布无显著统计学差异(P > 0.05)。结论:1) 青海地区H. pylori感染人群中CagA、VacA及尿素酶基因是本地检出率最高的基因型;2) H. pylori各基因型不能作为判断慢性胃炎是否能发展成胃癌及癌前病变的特异性标志。
关键词
幽门螺旋杆菌,基因分型,CagA,VacA,BabA,VacA
Study on the Relationship between Helicobacter pylori Associated Gastropathy and HP Genotyping in Qinghai Province
Huijun Yang, Xuehong Wang*
Department of Gastroenterology, The Affiliated Hospital of Qinghai University, Xining Qinghai
Received: May 28th, 2021; accepted: Jun. 19th, 2021; published: Jun. 30th, 2021

ABSTRACT
Objective: To study the genotypes of H. pylori strains in the gastric mucosa of patients with chronic non-atrophic gastritis (CSG), chronic atrophic gastritis (CAG) and gastric adenocarcinoma (GC) in Qinghai, and to explore the relationship between each genotype and the three groups of diseases. Methods: Collected patients who met the exclusion criteria in the Affiliated Hospital of Qinghai University from December 2019 to December 2020 were diagnosed with Helicobacter pylori infection by gastroscopy and histopathological examination through 13C or 14C urea breath test. A total of 78 patients with CAG, CAG, and GC were the subjects of this study. Gastric mucosal tissues were extracted by gastroscopy and cultured to obtain H. pylori strains. DNA was extracted from the H. pylori strains obtained above. Genes such as CagA, VacA (s1, s2, m1, m2), UreA, UreB, BabA were detected by PCR and sequencing. Results: 1) A total of 78 H. pylori strains were amplified in this experiment, including 24 cases in CSG group, 36 cases in CAG group and 18 cases in GC group. A total of 72 cases of CagA gene were detected, and the overall detection rate was 92.31% (P > 0.05). The positive rates of BabA, UreB and UreB genes were 64.1%, 64.10% and 79.49% (P > 0.05), respectively. The positive rates of VacAs1, VacA s2, VacA m1 and VacA m2 subtypes were 75.64%, 15.38%, 42.31% and 53.85% (P > 0.05), respectively. There was no significant statistical difference in the distribution of H. pylori genotypes among the three groups (P > 0.05). Conclusion: 1) CagA, VacA and urease genes were the genotypes with the highest local detection rate in H. pylori infected people in Qinghai. 2) H. pylori genotypes could not be used as a specific marker to determine whether chronic gastritis could develop into gastric cancer and precancerous lesions.
Keywords:Helicobacter pylori, Genotyping, CagA, VacA, BabA, VacA

Copyright © 2021 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY 4.0).
http://creativecommons.org/licenses/by/4.0/


1. 引言
幽门螺旋杆菌(Helicobacter pylori, H. pylori)系革兰阴性菌种,是一种能存活在人类胃黏膜组织中单级、末端顿圆且带有鞭毛、呈螺旋弯曲状的微需氧菌 [1]。它的发现使医学界对感染、炎症和癌症之间关系的有了初步了解。后来,关于它的研究风靡世界各地。众多研究发现这种细菌是导致包括慢性胃炎和消化性溃疡等胃十二指肠疾病的主要病原体,甚至有导致胃腺癌发生的可能,因此国际癌症研究中心(IARC)已把H. pylori归入确认致癌物之列 [2]。研究 [3] [4] 表明胃粘膜受到H. pylori感染后所表现出的不同临床结局除与宿主个体差异、免疫因素和环境因素有关外,还与H. pylori菌株的基因型有关,H. pylori不同基因型之间的致病性差异很大,因此感染者的临床结局也因型而异。H. pylori菌株能产生多种细胞毒素和毒力因子,最先被发现的是在致病性起主导作用的细胞毒素相关基因A蛋白(Cytotoxin-associated gene A, CagA)、空泡细胞毒素基因A蛋白(Vacuolating cytotoxin A, VacA),随后发现尿素酶基因(UreA)、血型组抗原结合黏附素(Blood group antigen-binding adhesin, BabA)等对疾病的发生皆有重要作用。但也有研究 [5] 发现H. pylori基因型与疾病进展及最终临床结局并无明显相关性,因此目前关于H. pylori的基因分型与相关疾病的关系依然存在争议,有待进一步研究澄清。因此研究本地区H. pylori基因分型及其与胃病发生关系研究对于各种上消化道疾病的防治具有重要意义。
2. 材料与方法
2.1. 一般资料
收集2019年12月至2020年12月期间在青海大学附属医院就诊的经尿素呼气实验证实存在H. pylori感染的患者共78例,其中男38例,女40例。选取经胃镜及病理组织学确诊为慢性非萎缩性胃炎(Chronic superficial gastritis, CSG) (24例)、慢性萎缩性胃炎(Chronic atrophic gastritis, CAG) (38例)、胃腺癌(Gastric cancer, GC) (18例)的患者为研究对象,行胃镜检查时在胃窦或胃体钳取胃粘膜2~3块,放入专用培养基保存。纳入标准:① 在我省有10年以上的居住史,年龄在18岁以上70周岁以内,不论性别。② 伴或不伴上消化道症状行13C或14C尿素呼气试验为阳性的患者,且未经H. pylori根除治疗。③ 所有研究对象经我院胃镜检查确诊为CSG、CAG以及初次诊断为GC者,被检测的患者均经病理组织学确诊。排除标准:① 接受过胃部手术(外科手术和微创等)、放射治疗或化学等治疗者;② 既往胃癌或恶性肿瘤,消化道出血、胃息肉患者;③ 近4周接受过抑酸药、抗菌药和保护胃黏膜药物及以及根除过H. pylori患者;④ 近1周有服用阿司匹林、华法林等抗凝药病史,不宜行活检者;⑤ 相关临床病史及病理检查资料不完整者。本实验经我院科研项目伦理委员会批准通过(批件编号:P-SL-201958)。所有参与者均签知情同意书。
2.2. 主要仪器及试剂
由上海芯超医学检验所有限公司提供的细菌微量生化反应管(尿素酶),哥伦比亚血琼脂培养基(上海科玛嘉微生物技术有限公司),心脑脊液(赛默飞世尔科技有限公司),快速尿素酶诊断试剂(广州贝思奇诊断试剂有限公司),三气培养箱(HF100,上海力申有限公司),PCR扩增仪(Veniti Dx,赛默飞世尔科技公司),高速离心机(Micro 21,赛默飞世尔科技公司)。
2.3. 方法
1) 菌株的培养及鉴定:将胃黏膜标本接种在哥伦比亚血琼脂培养基上,将胃粘液面与培养基接触进行旋转涂匀。在微需氧环境(5% O2,10% CO2,85% N2)温度在37℃培养3~4天。然后通过形态观察(革兰氏染色)以及生化反应如尿素酶、氧化酶及触酶等来鉴定。培养4天后观察平板,进行氧化酶试验,用氧化酶滤纸条分别蘸取平板菌落,滤纸顶端如立刻出现紫色,继而逐渐加深,证明该菌落为幽门螺杆菌,阴性不变色。
2) H. pylori基因的提取:使用广州美基生物科技有限公司提供的试剂盒Magen HiPure Bacterial DNA Kits进行H. pylori菌株DNA的抽提。
3) PCR引物设计:对H. pylori菌株中cagA、babA、ureA、ureB、vacA (s1/s2、m1/m2)等基因进行扩增,各基因序列来自NCBI,具体PCR扩增引物由上海芯超医学检验所有限公司设计合成,并进行测序。引物序列见表1。PCR反应体系:取5 ng总量的DNA定容在21 ul的ddH2O,加入25 ul 2xHieff PCR Master Mix (With Dye) (2XPCR扩增酶带染料),2 ul正向引物(浓度10 uM)与2 ul反向引物(浓度10 uM),总共定容在50 ul体系于0.2 ml离心管内。PCR反应条件:96℃预变性2 min,96℃变性10 s,50℃退火10 s,60℃延伸100 s,4℃终止1 min,共30个循环。PCR扩增产物用1.5%琼脂糖凝胶电泳,90 V电泳30 min,在紫外透射分析仪下观察,并用凝胶成像系统分析。
Table 1. Primers and sequences of H. pylori target gene amplification
表1. H. pylori目的基因扩增引物及序列
2.4. 统计学方法
统计软件使用SPSS 23.0版本,来进行数据的统计和分析。计数资料采用百分比(%)分析H. pylori基因型在受检对象中的检出率,采用c2检验或Fisher’s精确检验来分析各组疾病与所检测基因之间的关系。以P ≤ 0.05为差异有统计学意义。
3. 结果
1) 本实验采用PCR扩增技术对检测样本中提取的菌株DNA进行检测,目的基因包括vacA (s1/s2、m1/m2)基因、cagA基因、babA基因、ureA基因、ureB基因),PCR扩增产物用1.5%琼脂糖凝胶电泳,90 V电泳30 min,在紫外透射分析仪下观察,并用凝胶成像系统分析,如图1所示,vacA各亚型见图2。
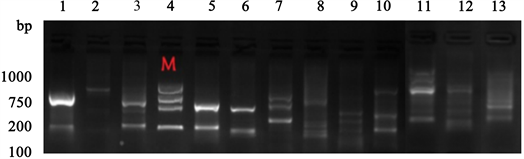 编码4泳道为Marker,从下至上大小分别为200 bp、500 bp、750 bp、1000 bp,1~3泳道和5~6泳道为cagA基因(715 bp),泳道7~9为babA (大小分别为691 bp、702 bp、750 bp);泳道10~12为ureB (大小分别为693 bp、746 bp、563 bp);泳道13为ureA (600 bp)。
编码4泳道为Marker,从下至上大小分别为200 bp、500 bp、750 bp、1000 bp,1~3泳道和5~6泳道为cagA基因(715 bp),泳道7~9为babA (大小分别为691 bp、702 bp、750 bp);泳道10~12为ureB (大小分别为693 bp、746 bp、563 bp);泳道13为ureA (600 bp)。
Figure 1. Gel electrophoresis images of PCR amplification fragments of some samples
图1. 部分样本的PCR扩增片段凝胶电泳图
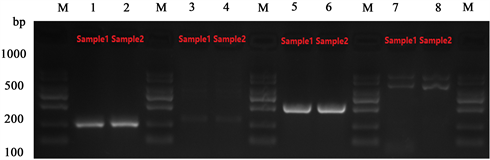 M为Marker,泳道1~2为vacA s1基因(大小259 bp),泳道3~4为vacA s2 (大小为199 bp);泳5~6为vacA m2 (大小352 bp);泳道7~8为vacA m1基因(大小570 bp)。
M为Marker,泳道1~2为vacA s1基因(大小259 bp),泳道3~4为vacA s2 (大小为199 bp);泳5~6为vacA m2 (大小352 bp);泳道7~8为vacA m1基因(大小570 bp)。
Figure 2. Gel electrophoresis of PCR amplified fragments of VacA gene
图2. VacA各基因亚型PCR扩增片段凝胶电泳图
2) H. pylori基因型中的检测结果
本实验共扩增78例H. pylori菌株,其中CagA基因共检测出72例,总体检出率为92.31% (72/78),BabA基因共扩增出50例阳性样本,检出率为64.1% (50/78)。UreA基因共检测出50例样本,检出率为64.10% (50/78)。UreB基因共检测出62例样本,检出率为79.49% (62/78)。VacA基因的各亚型检测情况:VacA s1、VacA s2、VacA m1、VacA m2基因总体检出率分别为:75.64%、15.38%、42.31%、53.85%。感染率最高的基因型依次为CagA、UreB、VacA s1。详见表2。
Table 2. Overall detection rate of H. pylori genotypes in subjects (n = 78)
表2. H. pylori各基因型在受检者中的总体检出率(n = 78)
3) H. pylori各基因型与三组疾病的关系分析
在测序成功的78例阳性菌株中,CagA基因在慢性非萎缩性胃炎、慢性萎缩性胃炎、胃癌三组中的检出率分别为100% (24/24)、88.89% (32/36)、88.89% (16/18),CagA在三组疾病中的分布无显著统计学差异(P > 0.05)。BabA基因共扩增出50例样本,在CSG、CAG、GC三组中的检出率分别为:83.33%、55.56%、55.56%,各组感染率无显著统计学差异(c2 = 6.022, P > 0.05)。UreA基因在CSG、CAG、GC三组疾病中的检出率分别为:75.00%、61.11%、55.56%,差异无统计学意义(c2 = 1.968, P > 0.05)。UreB基因在CSG、CAG、GC三组疾病中的检出率分别为:83.33%、83.33%、66.67%,差异无统计学意义(c2 = 2.177, P > 0.05)。UreA基因在CSG、CAG、GC三组疾病中的检出率分别为:75.00%、61.11%、55.56%,差异无统计学意义(c2 = 1.968, P > 0.05)。VacA基因的各亚型检测情况:VacA s1基因在CSG、CAG、GC三组疾病中的检出率分别为:79.17%、75.00%、72.22%,在各组中检出率无显著统计学差异(c2 = 2.445, P > 0.05)。VacA s2基因在CSG、CAG、GC三组疾病中的检出率分别为:12.50%、13.89%、22.22%,差异无统计学意义(P > 0.05)。VacA m1基因在CSG、CAG、GC三组疾病中的检出率分别为:58.33%、38.89%、27.78%,差异无统计学意义(c2 = 2.445, P > 0.05)。VacA m2基因在CSG、CAG、GC三组疾病中的检出率分别为:50.00%、61.11%、44.40%,其检出率亦无统计学差异。综合以上分析H. pylori基因分型vacA (s1/s2、m1/m2)基因、cagA基因、babA基因、ureA基因、ureB基因在CSG、CAG、GC等三组疾病中的分布均无显著统计学差异(P > 0.05)。见表3。
Table 3. Comparison of the detection rate of H. pylori genotype in the three groups of diseases [n(%)]
表3. H. pylori基因型在三组疾病中的检出率比较[n(%)]
注:#为Fisher’s Exact Test结果,无c2值。
4. 讨论
幽门螺旋杆菌慢性感染导致的胃黏膜炎症被称为H. pylori相关性胃炎 [6]。H. pylori通过诱发炎性活动和特异性免疫效应引发胃粘膜中性粒细胞浸润,导致胃黏膜急性活动性炎症,继而出现淋巴细胞和浆细胞浸润,导致胃粘膜慢性疾病,如持续感染,可出现萎缩、肠上皮化生及不典型增生,最终使部分宿主罹患GC [7]。CagA是目前已被证实是一种癌性蛋白,可参与GC形成。通过各种细胞激酶进行酪氨酸磷酸化,磷酸化的酪氨酸与Src同源磷酸酶2或适配蛋白GrB2相互作用,可以激活某些细胞信号传导通道,阻碍细胞间的粘附、细胞增殖、促进IL-8表达和细胞伸长 [8]。CagA也可以刺激上皮–间质转化、加速细胞增长,有促进胃黏膜上皮细胞发生变异的可能,最终导致GC形成 [9]。以往的研究报道了在世界不同地区收集的幽门螺杆菌分离株中VacA基因型和CagA基因的感染情况有较大差异。本实验共收集经证实有H. pylori感染的患者78例,其中CagA基因共检测出72例,总体检出率为92.31% (72/78),在慢性非萎缩性胃炎、慢性萎缩性胃炎、胃癌三组中的检出率分别为100% (24/24)例,88.89% (32/36)、88.89% (16/18)。据报道 [10] 在东亚至少90%的H. pylori阳性感染者是CagA阳性,本研究结果正验证了此观点。陈力等 [11] 对青岛地区患有胃黏膜病变的患者包括CSG、CAG、GC等进行H. pylori抗体分型研究三组疾病的检出率分别为85.7%、79.4%、96.2%,也与本研究结果相符合。根据本次实验CagA基因在CSG、CAG、GC三组疾病中的检出结果提示各组间检出率无统计学意义(P > 0.05)。因此CagA基因不能作为判断慢性胃炎是否有发展至胃癌的独立危险因素。
VacA构成使细菌在胃上皮细胞中定殖和存活的最关键的毒素之一。基本上所有菌株中都能检出VacA,它包括多种等位基因,在不同的亚型中具有不同程度的活性 [12],因此菌株之间的VacA毒素活性存在很大差异 [13]。基于特定区域中的序列异质性,VacA等位基因被分为s1、s2、m1、m2、s1m1、s1m2、s2m2和s2m1 [14]。VacA可能通过多种生物学机制促进胃癌的发生,首先VacA通过诱导胃上皮细胞改变可能潜在地改变胃环境以促进H. pylori定植和复制 [15]。有实验证实VacA还能抑制壁细胞的功能,使胃酸过少,从而促进硝酸盐的增殖而增加胃癌的风险 [13]。本实验中78例菌株中VacA s1基因共检测出59例,检出率为75.64%,VacA s2基因共检测出12例,检出率为15.38%,VacA m1基因共检测出33例,检出率为42.31%,VacA m2基因共检测出42例,检出率为53.85%。在本研究中,VacA s1比VacAs2多见,m2型比m1较为多见,且各基因型在三组疾病中分布无统计学差异(P > 0.05)。这与江海洋等 [16] 研究数据基本一致。蒋闵仁等 [17] 对上海地区消化道疾病患者菌株分型进行研究报道VacA s1/m2的阳性率最高,也与本研究结果相同。可以看出VacA s1m2组合基因型是本地的优势基因型。
BabA是一种血型抗原结合粘附素相关的毒力因子,可使幽门螺杆菌粘附于胃粘膜上皮并将毒素或其他毒力因子传递到宿主细胞中,从而促进对H. pylori的直接或间接损伤 [18]。BabA可粘附在胃上皮细胞,释放出过量的促炎因子,从而刺激癌变 [19]。BabA与严重胃病之间的关系尚有争议,但已知BabA2与Lewis B之间的相互作用可激活促炎细胞因子(CCL5、IL-8)和其他与癌前病变相关的分子的产生 [20]。众多研究表明大多数亚洲的幽门螺杆菌菌株BabA2阳性,本实验对青海地区H. pylori菌株进行基因扩增,结果显示有50例样本表达BabA基因,检出率为64.1%,这与其他研究相一致。Eshaghei等 [21] 对伊朗的一项研究中,BabA基因型的菌株感染率为71.6%,这与本研究所测数据基本一致。但低于Chomvarin等 [22] 人报道泰国消化不良患者中BabA2的患病率92%。相比之下,西方国家的患病率较低,据Olfat等 [23] 报道德国、瑞典、葡萄牙和芬兰的患病率分别为45%、45%、34%和60%。实验数据表明BabA基因型检出率在慢性非萎缩性胃炎组、慢性萎缩性胃炎和胃癌组,各组间差异无统计学意义(P > 0.05)。
尿素酶通过水解尿素产生氨和CO2,从而升高胃内的PH值,克服胃内的酸性环境,有利于H. pylori在胃粘膜的定植 [24]。尿素酶通过多种机制调节宿主免疫反应,包括改变调理作用、增强中性粒细胞和单核细胞的趋化性,由于与II类主要组织相容性复合体(MHC)受体结合而促进细胞凋亡,或增强促炎细胞因子的释放 [25]。尿素酶被认为是由于它可以刺激血管生成,而导致胃癌的进展的 [26]。我们的实验中UreA基因共检测出50例样本,检出率为64.10%,在CSG、CAG、GC三组疾病中的检出率分别为:75.00%、61.11%、55.56%。UreB基因共检测出62例样本,检出率为79.49%,在CSG、CAG、GC三组疾病中的检出率分别为:83.33%、83.33%、66.67%。三组疾病中尿素酶基因的检出率无显著差异(P > 0.05),我们的实验表明尿素酶基因也是青海本地人群H. pylori菌株分型中的主要基因型。但我省的检出率总体低于曹群奋等 [27] 对镇海地区84例H. pylori菌株进行的研究,据其报道该地UreA基因检出率为100%,且均在消化性溃疡及胃炎中的检出率更高。其有所出入其原因可能是样本量较小,或不同地区确实存在差异。
5. 结论
综上所述,通过本研究对青海部分地区HP感染人群基因分型的检测,使我们得知本地区H. pylori感染的基因型以CagA、VacA以及尿素酶基因型为主。但我们实验所测基因型在慢性萎缩性胃炎、慢性浅表性胃炎及胃癌中的分布并无显著统计学差异,目前的研究暂不能鉴别感染者中具体哪种基因型会有发展至胃癌的风险,故不能作为GC的特定标志物。本研究存在众多局限,首先纳入样本的数量较少,其次未对VacA基因的亚型如s1a、s1b、s1c以及oip的功能状态进行进一步的深入研究。再次胃癌组中样本比胃炎组少,导致结果存在一定偏差。今后的研究中需要进一步加大样本量的纳入,提高结果的可靠性。要使H. pylori基因型与上消化道疾病之间的关系研究真正应用于临床,在今后的研究中需要加大样本、多研究等进一步分析。
基金项目
青海省消化系统疾病临床医学研究中心(2019-SF-L3);规范化大型胃癌队列的建立及其可行性研究(2017YFC0908300)。
文章引用
杨慧君,王学红. 幽门螺旋杆菌相关性胃病与HP基因分型的关系研究
Study on the Relationship between Helicobacter pylori Associated Gastropathy and HP Genotyping in Qinghai Province[J]. 临床医学进展, 2021, 11(06): 2891-2899. https://doi.org/10.12677/ACM.2021.116419
参考文献
- 1. Fischbach, W. and Malfertheiner, P. (2018) Helicobacter pylori Infection. Deutsches Arzteblatt International, 115, 429-436. https://doi.org/10.3238/arztebl.2018.0429
- 2. Takahashi-Kanemitsu, A., Knight, C.T. and Hatakeyama, M. (2020) Molecular Anatomy and Pathogenic Actions of Helicobacter pylori CagA That Underpin Gastric Carcinogenesis. Cellular & Molecular Immunology, 17, 50-63. https://doi.org/10.1038/s41423-019-0339-5
- 3. 刘丹凤, 郑奎城. 幽门螺杆菌毒力因子CagA和VacA多样性与胃部疾病关系研究进展[J]. 海峡预防医学杂志, 2018, 2(4): 16-20.
- 4. 杨波, 杜利君, 魏剑林, 等. 幽门螺杆菌细胞毒素相关蛋白A、热休克蛋白60和细胞空泡毒素与慢性胃炎患者胃粘膜病理变化的关系[J]. 解放军预防医学杂志, 2018, 36(7): 832-835.
- 5. 张双红, 谢勇, 肖珍君, 等. 胃十二指肠疾病儿童幽门螺杆菌cagA、vacA和iceA基因型分布[J]. 中国当代儿科杂志, 2016, 18(7): 618-624.
- 6. 刘文忠, 谢勇, 陆红, 等. 第五次全国幽门螺杆菌感染处理共识报告[J]. 中国实用内科杂志, 2017, 37(6): 509-524.
- 7. 刘贤, 何帮顺, 林康, 陈杰, 彭红新, 王书奎. 幽门螺杆菌基因分型与胃病关系研究进展[J]. 现代生物医学进展, 2016, 16(7): 1390-1393.
- 8. Hatakeyama, M. (2014) Helicobacter pylori CagA and Gastric Cancer: A Paradigm for Hit-and-Run Carcinogenesis. Cell Host & Microbe, 15, 306-316. https://doi.org/10.1016/j.chom.2014.02.008
- 9. Román-Román, A., MartÍnez-Carrillo, D.N., Atrisco-Morales, J., et al. (2017) Helicobacter pylori vacA s1m1 Genotype but Not or Increase the Risk of Ulcer and Gastric Cancer in Patients from Southern Mexico. Gut Pathogens, 9, Article No. 18. https://doi.org/10.1186/s13099-017-0167-z
- 10. Yamaoka, Y., Orito, E., Mizokami, M., et al. (2002) Helicobacter pylori in North and South America before Columbus. FEBS Letters, 517, 180-184. https://doi.org/10.1016/S0014-5793(02)02617-0
- 11. 陈力, 张成栋, 殷洁, 等. 青岛地区胃黏膜病变患者幽门螺杆菌及其相关抗体的分析[J]. 胃肠病学和肝病学杂志, 2017, 26(6): 682-685.
- 12. Kim, I.-J. and Blanke, S.R. (2012) Remodeling the Host Environment: Modulation of the Gastric Epithelium by the Helicobacter pylori Vacuolating Toxin (VacA). Frontiers in Cellular and Infection Microbiology, 2, 37. https://doi.org/10.3389/fcimb.2012.00037
- 13. Mcclain, M.S., Beckett, A.C. and Cover, T.L. (2017) Helicobacter pylori Vacuolating Toxin and Gastric Cancer. Toxins, 9, 316. https://doi.org/10.3390/toxins9100316
- 14. Keikha, M., Ali-Hassanzadeh, M., & Karbalaei, M. (2020) Association of Helicobacter pylori vacA Genotypes and Peptic Ulcer in Iranian Population: A Systematic Review and Meta-Analysis. BMC Gastroenterology, 20, Article No. 266. https://doi.org/10.1186/s12876-020-01406-9
- 15. Tan, S., Noto, J., Romero-Gallo, J., et al. (2011) Helicobacter pylori Perturbs Iron Trafficking in the Epithelium to Grow on the Cell Surface. PLoS Pathogens, 7, e1002050. https://doi.org/10.1371/journal.ppat.1002050
- 16. 江海洋,赵春燕, 范聪聪, 等. 幽门螺杆菌毒力基因vacA多态性与胃肠道疾病的关系[J]. 胃肠病学和肝病学杂志, 2018, 27(2): 142-145.
- 17. 蒋闵仁, 黄英, 宋佳鸿, 等. 幽门螺杆菌细胞毒素相关蛋白A、细胞毒素相关蛋白E、细胞空泡毒素A基因型与上消化道疾病的关系[J]. 实用临床医药杂志, 2017, 21(13): 36-39.
- 18. Homan, M., Šterbenc, A., Kocjan, B., et al. (2014) Prevalence of the Helicobacter pylori babA2 Gene and Correlation with the Degree of Gastritis in Infected Slovenian Children. Antonie van Leeuwenhoek, 106, 637-645. https://doi.org/10.1007/s10482-014-0234-0
- 19. Ansari, S. and Yamaoka, Y. (2017) Helicobacter pylori BabA in Adaptation for Gastric Colonization. World Journal of Gastroenterology, 23, 4158-4169. https://doi.org/10.3748/wjg.v23.i23.4158
- 20. Ishijima, N., Suzuki, M., Ashida, H., et al. (2011) BabA-Mediated Adherence Is a Potentiator of the Helicobacter pylori Type IV Secretion System Activity. The Journal of Biological Chemistry, 286, 25256-25264. https://doi.org/10.1074/jbc.M111.233601
- 21. Safaei, H.G., Havaei, S.A., Tavakkoli, H., et al. (2010) Relation of babA2 Genotype of Helicobacter pylori Infection with Chronic Active Gastritis, Duodenal Ulcer and Non-Cardia Active Gastritis in Alzahra Hospital Isfahan, Iran. Jundishapur Journal of Microbiology, 3, 93-98.
- 22. Chomvarin, C., Namwat, W., Chaicumpar, K., et al. (2008) Prevalence of Helicobacter pylori vacA, cagA, cagE, iceA and babA2 Genotypes in Thai Dyspeptic Patients. International Journal of Infectious Diseases, 12, 30-36. https://doi.org/10.1016/j.ijid.2007.03.012
- 23. Olfat, F.O., Quing, Z., Monica, O., et al. (2005) Correlation of the Helicobacter pylori Adherence Factor BabA with Duodenal Ulcer Disease in Four European Countries. FEMS Immunology & Medical Microbiology, 44, 151-156. https://doi.org/10.1016/j.femsim.2004.10.010
- 24. Sidebotham, R., Worku, M., Karim, Q., et al. (2003) How Helicobacter pylori Urease May Affect External pH and Influence Growth and Motility in the Mucus Environment: Evidence from In-Vitro Studies. European Journal of Gastroenterology & Hepatology, 15, 395-401. https://doi.org/10.1097/00042737-200304000-00010
- 25. Schmalstig, A., Benoit, S., Misra, S., et al. (2018) Noncatalytic Antioxidant Role for Helicobacter pylori Urease. Journal of Bacteriology, 200, e00124-18. https://doi.org/10.1128/JB.00124-18
- 26. Olivera-Severo, D., Uberti, A.F., Marques, M.S., et al. (2017) A New Role for Helicobacter pylori Urease: Contributions to Angiogenesis. Frontiers in Microbiology, 8, 1833. https://doi.org/10.3389/fmicb.2017.01883
- 27. 曹群奋, 车海斌, 陈勇华, 等. 幽门螺杆菌的毒力基因vacA基因亚型与上消化道疾病关系的研究[J]. 中华医院感染学杂志, 2007, 17(12): 1491-1522.
NOTES
*通讯作者。
