Hans Journal of Agricultural Sciences
Vol.
09
No.
02
(
2019
), Article ID:
28967
,
12
pages
10.12677/HJAS.2019.92020
Mitochondrial Genome Sequence Analysis of Cerapachys xizangensis
Ye Zhong1,2, Shanyi Zhou1,2, Zhilin Chen1,2*
1Laboratory of Ecology of Rare and Endangered Species and Environmental Protection, Guangxi Normal University, Ministry of Education, Guilin Guangxi
2Guangxi Key Laboratory of Rare and Endangered Animal Ecology, Guangxi Normal University, Guilin Guangxi

Received: Feb. 3rd, 2019; accepted: Feb. 15th, 2019; published: Feb. 22nd, 2019

ABSTRACT
This paper reports mitochondrial genome of Cerapachys xizangensis based on high-throughput sequencing, and improves the method of primer design to circular DNA complete sequence creatively. Through the sequence analysis, we found that the mitochondrial genome of Cerapachys xizangensisis is a double-stranded DNA molecule, with total length of 16,769 bp, and two gene rearrangements occurred: one is gene shuffle; the other is gene inversion.
Keywords:Formicidae, Mitochondrial Genome, Gene Rearrangement, Primer Design
西藏粗角猛蚁线粒体基因组序列分析
钟烨1,2,周善义1,2,陈志林1,2*
1广西师范大学,珍稀濒危动植物生态与环境保护教育部重点实验室,广西 桂林
2广西师范大学,广西珍稀濒危动物生态学重点实验室,广西 桂林

收稿日期:2019年2月3日;录用日期:2019年2月15日;发布日期:2019年2月22日

摘 要
报道西藏粗角猛蚁Cerapachys xizangensis基于高通量测序的线粒体基因组全序列,创造性地改进了环状DNA缺口处的引物设计补全序列方法。通过序列分析发现,西藏粗角猛蚁线粒体基因组为闭合的双链DNA分子,总长为16,769 bp,发生了两处基因重排现象:一处为基因洗牌,另一处为基因倒置。
关键词 :蚁科,线粒体基因组,基因重排,引物设计

Copyright © 2019 by author(s) and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
昆虫线粒体基因组较小,为闭合的双链DNA分子,总长度为15~20 kb,含有37个基因,即13个蛋白编码基因(COI-III, cytb, ND1-6, ND4L, ATP6, ATP8)、2个核糖体RNA基因(srRNA, lrRNA)、以及22转运RNA基因(其中丝氨酸和亮氨酸有两个对应的tRNA,其它的18个tRNA分别对应一种氨基酸),此外还有一段最大非编码区,此区域AT含量极高,并控制着线粒体基因组的复制与转录,因此称之为控制区CR (Control Region),又称为AT丰富区。不同的物种控制区相差较大,它是造成基因组大小差异的主要原因 [1] 。线粒体基因组具有母系遗传的特点,进化速率快,重组水平低,很适合作为分子标记做较低阶元的系统发育分析 [2] [3] 。在线粒体基因组中,基因结构紧凑,蛋白编码基因中没有发现内含子(珊瑚亚纲种类除外) [4] [5] ,相邻基因间隔小,有较多的基因重叠现象,还存在着基因重排。由于线粒体DNA双链中均分布着一些基因,因此我们把有较多数目基因的链称为J链(majority strand),与之对应的链则称之为N链(minority strand) [6] 。此外,线粒体基因组高A + T含量是其显著特点 [7] 。蚂蚁线粒体基因组研究较晚,资料也较少。Gotzekr等 [8] 首次测定了切叶蚁亚科火蚁属Solenopsis中3种的线粒体基因组全序列,分别长15,549 bp、15,560 bp和15,552 bp,发现火蚁属线粒体基因组全序列中部分tRNA基因发生重排。Hasegawa等 [9] 测定了双针棱胸蚁Pristomyrmex punctatus正常型和变型的线粒体全序列,分析了正常型和变型之间的差异。Berman等 [10] 测定了臭蚁亚科的蜘蛛蚁Leptomyrmex pallens的线粒体基因组全序列,发现tRNA存在易位和倒置。Rodovalho等 [11] 测定了切叶蚁亚科Atta laevigata的线粒体基因组全序列。Babbucci等 [12] 测定了蚁亚科丝光褐林蚁Formica fusca和切叶蚁亚科粗角红蚁Myrmica scabrinodis的线粒体基因组全序列,首次证实其基因组与鳞翅目Dytrisia分支间共享同一基因排列顺序,同时分析了蚁科3类型的基因次序可能的进化机制。Kim等 [13] 测定了韩国一种弓背蚁Camponotus atrox的线粒体基因组全序列,发现tRNA中trnM发生移位,推测该重排方式可能是由复制/随机丢失所致。Yang等 [14] 测定了西利氏蚁Formica selysi的线粒体基因组全序列,证实其基因排序与丝光蚁F. fusca和蜘蛛蚁L. pallens的基因排序一致。西藏粗角猛蚁Cerapachys xizangensis是唐觉等 [15] 在《西藏昆虫》中描述的一个新种,也是国人首次命名的国内蚂蚁物种。测定该蚂蚁的线粒体基因组全序列,可以进一步丰富我国蚂蚁线粒体基因组数据库,为蚂蚁的系统发育研究提供基础资料。
2. 材料与方法
2.1. 研究材料
本实验所选取的蚂蚁标本采于西藏藏族自治区境内,根据《西藏昆虫》中所记述的西藏粗角猛蚁Cerapachys xizangensis的原始描述鉴定后确定。野外采集的标本放入盛有无水乙醇的收集管中,带回实验室置于−60℃的条件下保存。实验时,取同巢中的一个标本制作成针插标本,另取一个标本放置于吸水纸上,待酒精挥发后,用剪刀剪去头部以下部分,将头部放入2 mL的离心管中剪碎、研磨,按照试剂盒上的操作步骤提取总DNA,提取的总DNA经电泳检测合格后,一部分送生物公司做高通量测序,另保存一部分备用。
2.2. 引物设计补全序列
2.2.1. 高通量序列的获取
在生物公司官方网站下载目的DNA序列,用txt文件保存并命名该序列,然后用ClustalX(1.83)软件打开,获得总长度为16,741 bp的DNA序列。
2.2.2. 引物设计
引物设计按以下步骤进行:
① 测序公司返回的序列是线性的,还不能确认是否成环,因此需先将原始序列保存,然后打开一个副本文件,从序列尾部截取10 bp的序列放在另一个新建文本中并命名,用Clustal(1.83)软件的比对功能进行比对。若发现新截取的10 bp尾部序列与序列前端的某个区域不完全重合,则说明序列不能拼接成环,可能在前端与末端之间存在缺口。
② 根据高通量测序的特点及线粒体基因组的大小,可以估测缺口不长;又根据线粒体DNA呈环状的特点,可以用高通量测序末端的序列做正向引物和前端的序列做反向引物设计。本实验截取原序列副本的前端1200 bp,粘贴到序列的末端构成一个新序列并命名。经过计算后,定位缺口在15,541的位置。
③ 将第二步处理的序列导入Oligo软件中,根据引物设计的基本要求寻找最佳引物。因为PCR双向测序的起始和结尾序列可靠性较低,因此在设计引物时最好选在离缺口50 bp碱基外寻找。本实验最终选中了两组引物(见表1)用以扩增序列缺口。
④ 将设计的引物送生物公司合成。
Table 1. Primer sequence
表1. 引物序列
2.2.3. 扩增目的片段
首先将之前提取的DNA用表1中的引物做PCR扩增目的片段,然后对PCR产物进行琼脂糖凝胶电泳,实验发现两组引物均能扩增出清晰的条带且与maker的1000 bp条带相近,因此可以估计此条带即为目的片段。因为F2、R2这一对引物的条带更为清晰,所以选择相应的PCR产物送生物公司做双向测序。
2.2.4. 序列比对补齐缺口
从生物公司网站上下载目的序列测序结果并用txt文件保存,然后直接用ClustalX(1.83)软件导入目的序列和引物设计的第二步序列(即原始序列截取前端1200 bp碱基到尾部的序列)进行比对,在预先定位的15,541处发现序列有28 bp的缺口(见图1)。将缺口处的序列输入原始序列的尾部即获得西藏粗角猛蚁的线粒体DNA全序列,总长16,769 bp。

Figure 1. High-throughput sequencing gap
图1. 高通量测序缺口
3. 序列分析
3.1. 基因注释
3.1.1. tRNA基因定位及调零
将原始序列缺口补齐后放在tRNAscan-SE网站上搜索,得到15个tRNA基因(见表2)。由tRNA基因排序可知,生物公司给出的原始序列为J链,为了便于注释,首先将序列调零。本实验选择tRNAmet基因为起始基因,因此将补齐后的原始序列的前端1826 bp截取到序列尾部形成一个新序列,即序列调零。
Table 2. The tRNA genes were searched on tRNAscan-SE
表2. 在tRNAscan-SE上搜索得到的tRNA基因
3.1.2. tRNA二级结构
tRNA的二级结构一般由氨基酸接受臂、TΨC臂、TΨC环、反密码子臂、反密码子环、二氢尿嘧啶臂、二氢尿嘧啶环,及一个多变的额外环组成(见图2),有时某些tRNA会缺少二氢尿嘧啶臂。通常,这些tRNA都能形成倒L型的三级结构,一般反密码子臂和氨基酸接受臂比较保守,而二氢尿嘧啶臂、TΨC臂变异性较高,存在一些错配的情况。本文统计了搜索到的15个tRNA的二级结构(见图3)。

Figure 2. Schematic diagram of secondary structure of tRNA
图2. tRNA二级结构模式图


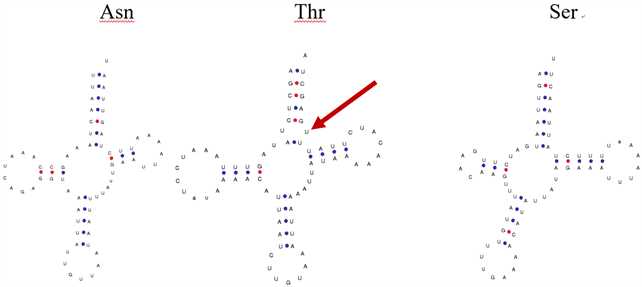

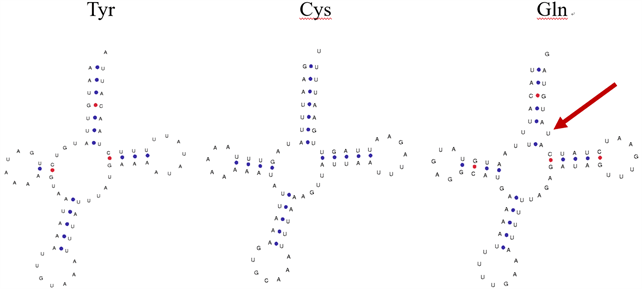
Figure 3. The secondary structure of the tRNA genes were searched on tRNAscan-SE
图3. 在tRNAscan-SE上搜索得到的tRNA基因二级结构
由图3可知,tRNAIle、tRNAThr、tRNAGln基因的氨基酸接受臂均发生了U-U错配(红色箭头标出)。
3.1.3. 相近种的线粒体基因组全序列获取及比对
为定位其它的tRNA基因、蛋白质基因和rRNA基因,从NCBI网站上用BLAST功能进行相似性搜索并选择相似性最高的序列。本研究从NCBI中选择了编号KT266831的双齿多刺蚁Polyrhachis dives的线粒体基因组全序列作为参考,用ClustalX(1.83)软件将其蛋白质基因、rRNA基因和7个tRNA基因(tRNAGly、tRNAAla、tRNAArg、tRNASer(TCT)、tRNAGlu、tRNALeu、tRNAval)截取保存。然后将西藏粗角猛蚁调零后的线粒体NDA序列导入ClustalX(1.83)软件中,再分别添加之前保存的双齿多刺蚁各基因序列进行比对,从而逐个确定西藏粗角猛蚁线粒体基因组其它的基因位置和序列,并最终得到其线粒体基因组注释结果(见表3)。
Table 3. The mitochondrial genome annotation of Cerapachys xizangensis
表3. 西藏粗角猛蚁线粒体基因组注释
3.1.4. 蛋白质基因翻译结果
西藏粗角猛蚁的13个蛋白质基因均是典型的ATN起始密码子,大多数蛋白基因以完全终止密码子ATT结尾,只有ND2基因以T,Cytp基因以TA作为不完全终止密码子,而ND6以TAG作为终止密码子。本文用MEGA4.1软件选择invertebrate mitichondrial密码子对13种蛋白质进行翻译(见表4)。
Table 4. Translation results of protein gene
表4. 蛋白质基因翻译结果
3.2. 蛋白质编码基因相对同义密码子使用
蛋白质编码基因相对同义密码子使用(Relative Synonymous Codon Usage, RSCU),是指对于某一特定的密码子在编码对应氨基酸的同义密码子间的概率,它除去了氨基酸组成对密码子使用的影响。理论上,如果没有选择压力,所有同义密码子的使用频率应该是相同的,即RSCU应该等于1,当某一密码子的RSCU大于1时,说明该密码子为使用较多的密码子,反之亦然。本研究用MEGA4.1软件统计了13种蛋白质基因的序列(终止密码子除外),总共10,938 bp,然后计算了它们的RSCU (见表5)。
Table 5. Frequency statistics of relative synonymous cod on usage
表5. 相对同义密码子使用频率统计
3.3. 基因重排
果蝇Drosophila yakuba为第一个被测序的昆虫线粒体基因组 [16] ,一般将果蝇的线粒体基因排列作为昆虫线粒体基因的模式排列(见图4)。随着分子生物学研究的逐步深入与成熟,越来越多的昆虫线粒体基因组被测序,基因重排事件也不断被发现,其中以tRNA基因重排为主,同时还存在着基因的重复 [17] - [22] 。基因重排事件主要有四种形式包括基因移位、基因洗牌、倒置和异位倒置,其中CR-ND2、ND2-COI、COII-ATP8、ND3-ND5是基因重排的热点区域 [23] [24] 。本研究中的西藏粗角猛蚁发生两处基因重排,即发生在CR-ND2区的基因洗牌和ND3-ND5区的基因倒置(见图5)。

Figure 4. Original arrangement of insect mitochondrial genome (speculation)
图4. 昆虫线粒体基因组原始排列(推测)

Figure 5. Gene rearrangement schematic diagram
图5. 基因重排示意图
4. 总结与讨论
本文测定了西藏粗角猛蚁的线粒体基因组全序列,丰富了我国蚁科昆虫的基因数据,还创造性地改进了环状DNA缺口处的引物设计方法,克服了昆虫线粒体基因组高A + T含量造成的扩增困难。通过注释和基因位置的分析,发现了两处基因重排现象。本研究发现的tRNA基因U-U错配问题,可能是因为测序误差而引起,也可能是因为基因突变,或者本身就是如此。这个问题还有待进一步的研究。
致谢
湖南师范大学博士研究生黄超洋为本实验引物设计提出了很好的建议,广西师范大学研究生唐祺玲介绍实验方法和数据分析软件,在此谨表谢意!
基金项目
国家自然科学基金项目(31672343)和科技基础性工作专项重点项目(2015FY210300)资助。
文章引用
钟 烨,周善义,陈志林. 西藏粗角猛蚁线粒体基因组序列分析
Mitochondrial Genome Sequence Analysis of Cerapachys xizangensis[J]. 农业科学, 2019, 09(02): 114-125. https://doi.org/10.12677/HJAS.2019.92020
参考文献
- 1. Boore, J.L. (1999) Animal Mitochondrial Genomes. Nucleic Acids Research, 27, 1767-1780. https://doi.org/10.1093/nar/27.8.1767
- 2. Wang, Y., Chen, J., Jiang, L.Y., et al. (2015) Hemipteran Mitochondrial Genomes: Features Structures and Implications for Phylogeny. International Journal of Molecular Sciences, 16, 12382-12404. https://doi.org/10.3390/ijms160612382
- 3. Amaral, D.T., Mitani, Y., Ohmiya, Y., et al. (2016) Organization and Comparative Analysis of the Mitochondrial Genomes of Bioluminescent Elateroidea (Coleoptera: Polyphaga). Gene, 586, 254-262. https://doi.org/10.1016/j.gene.2016.04.009
- 4. Beagley, C.T., Okada, N.A. and Wolstenholme, D.R. (1996) Two Mitochondrial Group I Introns in a Metazoan, the Sea Anemone Metridium senile: One Intron Contains Genes for Subu-nits 1 and 3 of NADH Dehydrogenase. Processdings of the National Academy of Sciences, 93, 5619-5623. https://doi.org/10.1073/pnas.93.11.5619
- 5. Fukami, H., Chen, C.A., Chiou, C.Y., et al. (2007) Novel Group I Introns Encoding a Putative Homing Endonuclease in the Mitochondrial Cox1 Gene of Scleractinian Corals. Journal of Molecular Evolution, 64, 591-600. https://doi.org/10.1007/s00239-006-0279-4
- 6. Simon, C., Frati, F., Beckenbach, A., et al. (1994) Evolution, Weighting, and Phylogenetic Utility of Mitochondrial Gene Sequences and a Compilation of Conserved Polymerase Chain Reaction Primers. Annals of the Entomological Society of America, 87, 651-701. https://doi.org/10.1093/aesa/87.6.651
- 7. Cameron, S.L., Dowton, M., Castro, L.R., et al. (2008) Mitochondrial Genome Organization and Phylogeny of Two Vespid Wasps. Genome, 51, 800-808. https://doi.org/10.1139/G08-066
- 8. Gotzek, D., Clarke, J. and Shoemaker, D. (2010) Mitochondrial Genome Evolution in Fire Ants (Hymenoptera: Formicidae). BMC Evolutionary Biology, 10, 1-13. https://doi.org/10.1186/1471-2148-10-300
- 9. Hasegawa, E., Kobayashi, K. and Yagi, N. (2011) Complete Mito-chondrial Genomes of Normal and Cheater Morphs in the Parthenogenetic Ant Pristomyrmex punctatus (Hymenoptera: Formicidae). Myrmecological News, 15, 85-90.
- 10. Berman, M., Austin, C.M. and Miller, A.D. (2014) Characterisa-tion of the Complete Mitochondrial Genome and 13 Microsatellite Loci through Next-Generation Sequencing for the New Caledonian Spider-Ant. Leptomyrmexpallens. Molecular Biology Reports, 41, 1179-1187. https://doi.org/10.1007/s11033-013-2657-5
- 11. de Melo Rodovalho, C., Lyra, M.L. and Ferro, M. (2014) The Mitochondrial Genome of the Leaf-Cutter Ant Atta laevigata: A Mitogenome with a Large Number of Intergenic Spacers. PLoS ONE, 9, 1-9.
- 12. Babbucci, M., Basso, A., Scupola, A., et al. (2014) Is It an Ant or a Butterfly? Convergent Evolution in the Mitochondrial Gene Order of Hymenoptera and Lepidoptera. Genome Biology and Evolution, 6, 3326-3343. https://doi.org/10.1093/gbe/evu265
- 13. Kim, M.J., Hong, E.J. and Kim, I. (2015) Complete Mitochondrial Ge-nome of Camponotus atrox (Hymenoptera: Formicidae): A New tRNA Arrangement in Hymenoptera. Genome, 59, 59-74. https://doi.org/10.1139/gen-2015-0080
- 14. Yang, S., Li, X., Cai, L.G. and Qian, Z.Q. (2016) Characteriza-tion of the Complete Mitochondrial Genome of Formica selysi (Insecta: Hymenoptera: Formicidae: Formicinae). Mito-chondial DNA, 27, 3378-3380. https://doi.org/10.3109/19401736.2015.1018229
- 15. 唐觉, 李参. 西藏昆虫[M]. 北京: 科学出版社, 1982: 371-374.
- 16. Clary, D.O. and Wolstenholme, D.R. (1985) The Mitochondrial DNA Molecule of Drosophila yakuba: Nucleotide Sequence, Gene Organization and Genetic Code. Journal of Molecular Evolution, 22, 252-271. https://doi.org/10.1007/BF02099755
- 17. Crozier, R.H. and Crozier, Y.C. (1993) The Mitochondrial Genome of the Honeybee Apis mellifera: Complete Sequence and Genome Organization. Genetics, 133, 97-117.
- 18. Szymura, J., Lunt, D. and Hewitt, G. (1996) The Sequence of the Meadow Grasshopper (Chorthippus parallelus) Mitochondrial srRNA, ND2, CO1, CO2, ATPase8 and 9 tRNA Genes. Insect Molecular Biology, 5, 127-139. https://doi.org/10.1111/j.1365-2583.1996.tb00047.x
- 19. Dowton, M., Belshaw, R., Austin, A.D., et al. (2002) Simultaneous Molecular and Morphological Analysis of Braconid Relationships (Insecta: Hymenoptera: Braconidae) In-dicates Independent mt-tRNA Gene Inversions within a Single Wasp Family. Journal of Molecular Evolution, 54, 210-226. https://doi.org/10.1007/s00239-001-0003-3
- 20. Dowton, M., Stephen, L., Cameron, J., et al. (2009) Characterization of 67 Mitochondrial tRNA Gene Rearrangements in the Hymenoptera Suggests That Mitochondrial tRNA Gene Position Is Selectively Neutral. Molecular Biology and Evolution, 26, 1607-1617. https://doi.org/10.1093/molbev/msp072
- 21. Tay, W.T., Elfekih, S., Court, L., et al. (2014) Complete Mitochondri-al DNA Genome of Bemisiatabaci Cryptic Pest Species Complex Asia II-5 (Hemiptera: Aleyrodidae). Mitochondial DNA, 27, 1-2.
- 22. Nishimoto, M., Okuyama, H., Kiyoshi, T., et al. (2017) Complete Mitochondrial Genome of the Japanese Bumblebee, Bombus Hypocrita Hypocrita (Insecta: Hymenoptera: Apidae). Mitochondrial DNA Part B, 2, 19-20. https://doi.org/10.1080/23802359.2016.1275849
- 23. Dowton, M. and Austin, A.D. (1999) Evolutionary Dynam-ics of a Mitochondrial Rearrangement “Hot Spot” in the Hymenoptera. Molecular Biology and Evolution, 16, 298-309. https://doi.org/10.1093/oxfordjournals.molbev.a026111
- 24. Dowton, M., Castro, L.R., Campbell, S.L., et al. (2003) Frequent Mitochondrial Gene Rearrangement at the Hymenopteran nad3-nad5 Junction. Journal of Molecular Evolution, 56, 517-526. https://doi.org/10.1007/s00239-002-2420-3
NOTES
*通讯作者。
