Computer Science and Application
Vol.06 No.06(2016), Article ID:17874,8
pages
10.12677/CSA.2016.66045
Generating AUTOLISP Code to Achieve the Diagram Drawing of Variable ID3 Genotyping Decision Tree Classifier by Compiling of MATLAB
Hongbin Li, Guangzhong He
Medical School, Xianyang Vocational and Technical College, Xianyang Shaanxi

Received: Jun. 14th, 2016; accepted: Jun. 24th, 2016; published: Jun. 28th, 2016
Copyright © 2016 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



ABSTRACT
Decision tree classifier is a kind of classification algorithm based on examples, which is widely used in the field of artificial intelligence. ID3 algorithm is the most classical decision tree construction algorithm. It constructs a decision tree by recursion, and the selection of the attribute which contains the most amount of information. The structure of one decision tree classifier sometimes is very large and complex, decision tree classification diagram is very intuitive, and some key information can be mined from the original data set, so the decision tree diagram drawing is very necessary. This study give us an example on genotyping decision tree drawing classifier in the domain of molecular biology, and talk about how to use the MALAB language compiler to generate AUTOLISP code, so as to achieve diagram drawing of a variable ID3 genotyping decision tree classifier.
Keywords:ID3 Decision Tree Classifier, Drawing Diagram, MATLAB, AUTOLISP

MATLAB编译生成AUTOLISP代码实现可变ID3基因分型决策树分类图的绘制
李宏彬,赫光中
咸阳职业技术学院医学院,陕西 咸阳
Email: leehbin@126.com
收稿日期:2016年6月14日;录用日期:2016年6月24日;发布日期:2016年6月28日

摘 要
决策树分类器,是一种基于实例的分类算法,广泛被应用于人工智能领域。ID3算法是最为经典的决策树建树算法,它通过递归和逐次挑选信息量最多的属性来构造决策树。决策树的结构有时非常庞大和复杂,而决策树分类图看起来非常直观,并且可以从建树的原始数据集中挖掘出一些关键的信息,因此决策树图的绘制是非常必要的。本研究从分子生物学领域中的基因分型决策树绘制为实例,浅谈如何使用MALAB语言编译生成AUTOLISP代码,从而实现可变ID3基因分型决策树分类图的绘制。
关键词 :ID3决策树,图的绘制,MATLAB,AUTOLISP

1. 引言
决策树分类器(decision tree classifier, DTC),简称决策树,是一种基于实例的归纳分类算法。它专注于从一些松散、零乱的事例中概括出树形式的分类规则。它选用从上到下的递归方法,在决策树的中间节点同其属性值进行比较,并根据对应的属性值选择向下的分支,最终在决策树的末端(叶节点)得到分类结论。由于每项决策都可能延伸出两个以上的选项,引出不同的推论,其外形类似树的枝干,故命名为决策树。决策树是使用最广泛的非参数分类方法,其基本思想是将一个复杂的决策分解成若干个简单的决策。相对其他人工智能算法,决策树有以下特点:决策树拥有最直观的知识表达,易于被人们理解和接受;决策树采用非参数方式,不需要使用者干预,非常适用于知识发现;决策树算法能够进行自我调整,当训练数据样本量突增后分类性能不会大幅度下降。目前,决策树分类器以其出色的分类性能,已广泛应用于人工智能领域,如数据挖掘、模式识别和图像分析等。
1.1. 决策树学习算法
决策树诞生于1966年Hunt等人提出的CLS学习算法 [1] ,它是早期的决策树学习算法。Quinlian于1979提出处理离散取值的ID3算法,和于1993 提出离散和连续取值均可处理的C4.5算法,以及上世纪八十年代初提出的基于二叉树的快速算法C5.0算法标志着决策树走向成熟 [2] - [6] 。下面我们以算法明确和学习能力强的ID3算法为例,看决策树是如何实现分类的。
ID3算法:
Procedure ID3 (Node n, Instances X, Feature f)
1) Best entropy = 1
2) for each feature f i inf
3) Calculation its conditional entropy H(X/f i)
4) if H(X/f i) < Best entropy
5) Best entropy= H(X/f i)
6) Best feature= f i
7) end if
8) end for
9) get the number of all possible values of feature Bestfeature, i.e. k
10) if k = 1
11) end procedure
12) else
13) Partition instance X into k subset
14) Split Node n into k child nodes
15) Call ID3 for each child node
16) end if
ID3算法是最为经典的分类决策树学习算法,ID3的基本思想是以信息熵即信息不确定性为度量,用于决策树节点的属性选择的判定条件,每次挑选信息量最多的属性,即能使熵值成为最小的属性来构造决策树,常用的判定方法有最小条件熵法(平均信息期望)或最大信息增益法。
1.2. 基因分型简介
随着生命科学研究的快速发展,大量的物种被测序,越来越多的物种基因的变种序列被发现,因此用于核酸序列功能研究的基因分型的任务量急剧增加。基因分型,或基因型分析,是指利用分子生物学实验方法确定样本基因型的技术。PCR-SSP (sequence specific primer)和PCR-SBT (sequence based typing)是目前最常用的基因分型技术,已广泛应用于遗传和法医领域。PCR-SBT是一种灵活可变的高分辨率的测序后使用软件进行基因分型的技术。同其它基因分型相比较,它的分型多态性位点不需要预先固定,人们通过分型不仅能识别已有等位基因,而且还能发现新的等位基因。PCR-SBT基因分型的一般技术路线是模板制备、PCR反应、DNA测序、外显子剪接、数据库比较。纯合子经简单的比较就可分型,杂合子分型需将目标序列在杂合位点的IUPAC码转化为各种可能的纯合子的组合序列对,并对可能组合对序列单独分型。然而通常的同数据库比对PCR-SBT基因分型需要占用大量的机器时间,本研究发现使用基于SNP (single nucleotide polymorphism)的决策树分类器可以使比较判断的次数大幅度降低从而显著提高基因分型的效率。
2. 材料和方法
某些简单的基因分型决策树可以用手工方法绘制,首先用多序列比对确定分型差异SNP位点,然后使用这些差异位点建立决策树,如图1所示。然而对于某些高度多态的基因如位于人类第六号染色体主要组织相容性复合体(MHC)区域的,拥有数十甚至成百上千个SNP的人类白细胞抗原基因(HLA)使用手工绘制分型决策树是困难的。这些基因执行关键的抗原提呈功能。现代医学研究发现,捐献者和接受者之间HLA基因的精确配型可以减少免疫并发症从而显著地提高器官移植的存活率,尤其是骨髓移植 [7] 。另外,HLA分型的结果经常被用于重要的法医学证据,例如亲子鉴定和犯罪鉴定。根据基因分型的特点,本研究选择侧重于离散值属性(核酸碱基“A”、 “T”、 “C”、 “G”及插入缺失位“-”)的ID3算法进行基因分型。当含有已知等位基因编码区序列的数据库建立以后,基因分型的第一步是等位基因差异位点特征SNP序列的构建,构建方法是采用多序列比对软件如BIOEDIT对包含各已定义等位基因的序列集进行多序列比对,如图2所示。
多序列比对后,SNP位点的碱基状态提取方法是,如果多序列比对文件某列数目占优的碱基数不等于多序列比对的序列数,则该列为一个SNP,从而可以通过MATLAB程序提取各序列在该SNP位点的

Figure 1. The building of genotyping decision tree
图1. 基因分型决策树的建立
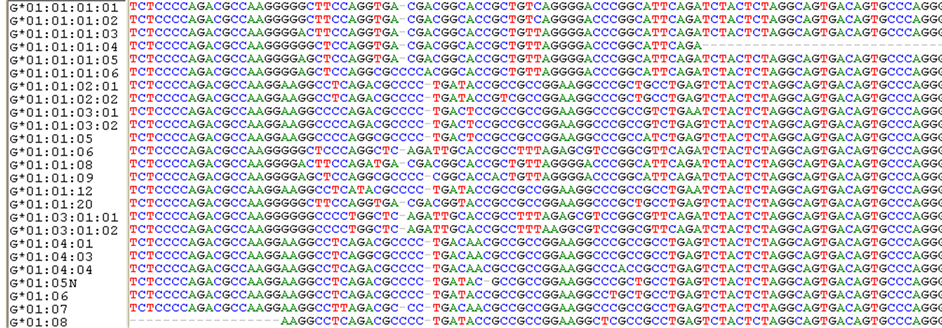
Figure 2. Multiple sequence alignment of allelic genotyping sequence (Gene HLA-G)
图2. 等位基因分型序列的多序列比对(基因HLA-G)
碱基模式。将各SNP位各序列的碱基模式及在比对文件中的列位置写入一个二维结构数组中(数组的行对应序列,列对应SNP位),将各序列的分类名称写在该结构数组的尾部, 这样等位基因差异位点特征SNP序列的构建已经完成。这些SNP位置相当于一个一个的属性,可以采用ID3算法依次选取条件熵最小的属性作为测试属性进行子树划分。倘若通过实验方法获得了一个样本各分型定义SNP位置的碱基状态数据,如SNP1 = “A”, SNP2 = “T”, SNP3 = “G”...... SNPN = “G”,如果想对该样本进行基因分型,可以通过同分型决策树比较来判断输入SNP位点序列的基因类别,若它是一个新发现的基因型,程序将提示第一个差异SNP的位置即碱基基因型。
基因分型决策树图的绘制
在MATLAB workspace中虽然可以浏览建立好的基因分型决策树,但仍旧不完全直观,因此基因分型决策树图的绘制是必要的。MATLAB中二维图形绘制仅限于简单的曲线,复杂图型的绘制是困难的,因此必须寻找其他可行的替代方式。LISP是美国科学家MacCarthy于上世纪五十年代创立的并广泛应用于人工智能领域的程序设计语言。Autodesk公司将LISP语言嵌于计算机辅助设计软件AUTOCAD内部,命名为AUTOLISP [8] ,将LISP语言和AUTOCAD各自的特点有机结合,使AUTOLISP即拥有一般高级语言的编程功能,又具备一般高级语言所欠缺的强大的图形功能。基因分型决策树的绘制基本思路是使用MATLAB发挥其科学计算的潜能完成决策树各节点(根节点、中间节点和叶节点)的节点属性参数计算、节点在图面的位置确定和AutoLISP代码的生成,然后在AUTOCAD中运行AUTOLISP代码完成最终的基因分型决策树的绘制。节点属性参数计算上一节已经涉及,下面介绍节点的位置确定算法。节点的图面位置确定算法有自顶向下递推和自底向上递推两种算法。自顶向下递推法是首先确定根节点的位置,递归确定根节点以下节点的位置,并通过树的自顶向下、先子后兄、从左到右的遍历将决策树转化为串联绘图节点队列,每个节点属性除原决策树节点属性外增加父节点坐标和本节点坐标四个属性参数,并依次绘制节点信息和进行父子连线。自顶向下进行决策树绘图的缺点是,当决策树的层数n增加后,幅面宽度wide成指数增加即:
 (1)
(1)
对于复杂的决策树若不压缩,即取接近5的值(对应最多五个分支),会出现幅面宽度过大超出AUTOCAD的允许范围而溢出的状况。若压缩,即取大于并接近1的值,有可能出现子树间隙小而交叠的情况如图3所示。
由于决策树的叶节点是有限的,如果幅面宽度正比于叶节点数目,节点的父节点的横坐标由子节点横坐标的均值决定而纵坐标由所处的层决定则可避免自顶向下递推法所产生的过宽或交叠的现象,称该算法为自底向上算法,自底向上需要以下步骤实现:
通过递归计算决策树的深度。首先将深度设置为1,通过递归计算各子树深度,如果子树的深度大于目前深度,则将其设置为决策树的深度,直到遍历完整个决策树。
将决策树按从自顶向下、先子后兄、从左到右的遍历将决策树转化为串联节点队列,串联节点的属性除原决策树节点属性外增加各节点的父节点属性。
扫描串联节点队列中叶节点,将叶节点总数设置为决策树的宽度,按从左到右的次序计算各叶节点位置横坐标值,纵坐标依据叶节点所在的层进行计算。
决策树节点的属性增加横、纵坐标属性,然后将串联节点的位置坐标数据反抄到决策树上,位置属性未设置的节点先将上述属性值置空。
通过递归自底向上、先子后兄、从左到右计算决策树各中间节点的位置属性。
向决策树补充各节点的父节点连线位置参数
决策树的完整位置信息转化为串联绘图队列数据输出
编译生成AutoLISP代码并存盘

Figure 3.Top-down compression may cause overlapping of subtrees
图3. 自顶向下压缩可能导致子树交叠
决策树是由节点图元和连线组成,如图4所示,图元是由框架部分(轮廓线)、连线和变动信息部分,即属性参数,包括位置(px, py)、父节点连线位置(xparents, yparents)、决策基因型(genetype)、SNP序号(colpos)、标识(data:无错误时当节点为叶节点时显示最终分类名,中间节点不显示;有错误则显示“error”)及SNP在原始多序列比对文件中的列位置(posinMSAfile),由于前面的程序已经将决策树的各节点生成绘图信息队列,下面需要做的是将这些信息转化为AUTOLISP代码,节点图元的绘图指令是一个含有8个参数的函数:(draw (list xparentsyparents) (list pxpy) “genetype” “colpos” “posinMSAfile” “data”),该函数以位置参数点(px, py)为基点绘制图元的框架部分,并连线基点(px, py)和父节点连线位置点(xparents, yparents),然后绘制变动信息基因型(genetype)、SNP序号(colpos)、基因型标识(data)及SNP在原始多序列比对文件中的列位置(posinMSAfile)。
EBI IMGT/HLA 数据库提供了人类主要组织相容性复合体系统(HLA)的的专业化数据库,本研究从该数据库中下载了多态性的MHC基因HLA-G、HLA-A、HLA-B (等位基因数分别为25、1729和2329)的序列数据,进行决策树分析,并绘制了基因分型决策树,经决策树分析后,节点数分别为46、3280和4407,如图5、图6(a)和图6(b)所示。高度多态基因HLA-A和HLA-B分型决策树非常庞大复杂,论文只显示轮廓,可在AUTOCAD中观察细节。基因分型决策树提供了一种通过与决策树比较进行基因分型的便捷途径,体现了基因各等位基因间DNA序列的直观的和最细致的共性和差异关系。共性是指等位基因间相似点如同属一个父族类或超族类、在哪些SNP位点基因型相同;而差异指等位基因间分属不同的族类或亚族类,在哪些SNP位点基因型不同,因此决策树可以用于多态基因的分类或聚类,HLA-A和HLA-B的决策树分类结果如表1和表2所示,它们的一级分类SNP在多序列比对文件中分别位于98和391。

Figure 4. The design of basic graphic element (node)
图4. 绘图基本图元(节点)设计
Table 1. Genotyping of gene HLA-A by DTC
表1. 基因HLA-A的决策树分型结果
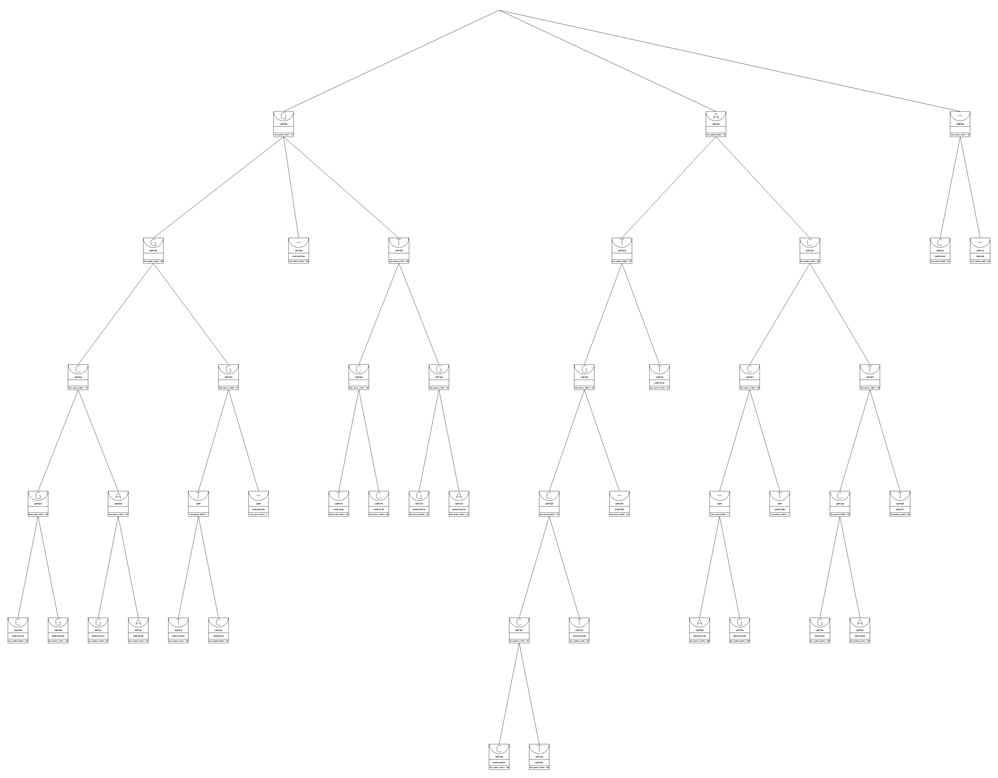
Figure 5. The genotyping decision tree of polymorphic gene HLA-G
图5. 多态基因HLA-G的基因分型决策树
 (a) HLA-A
(a) HLA-A (b) HLA-B
(b) HLA-B
Figure 6. The genotyping decision tree of polymorphic gene HLA-A and HLA-B
图6. 多态基因HLA-A和HLA-B的基因分型决策树
Table 2. Genotyping of gene HLA-B by DTC
表2. 基因HLA-B的决策树分型结果
3. 结论
本研究将ID3决策树建树算法应用到基因分型过程中,实现了建树、分型、识别新的基因型、决策树绘制等功能。该决策树可以用于多态基因的分类或聚类,还可以用来设计引物以构建聚合链式反应(PCR)实验,或设计基因分型试剂盒。本研究中作者构建了自底向上的决策树数字化程控绘图策略,使用MATLAB发挥其科学计算的潜能完成决策树各节点的属性参数计算、节点在图面的位置确定和AUTOLISP代码的生成,然后在AUTOCAD中运行AUTOLISP代码完成最终的基因分型决策树的绘制,实现了可变决策树分类图的程控生成,同时解决了MATLAB复杂图形绘制困难的问题。
文章引用
李宏彬,赫光中. MATLAB编译生成AUTOLISP代码实现可变ID3基因分型决策树分类图的绘制
Generating AUTOLISP Code to Achieve the Diagram Drawing of Variable ID3 Genotyping Decision Tree Classifier by Compiling of MATLAB[J]. 计算机科学与应用, 2016, 06(06): 368-375. http://dx.doi.org/10.12677/CSA.2016.66045
参考文献 (References)
- 1. 刘圣财. 基于决策树分类算法的研究与应用[D]: [硕士学位论文]. 长春: 长春理工大学, 2012.
- 2. Hunt, K.J. (1992) Induction of Decision Trees for Rule-Based Modelling and Control. Proceedings of the 1992 IEEE International Symposium on Intelligent Control, Glasgow, 11-13 August 1992, 306-311. http://dx.doi.org/10.1109/ISIC.1992.225108
- 3. Quinlan, J.R. (1979) Discovering Rules by Induction from Large Collections of Examples. Expert Systems in the Micro Electronic Age. Edinburgh University Press, 1979.
- 4. Quinlan, JR. (1986) Induction of Decision Trees. Machine Learning, 1, 81-106. http://dx.doi.org/10.1007/BF00116251
- 5. Quinlan, JR. (1996) Improved Use of Continuous Attributes in c4.5. Journal of Artificial Intelligence Research, 4, 77- 90.
- 6. Breiman, L., Friedman, J., Olshen, R., et al. (1984) Classification and Regression Trees. Chapman & Hall (Wadsworth, Inc.), New York.
- 7. Persijn, G.G., Cohen, B., Lansbergen, Q., et al. (1982) Effect of HLA-A and HLA-B Matching on Survival of Grafts and Recipients after Renal Transplantation. The New England Journal of Medicine, 307, 905-908. http://dx.doi.org/10.1056/NEJM198210073071501
- 8. 郭秀娟, 于全通, 范小殴, 主编. Autolisp语言程序设计[M]. 北京: 化学工业出版社, 2008.
