 Advance in Microbiology 微生物前沿, 2013, 2, 32-36 http://dx.doi.org/10.12677/amb.2013.21007 Published Online March 2013 (http://www.hanspub.org/journal/amb.html) Development of Genomic Structure and Function of Hepatitis E Virus* Yaxin Lei, Peng Liu, Ling Wang# Department of Microbiology and Center of Infectious Disease, Peking University Health Science Center, Beijing Email: #lingwang@bjmu.edu.cn Received: Feb. 20th, 2013; revised: Mar. 13th, 2013; accepted: Mar. 20th, 2013 Abstract: Hepatitis E virus (HEV) is one of the most important pathogens of acute viral hepatitis prevailing in developing countries. There are four major genotypes of mammalian HEV, genotype 1 and 2 are restricted to human infection, and induce epidemic hepatitis E which is mainly transmitted b y contaminated water; whereas genotype 3 and 4 are zoonotic causing sporadic cases which are common in developed countries. In recent years, much more attention has been paid to the structures of viral genes and the functions of viral proteins. The research progress achieved in such field will help us uncover the veil of HEV in pathogenesis and infection. Keywords: Hepatitis E Virus (HEV); Gene Structure; Open Reading Frame (ORF); Cis-Reactive Elements (CRE) 戊型肝炎病毒的基因组结构及生物学功能研究进展* 雷雅昕, 刘 鹏, 王 玲# 北京大学医学部病原生物学系和感染病中心,北京 Email: #lingwang@bjmu.edu.cn 收稿日期:2013 年2月20 日;修回日期:2013 年3月13 日;录用日期:2013 年3月20 日 摘 要:戊型肝炎病毒(HEV)是发展中国家引起急性病毒肝炎的重要病原体之一。HEV 有4个主要基因型,1 型和 2型只感染人,通过污染的水源引起暴发流行;3型和 4型属于人畜共患病原体,常引起散发病例。为进 一步阐明 HEV 的致病和跨种系传播机理,本文就近年来在 HEV 基因及其蛋白质的生物学特性和功能研究方面 取得的进展进行综述。 关键词:戊型肝炎病毒;基因结构;开放读码框;顺反应元件 1. 引言 戊型肝炎病毒(Hepatitis E virus, HEV)是1983 年 前苏联学者 Balayan 首次用免疫电镜观察到的病毒颗 粒[1],1989 年正式将其命名为 HEV。目前 HEV 有1、 2、3、4型和禽、鼠、兔 HEV。1型和 2型HEV 经粪 口途径传播,主要感染人类,导致流行性戊型肝炎; 3型和 4型HEV既可以感染人,也可以感染动物,猪 是主要动物宿主[2]。禽和鼠 HEV 不感染人,兔 HEV 可跨种系传播[3]。近年来不断有报道表明 3型和 4型 HEV 可使某些感染者出现慢性肝炎、肝硬化,慢性肝 病患者可出现急性肝衰竭[4]并进展为晚期肝病[5]。为 深入了解 HEV 的致病和跨种系传播机理,各国学者 对HEV 分子结构、蛋白质的生物学特性与功能进行 了更加深入的研究,并取得了一些新的进展,现综述 如下。 2. HEV基因组结构 *基金项目:国家自然科学基金资助项目(81271827);国家高校博士 点基金资助课题(20120001110098)。 #通讯作者。 HEV 是单股正链 RNA 病毒,哺乳动物的 HEV Copyright © 2013 Hanspub 32  戊型肝炎病毒的基因组结构及生物学功能研究进展 基因组全长约为 7.2~7.6 kb,而禽类 HEV 约为 6.7 kb[6]。HEV 的基因组结构主要由 5’端的非结构区(NS) 和3’端结构区(S)组成。编码三个开放读码框(open reading frame, ORF),其中 ORF1编码非结构蛋白[7], ORF2 编码衣壳蛋白[8],ORF3编码多功能的小蛋白[9]。 5’端和 3’端各有一个非编码区(NCR)。5’端有 7甲基 鸟甘酸帽结构,3’端含有150~200 个腺苷酸残基组成 的多聚腺苷(poly A)尾,详见(图1)[10]。 HEV基因组中的连接区域(Junction Region, JR) 将ORF1与ORF2、ORF3 分隔。两个顺反应元件(CRE) 均为茎环结构,第一个CRE 与3’末端的 ORF2及非 编码区重合;第二个 CRE 在JR区。 3. HEV的蛋白及其功能 3.1. 非结构蛋白(Non-Structural Proteins, NSPs) HEV的非结构蛋白由ORF1 编码。在三个开放读 码框中 ORF1最长,它起始于 5’端非编码区的末端, 共有 5082 个碱基。ORF1 可以编码一个含有1693 个 氨基酸的多肽链,参与病毒的复制和蛋白质加工[11]。 ORF1可以被分为若干个功能区段[7],包括甲基转移 酶区(methyltransferase, MT)、半胱氨酸蛋白酶区 (papain-like cysteine proteases, PCP)、高变区(hyper- variable region, HVR)、解旋酶区(helicase, Hel)、RNA 依赖的RNA 聚合酶区(RNA-dependent RNA poly- merases, RdRp)和X、Y区。其中 X、Y区的功能尚不 清楚。 3.1.1. MT区 MT 区编码甲基转移酶,参与5’端帽状结构的形 成[12]。5’端的帽状结构对于病毒侵染非人灵长类动物 至关重要,Emerson[11]等人的实验表明无帽状结构的 RNA 不能使黑猩猩发病或产生抗体。重组的HEV 解 图中标示三个开放读码框。MT:甲基转移酶;Y:Y区;PCP:木瓜蛋白酶 样的半胱氨酸蛋白酶;HVR:高变区;X:X区;Hel:解旋酶;RdRp:RNA 依赖的 RNA聚合酶;CRE:顺反应元件;SL:茎环结构;JR:连接区。 Figure 1. A schematic diagram of the HEV genome 图1. HEV基因组结构 旋酶可以催化帽状结构形成的第一步反应[13],因此编 码解旋酶的 Hel 区基因可能也参与了 5’端帽状结构的 形成。 3.1.2 Hel区 Hel 区编码解旋酶。解旋酶分为 6个超家族,SF1- SF6。SF1 和SF2 家族成员最多,它们都有7个标志 性模体(motif):I,Ia,II,III,IV,V,and VI。在大 肠杆菌中表达的 HEV 解旋酶具有双重解旋的活性, 既可以解旋 RNA(5’端至 3’端),又可以水解核酸。其 中motifⅠ或 motif Ⅱ的突变都会使解旋酶失去双重解 旋活性[14]。 3.1.3 PCP区 PCP 区编码半胱氨酸蛋白酶。在牛痘病毒表达系 统中,ORF1编码107 kDa和78 kDa的两种蛋白[15]。 在杆状病毒中,ORF1 编码很多小蛋白,加入半胱氨 酸蛋白酶抑制物后产物减少[16],但是对于 ORF1编码 蛋白的加工仍然缺乏直接的证据。此外,HEV的MT 与PCP 的重组酶具有去泛素化作用[17]。 3.1.4. Rd Rp区 RdRp 区合成RNA 依赖的 RNA聚合酶。HEV的 RdRp 可以结合到3’端的非编码区,以带有 Poly A尾 的HEV RNA做模板,在大肠杆菌中合成病毒 RNA[18]。RdRp 的表达定位于真核细胞的内质网,而 衣壳蛋白也在内质网中组装[19],因此,内质网是病毒 复制的场所之一。 3.1.5. HVR区 HVR 区富含脯氨酸。与 X区的 N端和 PCP 区的 C端富含脯氨酸的序列重合[7]。HVR 的长度和序列组 成在不同的 HEV 毒株中会有变化,但是其微小改变 对HEV的侵染性没有影响[20]。就病毒的复制和侵染 性而言,HVR 在HEV 四种基因型中具有相似的功能, 但是也有实验表明 HVR 对复制效率的影响具有基因 型特异性[21],所以,尽管 HVR的微小改变并不影响 病毒的侵染性,却可能通过与病毒或宿主的作用而改 变HEV 复制效率。 3.2. 衣壳蛋白(Capsid Protein) HEV的衣壳蛋白由ORF2 编码,含有 660 个氨基 酸,分子量为 72 kDa。其多肽序列富含精氨酸并有三 Copyright © 2013 Hanspub 33  戊型肝炎病毒的基因组结构及生物学功能研究进展 个N-糖基化位点[22],体外表达的衣壳蛋白大小不一 [23]。 3.2.1. 衣壳蛋白参与病毒的装配和与宿主的反应 N端的信号肽将衣壳蛋白引导至内质网,其中的 一部分会再转移至胞质[24]。在哺乳细胞中表达的衣壳 蛋白大部分会被糖基化,糖基化位点的变异可抑制侵 染性病毒颗粒的形成[25]。衣壳蛋白可能通过与病毒 RNA 5’端的包装信号序列结合进行病毒颗粒的装配 [26]。实验表明热休克蛋白 90(HSP90)[27]和葡萄糖调节 蛋白 78(Grp78)[28]参与病毒颗粒的胞内转运;硫酸肝 素糖蛋白(HSPGs)可能作为粘附受体促进HEV进入 Huh7 细胞[29]。 3.2.2. 衣壳蛋白的免疫原性 ORF2 C端的 578-607aa[30]和452-617aa[31]是线性 表位区域,458-607aa[32] 是构象表位区域。中国 HEV239(368-606aa)疫苗[33]与美国 NIH 研制的 rHEV (112-607aa)疫苗[34]具有相同的 C末端,两种疫苗均包 括HEV 构象表位区域(459-606aa),而大部分构象表 位都在 HEV 表面,能中和 HEV 并阻断病毒与宿主细 胞受体的结合。最近的一些研究表明只有能识别构象 表位的抗体可以中和抗原[35]。 3.3. 与细胞骨架结合的多功能磷酸蛋白 (Cytoskeleton-Associated Multifunctional Phosphor-Protein) ORF3 长约 360 nt,3’端与 ORF2重叠的部分约为 300 nt[36],编码一种与细胞骨架结合的磷酸蛋白。 ORF3 编码的蛋白 N端有两个疏水区 D1和D2,C端 有两个脯氨酸富集区P1 和P2[37]。ORF3 蛋白通过D1 区可与细胞骨架[38]、MAPK 磷酸酶[39]和微管[40]结合。 D2 区可作用于血色素结合蛋白,调节细胞内的铁离 子平衡[41]。 3.3.1. ORF3蛋白的起始密码 HEV的ORF3编码 112 个氨基酸,JR 区依次有 4 个AUG 密码子,其中第三个AUG 是ORF3 的起始密 码[42]。在哺乳动物细胞中表达的 ORF3 蛋白为 13 kDa, Ser71 氨基酸残基的磷酸化影响病毒颗粒的装配[43]。 3.3.2. ORF3蛋白的作用 为弄清 0RF3 蛋白的作用,Yamada[44]用ORF3 基 因突变的 cDNA 感染性克隆和HEV 野毒株,分别在 PLC/PKF/5细胞中培养。用免疫捕获 PCR法检测细胞 培养液中的 ORF3 蛋白。发现 ORF3 蛋白存在于从感 染细胞中释放出的HEV 野毒株粒子表面,而在 ORF3 突变体感染的细胞释放的病毒粒子表面未检测到 ORF3 蛋白。提示 HEV ORF3蛋白对于病毒从感染细 胞内释放具有重要作用。Surjit[ 45]等研究表明 ORF3 蛋 白与肿瘤易感基因 101(Tsg101)和α-1-微球蛋白相互 作用,参与内体分选复合物(ESCRT)的装配。因此, ORF3 蛋白与胞内蛋白作用也有利于病毒的复制和释 放。 3.3.3. ORF3蛋白与疫苗 基因 1、4型ORF3多肽与相同基因型的血清抗 体有更强的反应,因此1、4型ORF3蛋白的抗原性 存在基因型差异[46]。用 4型HEV ORF3蛋白免疫恒河 猴,可诱导产生较强的抗体反应,同时能部分抵抗 HEV感染[47]。ORF3 蛋白的这种抗原保护作用为疫苗 研制提供了新思路。 4. HEV的顺反应元件 (Cis-Reactive Elements, CREs) CREs 维持病毒RNA 的稳定,参与细胞间、细胞 内以及 HEV RNA之间的反应。一般来说,CREs存 在于病毒 5’和3’端的非编码区,但是HEV 5’端的非 编码区只有 26 nt,并且还有一个帽状结构,所以 HEV 的5’端不含有 CRE。 4.1. 第一个 CRE 第一个 CRE位于 3’端非编码区并与 ORF2 有重 合,可影响病毒的复制[11]。3’端的非编码区与其周围 区段形成茎环(stem-loop structures, SL)结构(图2(a)), 该结构与病毒的 RdRp 结合。若去除其中的 SL1、SL2 或Poly(A),则不能与 RdRp 结合形成复合物,因此推 测RdRp 可能具有识别这些结构的功能[18]。1型HEV 中SL2 第7106 位点碱基对的改变会大大降低病毒的 复制[48]。 4.2. 第二个 CRE 第二个 CRE 位于基因的连接区(junction region, JR)(图2(b))。体外实验表明 JR区4 nt或6 nt的突变 Copyright © 2013 Hanspub 34 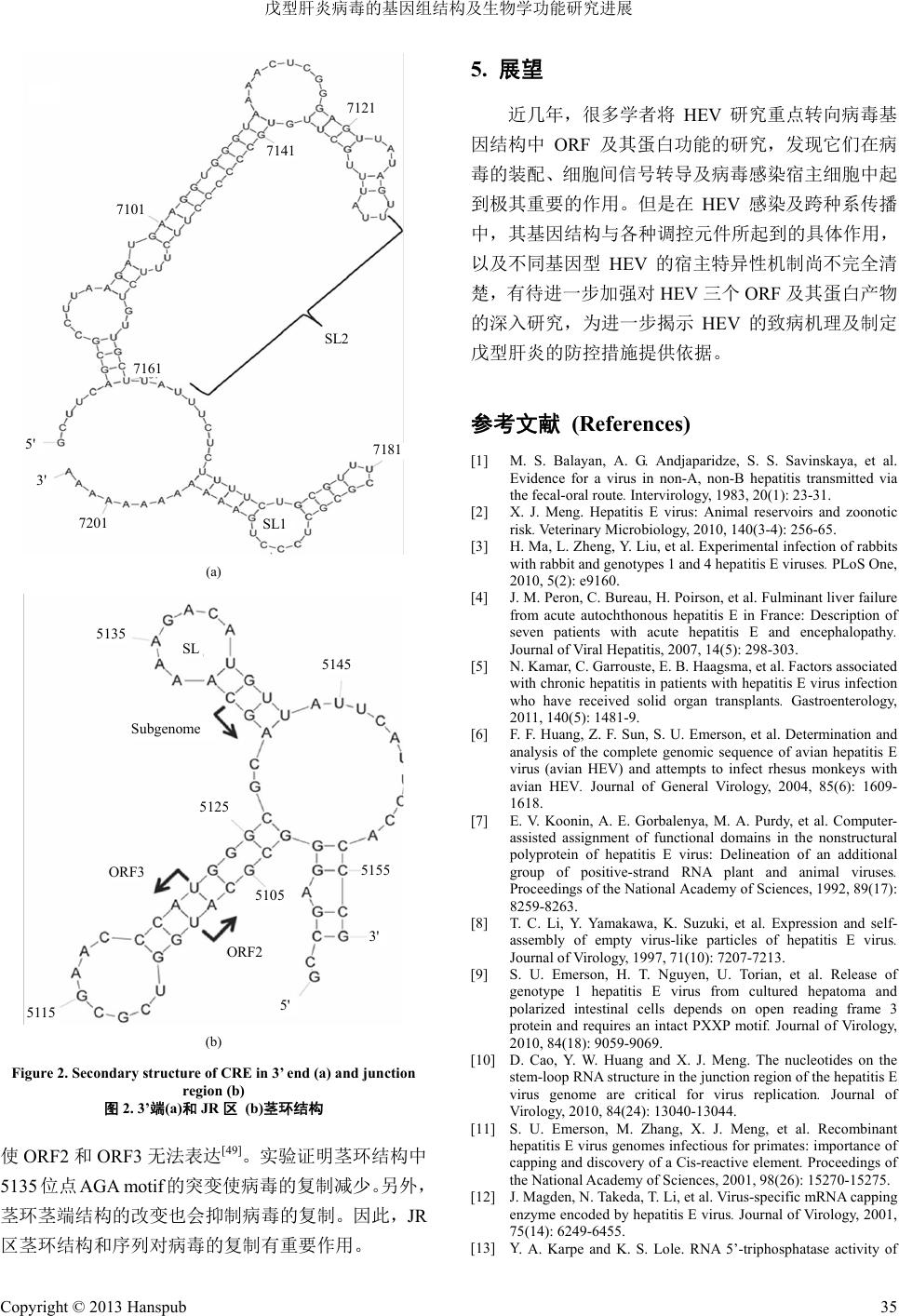 戊型肝炎病毒的基因组结构及生物学功能研究进展 7101 7121 7141 7181 7201 SL2 7161 SL1 5' 3' (a) 5125 5145 5135 5105 5115 5155 ORF3 SL 5' 3' ORF2 Subgenome (b) Figure 2. Secondary structure of CRE in 3’ end (a) and junction region (b) 图2. 3’端(a)和JR 区 (b)茎环结构 使ORF2 和ORF3 无法表达[49]。实验证明茎环结构中 5135位点 AGA motif的突变使病毒的复制减少。另外, 茎环茎端结构的改变也会抑制病毒的复制。因此,JR 区茎环结构和序列对病毒的复制有重要作用。 5. 展望 近几年,很多学者将 HEV 研究重点转向病毒基 因结构中 ORF 及其蛋白功能的研究,发现它们在病 毒的装配、细胞间信号转导及病毒感染宿主细胞中起 到极其重要的作用。但是在HEV 感染及跨种系传播 中,其基因结构与各种调控元件所起到的具体作用, 以及不同基因型 HEV 的宿主特异性机制尚不完全清 楚,有待进一步加强对 HEV三个 ORF 及其蛋白产物 的深入研究,为进一步揭示HEV 的致病机理及制定 戊型肝炎的防控措施提供依据。 参考文献 (References) [1] M. S. Balayan, A. G. Andjaparidze, S. S. Savinskaya, et al. Evidence for a virus in non-A, non-B hepatitis transmitted via the fecal-oral route. Intervirology, 1983, 20(1): 23-31. [2] X. J. Meng. Hepatitis E virus: Animal reservoirs and zoonotic risk. Veterinary Microbiology, 2010, 140(3-4): 256-65. [3] H. Ma, L. Zheng, Y. Liu, et al. Experimental infection of rabbits with rabbit and genotypes 1 and 4 hepatitis E viruses. PLoS One, 2010, 5(2): e9160. [4] J. M. Peron, C. Bureau, H. Poirson, et al. Fulminant liver failure from acute autochthonous hepatitis E in France: Description of seven patients with acute hepatitis E and encephalopathy. Journal of Viral Hepatitis, 2007, 14(5): 298-303. [5] N. Kamar, C. Garrouste, E. B. Haagsma, et al. Factors associated with chronic hepatitis in patients with hepatitis E virus infection who have received solid organ transplants. Gastroenterology, 2011, 140(5): 1481-9. [6] F. F. Huang, Z. F. Sun, S. U. Emerson, et al. Determination and analysis of the complete genomic sequence of avian hepatitis E virus (avian HEV) and attempts to infect rhesus monkeys with avian HEV. Journal of General Virology, 2004, 85(6): 1609- 1618. [7] E. V. Koonin, A. E. Gorbalenya, M. A. Purdy, et al. Computer- assisted assignment of functional domains in the nonstructural polyprotein of hepatitis E virus: Delineation of an additional group of positive-strand RNA plant and animal viruses. Proceedings of the National Academy of Sciences, 1992, 89(17): 8259-8263. [8] T. C. Li, Y. Yamakawa, K. Suzuki, et al. Expression and self- assembly of empty virus-like particles of hepatitis E virus. Journal of Virology, 1997, 71(10): 7207-7213. [9] S. U. Emerson, H. T. Nguyen, U. Torian, et al. Release of genotype 1 hepatitis E virus from cultured hepatoma and polarized intestinal cells depends on open reading frame 3 protein and requires an intact PXXP motif. Journal of Virology, 2010, 84(18): 9059-9069. [10] D. Cao, Y. W. Huang and X. J. Meng. The nucleotides on the stem-loop RNA structure in the junction region of the hepatitis E virus genome are critical for virus replication. Journal of Virology, 2010, 84(24): 13040-13044. [11] S. U. Emerson, M. Zhang, X. J. Meng, et al. Recombinant hepatitis E virus genomes infectious for primates: importance of capping and discovery of a Cis-reactive element. Proceedings of the National Academy of Sciences, 2001, 98(26): 15270-15275. [12] J. Magden, N. Takeda, T. Li, et al. Virus-specific mRNA capping enzyme encoded by hepatitis E virus. Journal of Virology, 2001, 75(14): 6249-6455. [13] Y. A. Karpe and K. S. Lole. RNA 5’-triphosphatase activity of Copyright © 2013 Hanspub 35  戊型肝炎病毒的基因组结构及生物学功能研究进展 Copyright © 2013 Hanspub 36 the hepatitis E virus helicase domain. Journal of Virology, 2010, 84(18): 9637-9641. [14] Y. A. Karpe and K. S. Lole. NTPase and 5’ to 3’ RNA duplex-unwinding activities of the hepatitis E virus helicase domain. Journal of Virology, 2010, 84(7): 3595-3602. [15] S. L. Ropp, A. W. Tam, B. Beames, et al. Expression of the hepatitis E virus ORF1. Archives of Virology, 2000, 145(7): 1321-1337. [16] D. Sehgal, S. Thomas, M. Chakraborty, et al. Expression and processing of the Hepatitis E virus ORF1 nonstructural polyprotein. Journal of Virology, 2006, 3: 38. [17] Y. A. Karpe and K. S. Lole. Deubiquitination activity associated with hepatitis E virus putative papain-like cysteine protease. Journal of General Virology, 2011, 92(9): 2088-2092. [18] S. Agrawal, D. Gupta and S. K. Panda. The 3’ end of hepatitis E virus (HEV) genome binds specifically to the viral RNA- dependent RNA polymerase (RdRp). Virology, 2001, 282(1): 87-101. [19] S. Rehman, N. Kapur, H. Durgapal, et al. Subcellular locali- zation of hepatitis E virus (HEV) replicase. Virology, 2008, 370(1): 77-92. [20] R. S. Pudupakam, Y. W. Huang, T. Opriessnig, et al. Deletions of the hypervariable region (HVR) in open reading frame 1 of hepatitis E virus do not abolish virus infectivity: Evidence for attenuation of HVR deletion mutants in vivo. Journal of Virology, 2009, 83(1): 384-395. [21] R. S. Pudupakam, S. P. Kenney, L. Cordoba, et al. Mutational analysis of the hypervariable region of hepatitis e virus reveals its involvement in the efficiency of viral RNA replication. Journal of Virology, 2011, 85(19): 10031-10040. [22] X. J. Meng. Recent advances in Hepatitis E virus. Journal of Viral Hepatitis, 2010, 17(3): 153-161. [23] S. Jameel, M. Zafrullah, M. H. Ozdener, et al. Expression in animal cells and characterization of the hepatitis E virus structural proteins. Journal of Virology, 1996, 70(1): 207-16. [24] M. Surjit, S. Jameel and S. K. Lal. Cytoplasmic localization of the ORF2 protein of hepatitis E virus is dependent on its ability to undergo retrotranslocation from the endoplasmic reticulum. Journal of Virology, 2007, 81(7): 3339-3345. [25] J. Graff, Y. H. Zhou, U. Torian, et al. Mutations within potential glycosylation sites in the capsid protein of hepatitis E virus prevent the formation of infectious virus particles. Journal of Virology, 2008, 82(3): 1185-1194. [26] M. Surjit, S. Jameel and S. K. Lal. The ORF2 protein of hepatitis E virus binds the 5’ region of viral RNA. Journal of Virology, 2004, 78(1): 320-328. [27] Z. Z. Zheng, J. Miao, M. Zhao, et al. Role of heat-shock protein 90 in hepatitis E virus capsid trafficking. Journal of Viral Hepatitis, 2010, 91(7): 1728-1736. [28] H. Yu, S. Li, C. Yang, et al. Homology model and potential virus-capsid binding site of a putative HEV receptor Grp78. Journal of Molecular Modeling, 2011, 17(5): 987-995. [29] M. Kalia, V. Chandra, S. A. Rahman, et al. Heparan sulfate proteoglycans are required for cellular binding of the hepatitis E virus ORF2 capsid protein and for viral infection. Journal of Virology, 2009, 83(24): 12714-12724. [30] D. J. Schofield, J. Glamann, S. U. Emerson, et al. Identification by phage display and characterization of two neutralizing chimpanzee monoclonal antibodies to the hepatitis E virus capsid protein. Journal of Virology, 2000, 74(12): 5548-5555. [31] J. Meng, X. Dai, J. C. Chang, et al. Identification and characterization of the neutralization epitope(s) of the hepatitis E virus. Virology, 2001, 288(2): 203-211. [32] Y. H. Zhou, R. H. Purcell and S. U. Emerson. An ELISA for putative neutralizing antibodies to hepatitis E virus detects antibodies to genotypes 1, 2, 3, and 4. Vaccine, 2004, 22(20): 2578-2585. [33] J. Zhang. Randomized-controlled phase II clinical trial of a bacterially expressed recombinant hepatitis E vaccine. Vaccine, 2009, 27: 1869-1874. [34] M. P. Shrestha. Safety and efficacy of a recombinant hepatitis E vaccine. The New England Journal of Medicine, 2007, 356: 895-903. [35] H. Zhang, X. Dai, X. Shan, et al. The Leu477 and Leu613 of ORF2-encoded protein are critical in forming neutralization antigenic epitope of hepatitis E virus genotype 4. Cellular & Molecular Immunology, 2008, 5(6): 447-456. [36] J. Graff, U. Torian, H. Nguyen, et al. A bicistronic subgenomic mRNA encodes both the ORF2 and ORF3 proteins of hepatitis E virus. Journal of Virology, 2006, 80(12): 5919-5926. [37] I. Ahmad, R. P. Holla and S. Jameel. Molecular virology of hepatitis E virus. Virus Research, 2011, 161(1): 47-58. [38] M. Zafrullah, M. H. Ozdener, S. K. Panda, et al. The ORF3 protein of hepatitis E virus is a phosphoprotein that associates with the cytoskeleton. Journal of Virology, 1997, 71(12): 9045-9053. [39] A. Kar-Roy, H. Korkaya, R. Oberoi, et al. The hepatitis E virus open reading frame 3 protein activates ERK through binding and inhibition of the MAPK phosphatase. The Journal of Biological Chemistry, 2004, 279(27): 28345-28357. [40] H. Kannan, S. Fan, D. Patel, et al. The hepatitis E virus open reading frame 3 product interacts with microtubules and interferes with their dynamics. Journal of Virology, 2009, 83(13): 6375-6382. [41] R. Ratra, A. Kar-Roy and S. K. Lal. The ORF3 protein of hepatitis E virus interacts with hemopexin by means of its 26 amino acid N-terminal hydrophobic domain II. Biochemistry, 2008, 47(7): 1957-1969. [42] Y. Zhu, Z. Li and C. Bu. The open reading frame 2 initiation site of genotype 4 hepatitis E virus (HEV). Infection, Genetics and Evolution, 2012, 12(3): 502-503. [43] S. Tyagi, H. Korkaya, M. Zafrullah, et al. The phosphorylated form of the ORF3 protein of hepatitis E virus interacts with its non-glycosylated form of the major capsid protein, ORF2. The Journal of Biological Chemistry, 2002, 277(25): 22759-22767. [44] K. Yamada, M. Takahashi, Y. Hoshino, et al. ORF3 protein of hepatitis E virus is essential for virion release from infected cells. Journal of General Virology, 2009, 90(8): 1880-1891. [45] M. Surjit, R. Oberoi, R. Kumar, et al. Enhanced alpha1 micro- globulin secretion from Hepatitis E virus ORF3-expressing human hepatoma cells is mediated by the tumor susceptibility gene 101. The Journal of Biological Chemistry, 2006, 281(12): 8135-8142. [46] H. Ma, X. Song, T. J. Harrison, et al. Hepatitis E virus ORF3 antigens derived from genotype 1 and 4 viruses are detected with varying efficiencies by an anti-HEV enzyme immunoassay. Journal of Medical Virology, 2011, 83(5): 827-832. [47] H. Ma, X. Song, T. J. Harrison, et al. Immunogenicity and efficacy of a bacterially expressed HEV ORF3 peptide, assessed by experimental infection of primates. Archives of Virology, 2009, 154(10): 1641-1648. [48] J. Graff, H. Nguyen, C. Kasorndorkbua, et al. In vitro and in vivo mutational analysis of the 3’-terminal regions of hepatitis e virus genomes and replicons. Journal of Virology, 2005, 79(2): 1017- 1026. [49] J. Graff, H. Nguyen, C. Yu, et al. The open reading frame 3 gene of hepatitis E virus contains a Cis-reactive element and encodes a protein required for infection of macaques. Journal of Virology, 2005, 79(11): 6680-6689. |