 Hans Journal of Co mpu tati on al Bi ology 计算生物学, 2012, 2, 34-41 doi:10.12677/hjcb.2012.24004 Published Online December 2012 (http://www.hanspub.org/journal/hjcb.html) Copyright © 2012 Hanspub 34 2-Dimensional HP Folding Structures of Caerin* Hejian Wang1, Shaomin Yan1, Guang Wu1,2# 1State Key Laboratory of Non-Food Biomass Enzyme Technology, Natio nal Engineer ing Resea rch C en ter for N o n-Food Biorefiner y, Guangxi Key Laboratory of Biorefinery, Gua ngxi Academy of Sciences, Nanning 2DreamSciTech Consulting, Shenzhen Email: #hongguanglishibahao@yahoo.com Received: Nov. 4th, 2012; rev ised : Nov. 26th, 2012; accepted: Dec. 1st, 2012 Abstract: The amino acid sequences of 7 Caerins were converted into 28 hydrophobic (H) or polar (P) sequences ac- cording to the normalized amino acid hydrophobicity index, and all of their possible folding structures were analyzed using 2D hydrophobic-pola r (HP ) model. The r esult s sho wed tha t Caeri ns ha ve man y nati ve stat es wi th the same mini- mal energy, which consist of various symmetric folding structures, and that the normalized amino acid hydrophobicity index can help further mor e distin guish native state s numeri ca lly. The study de monstrates the diver sity o f Caerin foldin g structures from hydrophobic-polar (HP) angle, which can shed light on understanding folding process of protein and implying possible ways to modify antimicrobial peptides through engineering. Keywords: Caerin; Foldi ng Structure; HP Model; H ydro phobicity Ind ex; Minima l Energy Caerin 的二维 HP 折叠结构* 王何健 1,严少敏 1,吴 光1, 2# 1广西科学院非粮生物质国家重点实验室,国家非粮生物质能源工程技术研究中心,广西生物炼制重点实验室,南宁 2深圳市追梦科技咨询有限公司,深圳 Email: #hongguanglishibahao@yahoo.com 收稿日期:2012年11 月4日;修回日期:2012年11 月26日;录用日期:2012 年12 月1日 摘 要:采用标准化氨基酸疏水性指数将 7种Caerins 的氨基酸序列转换为 28 种疏水性(H)或极性(P)的序列,通 过二维疏水性–极性(HP)模型分析其全部可能的折叠结构。结果显示 Caerins 有许多具有相同最小能量的天然形 态,这些形态有各种各样对称的折叠结构,而且这些天然形态可以根据标准化的氨基酸疏水性指标进一步从数 值上加以区分。这项研究从疏水性–极性角度揭示了 Caerin 折叠结构的多样性,有助于理解蛋白质的折叠过程, 并提示通过工程学方法修改抗菌肽的可能途径。 关键词:Caerin;折叠结构;HP 模型;疏水性指标;最小能量 1. 引言 抗生素抗药现象越来越普遍,已经成为公共健康的危 机。一些青蛙皮活性肽采用了 α螺旋构象并且能够使细菌 膜破裂,因此它们具有较小的细菌抗药性,已引起了广泛 的关注[1,2]。Caerin 是澳大利亚树蛙皮肤腺体中分泌的抗菌 阳离子短肽,它们通过像清洁剂一样引发跨膜小孔或者分 裂细胞膜的方式发挥作用[3]。通常这些包含二硫键的多 肽 可以在电喷雾离子化过程中和铜、镁、锌形成复离子。关 于金属(2+)连接成一个络合物的理论计算表明 MCU ( 2 +) 与 膜活性的 Caerin 络合,如 Caerin 1.8,是 4配位络合物且 近似正方形的平面[4], 因为 疏水性和亲水性的相 互作用 对 抗菌功能都很重要[5],这激发了我们使用疏水性–极性(HP) * 资助信息:广西自然科学基金项目(12 -071-10 and 12237022)。 # 通讯作者。  Caerin 的二维 HP 折叠结构 Copyright © 2012 Hanspub 35 模型研究折叠结构,此模型依据蛋白折叠的推动力,而氨 基酸的疏水性是形成小球状蛋白天然结构的主要动力[6]。 任何模型都是对现实生活中案例的近似,从本质上来 讲,模型之间的不同也就体现在它们提供的相似程度不 同。HP 模型看上去很简单,却有必要进行全面研究,因 为该模型需要高强度计算,它已经被列为 NP问题[7],并 且已被列为千禧年大奖难题(Millennium Prize P roblems:七 个由美国克雷数学研究所于 2000 年5月24日公布的数学 难题)的首要问题[8]。因此,研究 HP 模型能够帮助我们理 解NP 问题,开发最佳算法[9],处理生物学领域的其他高 强度计算问题,例如系统发生学和核糖核酸假结问题[10]。 HP 模型可用于二维和三维折叠结构。在二维平面或 三维立体结构中,每一个疏水性( H)或极性(P)的氨基酸都 沿着一条线走,采取自我回避的步骤,然后一个产生于非 连续的 H-H 连接被定义为负的单位能量,而能量最小的结 构被认为是一种天然的折叠结构。在二维 HP 模型中,结 构数量的公式是 3n−1,n是指一个蛋白质的氨基酸个数。 比如,2个氨基酸的结构数是 3(32−1),3个氨基酸则是 9(33−1),4个氨基酸是 81(34−1),以此类推有25 个氨基酸的 多肽则有 282,429,536,481 可能的折叠结构( 3 25−1)。采用中 央处理器是 2千兆赫的联想 ThinkPad 笔记本,每秒计算 200,000 到250,000 折叠结构,对于25 个氨基酸的 Caerin, 计算 282,429,536,481/250,000 到282,429,536,481/200,000 秒,也就是 1,129,718 到1,412,147 秒(大约 13~16 天)。本 研究致力于探索 Caerin 的所有二维 HP 模型折叠结构。 2. 材料和方法 2.1. 数据 在有关抗癌和抗肿瘤肽的网站中,总共找到7种 Caerin[11],并 从通 用蛋白 质 资源数 据库中 获取 它们 的氨 基 酸序列[12]。从阿德里奇化学网站获取的标准化氨基酸疏水 性指数[13],如表 1所示。 2.2. HP模型 HP 模型根据疏水性或极性将氨基酸分类,但并没说 明怎样处理中性氨基酸。首先,用表 1的标准化氨基酸疏 水性指数处理 Caerin 中的中性氨基酸;其次, 需要决定甘氨酸是疏水的还是极性的,因为这种标准化的 氨基酸疏水性指数假定甘氨酸为零。另一方面,氨基酸的 疏水性还取决于 pH 值(表1 ),因此分别按两个不同的 pH 值标准转换 Caerin 中的氨基酸。同时考虑这两种情形,在 HP 模型中每个 Caerin 有4个HP 序列,因此7种Caerins 共有 28个HP 序列需要分析(表2)。Caerins 分别有 24 和 25 个氨基酸,每个 HP 序列理论上分别有 94,143,178,827 和282,429,536,481个折叠结构。 3. 结果和讨论 尽管 HP 模型构造简易,除了已提到的巨大计算量之 外,仍然存在许多问题亟待解决,比如说,怎样处理中性 氨基酸的问题。确 实, 标准 化的氨 基酸 疏水性指标(表1) 为这个问题的解决提供了一种途径,但却导致计算量增加 了四倍(表2 ) ,因为每个 Caerin 的氨 Table 1. Normalized amino acid hydrophobicity index 表1. 标准化的氨基酸疏水性指数 At pH 2 At pH 7 Very Hydrophobic L 100 F 100 I 100 I 99 F 92 W 97 W 84 L 97 V 79 V 76 M 74 M 74 Hydrophobic C 52 Y 63 Y 49 C 49 A 47 A 41 Neutral T 13 T 13 E 8 H 8 G 0 G 0 S −7 S −5 Q −18 Q −10 D −18 Hydrophilic R −26 R −14 K −37 K −23 N −41 N −28 H −42 E −31 P −46 P −46 (used pH 2) D −55  Caerin 的二维 HP 折叠结构 Copyright © 2012 Hanspub 36 Ta ble 2. Seven Caeri ns and their HP sequences 表2. 7种Caerins 的氨基酸序列和 HP 序列 Caerin 分类 Classification 序列 Sequence Caerin 1.1 (AP00240) Amino acid GLLSVLGSVAKHVLPHVVPVIAEHL G = H at pH 2 HHHPHHHPHHPPHHPPHHPHHHHPH G = P a t pH 2 PHHPHHPPHHPPHHPPHHPHHHHPH G = H at pH 7 HHHPHHHPHHPHHHPHHHPHHHPHH G = P a t pH 7 PHHPHHPPHHPHHHPHHHPHHHPHH Caerin 1.3 (AP00242) Amino acid GLLSVLGSVAQHVLPHVVPVIAEHL G = H at pH 2 HHHPHHHPHHPPHHPPHHPHHHHPH G = P a t pH 2 PHHPHHPPHHPPHHPPHHPHHHHPH G = H at pH 7 HHHPHHHPHHPHHHPHHHPHHHPHH G = P a t pH 7 PHHPHHPPHHPHHHPHHHPHHHPHH Caerin 1. 5 (A P00244) Amino acid GLLSVLGSVVKHVIPHVVPVIAEHL G = H at pH 2 HHHPHHHPHHPPHHPPHHPHHHHPH G = P a t pH 2 PHHPHHPPHHPPHHPPHHPHHHHPH G = H at pH 7 HHHPHHHPHHPHHHPHHHPHHHPHH G = P a t pH 7 PHHPHHPPHHPHHHPHHHPHHHPHH Caerin 1.6 (AP00245) Amino acid GLFSVLGAVAKHVLPHVVPVIAEK G = H at pH 2 HHHPHHHHHHPPHHPPHHPHHHHP G = P a t pH 2 PHHPHHPHHHPPHHPPHHPHHHHP G = H at pH 7 HHHPHHHHHHPHHHPHHHPHHHPP G = P a t pH 7 PHHPHHPHHHPHHHPHHHPHHHPP Caerin 1.7 (AP00246) Amino acid GLFKVLGSVAKHLLPHVAPVIAEK G = H at pH 2 HHHPHHHPHHPPHHPPHHPHHHHP G = P a t pH 2 PHHPHHPPHHPPHHPPHHPHHHHP G = H at pH 7 HHHPHHHPHHPHHHPHHHPHHHPP G = P a t pH 7 PHHPHHPPHHPHHHPHHHPHHHPP Caerin 1.8 (AP00247) Amino acid GLFKVLGSVAKHLLPHVVPVIAEK G = H at pH 2 HHHPHHHPHHPPHHPPHHPHHHHP G = P a t pH 2 PHHPHHPPHHPPHHPPHHPHHHHP G = H at pH 7 HHHPHHHPHHPHHHPHHHPHHHPP G = P a t pH 7 PHHPHHPPHHPHHHPHHHPHHHPP Caerin 1.9 (AP00248) Amino acid GLFGVLGSIAKHVLPHVVPVIAEK G = H at pH 2 HHHHHHHPHHPPHHPPHHPHHHHP G = P a t pH 2 PHHPHHPPHHPPHHPPHHPHHHHP G = H at pH 7 HHHHHHHPHHPHHHPHHHPHHHPP G = P a t pH 7 PHHPHHPPHHPHHHPHHHPHHHPP G = H at pH 2 ,当pH为2时甘氨酸作为疏水性氨基酸 ;G = P at pH 2,当 pH为2时甘氨酸作为极性 氨基酸;G = H at pH 7,当pH 为7时甘氨酸作为疏水性氨 基酸;G = P at pH 7,当 pH 为7时甘氨酸作为极 性氨基酸。 基酸序列在转换成 HP 序列时必须考虑甘氨酸是疏水 还是极性以及两种 pH标准,而比较不同的 HP 序列 则显示了中性氨基酸的作用。例如,Caerin 1.1(表1, 第2行)的氨基酸序列转换成了不同的 HP 序列:第 1 位和第7位的甘氨酸(G)被转换成疏水性氨基酸 (G=H,第3行和第 5行)或极性氨基酸(G = P,第 4 行和第 6行);第12 、16、24 位的组氨酸(H)当pH 值 为2时被转换成极性氨基酸(第3行和第 4行),而当 pH值为 7时被转换成疏水性氨基酸(第5行和第 6行)。 可见 HP 序列可以根据蛋白质成分不同有不同程度的  Caerin 的二维 HP 折叠结构 Copyright © 2012 Hanspub 37 变化。 表2中每个 HP 序列具有 282,429,536,481 或者 94,143,178,827 可能的折叠结构,这对了解 Caerin 拥 有多少天然形态很重要。表 3列出了根据不同 HP 转 换所确定的 H-H 连接结果,包括天然形态的数量、最 小能量、疏水性指数总和的类型和范围。HP 模型揭 示了 Caerin 折叠结构的多样性,从中可以得出一些特 征:1) 除了 Caerin 1.9,其 它Caerins 天然形态在 pH2 的数量少于 pH7 的数量,在 pH7 时天然形态的数量 急剧增加到 8192 种;2) 在pH 值为 2时最小能量(圆 括号中的数字)低得多,此时甘氨酸被视为疏水的;3) 疏水性指数的类型(方括号中的数字)在pH值为 7时明 显增加;4) 疏水性指数总和(大括号中的数字)在pH 值为 2时更大。因此,我们的结果证实了 pH 值对多 肽折叠具有强烈影响,这有助于理解为什么许多抗微 生物蛋白在低 pH 值环境下会失去其功能。 根据吉布斯自由能的整体最低值[14],分子的天然 形态代表一个热力学稳定态。通过HP 模型发现 Caerins 有如此多的天然形态,说明了 Caerins 折叠机 理的灵活性,它们可以在特定的酶促反应中使用不同 的折叠结构,因为每个天然形态的最小能量都是同等 的。由于空间限制,图 1仅举例了假定甘氨酸为极性、 pH 值为7的情况下 Caerin 1.1 的8种天然形态。该图 中的每个结构都展示了 HP 模型是怎样将一个蛋白质 从位置 1到位置25 折叠到二维晶格中的。这些结构 组成了 11 个非连续的 H-H 连接,其中每个都是一个 负能量单位,如 3H-6H、3H-14H、6H-9H、9H-14H、 10H-13H、12H-17H、12-25H、13H-16H、18H-21H、 18H-25H和21H-24H。有趣的一点是,天然形态在左 手侧和右手侧的四个折叠结构之间是手性对称的,也 就是说由于它们不能在镜像图像中叠加,构建天然形 态的途径是手性的。由于对称性,蛋白质分子可以更 容易地通过不同的途径找到它的天然形态,使寻找其 天然形态的时间尽可能得缩短。 最小能量的天然型态种类随 pH 值的不同以及甘 氨酸的疏水或极性转换的不同而各异(表3),图 2展示 了这两种情况是如何影响 Caerin 1.6 的折叠结构。考 虑到两种 pH 值的影响,在 pH 值为 2时位置 12 和 25H24H23P 22H 21H 20H 19P 18H 17H 16H 15P 14H13H12H 11P 10H9H 8P 7P 6H 5H 4P3H 2H 1P Native state I 25H 24H 23P 22H21H 20H19P 18H17H16H15P 14H 13H 12H 11P10H9H 8P7P 6H5H 4P 3H 2H1P Native state II 25H 24H 23P 22H 21H 20H 19P18H 17H 16H 15P14H 13H 12H11P 10H 9H8P 7P 6H 5H 4P 3H 2H 1P Native state III 25H 24H 23P22H 21H20H 19P 18H 17H 16H 15P 14H 13H 12H 11P 10H 9H 8P 7P6H 5H4P 3H2H 1P Native state IV 25H 24H 23P 22H21H 20H19P 18H17H16H15P 14H 13H 12H 11P10H9H 8P7P 6H5H 4P 3H 2H1P Native state V 25H24H23P 22H 21H 20H 19P 18H 17H 16H 15P 14H13H12H 11P 10H9H 8P 7P 6H 5H 4P3H 2H 1P Native state VI 25H 24H 23P22H 21H20H 19P 18H 17H 16H 15P 14H 13H 12H 11P 10H 9H 8P 7P6H 5H4P 3H2H 1P Native state V I I 25H 24H 23P 22H 21H 20H 19P18H 17H 16H 15P14H 13H 12H11P 10H 9H8P 7P 6H 5H 4P 3H 2H 1P Native state V I II 虚线是非顺序的 HH 连接,每条虚线代表一个负 能量单位(−1 ),它 们的总和既 是最小能量(−11)。 Figure 1. Eight native states of folding str uctures of Caerin 1.1 under HP conversion of G = P a t pH 7 图1. 在pH7 甘氨酸作为极性氨基酸时 Caerin 1.1的8种天然形态 Table 3. Native state, minimal energy and sum of hydrophobicity inde x of Caerin H-H connection s 表3. Caerin H-H 连接的天然形态、最小能量、疏水性指数总和 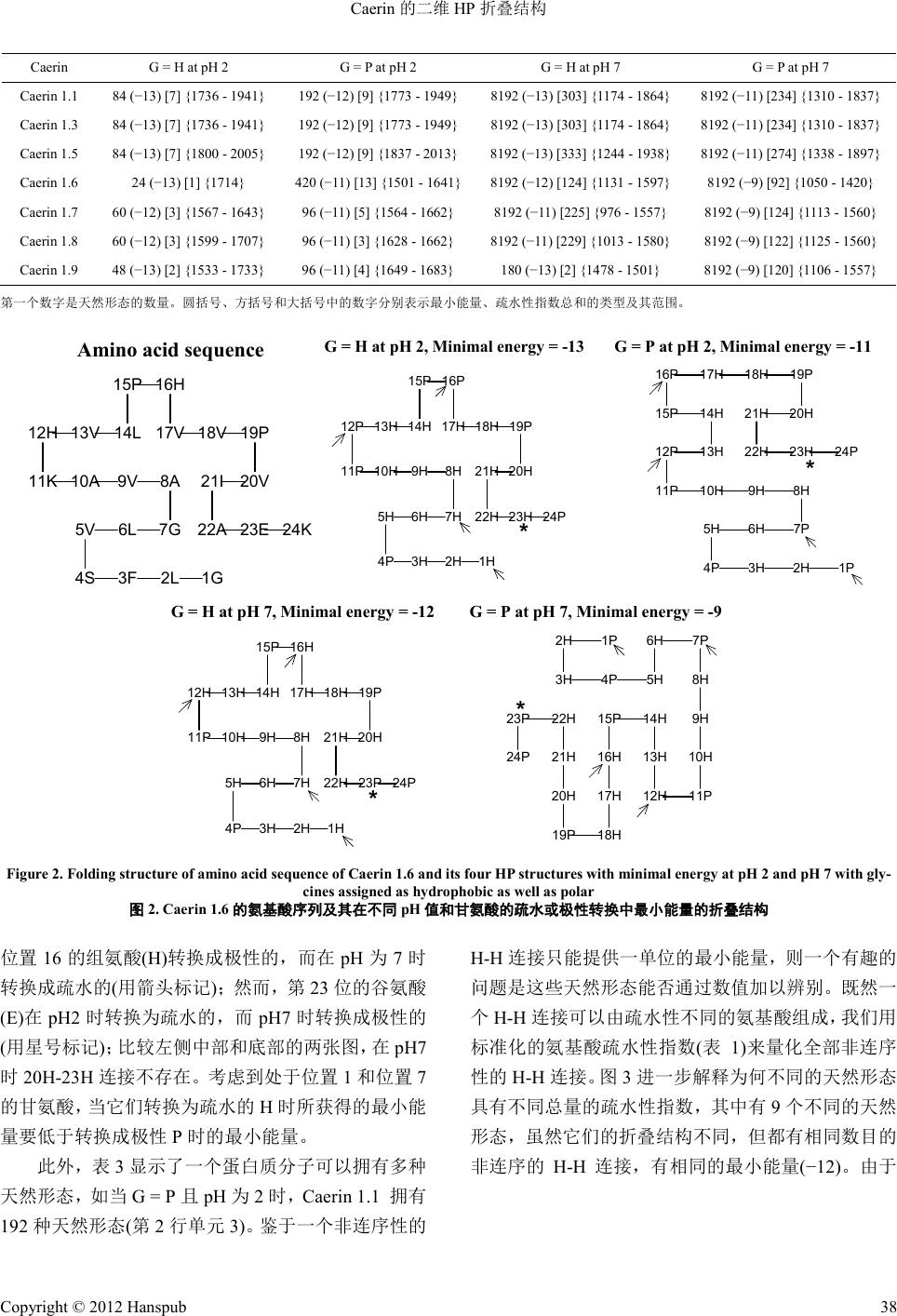 Caerin 的二维 HP 折叠结构 Copyright © 2012 Hanspub 38 Caerin G = H at pH 2 G = P at pH 2 G = H at pH 7 G = P at pH 7 Caerin 1.1 84 (−13) [7] {1736 - 1941} 192 (−12) [9] {1773 - 1949} 8192 (−13) [303] {1 174 - 1864} 8192 (−11) [234 ] {1310 - 1837} Caerin 1.3 84 (−13) [7] {1736 - 1941} 192 (−12) [9] {1773 - 1949} 8192 (−13) [303] {1 174 - 1864} 8192 (−11) [234 ] {1310 - 1837} Caerin 1.5 84 (−13) [7] {1800 - 2005} 192 (−12) [9] {1837 - 2013} 8192 (−13) [333] {1244 - 1938} 8192 (−11) [274] {1338 - 1897} Caerin 1.6 24 (−13) [1] {1714} 420 (−11) [13] {1501 - 1641} 8192 (−12) [124] {1131 - 1597} 8192 (−9) [92] {1050 - 1420} Caerin 1.7 60 (−12) [3] {1567 - 1643} 96 (−11) [5] {1564 - 1662} 8192 (−11) [225] {976 - 1557} 8192 (−9) [124] {1113 - 1560} Caerin 1.8 60 (−12) [3] {1599 - 1707} 96 (−11) [3] {1628 - 1662} 8192 (−11) [229] {1013 - 1580} 8192 (−9) [1 22] {1125 - 1560} Caerin 1.9 48 (−13) [2] {1533 - 1733} 96 (−11) [4] {1649 - 1683} 180 (−13) [2] {1478 - 1501} 8192 (−9 ) [120] {1106 - 1557} 第一个数字是天然形态 的数量。圆括 号、方括号 和大括号中的 数字分别表示 最小能量、 疏水性指数总 和的类型及其 范围。 24K23E22A 21I 20V 19P18V17V 16H15P 14L13V12H 11K 10A9V8A 7G6L5V 4S3F2L 1G Amino ac id sequence 24P23H22H 21H 20H 19P18H17H 16P15P 14H13H12P 11P 10H9H8H 7H6H5H 4P3H 2H 1H 24P23H22H 21H 20H 19P18H17H16P 15P 14H 13H12P 11P 10H9H8H 7P6H5H 4P 3H 2H1P G = H at pH 2, Minimal energy = -13 G = P at pH 2, Minimal energy = -11 * * 24P23P22H 21H 20H 19P18H17H 16H15P 14H13H12H 11P 10H9H8H 7H6H5H 4P3H 2H 1H 24P 23P 22H 21H 20H 19P 18H 17H 16H 15P 14H 13H 12H 11P 10H 9H 8H 7P6H 5H4P3H 2H 1P G = P at pH 7, Minimal energy = -9G = H at pH 7, Minimal energy = -12 * * Figure 2. Folding structure of amino acid sequence of Caerin 1.6 and its four HP structures with minimal ener gy at pH 2 and pH 7 with gly- cines assigned as hydrophobic as well as polar 图2. Caerin 1.6的氨基酸序列及其在不同 pH值和甘氨酸的疏水或极性转换中最小能量的折叠结构 位置 16 的组氨酸(H)转换成极性的,而在 pH 为7时 转换成疏水的(用箭头标记);然 而 ,第23位的谷氨酸 (E)在pH2 时转换为疏水的,而 pH7 时转换成极性的 (用星号标记);比较左侧中部和底部的两张图,在 pH7 时20H-23H 连接不存在。考虑到处于位置 1和位置 7 的甘氨酸,当它们转换为疏水的 H时所获得的最小能 量要低于转换成极性 P时的最小能量。 此外,表 3显示了一个蛋白质分子可以拥有多种 天然形态,如当 G = P且pH为2时,Caerin 1.1 拥有 192 种天然形态(第2行单元 3)。鉴于一个非连序性的 H-H 连接只能提供一单位的最小能量,则一个有趣的 问题是这些天然形态能否通过数值加以辨别。既然一 个H-H 连接可以由疏水性不同的氨基酸组成,我们用 标准化的氨基酸疏水性指数 (表1) 来量化全部非连序 性的 H-H连接。图 3进一步解释为何不同的天然形态 具有不同总量的疏水性指数,其中有9个不同的天然 形态,虽然它们的折叠结构不同,但都有相同数目的 非连序的 H-H 连接,有相同的最小能量(−12)。由于 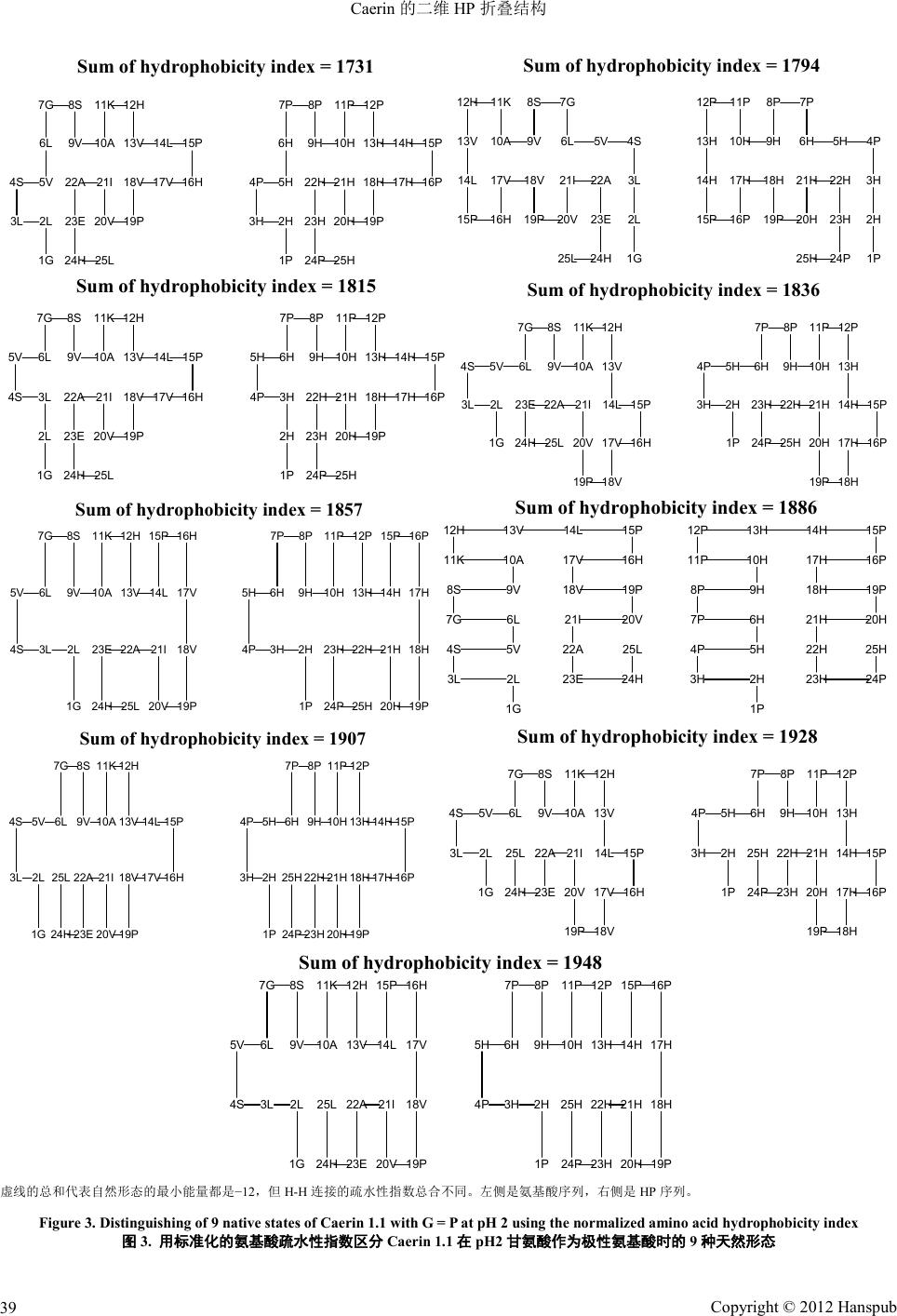 Caerin 的二维 HP 折叠结构 Copyright © 2012 Hanspub 39 25L24H 23E 22A 21I 20V 19P 18V 17V 16H 15P14L13V 12H11K 10A9V 8S7G 6L 5V4S 3L 2L 1G 25H24P 23H 22H 21H 20H 19P 18H 17H 16P 15P14H13H 12P11P 10H9H 8P7P 6H 5H4P 3H 2H 1P Sum of hydrophobic ity index = 1731 25L 24H 23E 22A21I 20V19P 18V17V 16H15P 14L 13V 12H 11K 10A 9V 8S 7G 6L5V 4S 3L 2L 1G 25H 24P 23H 22H21H 20H19P 18H17H 16P15P 14H 13H 12P 11P 10H 9H 8P 7P 6H 5H 4P 3H 2H 1P Sum of hydrophobic ity index = 1794 25L24H 23E 22A 21I 20V 19P 18V 17V 16H 15P14L13V 12H11K 10A9V 8S7G 6L5V 4S 3L 2L 1G 25H24P 23H 22H 21H 20H 19P 18H 17H 16P 15P14H13H 12P11P 10H9H 8P7P 6H5H 4P 3H 2H 1P Sum of hydrophobicity index = 1815 25L24H 23E 22A21I 20V 19P 18V 17V 16H 15P14L 13V 12H11K 10A9V 8S7G 6L5V4S 3L 2L 1G 25H24P 23H 22H 21H 20H 19P 18H 17H 16P 15P14H 13H 12P11P 10H9H 8P7P 6H5H4P 3H 2H 1P Sum of hydrophobic ity index = 1836 25L24H 23E 22A21I 20V 19P 18V 17V 16H15P 14L13V 12H11K 10A9V 8S7G 6L5V 4S 3L 2L 1G 25H24P 23H 22H21H 20H 19P 18H 17H 16P15P 14H13H 12P11P 10H9H 8P7P 6H5H 4P 3H2H 1P Sum of hydrophobic ity index = 1857 25L 24H23E 22A 21I 20V 19P18V 17V 16H 15P14L13V12H 11K 10A 9V8S 7G 6L 5V4S 3L 2L 1G 25H 24P23H 22H 21H 20H 19P18H 17H 16P 15P14H13H12P 11P 10H 9H8P 7P 6H 5H4P 3H 2H 1P Sum of hydrophobicity index = 1886 25L 24H 23E 22A 21I 20V 19P 18V 17V 16H 15P14L13V 12H11K 10A9V 8S7G 6L5V4S 3L 2L 1G 25H 24P 23H 22H 21H 20H 19P 18H 17H 16P 15P14H13H 12P11P 10H9H 8P7P 6H5H4P 3H 2H 1P Sum of hydrophobic ity index = 1907 25L 24H 23E 22A 21I 20V 19P 18V 17V 16H 15P14L 13V 12H11K 10A9V 8S7G 6L5V4S 3L 2L 1G 25H 24P 23H 22H 21H 20H 19P 18H 17H 16P 15P14H 13H 12P11P 10H9H 8P7P 6H5H4P 3H 2H 1P Sum of hydrophobicity index = 1928 25L 24H 23E 22A 21I 20V 19P 18V 17V 16H15P 14L13V 12H11K 10A9V 8S7G 6L5V 4S 3L 2L 1G 25H 24P 23H 22H 21H 20H 19P 18H 17H 16P15P 14H13H 12P11P 10H9H 8P7P 6H5H 4P 3H 2H 1P Sum of hydrophobicity index = 1948 虚线的总和代表自然形 态的最小能量 都是−12,但 H-H 连接的疏水性指数总合不同。左侧是氨基酸序列,右侧是 HP序列。 Figure 3. Distinguishing of 9 native states of Caerin 1.1 with G = P at pH 2 using the normalized amino acid hydrophobicity index 图3. 用标准化的氨基酸疏水性指数区分 Caerin 1.1在pH2 甘氨酸作为极性氨基酸时的 9种天然形态  Caerin 的二维 HP 折叠结构 Copyright © 2012 Hanspub 40 组成这 12 种H-H 连接的氨基酸不同,因此它们得以 区分。例如,图 2中最上面的图表显示了 12 种H-H 连接,2H-5H、2H-23H、5H-22H、6H-9H、9H-22H、 10H-13H、10H-21H、13H-18H、14H-17H、18H-21H、 20H-23H 和20H-25H(右侧图),与之相对应的连接是 2L-5V、2L-23E、5V-22A、6L-9V、9V-22A、10A-13V、 10A-21I、13V-18V、14L-17V、18V-21I、20V-23E和 20V-25L(左侧图)。这些氨基酸连接可以通过表 1进行 量化,它们的疏水性指数之和为 1773[(100 + 97) + (100 + 8) + (79 + 47) + (100 + 79) + (79 + 47) + (47 + 79) + (47 + 100) + (79 + 79) + (100 + 79) + (79 + 100) + (79 + 8) + (79 + 100)]。用这种方式,就可以辨别不同 的天然形态(见表 4)。 电荷状态分析和 H/D 与Cae rin 1. 8的交换实验表 明,络合作用涉及蛋白质辅助因子与钙离子钙调蛋白 的键合,这经历了一个结构更加紧凑的构象变化[15]。 采用分子动力学模拟方法,在溶液以及各种细胞膜环 境中检测某些抗微生物肽的结构性质,结果显示存在 一个键合于高膜面曲率区域的强烈倾向,而肽链的结 构正是基于局部曲率的程度[3]。 不同的模型从不同的角度揭示性质。例如,利用 二维核磁共振光谱学可以研究仿膜溶剂如三氟乙醇/ 水,而这种复合物的三维空间结构总是与之前确定的 单体模型肽的结构不同[17]。应用 HP模型的困难正在 于它巨大的计算量,过去的研究很少将该模型与实际 生活中的例子挂钩[16-18] 。本研究一个重要的启示就是 通过增加疏水氨基酸的数量来降低天然形态中的最 Table 4. Number of native states and their numerical distinctions determined by the normali zed amino acid hydrophobicity index for Caerin 1.1 with G = P at pH 2 表4. 在pH7甘氨酸作为极性氨基酸时 Caerin 1.1的天然形态数量 及其标准化氨基酸疏水性指数的区分结果 H-H连接的疏水性指数之和 天然形态的数量 1773 12 1794 24 1815 48 1836 24 1857 24 1886 24 1907 12 1928 12 1949 12 小能量,这有可能为肽链折叠产生更好的动力学效 应。如果某一天然形态和特定功能相关的话,则可以 使用一个疏水性氨基酸来替代一个中性氨基酸或者 亲水性氨基酸来降低该天然形态的最小能量值。 4. 结论 本研究使用二维疏水–极性模型来研究7种 Caerins 的折叠结构,将这些 Caerins 结构转化为 28 种HP序列,并使用标准化氨基酸疏水性指数来区分 它们的天然形态。结果显示:1) Caerisn 有着许多带有 相同最小能量的天然形态,这些形态可以适应不同的 反应;2) Caerins 有各种各样对称的折叠结构,这些结 构可以促进它们通过不同的途径找到自己的天然形 态;3) 标准化的氨基酸疏水性指标可以更好地帮助人 们从数字上区分天然形态,这就意味着有可能通过工 程学方法修改抗菌肽。可以通过用疏水氨基酸替换极 性氨基酸来修改 Caerins,也可以根据标准化的氨基酸 疏水性指数替换氨基酸来修改 Caerins。 参考文献 (References) [1] D. Bilusich, R. J. Jackway, I. F. Musgrave, et al. The host-de- fence skin peptide profiles of Peron’s tree frog Lito ria peronii in winter and summer. Sequence determination by electrospray mass spectrometry and activities of the peptides. Rapid Com- munic ations i n Mas s S pectro metry, 2009 , 23(17): 262 8-2636. [2] C . S. Chia, Y. Gong, J. H. Bowie, et al. Memb rane bin ding and perturbation studies of the antimicrobial peptides Caerin, citro- pin, a n d maculatin. Biopolymers, 2011, 96(2): 147-157. [3] R. Chen, A. E. Mark. The effect of membran e curvature on the conformation of antimicrobial peptides: implications for binding and the mechanism of action. European Biophysics Journal with Biophysics Letters, 2011, 40(4): 545 -553. [4] T. Wang, H. J. Andreazza, T. L. Pukala, et al. Histidine-cont ain- ing ho st-defence skin peptides of anurans bind Cu2+. An el ectro- spray ionisation mass spectrometry and computational model- ling study. Rapid Communications in Mass Spectrometry, 2011, 25(9): 1209-1221. [5] P. J. Sherman, R. J. Jackway, E. Nicholson, et al. Activities of seas onab ly variab le caerulein and rothein skin peptides from the tree frogs Litoria splendida and Litoria rothii. Toxicon, 2009, 54(6): 828-835. [6] K. F. Lau, K. A. Dill. A lattice statistical mechanics model of the con forma ti on and sequ enc e spac es of proteins. Macromolecules, 1989, 22(10): 3986-3997. [7] B. Berger, T. Leight. Protein folding in the hydrophobic-hydro- philic (HP) model is NP-complete. Journal of Computational Biology, 1998, 5(1): 27-40. [8] Wikimedia Foundation In c. Wikipedia, the free encyclopedia, 2012. http://en.wikipedia.org/wiki/Millennium_Prize_Problems [9] A. Shmygelska, H. H. Hoons. An ant colony optimisation algo- rithm for the 2D and 3D hydrophobic polar protein folding problem. BMC Bioinformatics, 2005, 6: 30. [10] R. B. Lyngsø, C. N. Pedersen. RNA pseudoknot prediction in energy-based models. Journal of Computational Biology, 2000,  Caerin 的二维 HP 折叠结构 Copyright © 2012 Hanspub 41 7(3-4): 409-427. [11] G. Wang, X. Li and Z. Wang. APD2: The updated an timicrobia l peptide database and its application in peptide design. Nucleic Acids Research, 20 0 9, 37(Database issue): D933-D937. [12] The UniProt Consortium. The universal protein resource (Uni- Prot) i n 2010. Nucleic Acids Research, 2010, 38(Database Issue): D142-D148. [13] Sigma-Aldrich Co. LLC. Hydrophobicity index for common amino acids, 2012. http://www.sigmaaldrich.com/life-science/metabolomics/learnin g-center/amino-acid-reference-chart.html [14] S. Govindarajan, R. A. Goldstein. On the thermodynamic hypo- thesis of protein folding. Proceedings of the National Academy of Sciences of the Uni ted States of Am erica, 1998, 95(10): 5545- 5549. [15] T. L. Pukala, T. Urathamakul, S. J. Watt, et al. Binding studies of nNOS-active amphibian peptides and Ca2+ calmodulin, using negative ion electrospray ionisation mass spectrometry. Rapid Communications in Mass Spectrometry, 2008, 22(22): 3501- 3509. [16] S. Yan, G. Wu. Analysis on folding of misgurin using 2-dimen- sional HP model. Proteins: Structure, Function, and Bioinforma- tics, 2012, 80(3): 764-773. [17] S. Yan, G. Wu. Detailed folding structures of M-lycotoxin-Hc1a and its mutageneses using 2-dimensional HP model. Molecular Simulation, 2012, 38(10): 809-822. [18] S. Yan, G. Wu. Detailed folding structures of Kappa-conotoxin RIIIJ and its mutageneses obtained from 2-dimensional HP model . Protein and Peptide Letters, 2 012, 1 9(5): 567-572. |