Hans Journal of Computational Biology
Vol.05 No.01(2015), Article ID:14888,9
pages
10.12677/HJCB.2015.51001
The Research on BLAST Accelerator with Parallel Multi-Seeds Detection and Extension
Fei Xia, Guoqing Jin, Chunlei Zhang
Electronic Engineering College, Naval Engineering University, Wuhan Hubei
Email: xcyphoenix@hotmail.com
Received: Feb. 6th, 2015; accepted: Feb. 21st, 2015; published: Feb. 28th, 2015
Copyright © 2015 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



ABSTRACT
BLAST adopts index-based approach to detect the matches between two substrings by looking up a large table and processing one match per query. In this paper, we propose a systolic array approach to detect string matches without using looking up tables. The pipelining systolic array is implemented as a multi-seeds detection and parallel extension engine to accelerate the first two stages of NCBI BLAST algorithm. Different from the index-based approach, our implementation con- sumes little memory resources and eliminates redundant string extensions. Our FPGA implementation achieves superior performance results in both of processing element number and clock frequency over related works in the area of FPGA BLAST accelerators. The experimental results show a speedup factor of more than 20× over the NCBI BLAST software running on a current main- stream PC platform.
Keywords:Multi-Seeds Detection, Hardware Acceleration, Bio-Sequence Searching, FPGA
具有同时多种子检测与并行扩展能力的BLAST算法加速器研究
夏飞,金国庆,张春雷
海军工程大学电子工程学院,湖北 武汉
Email: xcyphoenix@hotmail.com
收稿日期:2015年2月6日;录用日期:2015年2月21日;发布日期:2015年2月28日

摘 要
本文针对目前流行的BLAST算法存在的种子检测效率不高的问题,提出了一种不同于常规查询策略的并行多种子检测算法,并基于脉动阵列结构设计和实现了一种具备同时多种子检测与并行扩展能力的算法加速器,实现了对NCBI BLAST程序前两个阶段的加速。加速器阵列采用流水工作模式,能够动态计算所有单词的匹配点位置,避免了索引结构的存储开销;采用了阵列分组和并行种子收集策略减少了有效种子数量;采用组内种子合并策略避免了冗余扩展操作;采用多种子并行扩展策略实现了无阻塞的数据库搜索,显著提高了数据库搜索效率。实验结果表明,本设计在阵列规模和时钟频率上都优于相关研究工作,相对于目前主流的通用微处理器可获得20倍以上的加速效果。
关键词 :多种子并行检测,硬件加速,生物序列搜索,FPGA

1. 引言
BLAST (Basic Local Alignment Search Tool) [1] 是目前搜索效率最高、使用最广泛的启发式序列数据库搜索算法。目前大多数BLAST搜索工具运行于集群或并行计算机上,如Grid Blast [2] 、Cyberpara Blast [3] 、Blast Quest [4] 、Turbo Blast [5] 、Parallel Blast [6] 、Scala Blast [7] 等。随着生物序列数据库规模的急剧膨胀(GenBank的规模每18个月翻一番[8] ,目前数据库包含的序列数量超过1.7 × 108条,残基数量已接近1600亿),生物序列比对研究对计算能力的需求远远超过计算能力的增长。
FPGA (Field Programmable Gate Array)器件以其可编程特性、细粒度并行能力、丰富的计算资源、灵活的算法适应性和较低的硬件代价成为理想的算法加速平台。为了提高搜索效率,基于FPGA平台的并行BLAST算法成为研究热点。近年来备受关注的研究成果有Mercury BLASTn [9] -[11] 、Mercury BLASTp [12] 、RC-BLAST [13] 、Tree-BLAST [14] 、FPGA/FLASH [15] 、Multi-engine BLASTn [16] [17] ,并且产生了一批商用计算平台,如BEE2 [18] 、CLC Cube [19] 、Mitrion [20] 和DeCypher [21] 等。以上工作的共同特点是使用FPGA对算法中计算简单、数据密集的部分进行加速,将目标序列中的单词作为数据流,使其逐一通过算法加速器,在请求和目标序列之间寻找匹配的种子(seed),然后将其扩展为无空位的HSP片段(High Score Pairs),最后将HSP发送给通用微处理器执行进一步扩展和生成统计结果。
目前绝大多数硬件并行方案都采用基于索引表的搜索方法,通过建立表格来记录请求序列中单词出现的位置,然后通过查表的方法寻找种子。这种方法存在两个问题:首先,由于访存端口的限制,每次只能查询一个单词,也就意味着最多只能发现一个种子;其次,索引表的存储和访问开销将会成为系统的性能瓶颈。例如,Mercury BLASTn [10] 和Mitrion [20] 使用了基于Hash表的查找策略,根据请求序列建立Hash表,然后将目标序列中的单词作用于Hash函数并查表,匹配则说明找到了一个种子。由于FPGA的存储资源有限,Hash表通常存储在FPGA片外,这样外部存储器访问延迟将会成为处理过程的瓶颈。RC-BLAST [13] 和BEE2 [18] 采用了基于索引表方法:首先为请求序列中的单词建立位置索引,然后以目标序列中的单词作为地址查询索引表,如果找到匹配则返回相应的位置信息。由于片内存储容量的限制,RC-BLAST [13] 假设任意单词在请求序列中最多只出现三次,但实际上单词在序列中出现的位置和次数都是随机的,这样的假设并不合理。与以上两种索引表相比,FPGA/FLASH [15] 采用了更新颖的设计:它为请求序列和整个数据库都建立索引,对每个单词,通过查找索引表便可以直接得到其在数据库中出现的位置和上下文字符,避免了对数据库的全面搜索;同时减少了扩展阶段对数据库的随机访问次数。但是该方法会导致极大的存储开销(例如为人类基因组数据库建立索引要耗费超过150 GB存储空间,是原始数据的50倍[15] )。随着生物序列原始数据量的增长,这种方法带来的存储开销将难以承受。为了进一步提高搜索效率,Multi-engines BLASTn [17] 设计了64个独立的搜索引擎将请求序列与数据库中的64条目标序列同时进行比较,但就每一个引擎而言,仍采用与RC-BLAST和BEE2相同的基于位置索引的搜索方法。从本质上说,以上工作都采用了单种子检测方法,即每个时钟周期最多只能发现一个种子。最近,Mercury BLASTp [12] 使用了双种子搜索引擎来加速种子检测,但是该方法仍然基于位置索引表,通过复制逻辑模块(包括查找表和单词命中模块)来提高种子检测效率。
除了上述基于查表的方法外,还存在一类基于算术/逻辑运算的搜索方法,Tree-BLAST [14] 是其中的典型代表。Tree-BLAST没有采用基于种子的传统启发式搜索策略,而用直接打分的方法寻找高分片段对。由于需要给每4个处理单元分配一个存储模块实现替换矩阵的存储,片内存储资源限制了请求序列的规模,最大只能支持包含1024个碱基字符的请求序列。
2. BLAST算法并行计算结构
图1是硬件BLAST算法并行计算结构,算法核心逻辑由IO接口和PE阵列控制器、片内存储器、多种子检测阵列、种子收集与合并模块和并行扩展模块构成。IO接口和PE阵列控制器实现与主机的数据交互,PE阵列初始化以及数据格式转换,并控制目标序列以步进的方式进入PE阵列。片内存储器由多个存储体构成,用于保存请求和目标序列的多个副本,为并行扩展提供所需的序列片段。算法核心与两个外部存储器相连,从Memory#1中载入目标序列,输入多种子检测阵列,经过种子检测、合并和无空位扩展,将HSP列表写入Memory#2。
3. 多种子检测阵列
本阶段的功能是在请求序列和目标序列中搜索长度为3氨基酸字符的匹配单词串。假设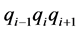 和
和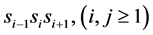 分别是查询和目标序列中长度为3的单词,如果条件
分别是查询和目标序列中长度为3的单词,如果条件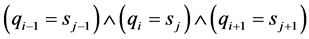 成立则意味着单词匹配成功,即种子被成功检测。
成立则意味着单词匹配成功,即种子被成功检测。
图2为PE并行多种子检测算法,L为PE阵列长度。种子检测和位置报告是PE的两个主要功能。处理阶段语句S3判断种子检测条件。种子的位置信息包括在请求和目标序列中的偏移以及目标序列在数据库中的编号,分别由初始化阶段语句S2、处理阶段的语句S1和S2动态生成。
图3是并行多种子检测阵列的结构。阵列由PE单元串联构成。请求序列寄存在阵列中(每个PE存储一个氨基酸字符),数据库中的序列依次流过PE阵列。PE[i] (阵列中的第i个PE)接收来自相邻PE发出的字符对 和
和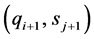 的比较结果,并比较字符对
的比较结果,并比较字符对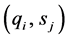 ,判断是否检测到种子。所有PE并行执行上述操作,因此阵列具备多种子检测能力(图3所示的阵列同时检测出两个种子:PE2发现AKL,PE3发现KLP)。
,判断是否检测到种子。所有PE并行执行上述操作,因此阵列具备多种子检测能力(图3所示的阵列同时检测出两个种子:PE2发现AKL,PE3发现KLP)。
图4是PE模块结构框图,中部虚线框内的3输入与门实现种子检测,是PE模块的核心。左右相邻PE产生的字符对匹配标志与当前PE产生的匹配结果一起送入与门的输入端。当这3个输入信号均为TRUE时,PE将单词匹配结果置为1。图中上下两部分所示的3个累加器负责生成匹配点(种子)位置。如果当前输入字符为序列结束标志,则将序列位置寄存器清零,同时目标序列ID寄存器加1。序列停顿信号和阵列停顿信号分别用于控制初始化和搜索阶段PE的工作状态。由于单词匹配结果依赖于3对氨基酸字符的比较结果,因此匹配结果的生成是PE的关键路径。时序分析表明,该路径的延时小于3 ns,不会
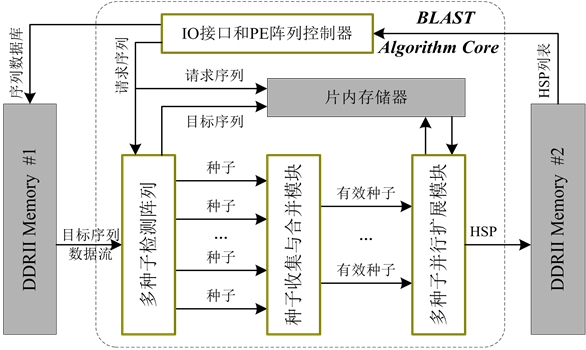
Figure 1. Parallel structure of BLAST algorithm
图1. BLAST算法并行计算结构

Figure 2. The seeds detecting algorithm for each PE
图2. 并行多种子检测算法

Figure 3. Structure of multi-seeds detection array
图3. 并行多种子检测阵列结构
成为FPGA实现的瓶颈。
4. 设计实现与优化
采用并行多种子检测有以下好处:1) 能够有效提高搜索程序的单词匹配能力。2) 阵列每次报告的种子都位于打分矩阵的同一条对角线上,便于对其进行过滤。3) 能提高数据吞吐率,有助于减少扩展阶段的空闲等待,提高资源利用率。另一方面,在有效提高种子检测效率的同时对后端处理能力提出了更高要求:1) 如何快速实现对多种子的收集将成为提高实际搜索效率的关键。2) 阵列同时报告的多个种子处于矩阵的同一条对角线上,这意味着可能存在连续的种子,对这些种子执行扩展将导致出现相同的结果。3) 单种子的扩展操作是串行的,扩展能力将成为搜索的瓶颈。为了提高阵列搜索效率,本文通过阵列分组实现并行种子搜集,通过组内种子合并实现种子过滤,通过多种子并行扩展匹配阵列的种子检测能力。
4.1. 阵列分组和并行种子收集
如图5所示,将多种子检测阵列划分为若干个PE组,同时将种子收集与合并模块分割为若干子模块,与PE组一一对应,并行收集PE组报告的种子,并将其写入局部FIFO。局部FIFO中的数据经过多级合并后进入Hit FIFO,随即执行无空位扩展。
分组策略的好处是控制了模块的逻辑量和互连端口的规模,优化了硬件逻辑设计。通过对PE阵列和种子合并模块进行分组,将两者之间庞大的多路选择器(MUX)拆分为规模较小的子模块(subMUX),从而消除了FPGA设计瓶颈。为了保证设计平衡,需要控制划分的粒度。实验表明由64个PE构成一组是最佳选择。由于将多路选择器的规模由512选1缩小为64选1,由8个组构成的PE阵列的时钟频率可由分组前的55 MHz提升至156 MHz,并且随着阵列规模的增长,频率不会出现明显降低。分组优化的代价是增加了多级局部FIFO和FIFO合并模块,局部FIFO使用FPGA片内存储资源实现,FIFO级数等于log2G,与庞大的多路选择器相比,FIFO合并模块消耗的逻辑资源可以忽略不计。
4.2. 组内种子合并
由于种子扩展过程是串行的,并行多种子检测对后端的扩展能力提出了很高的要求,本文通过组内
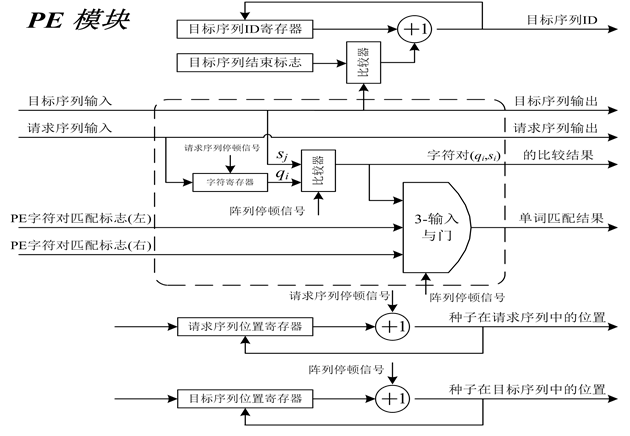
Figure 4. PE module structure
图4. 细粒度并行BLAST算法PE结构
种子合并策略来减少对扩展阶段的压力。而且由于PE阵列每次报告的多个种子都位于矩阵同一反对角线上。当请求和目标序列中的两个片段相似度很高时,组内连续的多个PE会同时发出种子命中信号,但这些种子在序列中所处的位置是连续的,属于同一个高分片断。如果将每个种子都传递给扩展模块将会导致不必要的重复扩展。为此,本章采用组内相邻种子合并策略减少有效种子的数量。
图6是种子收集与合并模块端口连接示意图。合并的好处是可以减少有效种子的数量,减轻扩展阶段的压力;同时通过消除冗余扩展来提高扩展效率。种子合并过程是:一旦PE阵列检测到单词匹配,便暂停目标序列的传递,并寄存标志位。根据单词匹配标志合并相邻种子,同时将合并后的有效种子传递给扩展模块,当所有匹配标志处理完后再重启搜索阵列。
图7是PE组内相邻种子合并算法。种子收集与合并子模块寄存对应PE组产生的单词匹配标志,并判断是否有种子被发现(S1)。语句S4寻找并记录匹配标志寄存器中“首1”的位置(1代表检测到匹配的单词)。S6中的循环将多个连续种子合并为一个有效种子。假设当前PE组处于图3所示的状态,PE2和PE3同时检测到种子(PE2发现AKL,PE3发现KLP)。如果不进行合并优化,种子收集与合并子模块会将这两个种子依次写入局部FIFO,随后将执行两次扩展。通过将两者合并为一个有效种子AKLP,则只需执行一次扩展。语句S7判断是否发现足够长的精确匹配片断。如果相邻种子的数量大于8 (对应10个
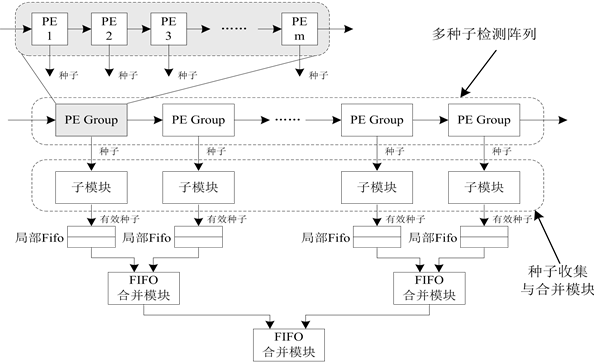
Figure 5. Array partition and hierarchical merging
图5. 多种子检测阵列分组与种子分级合并过程

Figure 6. Port connection module
图6. 种子收集与合并模块端口连接示意图

Figure 7. Successive seeds merging algorithm
图7. PE组内相邻种子合并算法
以上连续字符对的精确匹配),扩展模块将不会对该有效种子进行处理而直接将其输出。对于上述情况,如果不采用合并优化策略,记录匹配位置需要8个时钟周期,加上扩展开销,共需要72个时钟周期。而且扩展模块会报告发现8个HSP (而实际上只有1个有效HSP)。采用合并策略后只需要8个周期便可发现该HSP,减少了不必要的重复扩展。
4.3. 多路并行扩展
无空位扩展的过程是根据种子所处的位置读出两条长度为K的蛋白质子序列q和s,然后将子序列中的字符对 进行逐一比较,然后将分值累加。由于PE阵列具备并行多种子检测能力,而扩展阶段由于序列存储器访问的限制,每个周期只能读出一对氨基酸字符,导致种子的扩展能力与检测能力不匹配。即使通过合并策略减少了有效种子的数量,但当阵列规模较大时,仍可能由于扩展能力不足导致阵列出现阻塞。
进行逐一比较,然后将分值累加。由于PE阵列具备并行多种子检测能力,而扩展阶段由于序列存储器访问的限制,每个周期只能读出一对氨基酸字符,导致种子的扩展能力与检测能力不匹配。即使通过合并策略减少了有效种子的数量,但当阵列规模较大时,仍可能由于扩展能力不足导致阵列出现阻塞。
为了解决上述问题,本章采用了如图8所示的多路并行扩展策略平衡两阶段的处理能力。由于所有的扩展模块都需要同时访问序列存储器获取扩展所需的序列片断,因此为每个扩展模块都设置了独立的序列存储器副本(包括QryMem和Sub Mem,分别存储请求和目标序列),为并行扩展提供足够的存储端口。这样来自不同Hit FIFO中的多个种子便可以并行地执行扩展。
5. 实验与性能分析
5.1. FPGA实现
我们在XC5VLX330和XC4VLX160芯片上分别实现了包含3072和2048个PE的搜索阵列。与Tree- BLAST [14] 不同的是,阵列规模主要受限于逻辑而不是存储资源。从表1可以看到,使用同样的芯片XC4VLX160,本设计用更少的存储资源实现了更长的PE阵列,阵列规模为Tree-BLAST的2倍,同时设计频率也高于相关工作。
加速器的主要存储开销为序列存储器和种子合并FIFO。以规模为3072个PE的阵列为例,序列存储
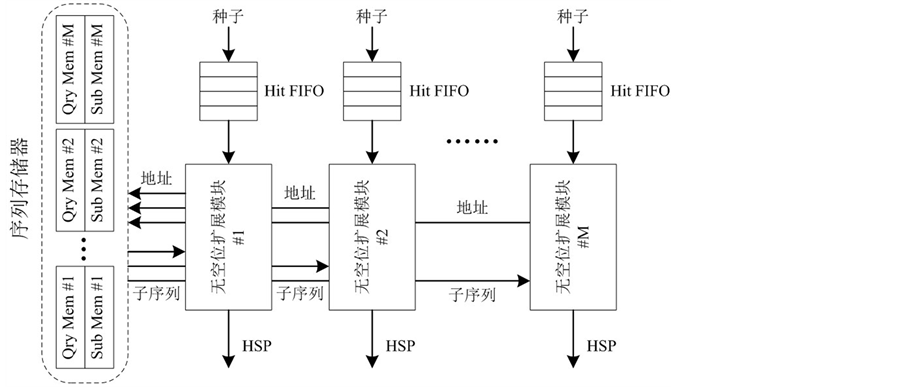
Figure 8. Structure of multi-channel parallel extension
图8. 多路并行扩展结构
Table 1. Performance results and comparison
表1. 基于线性阵列的BLAST算法加速器实现结果对比
开销为36 K × 5 bit,加上FIFO (16 K × 32 bit),共计692 Kbits,仅占XC5VLX330存储容量的6%。而基于索引表的搜索方法均受限于FPGA存储资源。RC-BLAST [13] 在Xilinx 4085XLA上实现了最大规模为64 K × 64 bits的索引表,但只能记录每个单词在请求序列中的3次出现。Mercury BLASTn [10] 和Mitrion BLAST [20] 将Hash表移至外部存储器中,导致存储访问延迟成为流水线设计的瓶颈。与基于索引表的RC-BLAST和基于线性阵列的Tree-BLAST相比,本设计对存储资源的需求分别降低约90%和50%。存储需求的降低减少了存储访问的复杂度,进而减小了FPGA布局布线的难度。
5.2. 加速性能
测试采用的搜索软件为NCBI BLAST [22] ,版本号2.2.16,使用Blosum62蛋白质替换矩阵。软件运行环境为Windows XP SP3操作系统,Visual Studio 2008 开发环境,Visual C++编译器,版本号为15.00.30729.01。
实验输入的请求序列取自Swiss-Prot数据库,长度为128~8192 bps,搜索对象为Swiss-Prot (从EBI下载[23] ,包含274,295条序列,共100,686,439个氨基酸字符)。BLASTn用于DNA数据库搜索,请求序列取自drosoph.nt数据库,长度为128~8192 bps,搜索对象为drosoph.nt [24] ,包含1170条DNA序列。每个输入长度随机选择10条序列,每条序列运行3次,取平均执行时间。表1中4 K和8 K长度下的硬件执行时间为模拟结果。
从表2可以看到,软件搜索时间随着序列规模的增长而急剧增加(BLASTp由128 bps时的1901 ms增加至8 Kbps时的61,162 ms,BLASTn由128 bps时的6225 ms增加至8 Kbps时的15,344 ms),而硬件
Table 2. Execution time (ms) and speed-up for different queries
表2. BLASTp和BLASTn算法加速效果(时间单位:毫秒)
搜索时间增长很缓慢。主要原因是随着序列长度的增加,软件建立索引表和执行搜索过程的开销都显著增加,而硬件搜索时间等于数据库流过阵列的时间加上停顿时间(即L + S + 停顿时间)。在这三个参数中,S为常量(等于数据库包含的字符数),与S相比,请求序列长度L的增加可以忽略不计,而停顿时间与种子数量直接相关。由于采用了多种优化策略施,在目标数据库确定的情况下,L的增加并不会导致种子数量和扩展开销急剧增加,所以加速比会相应增大。当输入序列长度为3072 bps时,使用算法加速器执行BLASTp和BLASTn程序搜索目标数据库,可分别获得约17倍和10倍的加速比;而模拟结果显示,当序列长度为8 Kbps时,可获得27倍和11倍的加速效果。
5.3. 性能功耗比
目前单个微处理器的平均功耗约为70 W~ 95 W [25] ,而使用Xilinx ISE Xpower工具的功耗分析结果表明,算法加速器功耗不超过10 W,XC5VLX330芯片的最大功耗小于30 W,仅为微处理器功耗的30%左右。因此就性能功耗比而言,基于FPGA平台的细粒度并行BLAST算法加速器方案明显优于传统的基于通用微处理器的解决方案。
6. 结论
本文提出一种基于脉动阵列结构的具备同时多种子检测与并行扩展能力的BLAST算法加速器,并构建原型系统实现了对NCBI BLAST程序前两个阶段的加速。
文章提出了同时多种子检测算法,并设计了基于线性结构的并行多种子搜索阵列;采用阵列分组和并行种子收集、组内种子合并和多种子并行扩展策略实现了无阻塞的数据库搜索,成功对BLAST算法实现硬件加速,相对于通用微处理器取得了20倍以上的加速效果。此外,本文提出的多种子并行检测方法同样适用于对多种启发式搜索算法的种子(单词)检测阶段实现硬件加速。
基金项目
国家自然科学基金,基于异构平台的高复杂度生物序列分析算法并行化研究(61202127),湖南省2013年学位与研究生教育专项基金(YB2013B008)。
文章引用
夏 飞,金国庆,张春雷, (2015) 具有同时多种子检测与并行扩展能力的BLAST算法加速器研究
The Research on BLAST Accelerator with Parallel Multi-Seeds Detection and Extension. 计算生物学,01,1-10. doi: 10.12677/HJCB.2015.51001
参考文献 (References)
- 1. Altschul, S.F., Gish, W., Miller, M., Myers, E.W. and Lipman, D.J. (1990) Basic local alignment search tool. Journal of Molecular Biology, 215, 403-410.
- 2. Kim, T.K., et al. (2005) HGBS: A hardware-oriented grid blast system. Proceedings of 5th International Symposium on Cluster Computing and the Grid, 1, 520-526.
- 3. Yutao, Q. and Feng, L. (2003) Cyberparablast: The parallelized blast web server. Proceedings of 2nd International Conference on Cyber-worlds (CW’03), 3-5 December 2003, 474-477.
- 4. Farmerie, W.G., Hammer, J., Liu, L. and Schneider, M. (2003) Having a blast: Analyzing gene sequence data with blastquest. Proceedings of 14th International Workshop on Database and Expert Systems Applications (DEXA’03), Prague, 1-5 September 2003, 37-41.
- 5. Bjornson, R., Sherman, A., Weston, S., Willard, N. and Wing, J. (2002) Turboblast: A parallel implementation of blast built on the turbo-hub. Proceedings of 16th International Parallel and Distributed Processing Symposium (IPDPS’02), Ft. Lauderdale, 15-19 April 2002, 183-190.
- 6. Lin, H., Ma, X., Chandramohan, P., Geist, A. and Samatova, N. (2005) Efficient data access for parallel blast. Proceedings of the 19th International Parallel and Distributed Processing Symposium (IPDPS’05), 1, 8 p.
- 7. Oehmen, C. and Nieplocha, J. (2006) Scalablast: A Scalable Implementation of BLAST for High-Performance Data- Intensive Bioinformatics Analysis. IEEE Transaction on Parallel and Distributed Systems, 17, 740-749.
- 8. National Center for Biotechnology Information (2014) Notes on GenBankstatistics. http://www.ncbi.nlm.nih.gov/genbank/statistics
- 9. Buhler, J.D., Lancaster, J.M., Jacob, A.C. and Chamberlain, R.D. (2007) Mercury Blastn: Faster DNA sequence comparison using a streaming hardware architecture. Proceedings of 3rd Annual Reconfigurable Systems Summer Institute (RSSI’07), Urbana, 17-20 July 2007, 10 p.
- 10. Krishnanurthy, P., Buhler, J., Chamberlain, R., et al. (2007) Biosequence similarity search on the mercury system. Proceedings of 15th IEEE International Conference on Application-Specific Systems, Architectures and Processors (ASAP’04), 365-375. Journal of VLSI Signal Process System, 49, 101-121.
- 11. Lancaster, J.M., Buhler, J.D. and Chamberlain, R.D. (2009) Acceleration of ungapped extension in Mercury BLAST. Journal of Microprocessors & Microsystems, 33, 281-289.
- 12. Jacob, A., Lancaster, J., Buhler, J. and Chamberlain, R.D. (2007) FPGA-accelerated seed generation in Mercury BLASTP. Proceedings of the 15th Annual IEEE Symposium on Field-Programmable Custom Computing Machines, Napa, 23-25 April 2007, 95-106.
- 13. Muriki, K. and Underwood, K.D. and Sass, R. (2005) RC-BLAST: Towards a portable, cost-effective open source hardware implementation. Proceedings of the 19th IEEE International Parallel and Distributed Processing Symposium, Denver, 4-8 April 2005, 196-203.
- 14. Herbordt, M.C., Model, J., Gu, Y.F., Sukhwani, B. and Van Court, T. (2006) Single pass, blast-like, approximate string matching on FPGAs. Proceedings of 14th Annual IEEE Symposium on Field-Programmable Custom Computing Machines, Napa, 24-26 April 2006, 217-226.
- 15. Lavenier, D., Xinchun, L. and Georges, G. (2006) Seed-based genomic sequence comparison using a FPGA/FLASH accelerator. Proceedings of IEEE International Conference on Field Programmable Technology, Bangkok, 13-15 December 2006, 41-48.
- 16. Sotiriades, E. and Dollas, A. (2007) Design space exploration for the BLAST algorithm implementation. Proceedings of the 15th Annual IEEE Symposium on Field-Programmable Custom Computing Machines, Napa, 23-25 April 2007, 323-326.
- 17. Sotiriades, E., Kozanitis, C. and Dollas, A. (2006) FPGA based architecture for DNA sequence comparison and database search. Proceedings of the 20th IEEE International Parallel and Distributed Processing Symposium, Rhodes Island, 25-29 April 2006, 186-193.
- 18. Chang, C. (2012) BLAST implementation on BEE2. Technical Report, Electrical Engineering and Computer Science University of California at Berkeley. http://bee2.eecs.berkeley.edu
- 19. CLC desktop Hardware-acceleration. White Paper on CLC Bioinformatics Cube, 2006. http://www.clccube.com
- 20. Mitrion. Inc. (2009) Ncbi blast accelerator. http://www.mitrionics.com
- 21. Timelogic Biocomputing Solisions (2010) Timelogic decypher BLAST engine intro-duction. http://www.timelogic.com/decypher_blast.html
- 22. NCBI BLAST software download, 2010. ftp://ftp.ncbi.nih.gov/blast/
- 23. European Bioinformatics Institute, EBI (2008) EBI database web. http://www.ebi.ac.uk/uniprot/database/download.html
- 24. National Center for Biotechnology Information (2009) BLAST database web. http://blast.ncbi.nlm.nih.gov/Blast.cgi
- 25. General purpose micro-processor power dissipation statistic. List of CPU power dissipation figures, 2014. http://en.wikipedia.org/wiki/ListofCPUpowerdissipation
