Hans Journal of Agricultural Sciences
Vol.07 No.03(2017), Article ID:21167,7
pages
10.12677/HJAS.2017.73033
Analysis of Genetic Diversity with SSR Markers on Winter Barley Germplasm Resources in Tibet
Xia Meng1, Xingquan Zeng2, Qimei Wangmo2, Nima Tashi2*
1College of Agriculture and Animal Husbandry, Tibet University, Linzhi Tibet
2Academy of Agriculture and Animal Husbandry, Lhasa Tibet
*通讯作者。

Received: Jun. 8th, 2017; accepted: Jun. 20th, 2017; published: Jun. 28th, 2017

ABSTRACT
In order to clarify the genetic diversity of the Tibetan winter barley germplasm resources, and provide bases for winter barley breeding, genetic diversity of 94 winter barley samples collected from Tibet were analyzed, based on the 32 pairs of SSR primers distributed in 7 chromosomes of barley. The results showed that, 115 polymorphic bands were detected from 32 pairs of primers totally, and in an average, every pair of primers had been amplified to be 3.6 polymorphic bands, which indicated that, the genetic basis of Tibet winter barley germplasm was wide, and multi different alleles existed, who owned a wealth of genetic diversities. Cluster analysis divided materials into six groups, and the sources of materials were independent to the related traits of SSR primer loci for study. There were no correlations between the source regions of materials and the clusters based on SSR polymorphism.
Keywords:Tibet Winter Barley, SSR, Genetic Diversity
西藏冬青稞种质资源的SSR标记遗传 多样性分析
孟霞1,曾兴权2,其美旺姆2,尼玛扎西2
1西藏农牧学院,西藏 林芝
2西藏农牧科学院,西藏 拉萨

收稿日期:2017年6月8日;录用日期:2017年6月20日;发布日期:2017年6月28日

摘 要
为了明确西藏冬青稞种质资源的遗传多样性,并为冬青稞育种提供依据,利用分布于青稞7条染色体上的32对SSR引物对来自西藏的94份冬青稞材料的遗传多样性进行了分析。结果表明,32对引物共检测出115条多态性条带,平均每对引物扩增出3.6条多态性带,表明西藏冬青稞种质资源遗传基础较广,蕴藏着多种不同的等位基因,具有丰富的遗传多样性。聚类分析将材料分为六组,材料的来源于所研究的SSR引物位点相关性状具有独立性,基于SSR多态性的聚类与材料来源地区无相关性。
关键词 :西藏冬青稞,SSR,遗传多样性

Copyright © 2017 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/


1. 引言
大麦(Hordeum vulgare L.)是世界上古老的粮食兼饲料作物之一,具有重要的经济价值。中国近缘野生大麦是产于青藏高原及其周边地区的一年野生大麦,是我国青藏高原特有的宝贵遗传资源 [1] [2] [3] [4] 。研究西藏近缘野生大麦的遗传多样性不仅对于揭示栽培大麦的起源与进化问题,而且在保护大麦遗传资源,以及指导大麦育种方面都有重要的意义。
简单序列重复(simple sequence repeat, SSR)也称微卫星DNA (microsatellite DNA),是一类由几个核苷酸(一般为1~6个)为重复单位组成的长达几十个核苷酸的串联重复序列 [5] 。其中重复单位数目及重复次数存在高度变异,而每个SSR两侧的序列一般是相对保守的单拷贝序列,根据两端序列的保守性,设计引物对基因组DNA进行PCR扩增,扩增产物经聚丙烯酰胺凝胶电泳分离获得扩增指纹图,分析微卫星序列多态性,从而揭示样本间的遗传多样性 [4] 。该标记具有多态性高、重复性好等优点,因此被作为分子标记广泛应用于遗传多样性分析、遗传图谱的构建及亲缘关系鉴定等方面 [4] 。
本研究从青稞7对染色体的不同位置随机选取了46对SSR引物,对94份不同的大麦品种进行遗传多样性分析,并从中筛选出能扩增出有效多态性的32对引物进行最终分析,探讨西藏近缘野生大麦的遗传多样性,为大麦相关研究提供分子水平上的理论依据。
2. 材料与方法
2.1. 材料
供试材料共94份,其中73份由西藏农牧科学院提供的,21份是林芝、昌都两地区收集的当地种植的冬青稞品种。
2.2. SSR引物
从Liu等(1996) [6] 发表的大麦7个不同的连锁群上随机选取46对SSR引物。产生多态性带的32对SSR引物的名称和序列见表1。引物由上海英俊生物技术有限公司合成。
2.3. 大麦基因组DNA的提取
每份大麦材料取幼苗叶片约0.3 g置于1.5 mL离心管中,加入液氮研磨成粉末,采用CTAB-氯仿抽提法提取DNA:取研磨好的样品,每管加入600 μL提取缓冲液(
Table 1. Name and series of 32 pairs of SSR primers
表1. 32对SSR引物名称及序列
2.4. SSR扩增及聚丙烯酰胺凝胶电泳
PCR反应总体积为10 μL,其中包括1 μL dNTPs (200 μmol/L),1 μL 10 × PCR Buffer (100 mmol/L Tris-HCl (pH 8.5)),1 μL MgCl2 (2.5 mmol/L),20 ng模板DNA,0.5 U rTaq DNA聚合酶,SSR引物(2 μmol/L) 1 μL,加水至10 μL。
PCR反应程序为:94℃预变性5 min;95℃变性30 s,56℃退火30 s,72℃延伸30s,共35个循环;最后一个循环结束后,在72℃延伸10 min。
SSR扩增产物用8%非变性聚丙烯酰胺(丙烯酰胺与甲叉双丙烯酰胺比例为39:1)凝胶电泳分离,0.1%硝酸银染色。
2.5. 数据统计分析
对筛选出的具有多态性的引物,统计相同迁移率位置上的SSR扩增产物,扩增条带存在时赋值为1,否则赋值为0。标准遗传距离(Genetic Distance, GD)计算公式为:GD = −ln[2Nij/(Ni + Nj)] [7] ,其中Ni和Nj分别为i和j两材料的总条带数,Nij为i和j两材料的共有条带数。遗传相似性聚类分析采用UPGMA方法(Unweighted Pair-Group Method with Arithmetic Mean,算术平均非加权配组法)。
3. 结果与分析
3.1. SSR分子标记多态性
本研究选取了分布于大麦7个染色群上的46对SSR引物对来自中国西藏的94份不同的冬青稞材料进行PCR扩增,其中32对能扩增出多态性DNA片段并且有很好的重复性,并最终用于遗传多样性分析。所用的32对引物共检测出115条多态性条带,平均每对引物扩增出3.6条多态性带。其中SCSSR09398、Bmac0126、SCSSR03907、Bmac0064这4对引物均检测出6条多态性带,为其中多态性较高的位点。SCSSR02093、SCSSR08238、GBM1252、GBM1419、SCSSR12344等8对引物检测出的多态性条带数最少,均只检测出2条多态性条带。32对引物分别检测出的多态性条带数目见表2。图1为引物SCSSR09398对其中94份大麦材料的基因组DNA经PCR扩增后聚丙烯酰胺凝胶电泳的结果。
3.2. 聚类分析
为了确定94份大麦材料基因型间的遗传关系,利用SSR遗传距离矩阵按UPGMA方法进行了聚类分析,构建了各供试基因型的亲缘关系图(图2)。
由图2可知,第一组1份材料,是亲本23;第二组4份材料,分别是亲本54、亲本56、亲本26、亲本12。第三组包含了87份材料,其中57份是冬青稞亲本种质,30份是收集来的冬青稞种质资源;第四组包含了来自拉萨72份,占该材料的76.60%,占拉萨材料的91.14%,林芝12份,昌都3份。第五组一份材料,是亲本15。第六组包含了2份材料,分别是亲本41和亲本44。
另外,在94份材料中,25份育成品种全部分布在第Ⅲ组。本实验选择的46对引物能较好地将供试材料区分,以上结果表明,品种来源与所研究的SSR引物位点相关性状具有独立性,基于SSR多态性的聚类与材料来源地区无相关性。
4. 讨论
本研究所选用的46对SSR引物中,有14对引物在94份材料中没有扩增出多态性条带,其他32对引物都扩增出了多态性带。表明所选标记位点存在丰富的变异,充分说明西藏冬青稞遗传基础较广,蕴藏着多种不同的等位基因,具有丰富的遗传多样性,可为冬青稞重要性状遗传特性、基因资源挖掘和品
Table 2. The chromosome locations of 32 pairs of SSR primers and the amplification results
表2. 32对SSR引物所在染色体及其扩增结果
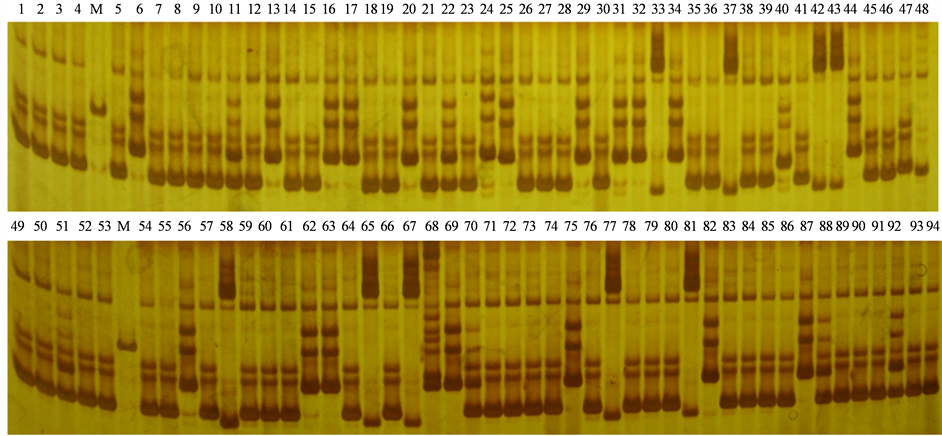
Figure 1. DNA amplification of SSR primer SCSSR09398 for 94 barely genomes
图1. SSR引物SCSSR09398对94份大麦基因组DNA的扩增结果
种选育等方面的研究提供参考。
聚类图显示了不同材料间的遗传距离,通过聚类分析将94份冬青稞材料分成5组,不同生态区参试材料SSR遗传多样性不同。拉萨是冬青稞遗传多样性最丰富的地区。另外育成品种全部分布在同一组群,可看出育成品种间的遗传距离较近,遗传基础较窄。说明冬青稞育种中加强亲缘关系更远的材料进行杂交组配使用,加强不同地区间资源的交换和基因交流,有利于拓宽冬青稞材料的遗传背景,以培养出变异类型更丰富的后代群体,选育新品种。

Figure 2. Genetic clustering diagram for the SSR marks of 94 Highland barley
图2. 94份青稞材料SSR标记的遗传聚类图
基金项目
科技厅。
文章引用
孟 霞,曾兴权,其美旺姆,尼玛扎西. 西藏冬青稞种质资源的SSR标记遗传多样性分析
Analysis of Genetic Diversity with SSR Markers on Winter Barley Germplasm Resources in Tibet[J]. 农业科学, 2017, 07(03): 245-251. http://dx.doi.org/10.12677/HJAS.2017.73033
参考文献 (References)
- 1. 胡颂杰. 西藏农业概论[M]. 第一版. 四川: 科学技术出版社, 1995: 345-428.
- 2. 马得泉. 中国西藏大麦遗传资源[M]. 第一版. 北京: 中国农业出版社, 2002.
- 3. 张亲. 两个大麦新矮杆基因的SSR标记[J]. 植物遗传资源学报, 2004, 5(2): 105-109.
- 4. 冯宗云, 张义正, 张立立, 凌宏清. 应用微卫星标记研究西藏野生二棱大麦的遗传多样性及地理分化[J]. 高技术通讯, 2003, 13(10): 46-53.
- 5. Russell, J.R., Fullrr, J.D., Macaulay, M., et al. (1997) Direct Comparison of Levels of Genetic Variation among Barley Accessions Detected by RFLPs, AFLPs, SSRs and RAPDs. Theoretical and Applied Genetics, 95, 714-722.
- 6. Liu, Z.W., Biyashev, R.M. and Saghai Maroof, M.A. (1996) Development of Simple Sequence Repeat DNA Markers and Their Integration into a Barley Linkage Map. Theoretical and Applied Genetics, 93, 869-876.
- 7. Nei, M. (1973) Analysis of Gene Diversity in Subdivided Populations. Proceedings of the National Academy of Sciences, 70, 3321-3323.
