Hans Journal of Biomedicine
Vol.05 No.03(2015), Article ID:15751,10
pages
10.12677/HJBM.2015.53005
Methods, Principles and Application of Gene Editing
Yuchang Zhu1, Xiaojiang Zheng1, Yibing Hu2*
1School of Biological Science and Technology, Hubei University for Nationalities, Enshi Hubei
2College of Resources & Environmental Sciences, Nanjing Agricultural University, Nanjing Jiangsu
*通讯作者。
Email: *huyb@njau.edu.cn
Received: Jul. 1st, 2015; accepted: Jul. 24th, 2015; published: Jul. 27th, 2015
Copyright © 2015 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



ABSTRACT
Fast development of gene editing technologies provides more powerful tools for gene function analysis. Now researchers can easily manipulate targeted gene with the Zinc Finger Nuclease (ZFN), Transcription Activation Like Effector Nuclease (TALEN) and Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR-associated proteins (CRISPR) technologies emerged in the last decade. These technologies revolutionized gene functional analysis and medical treatment. In this review, several typical gene editing technologies were listed, and their principles, characteristics and application were discussed.
Keywords:Gene Editing, Methods, Principles, Application

基因编辑技术的方法、原理及应用
朱玉昌1,郑小江1,胡一兵2*
1湖北民族学院生物科学与技术学院,湖北 恩施
2南京农业大学资源与环境科学学院,江苏 南京
Email: *huyb@njau.edu.cn
收稿日期:2015年7月1日;录用日期:2015年7月24日;发布日期:2015年7月27日

摘 要
基因编辑技术的飞速发展为基因功能研究工作提供了更多有力的工具。近十年来相继出现的锌指核酸酶(ZFN)、转录激活子样效应因子核酸酶(TALEN)以及规律性间隔的短回文序列重复簇(CRISPR)已经可以让研究人员很方便的对特定的目的基因进行突变、修复或替换等操作,从而为生物学研究及医学治疗领域带来革命性的变化。本文就有代表性的基因编辑技术的种类、原理及其研究进展进行了综述,并对它们各自的特点及应用前景和值得进一步研究的问题进行了探讨。
关键词 :基因编辑,方法,原理,应用

1. 引言
随着DNA测序技术的快速进步,越来越多的物种基因组序列成为已知,但鉴定这些基因的功能仍然是一项十分艰巨的任务。作为鉴定基因功能最重要的策略,获得其突变体是优先考虑的手段。然而,无论是依赖自发突变还是通过物理、化学手段如射线和化学诱变剂处理,或者利用转座子以及T-DAN插入,都只能被动的获得随机出现的基因突变体材料,并且基因出现突变的频率通常较低。因此,虽然过去运用这些手段对很多基因的功能鉴定提供了巨大的帮助,但它们目前已经难以满足日益扩大的基因功能研究的需求。令人鼓舞的是,近十年来基因编辑技术的出现为克服上述矛盾开辟了新的途径,特别是锌指核酸酶(ZFN)和转录激活子样效应因子核酸酶(TALEN)以及最近发展起来的规律性间隔的短回文序列重复簇(CRISPR)技术,为基因编辑提供了前所未有的方便和快捷,吸引了全世界研究人员的广泛关注。
基因编辑就是通过对细胞基因组中目的基因的一段核苷酸序列甚至是单个核苷酸进行替换、切除,增加或者是插入外源的DNA序列,使之产生可遗传的改变 [1] - [3] 。与射线或化学诱变剂导致的DNA随机突变不同的是,基因编辑技术是定向改变基因的组成和结构,具有高效、可控和定向操作的特点。因此,基因编辑技术被麻省理工科技评论评为2014年十大突破性科学技术 (http://www.technologyreview.com/lists/technologies/2014/),其中CRISPR还获得2015年度生命科学突破奖(https://breakthroughprize.org/News/21)。
虽然各种基因编辑技术的原理及作用方式并不相同,但它们的共同之处是基因编辑都建立在使目标基因DNA产生双链断裂(Double Strand Breaking, DSB)的基础上。因为DNA分子单链断裂或缺失后容易被细胞内的各种修复机制所修复而不产生任何改变 [4] ,但DNA双链断裂的结果则有很大的不同。细胞内DNA双链断裂的修复有两种方式,即同源重组修复(Homologous Recombination, HR)和非同源末端链接修复(Non-Homologous End Join, NHEJ) [5] [6] 。当DNA发生双链断裂后,如果细胞内存在与裂口两端同源的序列,则进行同源重组修复,外源DNA片段可借此插入断裂序列中对原来的基因进行敲除或将外源基因插入基因组DNA而形成所谓的基因敲入;若细胞内无同源序列存在,双链断裂的DNA分子则通过NHEJ连接,由于无模板可以利用,这种“硬”连接容易导致碱基的缺失、增加或改变而引起突变。实际上,无论是HR修复还是NHEJ修复,都可以产生较于原来基因的不同变化。虽然DNA双链断裂对基因组而言是严重的伤害[7] ,但这种变化正是研究基因功能所需要的。下面我们就基因编辑技术的种类及其特点分别进行简单的介绍。
2. 同源重组(Homologous Recombination, HR)
从使基因发生定向改变这个角度而言,早期的基因编辑技术可以追溯到同源重组这种方法。基于同源重组的基因编辑被称为基因打靶或基因靶向技术(gene targeting),例如依赖大肠杆菌细胞内的Rec A或酵母的RAD54重组酶,基因靶向技术可以对目标基因或基因的部分序列进行替换、删除,或在细胞内存在同源序列的情况下插入外源DNA序列。运用同源重组对基因进行编辑的方法已经在微生物、植物和动物中取得了成功[2] 。不过,该方法的一个主要局限在于重组频率低,约为10−4~10−5 [2] 。虽然对微生物及培养的动物细胞系、甚至对一些苔藓植物如小立碗藓来说,同源重组技术可以获得满意的基因编辑效率,但对高等植物或动物,基于同源重组的基因靶向技术由于效率太低,在实践上难以广泛应用,尽管也有少数成功的例子[8] [9] 。据分析,高等动、植物中同源重组效率低下的原因可能在于这些细胞内同源重组酶的效率不高,因为有研究显示,转入外源的同源重组酶基因后,这些物种中的同源重组效率提高了一个数量级[8] [9] 。
3. 锌指核酸酶(Zinc Finger Nuclease, ZFN)
人工核酸酶ZFN可以算是第一种具有普遍适用性的基因编辑技术。我们知道,分子生物学中广泛使用的工具酶主要是II型限制性核酸内切酶,绝大多数情况下这些酶的切割位点位于其DNA识别序列之中。不过,IIS型的核酸内切酶(如Fok I)则不同,其识别序列和切割序列相距9个核苷酸,并且切割和识别功能分别由酶蛋白的不同结构域完成。人工核酸酶正是利用了IIS型核酸内切酶的这一特点,在保留它的非特异的酶切割功能结构域的基础上,将其DNA识别结构域用能够识别特定核苷酸序列的、人工合成的结构域取代,这样就构成了能够根据人们的需要而识别特定DNA序列并进行切割的人工核酸酶。
按此原理构建的锌指核酸酶ZFN就是由一系列锌指结构单元与Fok I的核酸酶切活性区域组合而成的(图1),它具有对特定的DNA序列进行识别和切割的能力。ZFN对不同DNA序列识别的机理在于组成其锌指蛋白基元(Motif)的种类及排列方式。锌指结构是很早就发现的一类能与DNA分子相互作用的蛋白结构基元。在人类基因组中,约有3%蛋白含有这样的结构[10] 。锌指结构基元一般由30个左右的氨基酸组成,其结合锌离子的保守氨基酸为四个半胱氨酸残基(Cys-)或两个半胱氨酸残基和两个组氨酸残基(His-)。空间结构上,锌指结构从N端到C端由两个反向的β平行结构和一个α螺旋组成。α螺旋的1、3、6位氨基酸残基分别特异性的结合其识别DNA分子中三个连续的碱基[11] [12] ,亦即不同的锌指结构基元中其α螺旋的1、3、6位上氨基酸残基是不同的,因此,将不同的锌指结构单位(通常3~6个)组合起来就可以形成识别18~36 bp的一段双链DNA序列。所以,选择不同的锌指结构单位组合起来并且和Fok I连接,就构成了能针对特定的目标序列进行切割的人工核酸酶(图1)。不仅如此,Fok I需要形成二聚体才具有酶切活性[13] ,虽然Fok I自身二聚化也能产生对DNA的切割作用,但切割效率极低并且容易产生非特异切割,所以在设计ZFN时,还需要对Fok I进行突变,使之不能形成同源二聚体;并且研究显示,当两个结合不同靶序列的突变Fok I相距5~6 bp就可以形成异源二聚体而具有酶切功能,这样设计的另一个优点是可以增加ZFN识别的特异性[14] 。
ZFN这种基因编辑方法在一系列的模式生物中都获得了成功[15] ,研究显示ZFN基因编辑效率约为30%,相对于同源重组,其编辑效率无疑具有了质的提高。不过,目前它还存在一些不足,一个突出的问题是所谓的脱靶效应,即合成的核酸酶并没有对预先设定的目标DNA序列进行识别和切割。出现这种情况的原因在于组成人工核酸酶的各个锌指结构单元之间存在相互影响,即存在所谓上下文依赖(Context-dependant),也就是说将不同锌指结构单位连接起来后,其DNA识别序列并不是它们单独存在时分别识别的核苷酸组序列的简单相加[16] [17] 。因此,这种脱靶效应很大程度上还是目前对锌指结构单

Figure 1. A schematic figure of ZFN components and two joining models of double strand breaks after ZFN nuclease cleavage (based on reference [15] )
图1. ZFN核酸酶结构及其工作原理示意图。图中显示ZFN的组成以及目标DNA经锌指核酸酶切割后的两种不同的连接(修复)方式(参考文献[15] )
元之间的相互影响缺乏足够了解造成的。由于受专利等因素的影响,不同的研究机构对他们研究出的结果并没有充分共享。相信随着以后对锌指结构单元之间相互影响的深入了解,ZFN脱靶问题可能会得到较好的解决。
4. 转录激活子样效应因子核酸酶TALEN (Transcription Activation Like Effector Nuclease)
在ZFN问世不久,另一种人工核酸酶TALEN也被开发出来。与ZFN一样,TALEN也是利用一种对DNA分子特异识别的蛋白结构与Fok I的酶切活性结构域组合形成的。与ZFN不同的是,TALEN利用的是黄杆菌(Xanthomonas)的转录因子激活样效应因子(Transcription Activation Like Effector)中DNA序列识别模块作为特异识别DNA序列的基础[18] 。TALEN识别模块一般由34个氨基酸组成(图2),其组成的氨基酸除了第12和13位外其余都是保守的,因此第12/13位氨基酸被称为可变的双氨基酸残基RVD (Repeat Variable Di-residue),正是这两个氨基酸决定了TALEN结合DNA上核苷酸的种类[19] -[21] 。实际上,组成DNA分子的四种不同碱基都有与之对应的TALEN识别模块(图2),所以在构建TALEN人工核酸酶时原理相对简单,只需将表达不同的TALEN识别模块序列按目标序列的顺序连接起来,再与Fok I酶的编码序列融合即可完成。不过,由于目标序列的每个碱基都需要一个TALEN识别模块,所以整个构建过程工作量较大。目前,在拟南芥等多种生物中其基因编辑的作用已经得到验证[18] [22] 。
除了原理简单,TALEN的另一个优势在于,理论上说对任意的核苷酸序列,都能以它为靶标构建一个特异的TALEN核酸酶。在编辑效率方面,TALEN虽然也还没有达到理想的程度,但相对于ZFN还是有所提高[23] ,并且有更好的特异性,使其在医疗用途中更加安全,不过脱靶效应仍然存在。实际上所谓“脱靶”产生的影响并不只是降低了基因编辑的成功率,因为无论是ZFN还是TALEN,它们识别的目标基因的序列长度一般只有十几个碱基,而在整个基因组中,不可避免的有一些与目标序列相似的序列存在,所以,脱靶后更大的危害在于人工核酸酶对基因组中具有与靶序列相似的基因进行编辑,从而产生不可预测的后果。在医学治疗领域的基因编辑运用中,这种情况尤其需要避免。不过,最近的研究表明,通

Figure 2. A cartoon figure of TALEN components and structure (https://www.addgene.org/talen/). Red and purple ellipses represent TALEN recognition modules
图2. TALEN结构及组成示意图(https://www.addgene.org/talen/)。图中红色和紫色的椭圆形结构为TALEN的识别模块
过运用一套扩展的RVD有效的增强了TALEN的特异性及编辑效率,这种新的技术有望在生物及医学领域得到更广泛的应用 [24] 。
对于其构建复杂的问题,目前已有公司开发出商业化的试剂盒,特别是采用新的克隆技术如GOLDEN GATE CLONING [25] ,最快一天左右的时间就可以构建针对一个目标基因特定序列的TALEN核酸酶,不过这种技术使用成本仍然较高。
5. 规律性间隔的短回文序列重复簇(Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR-Associated Proteins, CRISPR)
如果说ZFN和TALEN技术的出现使基因编辑研究获得了很大的成功,那么最近的CRISPR则让这一领域的发展产生了飞跃。CRISPR目前还没有较为统一的中文名称,这里我们将其译为“规律性间隔的短回文序列重复簇”。
实际上早在1987年,日本的研究人员就发现E. coli中存在一些29 bp的重复序列,这些重复序列被一些长度与重复序列类似(32 bp)的但彼此不同的间隔序列(spacer)分开,不过他们当时并不清楚这些序列存在的意义 [26] 。2002年,Jansen等 [27] 将这些间隔排列的重复序列命名为CRISPR。直到2007年,CRISPR和Cas核酸酶才被发现与细菌的适应性免疫有关,并且这种机制在细菌和古细菌中普遍存在 [28] 。随后的研究显示CRISPR-Cas系统的种类及组成成分较多,但一个来自产脓链球菌(Streptococcus pyogenes)、由Cas9蛋白组成的CRISPR系统由于其组成简单引起了研究人员的深入探索,该系统只有三个必须的组成部分,即tracrRNA,CrRNA和Cas9核酸酶 [6] 。
根据Jiang等 [29] 的研究,CRISPR/Cas9系统发挥作用的基本过程可分为三个阶段,即间隔序列获得期、CRISPR/Cas表达期和DNA干扰期(图3)。在间隔序列获得期病毒或质粒侵染细菌时,病毒或质粒DNA被宿主的核酸酶切割成短的DNA片段,符合条件的切割片段作为protospacers而被整合进宿主基因组的CRISPR基因座中成为新的spacer。CRISPR中已经存在的spacers则是以前外源DNA入侵时留下的“记录”。彼此不同的spacer被重复序列(repeat)隔开。在间隔序列和重复序列附近还存在编码Cas9核酸酶的区域以及转录生成traCrRNA的非编码区。
随后CRISPR/Cas9的表达期内,spacers和repeats序列一起被转录成长链的pre-CrRNA,同时tracrRNA和Cas9也被转录出来(图3)。当tracrRNA与pre-CrRNA中由repeat序列转录形成的区域互补结合形成双链RNA (guide RNA)后,引发双链RNA特异的RNaes III对pre-CrRNA进行剪切形成成熟的CrRNA,这个过程同时还需要Cas9的参与 [6] 。每个成熟的CrRNA中包含由spacer转录出来的长20
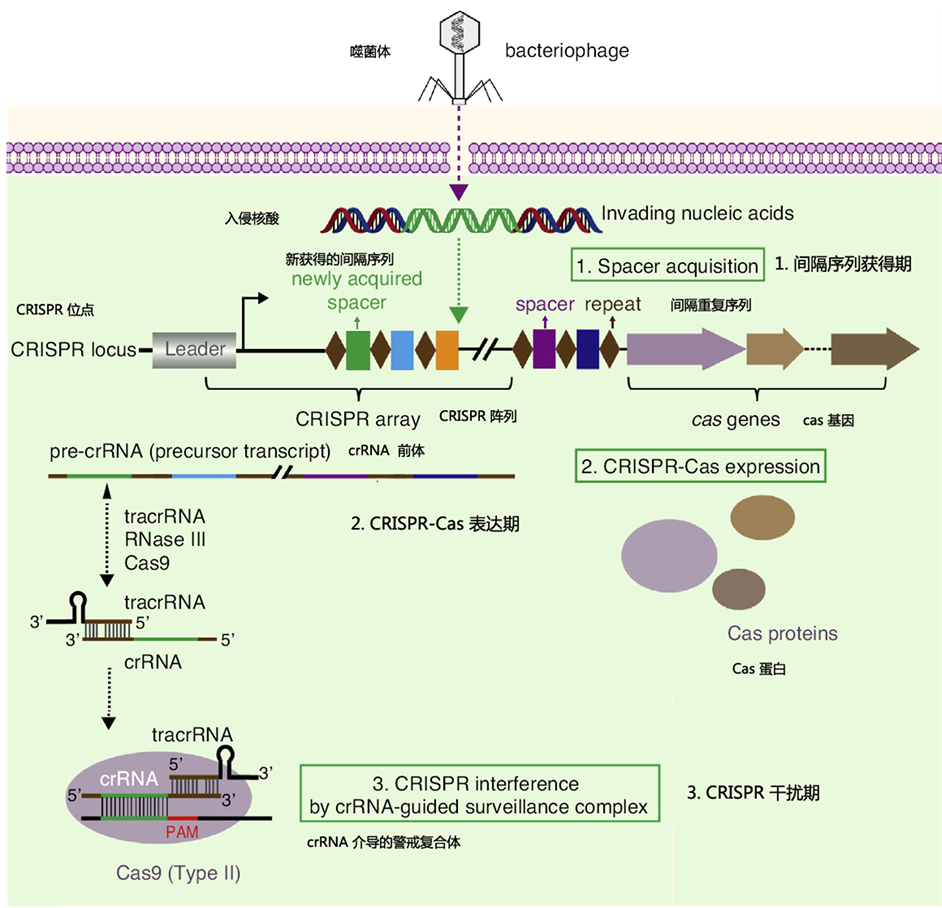
Figure 3. A cartoon figure of CRISPR components and working model (revised on reference [29] )
图3. CRISPR/Cas9组成及工作原理示意图(根据参考文献 [29] ,有修改)
个核苷酸的引导序列和由repeat转录形成的、与tracrRNA互补的区域,其中引导序列是能与入侵DNA互补结合的区域。
在DNA干扰期,CrRNA与tracrRNA结合后形成的guide RNA (gRNA)引导Cas9一起与入侵病毒或质粒DNA结合,导致Cas9核酸酶在入侵DNA核苷酸中PAM (核苷酸组成为5’-NGG-3’或5’-NAG-3’)5’端上游3个碱基处对入侵DNA进行双链切割产生断裂,从而破坏入侵DNA使细菌免受侵害 [6] [30] 。由于PAM的5’端与目标序列(即与引导序列互补的外源DNA区域)的3’端相连,所以切割实际上发生在引导序列结合的范围内。
在CRISPR/Cas9系统对入侵DNA序列的识别过程中,除了gRNA中的引导序列与靶DNA通过碱基配对识别以外,外源DNA序列上的PAM序列也发挥着至关重要的作用 [30] ,因为外源入侵DNA的PAM序列诱变后即可躲过CRISPR/Cas9系统的警戒而免受切割 [31] 。简单地说,PAM是CRISPR/Cas9系统识别“自己”或“异己”的策略,因为CRISPR/Cas9操纵子的DNA中虽有能与引导序列配对的spacer序列,但其邻近的repeat序列中无PAM序列,所以CRISPR/Cas9不会对其产生切割。
同时,详细的研究还显示,Cas9蛋白实际上包含与HNH和RuvC这两个内切酶同源的结构域,HNH结构域特异地切割靶DNA中与CrRNA互补的DNA链,RuvC酶类似的结构域则切割非互补链。诱变这两个区域中的任何一个都会导致Cas9与靶DNA结合后只能产生具有单链切割的环状而非双链断裂的线性DNA分子 [6] [32] 。不仅如此,Jinek等 [6] 还发现在引导序列与目标DNA互补结合过程中,远离PAM的一端能容忍一些碱基的错配,但这种情况可能导致脱靶效应的产生,Hsu等 [33] 报道在人类细胞系中运用CRISPR/Cas9的确有脱靶现象出现。
针对CRISPR/Cas 9系统这一问题,Ran等 [34] 对CRISPR/Cas9进行了深入的研究,他们发现,两个适当位移的不同gRNA (即两个不同gRNA与各自的目标序列结合后,它们之间的距离保持在一定范围以内)与Cas9n (突变Cas9的RuvC类似的结构域使之形成只有HNH酶活性的切割酶)一起能在靶DNA上产生双链切割的同时大大降低可能出现的脱靶效应,在培养的鼠受精卵中实验表明这种策略具有良好的应用潜力 [34] 。这种策略成功的原因可能在于用两个sgRNA的方式相当于将CRISPR/Cas9系统中特异识别序列的长度增加了一倍,因此其识别特异性大大增加。
在高等植物领域,运用CRISPR/Cas9对水稻中不同靶基因的研究表明,约一半的胚细胞中靶基因被该系统进行了编辑,并且在T0代就可以比较容易的获得纯合突变体植株。尤其重要的是其脱靶效应非常低,因此,CRISPR/Cas9是水稻研究的一个有效工具 [35] 。不仅如此,在高等动物中对灵长类动物进行CRISPR/Cas9基因编辑的研究也表明,通过对单细胞胚进行sgRNA和Cas9的mRNA共同注射,研究人员成功获得了同时对两个靶基因进行了修饰的食蟹猴,并且他们没有检测到脱靶现象的发生 [36] ,这个结果说明CRISPR技术在医学上有巨大的应用潜力。
此外,研究还发现将转录tracrRNA和CrRNA的DNA编码序列连接在一起,转录后形成一个人工设计的gRNA同样可以有效引导Cas9对靶基因进行编辑 [6] [32] 。因此,整个CRISPR/Cas9系统使用起来非常简单:只需要根据目标序列设计引物扩增spacer,再将其整合进含有tracRNA和Cas9表达盒的载体,就完成了基因编辑载体的准备工作。此外,为适应真核生物的细胞结构,一般在设计编辑载体时要在Cas蛋白编码序列一端或两端加上核定位信号。目前很多实验室已经构建了专门用于基因敲除的CRISPR/Cas9载体 [37] [38] 。
6. 结语
人工核酸酶ZFN和TALEN都是依靠蛋白质对核苷酸序列的识别,其切割的机制更是直接利用了天然存在的核酸酶的结构域,从原理上说,人工核酸酶和天然存在的限制性核酸内切酶并无本质区别,尤其是天然的II型核酸内切酶多数为同源二聚体结构,所以通常它识别和切割具有回文结构的序列,这一点与人工核酸酶采用的工作原理也有相似之处。那么,天然的限制性核酸内切酶对DNA的识别是否同样依靠锌指结构或TALEN等类似的机制?尽管目前还不清楚,但弄清楚这个问题可能会带来更多新的发现。
有趣的是,与十多年前发现并得到广泛应用的RNAi机制类似,CRISPR/Cas9也是利用短的双链RNA介导目标基因的沉默 [39] 。不同的是,RNAi是在转录水平干扰外源入侵基因的表达,而CRISPR/Cas9是破坏外源入侵生物DNA组成和结构从而导致外源基因被彻底破坏。尽管机制存在区别,但它们都是微生物防御体系的重要组成部分。由此可见,短的双链RNA在生物的防御体系中扮演着极其重要的角色 [29] 。然而,目前发现细菌中大约只有40%的种类存在CRISPR这种防御机制,因此研究具备和不具备CRISPR体系的细菌的防御策略有何不同也是一个值得深入关注的问题。
对比人工核酸酶和CRISPR/Cas9系统可以发现,CRISPR/Cas9的优势主要体现在两个方面:一是用于基因敲除的CRISPR载体构建极其简单,只需要根据推荐的序列合成spacer并将其整合进载体,就完成了基因编辑载体体外的操作过程;二是CRISPR/Cas9与DNA靶序列结合有更高的特异性 [40] ,原因是ZFN和TALEN对靶DNA的识别是依靠蛋白质与DNA的相互作用(实际上是组成蛋白质的氨基酸残基在空间结构上与DNA碱基侧链基团之间的相互作用),其特异性相对较低;而CRISPR/Cas9系统对靶DNA序列的识别则是RNA和DNA按照碱基互补配对原则进行的,因此特异性更高。除此之外,相对于同源重组,CRISPR不仅有更高的效率,适应范围也更加广泛。目前的研究表明该技术适合从原核生物到高等动植物在内的所有生物类群,而且,对CRISPR的酶切基团进行诱变后可以对DNA进行单链切割以及可以同时进行多基因或单基因的多靶点编辑[34] 。
对生物学领域的研究者来说,CRISPR的优势在于它能非常方便的对感兴趣的基因进行编辑使之产生突变,从而极大的方便了阐明每个基因的功能。然而,医学领域的专家更关注运用该技术进行基因治疗的潜力。尽管目前还只是处于初始阶段,但可以预料,CRISPR将给生命科学和医学领域带来难以估量的重要影响。
基金项目
本研究获得了湖北省自然科学基金指导项目(2015CFC879)、湖北省教育厅B类项目(B20082901)及湖北民族学院博士启动基金项目(MY2014B008)资助。
文章引用
朱玉昌,郑小江,胡一兵. 基因编辑技术的方法、原理及应用
Methods, Principles and Application of Gene Editing[J]. 生物医学, 2015, 05(03): 32-41. http://dx.doi.org/10.12677/HJBM.2015.53005
参考文献 (References)
- 1. Esvelt, K.M. and Wang, H.H. (2013) Genome-scale engineering for systems and synthetic biology. Molecular Systems Biology, 9, 641. http://dx.doi.org/10.1038/msb.2012.66
- 2. Puchta, H. and Fauser, F. (2013) Gene targeting in plants: 25 years later. The International Journal of Developmental Biology, 57, 629-637. http://dx.doi.org/10.1387/ijdb.130194hp
- 3. Tan, W.S., Carlson, D.F., Walton, M.W., Fahrenkrug, S.C. and Hackett, P.B. (2012) Precision editing of large animal genomes. Advances in Genetics, 80, 37-97. http://dx.doi.org/10.1016/B978-0-12-404742-6.00002-8
- 4. Dianov, G.L. and Hubscher, U. (2013) Mammalian base excision repair: The forgotten archangel. Nucleic Acids Research, 41, 3483-3490. http://dx.doi.org/10.1093/nar/gkt076
- 5. Händel, E.M. and Cathomen, T. (2011) Zinc-finger nuclease based ge-nome surgery: It’s all about specificity. Current Gene Therapy, 11, 28-37. http://dx.doi.org/10.2174/156652311794520120
- 6. Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J.A. and Charpentier, E. (2012) A programmable dual- RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 337, 816-821. http://dx.doi.org/10.1126/science.1225829
- 7. Ragg, H. (2011) Intron creation and DNA repair. Cellular and Molecular Life Sciences, 68, 235-242. http://dx.doi.org/10.1007/s00018-010-0532-2
- 8. Reissb, B., Schubert, I., Kopchen, K., Wendeler, E., Schell, J. and Puchta, H. (2000) A stimulates sister chromatid exchange and the fidelity of double-strand break repair, but not gene targeting, in plants transformed by agrobacterium. Proceedings of the National Academy of Sciences of the United States of America, 97, 3358-3363. http://dx.doi.org/10.1073/pnas.97.7.3358
- 9. Lieberman-Lazarovich, M. and Levya, A. (2011) Homologous re-combination in plants: An antireview. Methods in Molecular Biology, 701, 51-65. http://dx.doi.org/10.1007/978-1-61737-957-4_3
- 10. Klug, A. (2010) The discovery of zinc fingers and their ap-plications in gene regulation and genome manipulation. Annual Review of Biochemistry, 79, 213-231. http://dx.doi.org/10.1146/annurev-biochem-010909-095056
- 11. Beerli, R.R. and Barbas III., C.F. (2002) Engi-neering polydactyl zinc-finger transcription factors. Nature Biotechnology, 20, 135-141. http://dx.doi.org/10.1038/nbt0202-135
- 12. Dreier, B., Segal, D.J. and Barbas III., C.F. (2000) Insights into the molecular recognition of the 5′2 GNN23′ family of DNA sequence by zinc finger domains. Journal of Molecular Biology, 303, 489-502. http://dx.doi.org/10.1006/jmbi.2000.4133
- 13. Bitinaite, J., Wah, D.A., Aggarwal, A.K. and Schildkraut, I. (2002) FokI dimerization is required for DNA cleavage. Proceedings of the National Academy of Sciences of the United States of America, 95, 10570-10575. http://dx.doi.org/10.1073/pnas.95.18.10570
- 14. Miller, J.C., Holmes, M.C., Wang, J., Guschin, D.Y., Lee, Y.L., Rupniewski, I., Beausejour, C.M., Waite, A.J., Wang, N.S., Kim, K.A., Gregory, P.D., Pabo, C.O. and Rebar, E.J. (2007) An improved zinc-finger nuclease architecture for highly specific genome editing. Nature Biotechnology, 25, 778-785. http://dx.doi.org/10.1038/nbt1319
- 15. Carroll, D. (2011) Genome engineering with zinc-finger nucleases. Genetics, 188, 773-782. http://dx.doi.org/10.1534/genetics.111.131433
- 16. Isalan, M., Choo, Y. and Klug, A. (1997) Synergy between adjacent zinc fingers in sequence-specific DNA recognition. Proceedings of the National Academy of Sciences of the United States of America, 94, 5617-5621. http://dx.doi.org/10.1073/pnas.94.11.5617
- 17. Imanishi, M., Nakamura, A., Morisaki, T. and Futaki, S. (2009) Positive and negative cooperativity of modularly assembled zinc fingers. Biochemical and Biophysical Research Communications, 387, 440-443. http://dx.doi.org/10.1016/j.bbrc.2009.07.059
- 18. Miller, J.C., Tan, S., Qiao, G., Barlow, K.A., Wang, J., Xia, D.F., Meng, X., Paschon, D.E., Leung, E., Hinkley, S.J., Dulay, G.P., Hua, K.L., Ankoudinova, I., Cost, G.J., Urnov, F.D., Zhang, H.S., Holmes, M.C., Zhang, L., Gregory, P.D. and Rebar, E.J. (2011) A TALE nuclease architecture for efficient genome editing. Nature Biotechnology, 29, 143-148. http://dx.doi.org/10.1038/nbt.1755
- 19. Boch, J., Scholze, H., Schornack, S., Landgraf, A., Hahn, S., Kay, S., Lahaye, T., Nickstadt, A. and Bonas, U. (2009) Breaking the code of DNA binding specificity of TAL-type III effectors. Science, 326, 1509-1512. http://dx.doi.org/10.1126/science.1178811
- 20. Moscou, M.J. and Bogdanove, A.J. (2009) A simple cipher go-verns DNA recognition by TAL effectors. Science, 326, 1501. http://dx.doi.org/10.1126/science.1178817
- 21. Streubel, J., Blücher, C., Landgraf, A. and Boch, J. (2012) TAL effector RVD specificities and efficiencies. Nature Biotechnology, 30, 593-595. http://dx.doi.org/10.1038/nbt.2304
- 22. Joung, J.K. and Sander, J.D. (2013) TALENs: A widely applicable tech-nology for targeted genome editing. Nature Reviews Molecular Cell Biology, 14, 49-55. http://dx.doi.org/10.1038/nrm3486
- 23. Chen, S., Oikonomou, G., Chiu, C.N., Niles, B.J., Liu, J., Lee, D.A., An-toshechkin, I. and Prober, D.A. (2013) A large-scale in vivo analysis reveals that TALENs are significantly more mu-tagenic than ZFNs generated using context- dependent assembly. Nucleic Acids Research, 41, 2769-2678. http://dx.doi.org/10.1093/nar/gks1356
- 24. Miller, J.C., Zhang, L., Xia, D.F., Campo, J.J., Ankoudinova, I.V., Guschin, D.Y., Babiarz, J.E., Meng, X., Hinkley, S.J., Lam, S.C., Paschon, D.E., Vincent, A.I., Dulay, G.P., Barlow, K.A., Shivak, D.A., Leung, E., Kim, J.D., Amora, R., Urnov, F.D., Gregory, P.D. and Rebar, E.J. (2015) Improved specificity of TALE-based genome editing using an expanded RVD repertoire. Nature Methods, 12, 465-471. http://dx.doi.org/10.1038/nmeth.3330
- 25. Cermak, T., Doyle, E.L., Christian, M., Wang, L., Zhang, Y., Schmidt, C., Baller, J.A., Somia, N.V., Bogdanove, A.J. and Voytas, D.F. (2011) Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Research, 39, 82. http://dx.doi.org/10.1093/nar/gkr218
- 26. Ishino, Y., Shinagawa, H., Makino, K., Amemura, M. and Nakata, A. (1987) Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology, 169, 5429-5433.
- 27. Jansen, R., Embden, J.D., Gaastra, W. and Schouls, L.M. (2002) Identification of genes that are associated with DNA repeats in prokaryotes. Molecular Microbiology, 43, 1565-1575. http://dx.doi.org/10.1046/j.1365-2958.2002.02839.x
- 28. Barrangou, R., Fremaux, C., Deveau, H., Richards, M., Boyaval, P., Moineau, S., Romero, D.A. and Horvath, P. (2007) CRISPR provides acquired resistance against viruses in prokaryotes. Science, 315, 1709-1712. http://dx.doi.org/10.1126/science.1138140
- 29. Jiang, F. and Doudna, J.A. (2015) The structural biology of CRISPR-Cas systems. Current Opinion in Structural Biology, 30, 100-111. http://dx.doi.org/10.1016/j.sbi.2015.02.002
- 30. Mali, P., Aach, J., Stranges, P.B., Esvelt, K.M., Moosburner, M., Kosuri, S., Yang, L. and Church, G.M. (2013) CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. Nature Biotechnology, 31, 833-838. http://dx.doi.org/10.1038/nbt.2675
- 31. Deveau, H., Barrangou, R., Garneau, J.E., Labonte, J., Fremaux, C., Boyaval, P., Romero, D.A., Horvath, P. and Moineau, S. (2008) Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. Journal of Bacteriology, 190, 1390-1400. http://dx.doi.org/10.1128/JB.01412-07
- 32. Cong, L., Ran, F.A., Cox, D., Lin, S., Barretto, R., Habib, N., Hsu, P.D., Wu, X., Jiang, W., Marraffini, L.A. and Zhang, F. (2013) Multiplex genome engineering using CRISPR/Cas sys-tems. Science, 339, 819-823. http://dx.doi.org/10.1126/science.1231143
- 33. Hsu, P.D., Scott, D.A., Weinstein, J.A., Ran, F.A., Konermann, S., Agarwala, V., Li, Y., Fine, E.J., Wu, X., Shalem, O., Cradick, T.J., Marraffini, L.A., Bao, G. and Zhang, F. (2013) DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology, 31, 827-832. http://dx.doi.org/10.1038/nbt.2647
- 34. Ran, F.A., Hsu, P.D., Lin, C.Y., Gootenberg, J.S., Konermann, S., Trevino, A.E., Scott, D.A., Inoue, A., Matoba, S., Zhang, Y. and Zhang, F. (2013) Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 154, 1380-1389. http://dx.doi.org/10.1016/j.cell.2013.08.021
- 35. Zhang, H., Zhang, J., Wei, P., Zhang, B., Gou, F., Feng, Z., Mao, Y., Yang, L., Zhang, H., Xu, N. and Zhu, J.K. (2014) The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation. Plant Biotechnology Journal, 12, 797-807. http://dx.doi.org/10.1111/pbi.12200
- 36. Niu, Y., Shen, B., Cui, Y., Chen, Y., Wang, J., Wang, L., Kang, Y., Zhao, X., Si, W., Li, W., Xiang, A.P., Zhou, J., Guo, X., Bi, Y., Si, C., Hu, B., Dong, G., Wang, H., Zhou, Z., Li, T., Tan, T., Pu, X., Wang, F., Ji, S., Zhou, Q., Huang, X., Ji, W. and Sha, J. (2014) Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 156, 836-843. http://dx.doi.org/10.1016/j.cell.2014.01.027
- 37. Miao, J., Guo, D., Zhang, J., Huang, Q., Qin, G., Zhang, X., Wan, J., Gu, H. and Qu, L.J. (2013) Targeted mutagenesis in rice using CRISPR-Cas system. Cell Research, 10, 1233-1236. http://dx.doi.org/10.1038/cr.2013.123
- 38. Ma, X., Zhang, Q., Zhu, Q., Liu, W., Chen, Y., Qiu, R., Wang, B., Yang, Z., Li, H., Lin, Y., Xie, Y., Shen, R., Chen, S., Wang, Z., Chen, Y., Guo, J., Chen, L., Zhao, X., Dong, Z. and Liu, Y.G. (2015) A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants. Molecular Plant, in Press. http://dx.doi.org/10.1016/j.molp.2015.04.007
- 39. Wiedenheft, B., Sternberg, S.H. and Doudna, J.A. (2012) RNA-guided genetic silencing systems in bacteria and archaea. Nature, 482, 331-338. http://dx.doi.org/10.1038/nature10886
- 40. Gaj, T., Gersbach, C.A. and Barbas III., C.F. (2013) ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 31, 397-405. http://dx.doi.org/10.1016/j.tibtech.2013.04.004