Hans Journal of Computational Biology
Vol.4 No.02(2014), Article
ID:13727,10
pages
DOI:10.12677/HJCB.2014.42005
Global Docking Method for Flexible Peptide Segment-Protein Interactions
School of Life Sciences, Fudan University, Shanghai
Email: rlai11@fudan.edu.cn, huangqiang@fudan.edu.cn
Copyright © 2014 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



Received: Jun. 3rd, 2014; revised: Jun. 9th, 2014; accepted: Jun. 13th, 2014
ABSTRACT
Protein-peptide binding plays various important roles in living cells. In many cases, the peptidebinding sites of proteins are not known in prior. Then, computational prediction of the peptidebinding sites is desirable. Popular programs for protein-peptide docking usually depend strongly on the initial positions of peptides, such as Rosetta. To overcome this limitation, here we develop a global docking approach in which the peptide is initially distributed evenly on 26 surface locations of a virtual sphere around the protein, and define a selection parameter for discriminating native-like binding site from non-native sites. We used this approach to predict the native-like binding conformations of peptide-protein complexes, and in most cases the peptide-binding sites were correctly predicted, with Cα-RMSDs below 5.5 Å with respect to the crystal structures of peptides. The results of this study suggested that our approach may be very useful for the identification of peptide-binding sites of proteins.
Keywords:Protein-Peptide Interaction, Peptide-Binding Site, Molecular Docking

柔性多肽片段–蛋白质相互作用的
全局对接方法
来瑞颖,万 波,黄 强
复旦大学生命科学学院,上海
Email: rlai11@fudan.edu.cn, huangqiang@fudan.edu.cn
收稿日期:2014年6月3日;修回日期:2014年6月9日;录用日期:2014年6月13日

摘 要
多肽–蛋白质的相互作用在生物细胞中发挥着各种各样重要的作用。通常情况下,它们之间的结合信息是未知的。所以,利用计算方法预测结合位点具有重要意义。而以Rosetta为代表的常用对接软件通常具有很强的初始位置依赖性。为克服这一局限性,本研究提出了一种全局对接的方法,以受体蛋白为球状系统的中心,将多肽平均地分布在球面26个位置上;同时定义了一个区分天然结合构象和非天然结合构象的筛选参数。用上述方法预测多肽–蛋白质的结合构象,结果显示该方法能成功预测蛋白质的结合位点,且多数多肽的预测构象的Cα-RMSD在5.5 Å以下。因此,研究结果表明,所发展的方法在蛋白质多肽结合位点预测方面有很好的应用价值。
关键词
多肽–蛋白质相互作用,多肽结合位点,分子对接

1. 引言
新药设计通常基于体内重要的蛋白质–蛋白质相互作用位点,其中相当一部分是由柔性多肽所介导的,在活细胞中,它们控制了众多至关重要的生理过程[1] 。目前,蛋白质–多肽相互作用已经在制药、生物技术(如蛋白质功能检测) [2] 、生物标记物、生物传感器[3] 以及多肽药物治疗等领域得到广泛应用[4] 。因此,预测和设计多肽–蛋白质相互作用对于生物技术和新药发现具有重要的意义。高分辨率的结构解析法已经在多肽–蛋白质的晶体结构方面得到了广泛应用,包括:核磁共振法和X射线衍射法[5] 。然而,蛋白质结晶过程(包括蛋白质纯化和条件选择)仍存在很大的困难和挑战,而且实验周期长,花费昂贵。
为解决上述问题,可以通过计算模拟的方法来直接预测蛋白质–多肽相互作用。分子对接是预测蛋白质–多肽相互作用最重要的计算技术之一。目前,相关的对接软件包括:AutoDock[6] ,RosettaDock[7] 和DOCK[8] 等等。但是在完全未知互作信息的前提下,进行蛋白质–多肽的相互作用预测尚没有较为系统的方法。AutoDock是盲对接常用的一种软件,其所适用的多肽全长上限仅仅为四个[9] 。另外一种盲对接方法能够克服上述缺陷,然而在入选例子中,互作位点一般均在最大和次大的口袋中[10] 。另外,在许多例子中,非结合态的蛋白质结构通常没有明显的口袋出现在结合位点附近[11] 。
此外,在计算结构生物学中,预测多肽的结合构象是最具有挑战性的,主要因为,多肽具有很大的自由度[12] ,再加上有很多自然状态下的蛋白质并没有稳定的构象[13] ,其内部构象也可能会迅速地发生转变[14] 。有研究表明,考虑多肽的柔性能够增加对接预测的准确性[15] [16] 。同时,相关研究显示,Rosetta FlexPepDock模块能够边实现多肽的从头预测,边实现对接,但是必须已知部分互作信息[7] 。为了解决上述难题,我们提出了一种新的盲对接方法,即在未知互作信息的前提下,用Rosetta FlexPepDock程序实现相互作用位点的系统预测,其整个研究流程见图1A。
2. 材料与方法
2.1. 多肽–蛋白质复合物结构的准备
用于对接的蛋白质–多肽复合物的结构均来自PDB (Protein Data Bank)蛋白质库(见表1)。对于每一
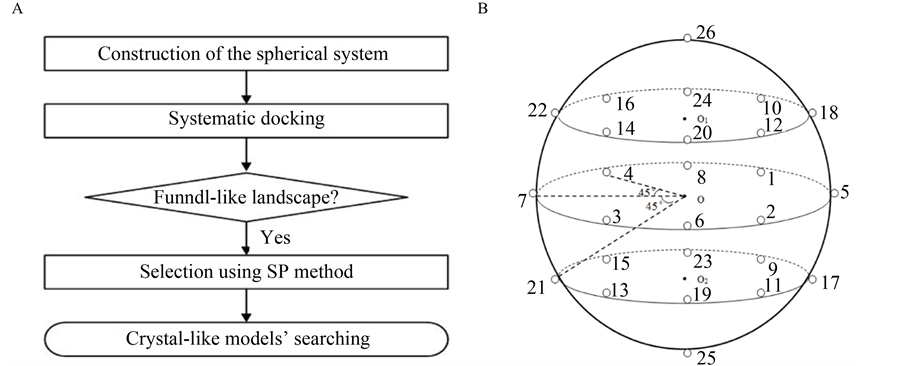

Figure 1. (A) Flowchart of the global docking approach for protein-peptide interactions; (B) The sketch map of the systematic docking with 26 initial locations; (C) The bindingscore-RMSD landscapes with the example of Calmodulin-MLCK docking pair
图1. (A) 多肽–蛋白质相互作用的全局对接流程图;(B) 全局对接系统示意图(26个初始位置);(C) 以Calmodulin-MLCK为例的bindingscore-RMSD散点图
表1. 系统对接的多肽–蛋白质复合物结构
1表示在实现对接时,将来自3MI9的Tat多肽也考虑进来,因为有关研究显示,Tat多肽和AFF4也有直接的相互作用[18] 。
个结构,首先将其离子、配体以及水分子去掉。另外,如果蛋白质中存在不连续的片段,使用经典建模程序MODELLER将其补齐。从蛋白质–多肽的复合物结构中,选取连续9个氨基酸片段的多肽作为对接多肽片段。原因如下:在Rosetta FlexPepDock中,用于对接的多肽平均长度是9个[7] ,另外,在分析蛋白质序列的疏水性时,其默认的窗口序列长度也是9[17] 。那么,9个连续的多肽片段可以视为性质较为稳定的结构单元。
2.2. 球状对接系统的构建
在未知互作信息的前提下,Rosetta程序通常不能准确预测蛋白质–多肽相互作用位点。为了找到对接的最佳初始位置,通过编写Python脚本来实现全局对接系统的构建:以蛋白质为球心,将多肽均匀地放置在球面26个初始位置上。Rosetta程序中的FlexPepDock模块可以同时实现对接和从头预测,为简化模型,同一多肽在26个初始位置上的构象均设置为相同(图1B)。
2.3. 构象筛选的经验参数法
基于上述构建的系统实现平行对接,并设置相关的参数[19] ,每个位置对接产生5000个构象,并把这些对接构象按其结合自由能(即结合亲和性)大小进行排序:结合自由能越低,排序越前。在这里,结合自由能值直接使用Rosetta经验能量函数值表示,即Rosetta程序计算所得的binding score。按常规的配体–蛋白对接分析过程[7] [20] ,以最低binding score的构象为参考构象,计算得到其它构象对参考构象的RMSD (均方根偏差)值(参考构象的RMSD = 0)。以构象的RMSD值为横坐标,其binding score为纵坐标,获得关于5000个对接构象的binding score-RMSD散点图。
为从26个初始位置中确定出多肽的最佳对接初始位置,我们对binding score-RMSD散点图进行了深入分析,不断摸索与总结,获得了一个筛选最佳初始位置的经验公式。其过程是:对从某一初始位置出发对接所得的5000个构象,把binding score排在前20、且其RMSD值大于0且小于7Å的构象取出,构成一个构象总数为N (0 £ N < 20)的子集, 并设子集中最小RMSD值RMSDmin,对应构象在子集中binding score排序为Rankmin。我们发现,这些从散点图中获得的特征参数存在以下规律:N、RMSDmin和Rankmin越小,binding score也越小,越有利于筛选出好的初始对接位置。因此,有如下用于筛选最佳初始位置的经验公式:
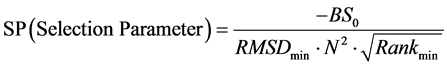 (1)
(1)
上式中的BS0是5000个构象中的最低binding score值,因为5000个对接构象中绝大部分的binding score为负值,所以这个最小值是小于零的,为负值。还有,我们发现上式中的N 对于最佳位置的筛选作用较大,为强化它的作用,将其做平方处理;类似地,Rankmin对于最终结果作用较小,将其进行开方处理。
这样,利用每一个对接初始位置所获得的5000个构象,如果其N > 0,可用上式确定计算该位置对应的SP值,最大SP值的位置即为最佳对接初始位置。对N = 0的位置,不需计算而直接认定其为非最优位置。另外,在与晶体结构对比中,对接能量排在前10低的构象当中,其Cα-RMSD在5.5 Å以内的构象被视为成功预测构象。
2.4. 应用于小分子的ISR法
ISR的全称即固有专一率,ISR法是一种适用于小分子–蛋白质全局对接的定量虚拟筛选法[21] ,它表示对接双方的特异性高低。这种ISR方法适用于小分子化合物与蛋白质的对接[22] 。计算公式如下[21] :
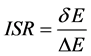 (2)
(2)
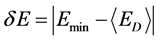 (3)
(3)
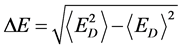 (4)
(4)
δE指的是能隙,即最小能量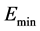 与构象的平均能量
与构象的平均能量 的差值,
的差值,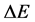 指的是构象的能量波动。
指的是构象的能量波动。
ISR法通过ISR-Affinity散点图来进行直观展示,将本研究提出的SP法与ISR法进行比较,进一步证明其可靠性。
3. 结果与讨论
3.1. Bindingscore-RMSD散点图的经验参数筛选
为了找出正确的或者近似的初始位置,基于收敛状散点图(以Calmodulin-MLCK为例,见图1C),按照经验公式计算筛选参数。根据经验公式(1),将符合公式适用条件的初始位置筛选出来,具体地包括:位置2,4,8,10,12,18,25 (PKA-PKI),位置7,9,10,12 (CycT1_Tat-AFF4),位置2,5,8,11,13,17,18,23 (LAS17-SLA1),位置4,5,7,10,14,15,19 (Androgen-FxxLF),位置2,6,12,16,17,18,19 (Chymotrypsin A-TATI),位置1,2,3,4,5,8,11,12,15,16,18,23,24,26 (ABL1-7C12),位置1,2,3,4,8,12,21,24 (Calmodulin-MLCK),位置2,5,10,12,18,24,26 (SLAM-CD150)。对于上述每个对接例子的粗略筛选位置,计算相应的SP值,最大SP值所在的初始位置即最佳初始位置,见表2。结果显示,PKA-PKI的最佳初始位置是10 (SP value =1.646),CycT1_Tat-AFF4的最佳初始位置是7 (SP value = 0.800),LAS17-SLA1的最佳初始位置是2 (SP value = 1.487),Androgen-FxxLF的最佳初始位置是15 (SP value = 1.543),Chymotrypsin A-TATI的最佳初始位置是19(SP value = 1.751),ABL1-7C12的最佳初始位置是23 (SP value = 1.021),Calmodulin-MLCK的最佳初始位置是24 (SP value = 1.142),SLAM-CD150的最佳初始位置是26 (SP value = 1.376)。
3.2. 基于初始位置的构象选择
基于筛选得到的初始位置,计算结合自由能排在前10低的构象的Cα-RMSD值(与晶体结构相比,见表3)。与晶体结构相比,其中6个例子的最终Cα-RMSD值在5.5Å以内,被视为正确的预测构象,包括:Chymotrypsin A-TATI,Androgen-FxxLF,LAS17-SLA1,Calmodulin-MLCK,ABL1-7C12和CycT1_TatAFF4。只有PKA-PKI和SLAM-CD150 (Cα-RMSD分别为7.71 Å和8.05 Å)不在正确预测的范畴内。总之,
表2. 每对蛋白质–多肽基于26个初始位置的SP值
Table 3. The lowest Cα-RMSD with respect to the crystal structure, binding score and rank of the binding score
表3. 与晶体结构相比的最小Cα-RMSD值,binding score及其排序
大多数例子的预测结果是成功的,见图2A。
统计数据显示(见图2B),在结合自由能TOP1的构象中,预测正确率为25%,包括CycT1·Tat-AFF4和Calmodulin-MLCK,对应的Cα-RMSD值分别是2.10 Å (Rank1)和3.55 Å (Rank1)。在结合自由能TOP2的构象中,预测正确率为50%,包括CycT1·Tat-AFF4,Androgen-FxxLF,Chymotrypsin A-TATI,Calmodulin-MLCK,对应的Cα-RMSD值分别是2.10 Å (Rank1),4.03 Å (Rank2),5.24.Å (Rank2)和3.55 Å (Rank1)。在结合自由能TOP5的构象中,预测正确率为50%,包括CycT1·Tat-AFF4,Androgen-FxxLF,Chymotrypsin A-TATI,Calmodulin-MLCK,对应的Cα-RMSD值分别是2.10 Å (Rank1),4.03 Å(Rank2),5.24.Å (Rank2)和3.55 Å (Rank1)。在结合自由能TOP10的构象中,预测正确率在75%,包括CycT1·Tat-AFF4,LAS17-SLA1,Androgen-FxxLF,Chymotrypsin A-TATI,ABL1-7C12,Calmodulin-MLCK,对应的Cα-RMSD值分别是2.10 Å (Rank1),5.35 Å (Rank9),4.03 Å (Rank2),5.24 Å (Rank2),5.16 Å (Rank10)和3.55 Å (Rank1)。因此,SP法能够成功识别天然结合构象。当然,为了能够更加全面地检验该方法的可靠性,需要更多的例子加以验证。

Figure 2. (A) The crystal and predicted conformations. Shown are the native binding pose (magenta) and the final predicted binding pose (blue); (B) The percentages of near-native models of Cα-RMSD values below 5.5 Å with respected to crystal structure in binding score poses of top 1, top 2, top 5 and top 10; (C) ISR-Affinity plots of the eight docking pairs
图 2. (A) 晶体构象(紫色)和预测构象(蓝色);(B) 结合自由能的排序分别在top 1, top 2, top 5 和top 10,近似天然结合构象(与晶体结构相比,Cα-RMSD在5.5 Å以内的构象)的预测成功率;(C) 八个例子的ISR-Affinity散点图
表4. SP法与ISR法比较
注:粗体划线的位置表示SP法与ISR法的精准匹配,斜粗体位置表示SP法与ISR法的模糊匹配。
3.3. 与ISR法的比较
最终结果显示,SP筛选法能够在大多数例子中找到最优构象。其他相似的研究揭示,ISR值能够反映小分子–蛋白质对接的特异性。通过对比SP筛选法与ISR筛选法,我们找到了两者间存在的合理关系。在ISR-Affinity的散点图中,处于右上方的点,其ISR值以及亲和性都是最大的。在8个例子当中,SP法选出的点分别在ISR-Affinity散点图的右上方能够找到准确或者模糊的对应(图2C,表4)。与ISR法相比,准确预测结果包括:CycT1_Tat-AFF4(位置7),Androgen-FxxLF(位置15),Chymotrypsin A-TATI(位置19),Calmodulin-MLCK(位置24),SLAM-CD150(位置26)。模糊预测结果包括:PKA-PKI(位置10与ISR法预测的位置1相邻),LAS17-SLA1 (位置2与ISR法预测的位置12相邻),ABL1-7C12 (位置23与ISR法预测的位置15相邻)。结果显示,SP法与ISR法具有62.5%的精准匹配率和100%的模糊匹配率。此外,与SP筛选法相比,ISR筛选法得到的结果并不精准,它能够在前5个结果中找到较为匹配的位置,无法直接找到最优的位置。因此,SP筛选法具有更大的精准度和优势。
4. 结论
本研究提出了一种多肽–蛋白质系统盲对接法,将多肽均匀地分布到以蛋白质为中心的球面上实现平行对接,并提出了经验参数筛选法(即SP法)来筛选预测构象,最终结果显示,多数多肽的预测构象的Cα-RMSD在5.5 Å以下,得以成功识别和预测多肽–蛋白质的相互作用,得到成功的预测结合构象。另外已被证明,我们所提出的这种新的研究方法,与适用于小分子–蛋白质对接的ISR法相比,具有高度的一致性,而且可以将预测结果的范围更进一步地缩小。综上,在未知任何互作信息的前提下,利用该方法能够成功预测蛋白质–多肽的结合位点,在蛋白质与多肽结合位点的预测方面,所发展的方法将有很好的应用价值。
项目基金
上海市重点学科建设项目(B111)。
参考文献 (References)
- [1] Petsalaki, E. and Russell, R. (2008) Peptide-mediated interactions in biological systems: New discoveries and applications. Current Opinion in Biotechnology, 19, 344-350.
- [2] Karanicolas, J. and Kuhlman, B. (2009) Computational design of affinity and specificity at protein-protein interfaces. Current Opinion in Structural Biology, 19, 458-463.
- [3] Hao, J., Serohijos, A., Newton, G., Tassone, G., Wang, Z., Sgroi, D., Dokholyan, N. and Basilion, J. (2008) Identification and rational redesign of peptide ligands to CRIP1, a novel biomarker for cancers. PLOS Computational Biology, 4, e1000138.
- [4] Vlieghe, P., Lisowski, V., Martinez, J. and Khrestchatisky, M. (2010) Synthetic therapeutic peptides: Science and market. Journal of Neuroscience Methods, 15, 40-56.
- [5] Doyle, D., Lee, A., Lewis, J., Kim, E., Sheng, M. and MacKinnon, R. (1996) Crystal structures of a complexed and peptide-free membrane protein-binding domain: Molecular basis of peptide recognition by PDZ. Cell, 85, 1067-1076.
- [6] Morris, G., Goodsell, D., Huey, R. and Olson, A. (1996) Distributed automated docking of flexible ligands to proteins: Parallel applications of AutoDock 2.4. Journal of Computer-Aided Molecular Design, 10, 293-304.
- [7] Raveh, B., London, N., Zimmerman, L. and Schueler-Furman, O. (2011) Rosetta FlexPepDock ab-initio: Simultaneous folding, docking and refinement of peptides onto their receptors. Plos One, 6, e18934.
- [8] Shoichet, B. and Kuntz, I. (1993) Matching chemistry and shape in molecular docking. Protein Engineering Design & Selection, 6, 723-732.
- [9] Hetényi, C. and van der Spoel, D. (2009) Efficient docking of peptides to proteins without prior knowledge of the binding site. Protein Science, 11, 1729-1737.
- [10] Aita, T., Nishigaki, K. and Husimi, Y. (2010) Toward the fast blind docking of a peptide to a target protein by using a four-body statistical pseudo-potential. Computational Biology and Chemistry, 34, 53-62.
- [11] Coleman, R. and Sharp, K. (2010) Protein pockets: Inventory, shape, and comparison. Journal of Chemical Information and Modeling, 50, 589-603.
- [12] Dagliyan, O., Proctor, E., D’Auria, K., Ding, F. and Dokholyan, N. (2011) Structural and dynamic determinants of protein-peptide recognition. Structure, 19, 1837-1845.
- [13] Vallee-Belisle, A., Ricci, F. and Plaxco, K. (2009) Thermodynamic basis for the optimization of binding-induced biomolecular switches and structure-switching biosensors. Proceedings of the National Academy of Sciences India Section B, 106, 13802-13807.
- [14] Uversky, V. and Dunker, A. (2010) Understanding protein non-folding. BBA-Proteins Proteom, 1804, 1231-1264.
- [15] Humphris, E. and Kortemme, T. (2008) Prediction of protein-protein interface sequence diversity using flexible backbone computational protein design. Structure, 16, 1777-1788.
- [16] Ding, F., Yin, S. and Dokholyan, N. (2010) Rapid flexible docking using a stochastic rotamer library of ligands. Journal of Chemical Information and Modeling, 50, 1623-1632.
- [17] Chou, S., Upton, H., Bao, K., Schulze-Gahmen, U., Samelson, A., He, N., Nowak, A., Lu, H., Krogan, N., Zhou, Q. and Alber, T. (2012) HIV-1 tat recruits transcription elongation factors dispersed along a flexible AFF4 scaffold. Proceedings of the National Academy of Sciences India Section B, 110, E123-E131.
- [18] Schulze-Gahmen, U., Upton, H., Birnberg, A., Bao, K., Chou, S., Krogan, N. and Zhou, Q. (2013) Building a super elongation complex for HIV. Elife, 2, e00577.
- [19] Bradley, P. (2005) Toward high-resolution de novo structure prediction for small proteins. Science, 309, 1868-1871.
- [20] Zhu, J., Yang, Q., Dai, D. and Huang, Q. (2013) X-ray crystal structure of phosphodiesterase 2 in complex with a highly selective, nanomolar inhibitor reveals a binding-induced pocket important for selectivity. Journal of the American Chemical Society, 135, 11708-11711.
- [21] Yan, Z., Zheng, X., Wang, E. and Wang, J. (2013) Thermodynamic and kinetic specificities of ligand binding. Chemical Science, 4, 2387.
- [22] Yan, Z. and Wang, J. (2012) Specificity quantification of biomolecular recognition and its implication for drug discovery. Science Report-UK, 2, srep00309.
