Botanical Research
Vol.3 No.06(2014), Article ID:14385,10 pages
DOI:10.12677/BR.2014.36030
Study on Genetic Diversity of 7 Populations of Actinidia by Using Chloroplast Genome Fragment
Engineering Research Center for Protection and Utilization of Bioresource in Ethnic Area of Southern China, South-Central University for Nationalities, Wuhan
Email: *liuhong@mail.scuec.edu.cn
Copyright © 2014 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



Received: Sep. 14th, 2014; revised: Oct. 16th, 2014; accepted: Oct. 27th, 2014
ABSTRACT
Actinidia arguta is one of the most complex species in the genus of Actinidia with different ploidy. In this paper, we choose seven representative wild populations of A. arguta in four provinces of China. We use 21 pairs of chloroplast genome hypervariable segments to analyse genetic diversity of seven populations. The result shows that seven pairs within twenty one pairs of primer fragment can amplify samples successfully. The No. 13 fragments rpl32-trnL had the best resolution ratio. We used the fragment rpl32-trnL to build the phylogenetic tree of seven populations with MP algorithm and NJ method. The experimental results show that two populations from Changbai Mountain are located at the base of the tree. Three populations from the Dabie Mountains are not in the same branch. At last, the average polymorphic locus of 21 fragments is 27.16. This study provided basis for the development and application of chloroplast genome in Actinidia species.
Keywords:Actinidia arguta, Chloroplast Genome, Hypervariable Segment, Phylogenetic Tree

利用叶绿体基因组高变片段对7个软枣猕猴桃居群遗传多样性的研究
刘 虹*,刘锡红,刘秋宇,李 刚,覃 瑞
中南民族大学,南方少数民族地区生物资源保护与综合利用工程中心,武汉
Email: *liuhong@mail.scuec.edu.cn
收稿日期:2014年9月14日;修回日期:2014年10月16日;录用日期:2014年10月27日

摘 要
软枣猕猴桃是猕猴桃属内倍性最复杂的物种之一,本研究针对分布在国内4个省份具有代表性的7个野生软枣猕猴桃居群,选用21对叶绿体基因组高变片段对7个居群的软枣猕猴桃进行序列比对、系统构建和多态性分析。研究发现,21对引物中有7对引物在7个软枣猕猴桃居群样本中均能扩增成功,第13号片段rpl32-trnL的分辨率最好。利用片段rpl32-trnL对7个软枣猕猴桃居群分别采用MP算法和NJ法构建的系统进化树表明,来自长白山的2个居群位于系统树最基部,来自大别山的3个居群并未处于同一支中,21个片段的平均多态位点数达到27.16。本研究为猕猴桃属叶绿体基因组的开发和应用奠定了基础。
关键词
软枣猕猴桃,叶绿体基因组,高变片段,系统进化树

1. 引言
叶绿体是绿色植物进行光合作用的重要细胞器,具有完整的遗传信息系统,其遗传物质被称作叶绿体基因组[1] 。对叶绿体基因组的比较分析从上世纪90年代首次获得烟草[2] 和地钱[3] 叶绿体基因组的完整序列以后得到迅速发展,越来越多物种的叶绿体基因组被测定。目前叶绿体系统分析被广泛应用于比较基因组学系统发育研究,在裸子植物中,叶绿体基因组一般是父系遗传,而大多数被子植物的叶绿体基因组为母系遗传[1] 。现有的叶绿体基因组研究的层次主要在目和科间水平进行,在亚种间的变异及进化的研究相对较少[4] -[6] 。
叶绿体基因组DNA (chloroplast DNA, cpDNA)一般为双链环状结构,由四部分组成:两个序列完全相同、单方向相反的重复序列(inverted repeat sequence) IRA和IRB,大的单拷贝区(large single-copy region, LSC)和小的单拷贝区(small single-copy region, SSC)。LSC和SSC位于两个重复区之间[1] 。由于叶绿体基因组是仅次于核基因组的第二大基因组,且叶绿体的核酸置换率适中,故叶绿体基因组在种级以上较高阶元的系统发育研究有着显著的优势,加上被子植物叶绿体基因组较小,长度在115~165 kb之间,总量约占植物总DNA量的10%~20% [7] ,且多为单拷贝闭合环状双链DNA,在分子水平上差异明显,无论是在序列上还是在结构上都是相当保守的,分子性状的同源关系更容易确定。
软枣猕猴桃(Actinidia arguta)为猕猴桃科猕猴桃属多年生落叶大藤本,雌雄异株。其典型特征是茎叶不具毛,浆果表面无斑点,属9种光果猕猴桃种类之一,软具有很高的营养价值[8] 。软枣猕猴桃广布于东北、华北、西北及长江流域,是猕猴桃属中在中国地域分布最广泛的种类之一,在朝鲜、日本、俄罗斯亦有分布[9] 。对软枣猕猴桃的开发利用中,日本的研究处于领先地位,片冈等人[10] 对日本境内分布的软枣猕猴桃资源进行了详细的调查,发现日本各州县分布的软枣猕猴桃有2、4、6、7和8倍体,倍性变化差异较大。我国软枣猕猴桃资源非常丰富,但一直以来有关猕猴桃属植物的研究多集中在新品种的选育及种间系统发育的研究[11] -[13] ,种下特别是软枣猕猴桃种内遗传多样性的研究比较少,利用叶绿体基因组标记对软枣猕猴桃的研究尚未见报道。本文选择全国不同地域的软枣猕猴桃作为研究对象,通过分析7个软枣猕猴桃居群叶绿体基因组的位点变异来探讨猕猴桃属植物种内遗传多样性的变化,以及叶绿体基因组序列比对是否对猕猴桃系统发育的适用性。
2. 材料和方法
2.1. 材料
DNA纯化试剂盒:上海百蕊生物科技有限公司生产;
PCR仪:上海伯乐生命医学产品有限公司。
7个居群的软枣猕猴桃实验材料见表1,随机取样,叶片分子材料由中科院武汉植物园王彦昌提供。CTAB法提取基因组总DNA,用DNA纯化试剂盒进行纯化。
2.2. 引物的选择、PCR扩增和测序
本文选用的叶绿体基因间隔区21对引物编号及其结构见图1,每对引物扩增大小在0.75 kb~2.0 kb。其中编号4-5号引物为内含子区,7号和18号引物为编码区,其余的17对均为基因间隔区[14] 。图中大的方框代表编码基因,小的空心代表基因间隔区,小的实心区域代表内含子区,数字代表对应片段长度。
PCR反应程序为94℃ 3 min,然后34个循环,94℃ 30 s,52℃ 30 s,72℃ 2 min,最后72℃ 5 min。PCR扩增产物经1%琼脂糖凝胶电泳分离,割取目的条带,经凝胶回收试剂盒纯化,纯化产物送至上海桑尼测序公司进行测序。
2.3. 比对方法
多序列的比对采用软件ClustalX 2.0做初步比对[15] ,然后用BioEdit Se-Al 2.0对比对结果进行手工调整,再用MEGA 5.10的邻接法(neighbor-joining)中的Kimura 2-parametery遗传距离[16] ,步展值(Bootstrap)的可靠性设置为1000次。Maximum parsimony(最大简约法)采取Subtree-Pruning-Regrafting法进行搜索,步展值(Bootstrap)的可靠性设置为1000次。
2.4. 序列单核苷酸多态性和Indel的检测
根据Caicedo等人的方法[17] ,将长度大于一个碱基的插入\缺失作为单碱基的插入\缺失处理。用DnaSP ver. 5.10软件对核苷酸的多样性(π)、多态性位点数(S)、插入\缺失(Indel)和单倍型多态性(h)进行分析[18] 。
表1. 软枣猕猴桃采集信息

Figure 1. The structure of twenty one pairs of primer fragments (From Dong Wen-pan)
图1. 21对片段引物编号及其结构(引自董文攀[14] )
3. 实验结果
3.1. 引物通用性的验证
利用上述21对引物对7个居群的软枣猕猴桃扩增的琼脂糖电泳如图2所示。从实验结果来看,21对引物中有7对引物第9、10、11、13、17、19、20对引物在7个软枣猕猴桃居群样本中均能扩增成功,第5对引物在7个样本中成功扩增6个,第4、7对引物则在7个样本中均没有扩增成功。
3.2. 居群间的遗传多样性分析
7个软枣猕猴桃种内居群间的遗传多样性分析见表2和表3。从表2可以看出,21个高变叶绿体基因组片段扩增结果在7个软枣猕猴桃样本中差异较大。
从多态性位点(polymorphic sites)S值来看,变异最大的片段为第13号引物rpl32-trnL,其S值为111,其次为第20号trnSGCU-trnGGCC和第2号accD-psal。根据核苷酸多样性π值的大小判断,第1 (rbcL-accD)、2 (accD-psal)、6 (ndhC-trnV)、8 (petA-psbJ)、12 (psbE-petL)、13 (rpl32-trnL)、14 (rpoB-trnC)、16 (trnT-
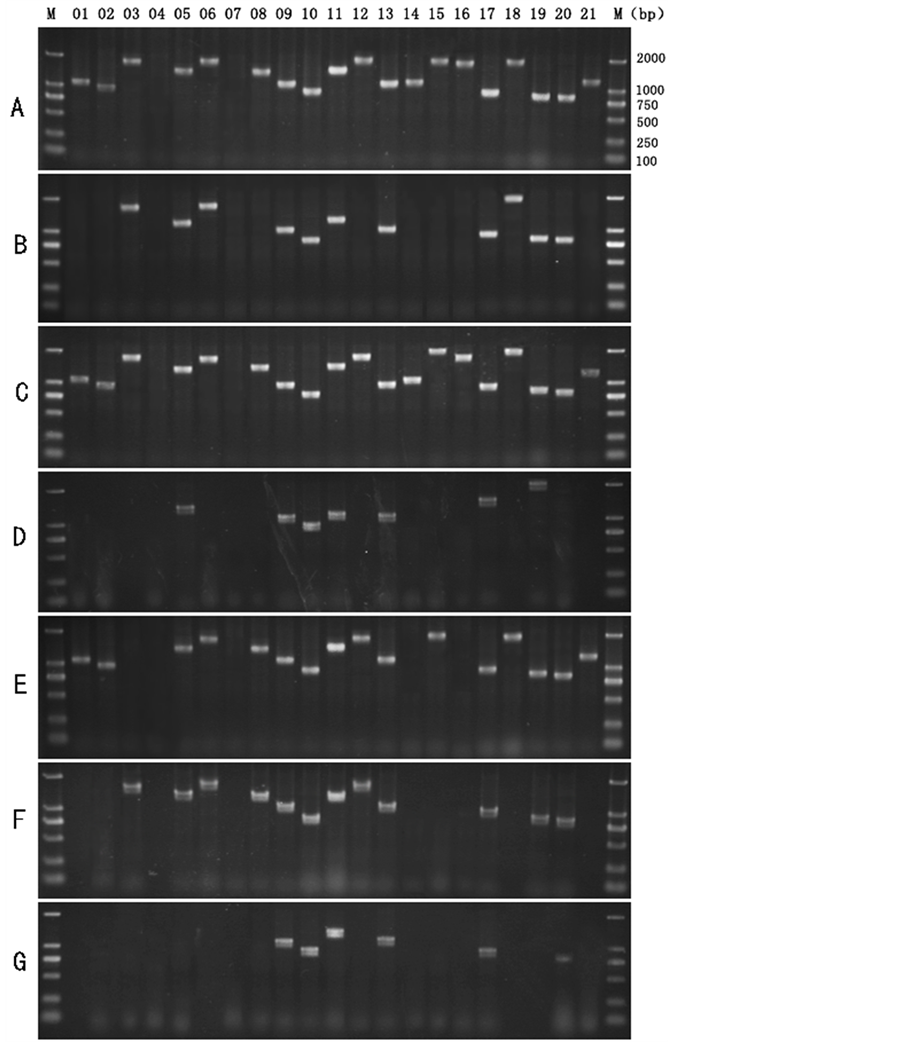
Figure 2. Agarose gel electrophoresis of twenty one pairs primer amplified to seven populations of Actinidia arguta. The top numbers are the serial numbers. The right numbers represent the size of marker (bp). Letters represent different populations
图2. 21对引物7个居群的软枣猕猴桃扩增的琼脂糖电泳图;顶端数字表示引物序号,右侧数字表示marker的大小(bp)。字母表示软枣猕猴桃居群编号
psbD)、20 (trnSGCU-trnGGCC)是变异比较大的位点,其π均值大于0.01,剩下的片段的平均π值为0.0049。其中第11号petB-petD片段中发生插入\缺失的信息最多。
从表3的结果可知,单倍型多态性水平(h)的变化范围为0.076~0.050,而核苷酸多态性水平(π)变化范围为0.00066~0.12003。
表2. 7个软枣猕猴桃样本中的21个高变片段
L:片段长度;π:同一片段的核苷酸多态性;I:片段中插入\缺失的总数;GC%:片段中的GC含量。
表3. 软枣猕猴桃样品21个基因序列信息
3.3. 七个软枣猕猴桃居群的系统发育关系
从成功扩增、测序包含7个样本的21个高变片段的基因序列来看(表3),第13号片段rpl32-trnL的分辨率最好。我们使用片段rpl32-trnL对七个软枣猕猴桃居群分别采用MP算法和NJ法构建了系统进化树(图3)。从图3可以看出,居群A和B位于7个居群中最基部位置,它们均来自于长白山,与其他的居群呈姐妹类群关系。居群F、D、G关系密切,聚为一支。D与C、E都取自于大别山,但并未处于同一支中。
4. 分析与讨论
叶绿体基因组常被看作是一个独立的遗传单位,由于其不经过遗传重组,所以有利于进行遗传分析。一些叶绿体基因组的非编码区具有较快的进化速率,可区分出种内多种单倍型,适于植物近缘种间和种内的亲缘关系和遗传多样性的研究[19] 。叶绿体基因组在绝大多数被子植物中为母系遗传,其叶绿体基因组只能通过种子进行传播,能真实地反映由于种子扩散而引起的遗传结构差异[20] 。
前人利用其他的分子标记,对不同地域的软枣猕猴桃遗传结构进行了研究,黄岳等[21] 以长白山区多个取样点采集的软枣猕猴桃样品叶片为试验材料,利用RAPD技术对其进行遗传多样性分析,聚类分析结果表明,长白山区野生软枣猕猴桃各居群存在一定程度的地理区域聚类趋势,表明遗传变异与地理距离间没有显著的相关性。本研究则是基于叶绿体基因组的核苷酸变异对软枣猕猴桃的居群遗传结构进行分析。由于叶绿体基因组的有效群体大小(effective population size)远小于核基因组,遗传漂变对叶绿体基
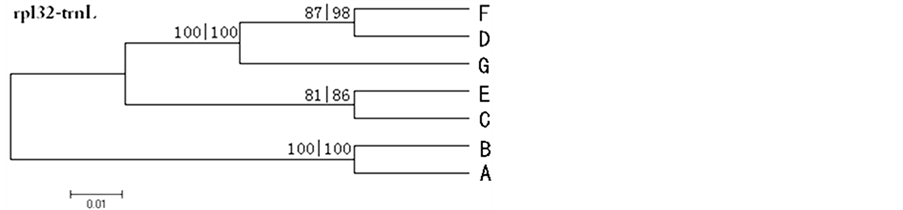
Figure 3. Phylogenetic tree of seven populations of Actinidia arguta by using rpl32-trnL. The different numbers represent NJ and MP calculation method
图3. 利用rpl32-trnL片段构建的7个软枣猕猴桃居群间系统发育树;树枝上的数值分别表示NJ和MP法的支持率
因组居群遗传结构的作用更为强烈。因此,基于叶绿体基因组核苷酸变异的检测往往表现出更高的居群分化水平。
本研究发现叶绿体基因组中分辨率最高的片段位于第13号rpl32-trnL基因上,说明位于基因间隔区的rpl32-trnL片段属于叶绿体基因组高变区。根据Shaw等[22] 通过对不同植物叶绿体基因组上的34个非编码区域的序列进行比对,发现rpl32-trnl区段的变异最为丰富,非常适合对较低分类单元植物的亲缘关系进行研究。刘阳等通过利用rpl32-trnl区段对云南地区栽培的大叶茶9个居群和大理茶3个居群的遗传多样性和居群遗传结构进行比较,发现栽培大理茶和栽培大叶茶的rpl32-trnL片段的遗传变异都主要存在于居群间[19] 。该片段在种内水平上表现了很好的多态性,也有研究表明rpl32-trnL片段在物种水平上也具有很好的多态性。本研究恰好验证了rpl32-trnl区段也可以在软枣猕猴桃居群间起到很好的分类效果,为获得叶绿体基因组在猕猴桃不同居群的比较分析的普通引物奠定了基础。
值得注意的是选用的21对引物高变片段中,第4号(alpP)和第7号(ndhF)片段在7个居群中均未扩增成功,说明这两个片段在软枣猕猴桃居群内全部发生丢失。而在董文攀[14] 的研究中,这2个片段在蜡梅科大部分样本中都成功扩增,说明这两个片段在不同科的植物中存在较大的差异。有研究显示第17号trnH-psbA片段也具有较好的多态性,被建议列为DNA条形码的候选片段[23] ,在蜡梅科叶绿体基因组研究中发现该片段在各个类群中的长度差异很大[14] ,太大的差异可能带来比对结果的不准确,但在本研究中,trnH-psbA片段长度的变化在7个居群中较为稳定,但核苷酸多态性水平较低,不太适合软枣猕猴桃种内的遗传变异的比对。此外,第5号(ndhA)、6 (ndhC-trnV)、9 (petN-psbM)、10 (psbM-trnD)、11 (petB-trnD)和17 (trnH-psbA)片段在不同的类群中表示的多态性也不同,其中第9和第10两个片段受到的关注度比较高,乐霁培等将两个核基因组片段(LEAFY和G3PDH)和第9~10号两个叶绿体基因间间隔区片段与现有的标记结合,以提高系统发育结果的准确性,结果发现根据叶绿体基因组构建的系统进化树与核基因组系统进化树结果不一致性[24] ,也许是由于祖多态性不完整的谱系分类所致。从序列多态性比对结果来看,第20号(trnSGCU-trnGGCC)的序列多态性比21号(trnSUGA-trnGUCC)要高,在第21号中存在poly(T)结构,而在第20号中存在(AT)n的重复。根据权旭等[25] 的研究发现,在某些种内的trnSUGA-trnGUCC片段中甚至会出现大片段的插入和缺失。在很多类群中trnSUGA基因被认为是起源于线粒体基因[25] ,但是属于基因间隔区的trnSUGA-trnGUCC是仅在叶绿体基因组中所有的[14] 。
本研究的不足之处在于居群样本数量稍偏小,此外,如果选取样本的地域分布更广泛的话,其分析更加能够体现软枣猕猴桃的居群变异情况。中国是全球野生猕猴桃资源最丰富的国家,软枣猕猴桃是分布最广泛的猕猴桃之一,也是猕猴桃属植物中应用开发比较多的种类。本研究利用基于叶绿体基因组的21对引物高变片段,对来自于吉林、安徽、四川和陕西四省份的7个软枣猕猴桃居群进行了遗传结构分析,探讨了猕猴桃属植物种内遗传多样性的变化,为猕猴桃属植物叶绿体基因组的进化研究提供基础依据。
基金项目
国家自然科学基金(31370253);武汉市青年科技晨光计划项目(201271031395)。
参考文献 (References)
- [1] 王玲, 董文攀, 周世良 (2012) 被子植物叶绿体基因组的结构变异研究进展. 西北植物学报, 6, 1282-1288.
- [2] Shinozaki, K., Ohme, M., Tanaka, M., et al. (1986) The complete nucleotide sequence of the tobacco chloroplast genome: its gene organization and expression. The EMBO Journal, 5, 2043-2049.
- [3] Ohyama, K., Kohchi, T., et al. (1986) Chloroplast gene organization deduced from complete sequence of Liverwort Marchantia polymorpha chloroplast DNA. Nature, 322, 572-574.
- [4] Wu, C.S., Lai, Y.T., Lin, C.P., Wang, Y.N. and Chaw, S.M. (2009) Evolution of reduced and compact chloroplast genomes (cpDNAs) in gnetophytes; selection toward a lower-cost strategy. Molecular Phylogenetics and Evolution, 52, 115-124.
- [5] Leseberg, C.H. and Duvall, M.R. (2009) The complete chloroplast genome of Coix lacryma-Jobi and a comparative molecular evolutionary analysis of plastomes in cereals. Journal of Molecular Evolution, 69, 311-318.
- [6] Guisinger, M.M., Kuehl, J.V., Boore, J.L. and Jansen, R.K. (2011) Extreme reconfiguration of plastid genomes in the angiosperm family Geraniaceae: Rearrangements, repeats, and codon usage. Molecular Biology and Evolution, 28, 583-600.
- [7] Jansen, R.K., Raubeson, L.A., Boore, J.L., et al. (2005) Method for obbtaining and analyzing whole chloroplast genome sequences, methods in enzymology. Academic Press, 348-384.
- [8] 曹家树, 秦岭 (2005) 园艺植物种质资源学. 中国农业出版社, 北京, 140.
- [9] 李坤明, 胡忠荣, 陈伟 (2006) 昭通地区野生猕猴桃资源及其利用评价. 中国野生植物资源, 2, 39-41.
- [10] Kataoka, I., Mizugami, T., Kim, J.G., et al. (2006) Distribution and character of ploidy variance in Actinidia arguta. 园学杂志, 2, 121.
- [11] 李作洲 (2006) 猕猴桃属植物的分子系统学研究. 中科院武汉植物园, 武汉.
- [12] 邹游, 丁建, 申瑛, 赵建, 杨志荣, 吴成 (2007) 11个猕猴桃品种间的遗传多样性分析. 应用与环境生物学报, 2, 172-175.
- [13] 刘磊, 姚小洪, 黄宏文 (2013) 猕猴桃EPIC标记开发及其在猕猴桃属植物系统发育分析中的应用. 园艺学报, 6, 1162-1168.
- [14] 董文攀 (2012) 蜡梅科叶绿体基因组进化及被子植物高变叶绿体基因标记开发与应用. 东北林业大学, 哈尔滨, 43.
- [15] Thompson, J.D., Gibson, T.J., Plewniak, F., Jeanmougin, F. and Higgins, D.G. (1997) The CLUSTAL_X windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 25, 4876-4882.
- [16] Tamura, K., Peterson, D., Stecher, G., Nei, M. and Kumar, S. (2011) MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 28, 2731-2739.
- [17] Caicedo, A.L. and Schaal, B.A. (2004) Population structure and phylogeography of Solanum pimpinellifolium inferred from a nuclear gene. Molecular Ecology, 13, 1871-1882.
- [18] Rozas, J., Sánchez-DelBarrio, J.C., Messeguer, X. and Rozas, R. (2003) DnaSP, DNA polymorphism analyses by the coalescent and other methods. Bioinformatics, 19, 2496-2497.
- [19] 刘阳, 杨世雄, 高立志 (2010) 云南古茶园栽培大叶茶和大理茶群体的叶绿体RPL32-TRNL核苷酸变异和遗传分化. 云南植物研究, 5, 427-434.
- [20] Chen, S.Y., Wu, G.L., Zhang, D.J., Gao, Q.B., Duan, Y.Z., Zhang, F.Q. and Chen, S.L. (2008) Molecular phylogeography of alpine plant Metagentiana striata (Gentianaceae). Journal of Systematics and Evolution, 46, 573-585.
- [21] 黄岳, 朴一龙, 王琳 (2009) 长白山区野生软枣猕猴桃种质RAPD分析. 延边大学农学学报, 2, 119-123.
- [22] Shaw, J., Lickey, E.B., Schilling, E.E. and Small, R.L. (2007) Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms: The tortoise and the hare III. American Journal of Botany, 94, 275-288.
- [23] Kress, W.J., Wurdack, K.J., Zimmer, E.A., Weigt, L.A. and Janzen, D.H. (2005) Use of DNA barcodes to identify flowering plants. Proceedings of the National Academy of Sciences of the United States of America, 102, 8369-8374.
- [24] Yue, J.P., Sun, H., Baum, D.A., Li, J.H., Al-Shehbaz, I.A. and Ree, R. (2009) Molecular phylogeny of Solms-laubachia (Brassicaceae) s.l., based on multiple nuclear and plastid DNA sequences, and its biogeographic implications. Journal of Systematics and Evolution, 47, 402-415.

NOTES
*通讯作者。
